自己如何制作动图表情包

自己如何制作动图表情包
大家好,我是小夏,在日常生活娱乐中,我们可能会有这样的需求:自己制作独创蜜汁表情包从而能在微信群中脱颖而出。而一般这种需求我们常规的做法都是通过借用现成的表情包去满足。但是我们都知道,很多出自他人之手的表情包有时候并不能淋漓尽致地表达自己内心地所思所想,而且能被找到并借用的表情包很多人在用,难免有时候会同他人撞上同款表情包的境况,所以,考虑到这些情况,自己制作表情包的想法势在必行。那么,我们一起来看看如何自制动图表情包吧!
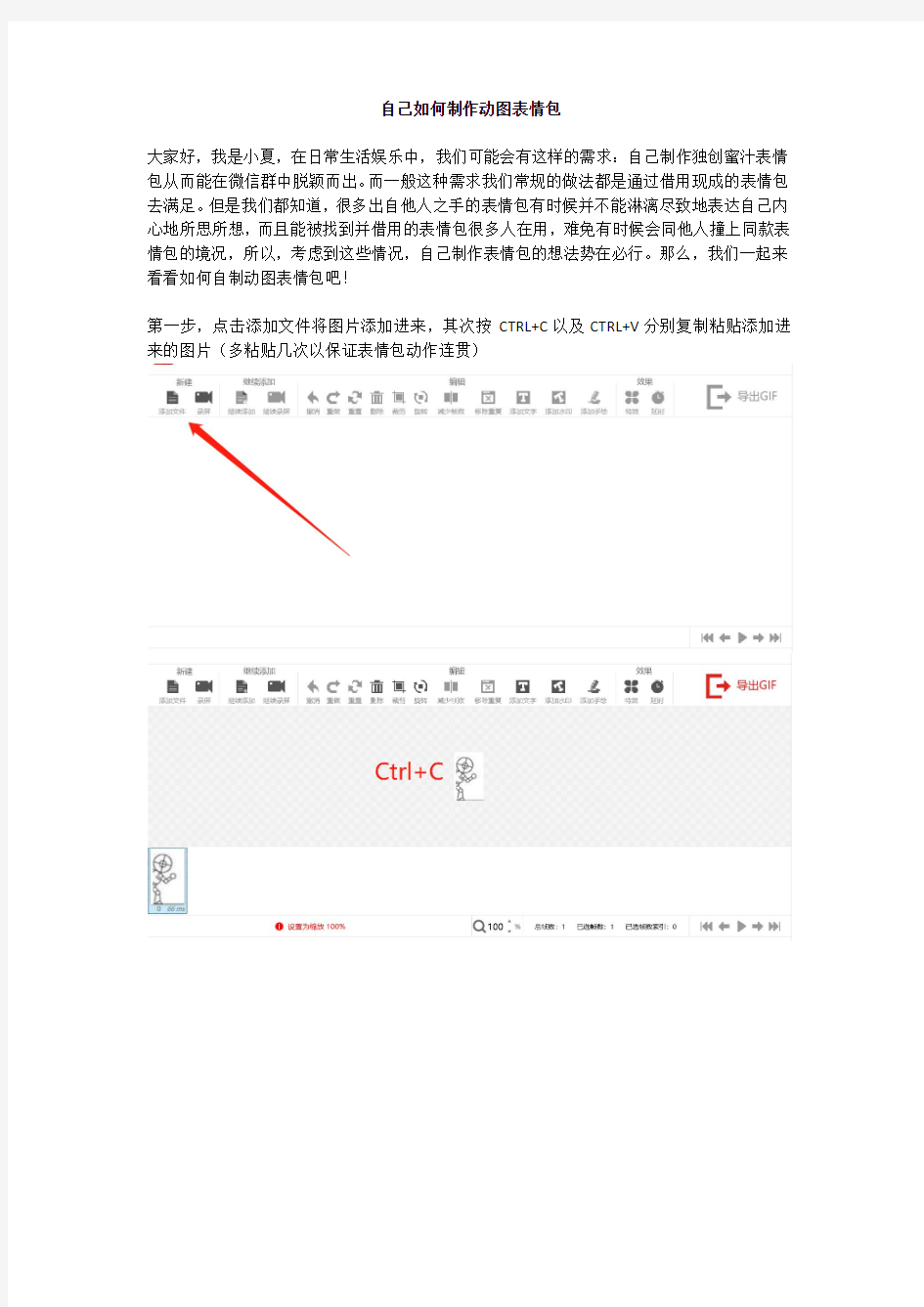
第一步,点击添加文件将图片添加进来,其次按CTRL+C以及CTRL+V分别复制粘贴添加进来的图片(多粘贴几次以保证表情包动作连贯)
第二步,继续添加,在弹出窗口调整帧数尺寸,其余步骤与第一步相同。(注意第二次添加图片时需将鼠标定位在粘贴的最后一张图片处)
第三步,如图,可以给表情包增加一些特效、文字等。
第四步,点击导出GIF,在弹出窗口处设置文件格式。
第五步,下拉滚动条设置文件储存路径后点击确定即可。
APBS教程
目录 1.怎样阅读教程 2.怎样准备结构来进行静电势计算 2.1PQR格式 2.2XML格式 2.3由PDB文件生成PQR文件(PDB2PQR) 3.怎样观察生物大分子周围的静电势 3.1VMD 3.2PyMOL 3.3PMV 4.怎样计算溶剂化能 4.1极性溶剂化 4.2非极性溶剂化 5.怎样计算结合能 5.1溶剂化能对结合的贡献 5.2包括库仑力贡献 5.3不行!配体没有设置参数 5.4一个配体结合的例子 6.怎样计算溶剂化力 7.怎样计算PKa? 7.1概况 7.2介绍 7.3应用于溶菌酶 8.我的计算需要的内存太大了! 8.1并行计算:一个例子 8.2异步时序计算 9.如何将APBS用于分子动力学软件(MM/PBSA等)? 10.怎样通过网络运行APBS?(Gemstone) 10.1得到Gemstone 10.2用PDB2PQR来准备结构 10.3用APBS进行静电计算 11.更多例子…… 12.所有这些没有回答我的问题-…… 图像清单 3.1 .弓形阻遏物的等势线(在VMD中) 3.2.弓形阻遏物表面势能(在VMD中) 3.3.FAS2静电势能等势线(+/- KT/e)(在PyMOL中) 3. 4.FAS2静电表面势能(+/- 5KT/e)(在PyMOL中) 3. 5.全溶剂化能循环 5.1. 结合自由能循环 7.1. pK a摄动自由能循环原理图 7.2. HEWL活性位点
8.1.并行计算得到的肌动蛋白二聚体等势线 10.1. Gemstone PDB2PQR 计算 10.2. Gemstone APBS/Calculation 屏幕 10.3. Gemstone APBS/Grid屏幕 10.4. Gemstone APBS/Physics屏幕 10.5. Gemstone APBS/File I/O屏幕 10.6. Gemstone APBS/Calculation屏幕(运行完成) 表格清单 7.1. 常见可滴定基团的模型氨基酸pKa 值; 数据来自Nielsen et al (见注脚) List of Examples 4.1. 玻恩离子PQR 4.2.玻恩输入文件示例 方程式清单 4.1. 玻恩离子极性溶剂化能 5.1. 结合自由能方程 5.2. 溶剂化对结合自由能的贡献 5.3. 库仑力对结合自由能的贡献 5.4. 结合自由能 7.1. 酸解离自由能 7.2. 迁移自由能 Chapter 1.怎样阅读教程 这本教程是以"怎样做"的形式设计的,使读者能熟悉使用APBS进行静电计算。读者需要最新版本的APBS((https://www.360docs.net/doc/289002630.html,)来演示本教程中提供的实例。需要的其他文件列在Individual section里。 重要信息 注意本教程中的许多实例操作也可以通过网络Gemstone实现,而不需要在本地装载APBS 软件。更多信息见Chapter 10, 怎样通过网络运行APBS ? (Gemstone) 。 提示 本教程仍在完善之中,并且会在下一版本的APBS 开 发出之前完成。未完成部分涵盖的许多课题在APBS 实例目录中有所展示。
pymol使用教程
简介&安装 Pymol是一个开放源码,由使用者赞助的分子三维结构显示软件,由Warren Lyford DeLano编写,并且由DeLano Scientific LLC负责商业发行。 Pymol被用来创作高品质的分子(特别是生物大分子如蛋白质)三维结构。据软件作者宣称,在所有正式发表的科学论文中的蛋白质结构图像中,有四分之一是使用Pymol来制作的。 Pymol名字的来源:“Py”表示该软件基于python这个计算机语言,“Mol”则是英文分子(molucule)的缩写,表示该软件用来显示分子结构。 由于实验需要,本人正在学习该软件,在这里把学习过程记录下来,希望对有需要的朋友有所帮助。今天先来说说安装吧。 自2006年8月1日起,DeLano Scientific 对事先编译好的PyMOL执行程序(包括beta版)采取限定下载的措施。目前,只有付费用户可以取得。不过源代码目前还是可以免费下载,供使用者编译。如果你和我一样,不想为此花钱的话: 1.如果你是Windows用户,首先下载Pymol的源代码。 然后安装CygWin,并且确保正确安装以下模块: ?C++ (gcc or g++ package name) ?Python ?OpenGL ?PNG 然后在源代码目录里面依次运行: 2.如果你是Linux用户,首先确保以下东东已安装: ?Python ?Pmw ?OpenGL driver(我用的是NVdia) ?libpng ?Subversion client(下载源代码需要) 然后下载Pymol的源代码 $ mkdir pymol-src $ svn co https://https://www.360docs.net/doc/289002630.html,/svnroot/pymol/trunk/pymol pymol-src 然后进入源代码目录 # cd pymol-src 开始依次编译
最新pymol作图的一个实例
Pymol作图的实例 这是一个只是用鼠标操作的初步教程 Pdb文件3ODU.pdb 打开文件 pymol右侧 All指所有的对象,2ODU指刚才打开的文件,(sele)是选择的对象 按钮A:代表对这个对象的各种action, S:显示这个对象的某种样式, H:隐藏某种样式, L:显示某种label, C:显示的颜色 下面是操作过程: 点击all中的H,选择everything,隐藏所有 点击3ODU中的S,选择cartoon,以cartoon形式显示蛋白质 点击3ODU中的C,选择by ss,以二级结构分配颜色,选择 点击右下角的S,窗口上面出现蛋白质氨基酸序列,找到1164位ITD,是配体 点击选择ITD ,此时sele中就包含ITD这个残基,点击(sele)行的A,选 择rename selection,窗口中出现,更改sele 为IDT,点击(IDT)行的S选择sticks,点击C,选择by element,选择,,调 整窗口使此分子清楚显示。 寻找IDT与蛋白质相互作用的氢键: IDT行点击A 选择find,选择polar contacts,再根据需要选择,这里选择to other atoms in object ,分子显示窗口中出现几个黄色的虚线,IDT行下面出现了新的一行,这就是氢键的对象,点击这一行的C,选择 red red,把氢键显示为红色。 接着再显示跟IDT形成氢键的残基 点击3ODU行的S,选择lines,显示出所有残基的侧链,使用鼠标转动蛋白质寻找与IDT 以红色虚线相连的残基,分别点击选择这些残基。注意此时selecting要是
residures。选择的时候要细心。取消选择可以再次点击已选择的残基。使用上述的方法把选择的残基(sele)改名为s1。点击S1行的S选择sticks,C选择by elements,点击L选择residures显示出残基名称.在这个例子中发现其中有一个N含有3个氢键有两个可以找到与其连接的氨基酸残基,另一个找不到,这是因为这个氢键可能是与水分子形成的,水分子在pdb文件中只用一个O表示,sticks显示方式没有显示出来水分子,点击all行S选择nonbonded,此时就看到一个水与N形成氢键 ,点击分子空白处,然后点击选择这个水分子,更改它的名字为w。在all 行点击H,选择lines,在选择nonbonded,把这些显示方式去掉只留下cartoon。点击w行s 选择nb-spheres. 到目前为止已经差不多了 下面是一些细节的调整 残基名称位置的调整:点击右下角是3-button viewing 转变为3-button viewing editing,这样就可以编辑修改 pdb文件了,咱们修改的是label的位置。按住ctrl键点击窗口中的残基名称label,鼠标拖拽到适合的部位,是显示更清晰。 然后调整视角方向直到显示满意为止,这时就可以保存图片了,file>>save image as>>png
cad里面动态块的制作教程
cad里面动态块的制作教程 创建CAD动态块的一般步骤 将一个或多个单一的实体对象整合为一个对象,这个对象就是图块。图块中的各实体可以具有各自的图层、线性、颜色等特征。在应用时,图块作为一个独立的、完整的对象进行操作,可以根据需要按一定比例和角度将图块插入到需要的位置。向块中添加参数集与添加参数所使用的方法相同。参数集中包含的动作将自动添加到块定义中,并与添加的参数相关联。接着,必须将选择集(几何图形)与各个动作相关联。 首次向动态块定义添加参数集时,每个动作旁边都会显示一个黄 色警告图标。这表示需要将选择集与各个动作相关联。可以双击该黄色警示图标,然后按照命令行上的提示将动作与选择集相关联。 如果插入的是查寻参数集,双击黄色警示图标时将会显示“特性 查寻表”对话框。与查寻动作相关联的是添加到此表中的数据,而不是选择集。 为了得到高质量的动态块,提高块的效率,避免重复修改,我们 一般可以通过以下几个步骤完成动态块的创建。
步骤1:规划 在创建动态块之前,有必要对动态块进行必要的规划,规划动态块要实现的功能、外观,在图形中的使用方式,以及要实现预期功能需要使用哪些参数和动作。 步骤2:绘制几何图形 绘制动态块中所包含的基本图元,当然,这些图元也可以在块器中绘制。步骤3:添加参数和动作 这是动态块创建过程中最关键的环节,参数和动作的不但要考虑到动态块功能的实现,同时也要考虑到动态块的可读性及修改的方便性,尽可能将参数的作用点吸咐在对应的图元上,且动作应摆放在其关联参数附近,参数和动作较多时还需要为其重命名,以便理解、和修改。 步骤4:测试动态块 保存并退出块器后,对动态块进行效果测试,检测是否能达到预期的效果。
pymol作图的一个实例
Pymol作图的实例 这就是一个只就是用鼠标操作的初步教程 Pdb文件3ODU、pdb 打开文件 pymol右侧 All指所有的对象,2ODU指刚才打开的文件,(sele)就是选择的对象 按钮A:代表对这个对象的各种action, S:显示这个对象的某种样式, H:隐藏某种样式, L:显示某种label, C:显示的颜色 下面就是操作过程: 点击all中的H,选择everything,隐藏所有 点击3ODU中的S,选择cartoon,以cartoon形式显示蛋白质 点击3ODU中的C,选择by ss,以二级结构分配颜色,选择 点击右下角的S,窗口上面出现蛋白质氨基酸序列,找到1164位ITD,就是配体 点击选择ITD ,此时sele中就包含ITD这个残基,点击(sele)行的A,选择rename selection,窗口中出现,更改sele为IDT,点击 (IDT)行的S选择sticks,点击C,选择by element,选择,,调整窗口使此分子清楚显示。 寻找IDT与蛋白质相互作用的氢键: IDT行点击A 选择find,选择polar contacts,再根据需要选择,这里选择to other atoms in object , 分子显示窗口中出现几个黄色的虚线,IDT行下面出现了新的一行 ,这就就是氢键的对象,点击这一行的C,选择red red,把氢键显示为红色。 接着再显示跟IDT形成氢键的残基 点击3ODU行的S,选择lines,显示出所有残基的侧链,使用鼠标转动蛋白质寻找与IDT以红色虚线相连的残基,分别点击选择这些残基。注意此时selecting要就是 residures。选择的时候要细心。取消选择可以再次点击已选择
pymol 基本操作
简介&安装 Pymol是一个开放源码,由使用者赞助的分子三维结构显示软件,由Warren Lyford DeLano编写,并且由DeLano Scientific LLC负责商业发行。 Pymol被用来创作高品质的分子(特别是生物大分子如蛋白质)三维结构。据软件作者宣称,在所有正式发表的科学论文中的蛋白质结构图像中,有四分之一是使用Pymol来制作的。 Pymol名字的来源:“Py"表示该软件基于python这个计算机语言,“Mol”则是英文分子(molucule)的缩写,表示该软件用来显示分子结构。 由于实验需要,本人正在学习该软件,在这里把学习过程记录下来,希望对有需要的朋友有所帮助。今天先来说说安装吧. 自2006年8月1日起,DeLano Scientific 对事先编译好的PyMOL执行程序(包括beta版)采取限定下载的措施。目前,只有付费用户可以取得.不过源代码目前还是可以免费下载,供使用者编译。如果你和我一样,不想为此花钱的话: 1.如果你是Windows用户,首先下载Pymol的源代码。 然后安装CygWin,并且确保正确安装以下模块: ?C++ (gcc or g++ package name) ?Python ?OpenGL ?PNG 然后在源代码目录里面依次运行: 2.如果你是Linux用户,首先确保以下东东已安装: ?Python ?Pmw ?OpenGL driver(我用的是NVdia) ?libpng ?Subversion client(下载源代码需要) 然后下载Pymol的源代码 $ mkdir pymol—src $ svn co pymol-src 然后进入源代码目录 # cd pymol-src 开始依次编译 # python setup.py install # python setup2。py install
Gromacs教程-水中的溶菌酶
GROMACS 教程水中的溶菌酶 Justin Lemkul Department of Biochemistry, Virginia Tech YongMa2008@小木虫译
y o n g m a 2008@小木虫 写在前面: 1 本人没有系统学习过MD, 本教程是本人开始自学GROMACS 的入门教程,在课余时间翻译的,对于很多专有词汇,不是很懂,翻译的不尽正确,欢迎批评指正 2 由于版本问题,本教程中某些命令行或需要输入的变量可能会有不同,请自行斟酌 3 本教程中红色为命令行,蓝色为超级链接,橙黄色为程序运行时终端显示文字或文件内部文字 4 由于Word 排版问题,某些命令行中的空格不是很明显,请注意;,同时由于时间仓促难免出现排版错误,请见谅 5 原版教程链接如下,强烈建议有兴趣的童鞋学习原版https://www.360docs.net/doc/289002630.html,/Pages/Personal/justin/gmx ‐tutorials/lysozyme/index.html
y o n g m a 2008@小木虫 本例将指导新用户进行设置一个蛋白质(lysozyme)加上离子在水盒子里的模拟过程。每个步骤将包含对输入输出文件一般应用的典型设置的解释。 这个教程假定你正在用gromacs 的4.5.x 版本。 第一步,准备拓扑 我们必须先下载我们要用的蛋白质结构。在这个教程中,我们将用鸡蛋清溶菌酶(PDB 代码1AKI). 去RCSB (https://www.360docs.net/doc/289002630.html,/pdb/home/home.do ) 网页下载PDB 文本格式的结构。 下载结构之后,可以利用VMD,chimera, PyMOL 等可视化程序看一下蛋白质结构。看了这个分子之后,你要去掉结晶水。用纯文本编辑器,比如vi, emacs(Linux/Mac)或者notepad(Windows)。不要用文字处理软件!删掉那些相关行(PDB 文件中residue“HOH”). 注意这个过程不是必须的(比如在水分子的活性部位结合案例中)。我们强调我们这里不需要结晶水。 检查你的.pdb 文件看MISSING 下面列的条目。因为这些条目显示了不存在于该晶体结构中的原子或者残基。终端可能没有或者不显示关于动力学的问题。任何不完整的内部序列或氨基酸残基的原子的缺失将导致pdb2gmx 失败。这些丢失的原子/残基必须通过其他软件来弥补。还要注意pdb2gmx 不是万能的,不能为任意分子产生拓扑文件,而只针对力场库中已经定义的残基(在*.rtp 文件中的一般的蛋白质,核酸和一些有限的辅助因子,像是NAD(H) 和 ATP). 现在结晶水已经没了,我们需要验证那些需要的原子还都在,PDB 文件应该只含有蛋白质原子,而且准备好了迎接第一个gromacs 工具pdb2gmx. Pdb2gmx 的目的是产生三个文件。 1. 分子的拓扑文件 2. 位置限定文件 3. 后处理文件 拓扑文件(默认为topol.top)包含定义分子模拟的所有信息。这些信息包含非成键参数(原子类型和电荷)和成键信息(键长,键角和二面角)。拓扑文件形成后,我们要仔细看一下。 用如下命令执行pdb2gmx 命令: pdb2gmx -f 1AKI.pdb -o 1AKI_processed.gro -water spce 结构将被pdb2gmx 处理,你要选择力场: Select the Force Field: From '/usr/local/gromacs/share/gromacs/top': 1: AMBER03 force field (Duan et al., J. Comp. Chem. 24, 1999-2012, 2003) 2: AMBER94 force field (Cornell et al., JACS 117, 5179-5197, 1995)
制作高清GIF图教程【GIF制作教程】
制作高清GIF图教程【GIF制作教程】 首先,需要两个软件,分别是Potplayer,这是个播放器,免费的,可以去百度一下,很容 易找到,找到合适自己电脑的版本,就装进电脑吧。主要是分64位和32位两个版本,楼 主我就是苦逼的XP。。。。。。←就是它 另一个是Photoscape,这软件也很好找,是个适合初学者用的简单的GIF合成软件。 ←就是它。 首先我们用Potplayer打开要截取的视频。 打开视频之后找到要截取的画面后,按Ctrl+G可以打开这个截图的文件夹,顶上可以设置
截取的图片路径,其他可以和我做一样的设置,尺寸设置一栏可以选择原始尺寸,但是如果截取很卡,可以改为自定义尺寸。需要字幕的话可以点上同时截取字幕。 并在右键菜单字幕设置中选择非图层输出。
这里还有一点很重要的是帧设置,截取帧数可以设置大一点,因为可以随时开始停止所以设置1000或10000都没关系,重要的是截取间隔,如果想要时间截取的长一点,可以像楼上那张图一样设置成100,要让画面更流畅,可以设置成70-80左右。 PS,不推荐50或者以下,因为如果间隔太小,一个是会大大缩减截取时间,一个是会截取很多相同的帧,而且帧数过高经证明会有浏览器出现卡顿现象。 点击开始就可以开始截图了,点击停止就能停止连续截图,这里就不上图解释了。 接下来我们打开截图文件夹。
就像这样,按住Ctrl来多选。选中所有的要做成GIF的图片。 接下来打开Photoscape来合成GIF图片。 找到四点钟方向的GIF动画,打开。 然后打开截图文件夹,点住要做GIF的第一张(也就是楼上那张图里面第一行从右往左数第二张,这很重要。)拖入下图所示的位置。
pymol做图简单教程
1.将对接好的ligand和蛋白merge(好像每次只能用一个ligand和一个蛋白,否则pymol里会 把几个ligand当作一个 2.Merge后export structure,存成pdb格式 3.在pymol中打开.pdb 4.在里面找出ligand,点sele的A(action)/rename Rename这个ligand,这里命名为lig 5.选all,H(hide)everthing 6.选lig,S(show)sticks 7.2j50(就是那个蛋白)S/cartoon 8.在后面的color调整颜色,这里ligand选by element
2j50选white 9.下面显示氢键,点击2j50的Action,选择find/polar contacks/to other atoms in object,氢键就出来了
10.然后再找氢键作用的残基,可以在上面直接点选,但最好找出氨基酸代码来选取(因为
这里面点不准) Rename它们,以便以后好找,这里rename为res,show它们为line,color为by element 11.再选display,将background改成white 12.然后分别点选这几个res(因为颜色不同,所以很容易找出来),右键label/residues
https://www.360docs.net/doc/289002630.html,bel出来后,还可以调整它们的位置,点一下左图viewing,就变成了右图的editing,这时就可以按住ctrl键拖动label的位置了 12.大体已经完成了,再进行一些修饰
Setting/transparency/cartoon/50% 蛋白的透明度Setting/label/size 18调节字体的大小
APBS教程
APBS(分析处理测试)教程目录 1.怎样阅读教程 2.怎样准备结构来进行静电势计算 2.1PQR格式 2.2XML格式 2.3由PDB文件生成PQR文件(PDB2PQR) 3.怎样观察生物大分子周围的静电势 3.1VMD 3.2PyMOL 3.3PMV 4.怎样计算溶剂化能 4.1极性溶剂化 4.2非极性溶剂化 5.怎样计算结合能 5.1溶剂化能对结合的贡献 5.2包括库仑力贡献 5.3不行!配体没有设置参数 5.4一个配体结合的例子 6.怎样计算溶剂化力 7.怎样计算PKa? 7.1概况 7.2介绍 7.3应用于溶菌酶 8.我的计算需要的内存太大了! 8.1并行计算:一个例子 8.2异步时序计算 9.如何将APBS用于分子动力学软件(MM/PBSA等)? 10.怎样通过网络运行APBS?(Gemstone) 10.1得到Gemstone 10.2用PDB2PQR来准备结构 10.3用APBS进行静电计算 11.更多例子…… 12.所有这些没有回答我的问题-……
图像清单 3.1 .弓形阻遏物的等势线(在VMD中) 3.2.弓形阻遏物表面势能(在VMD中) 3.3.FAS2静电势能等势线(+/- KT/e)(在PyMOL中) 3.4.FAS2静电表面势能(+/- 5KT/e)(在PyMOL中) 3.5.全溶剂化能循环 5.1. 结合自由能循环 7.1. pK a摄动自由能循环原理图 7.2. HEWL活性位点 8.1.并行计算得到的肌动蛋白二聚体等势线 10.1. Gemstone PDB2PQR 计算 10.2. Gemstone APBS/Calculation 屏幕 10.3. Gemstone APBS/Grid屏幕 10.4. Gemstone APBS/Physics屏幕 10.5. Gemstone APBS/File I/O屏幕 10.6. Gemstone APBS/Calculation屏幕(运行完成) 表格清单 7.1. 常见可滴定基团的模型氨基酸pKa 值; 数据来自Nielsen et al (见注脚) List of Examples 4.1. 玻恩离子PQR 4.2.玻恩输入文件示例 方程式清单 4.1. 玻恩离子极性溶剂化能 5.1. 结合自由能方程 5.2. 溶剂化对结合自由能的贡献 5.3. 库仑力对结合自由能的贡献 5.4. 结合自由能 7.1. 酸解离自由能 7.2. 迁移自由能
pymol作图的一个实例演示教学
p y m o l作图的一个实 例
Pymol作图的实例 这是一个只是用鼠标操作的初步教程 Pdb文件3ODU.pdb 打开文件 pymol右侧 All指所有的对象,2ODU指刚才打开的文件,(sele)是选择的对象 按钮A:代表对这个对象的各种action, S:显示这个对象的某种样式, H:隐藏某种样式, L:显示某种label, C:显示的颜色 下面是操作过程: 点击all中的H,选择everything,隐藏所有 点击3ODU中的S,选择cartoon,以cartoon形式显示蛋白质 点击3ODU中的C,选择by ss,以二级结构分配颜色,选择 点击右下角的S,窗口上面出现蛋白质氨基酸序列,找到1164位ITD,是配体点击选择ITD ,此时sele中就包含ITD这个残基,点击(sele)行的A,选择rename selection,窗口中出现
,更改sele为IDT,点击(IDT)行的S选择sticks,点击C,选择by element,选择,,调整窗口使此分子清楚显示。 寻找IDT与蛋白质相互作用的氢键: IDT行点击A 选择find,选择polar contacts,再根据需要选择,这里选择to other atoms in object ,分子显示窗口中出现几个黄色的虚线 ,IDT行下面出现了新的一行 ,这就是氢键的对象,点击这一行的C,选择red red,把氢键显示为红色。 接着再显示跟IDT形成氢键的残基 点击3ODU行的S,选择lines,显示出所有残基的侧链,使用鼠标转动蛋白质寻找与IDT以红色虚线相连的残基,分别点击选择这些残基。注意此时selecting要是residures。选择的时候要细心。取消选择可以再次点击已选择的残基。使用上述的方法把选择的残基(sele)改名为s1。点击S1行的S选择sticks,C选择by elements,点击L选择residures显示出残基名称.在这个例子中发现其中有一个N含有3个氢键有两个可以找到与其连接的氨基酸残基,另一个找不到,这是因为这个氢键可能是与水分子形成的,水分子在pdb文件中只用一个O表示,sticks显示方式没有显示出来水分子,点击all行S选择nonbonded,此时就看到一个水与N形成氢键
pymol教程-09.7.23
PyMOL用户指南 目录 一、鼠标操作入门4 (这个数字是超链接,ctrl+左键) 1.启动4 1)通过鼠标4 2)通过命令行4 2.PyMOL窗口4 1)Virewer窗口4 2)外部GUI窗口5 3.下载PDB文件5 4.操控视图6 1)基本鼠标控制6 2)虚拟滚动球旋转7 3)移动截面7 4)改变旋转中心点8 5)简单回顾9 二、命令行操作入门9 1.记录结果9 2.载入数据9 3.操控对象(Object)10 1)原子选择11 2)对象和选择的着色12 3)对象和选择的on/off12 4.改变视点13 5.保存工作13 1)脚本和日志文件13 2)图像文件14 3)会话文件14 6.命令行快捷键14 1)用TAB键完成命令15 2)用TAB键完成文件名15 3)自动推理15 7.其他命令和帮助15
三、命令句法和原子选择16 1.语法16 1)选择表达16 2)原子选择命名16 3)单字选择符 4)属性选择符18 5)选择代数20 6)宏指令21 2.从PyMOL中读取Python 22 四、卡通表示23 1.背景23 1)可达性23 2)美化和精确23 2.定制化25 1)卡通类型25 2)精美螺旋28 3.二级结构归属29 五、光线追踪30 1.重要设置30 2.保存图片31 六、立体效果31 1.支持的立体模式31 2.制作立体图片31 3.相关命令31 七、动画32 1.概念32 2.重要命令32 1)Load 2)Mset 3)Mdo 4)Mmatrix 3.简单举例33 4.复杂举例33 5.预览ray-traced动画图片34 1)Cache_frames
mg动画教程 mg动画如何制作
mg动画教程,mg动画如何制作 万彩动画大师-比AE、Flash简单的MG动画视频制作软件。平时制作一些动画宣传片、类似于《飞碟说》这样MG动画的时候,大家就会想到AE、flash等专业的软件,但是无奈这些软件过于专业,要花费大量时间和精力去学,非常复杂。而有些朋友会说,用PPT也可以做啊,做完再转视频,其实更复杂,做一页动画添加几十个动画,还要花几十分钟时间去调试,这是不是可以说是自虐? 这里给大家介绍一个免费的,专门制作MG动画视频制作的软件——万彩动画大师(官网:https://www.360docs.net/doc/289002630.html,),它比AE、Flash简单得多,运行流畅,布局简洁实用,比较扁平化,符合大多数人的使用习惯。它适用于制作企业宣传动画,产品介绍短片、趣味课件视频、微课视频、多媒体课件、演示演讲动画视频、动画广告,营销动画等。
使用万彩动画大师,从新建场景->设置背景->编辑时间轴->输出视频,所见所得,轻松上手;内置素材丰富,包含精美的动画模板、种类多样的各种动画角色、矢量图库、丰富的场景选择;强大的时间轴工具:支持应用不同的动画效果,调整对象进出场时间、速度及停留时间;拥有炫酷的镜头特效:可以添加旋转、缩放、平移等特效镜头,能够轻松制作出专业的MG动画。 Mg动画教程大纲: 一、创建新工程文件 二、新建场景、选择场景 三、如何选中、编辑物体
四、添加镜头 五、添加字幕 六、添加背景图片 七、添加动画效果 八、预览、保存及发布动画视频 一、mg动画教程-创建新工程文件 万彩动画大师提供两种方式来创建新工程文件。 1.选择“新建空白项目”
开始编辑工程。 2.打开一个在线模板。
教你简单制作动画图片的小方法
有朋友问我,如何做作有个性化的动画图?做动画的方法有很多种,我们就从简单学起,介绍如何用Photoshop和Imageready制作动画。 效果图 1)新建文件 点击“文件”—“新建”—“FUPING111”,设定宽度、高度、名称,“背景内容”选透明”,新建一个300(宽)×150(高)象素即可,具体尺寸可以按自己需要设定。 2)制作所需的图片或素材 在photoshop里打开所需的图片,这里只需要一张背景图,再输入您所要显示的文字如图1。鼠标左键点击背景图片,按“ Ctrl+A”,图四周出现流动的虚线框,再 Ctrl+C拷贝该图片。鼠标左键点击刚才新建的“FUPING111”文件,按“Ctrl+V”,就将图片粘贴进“FUPING111”中了。(如果图片不是300×150象素,那么还需要把图片制作成300×150象素) 图1 3)进入动画软件
点击photoshop的工具条最下方的图标。进入Imageready软件,如图2。 图2 如果这个步骤无法完成,表示您安装的是 photoshop迷你版本,请重新下载完整版的 photoshop。正常情况下, Imageready在左下角有个“动画窗口”,右下角有“图层窗口”。如果找不到这 2个窗口,请在软件菜单“窗口”菜单中勾选显示出来。 图3 4)动画制作
进入 Imagereadym,在“动画”对话框内默认为一帧。我们初步的设计为文字从左侧进入图片中央,停顿5秒,再从右侧滚出。所以先将第一桢的文字使用移动工具,将文字拖到左侧图框外。点“复制桢”按钮,复制第二桢,在第二桢把文字拖到图框中央,见图4 图4 按住“SHIFT”键,选取1、2桢,点“过渡”按钮,出现“过渡”对话框,在“要添加的桢”狂内,填入“10”,增加10桢的过渡。添加的过渡桢数越多,动画过渡的越自然,但动画文件越大。见图5
MD基本教程-溶菌酶的水溶液的MD模拟
溶菌酶水溶液的简单分子动力学模拟教程 GROMACS是一个使用经典分子动力学理论研究蛋白质动力学的工具[1]。这个软件包遵守GNU协议,因此可以免费从其主页(https://www.360docs.net/doc/289002630.html,)上下载。GROMACS可以在linux、unix和Windows上使用。 第一步建立拓扑结构 本教程使用鸡蛋清溶菌酶(PDB code:1AK1)作为目标蛋白质。首先需要从RCSB(https://www.360docs.net/doc/289002630.html,/pdb/home/home.do)网站下载其三维结构文件。然后通过诸如SYBYL、Discovery Studio、VMD、Chimera和PyMOL等图形显示软件分析其三维结构。根据模拟的需要去除或保留该文件中的结晶水分子。例如,在研究蛋白质和配基之间的亲和作用时,结合在活性位点附近的水分子就必须保留。但如果利用MD模拟研究蛋白质的构象转换时,蛋白质中的结晶水就可以删除。删除PDB文件中分子可以利用简单的文本编辑软件如vi(Linux)、emacs (Linux/Mac)和Notepad (Windows) 删除这些分子所对应的条目。例如,如果要删除文件中高的结晶水就可以将PDB文件中的所有“HOH”残基删除。切记不要用文字处理软件Word处理该文件。 在运行pdb2gmx程序之前,一定要检查pdb文件中的MISSING条目。这些条目会标注出该晶体结构中缺失哪些原子或残基。这一步是必须做的,因为不完全氨基酸残基或分子都会导致pdb2gmx命令的失败。这些遗失的原子或残基必须利用其他的软件补充完整。但末端的遗失可能不会对动力学模拟的顺利进行造成影响,但有可能会对结果有影响。此外,还要注意pdb2gmx程序并不是万能的,它并不能为所有的分子建立拓扑结构,仅能对一些力场中定义好的分子才有效,例如大部分蛋白质,核酸,还有一些辅因子如NAD、NADH 、ATP和ADP 等。此外,一些糖类(海藻糖等)的力场也都定义好,可以从力场文件中获得。因此,如果目标蛋白质含有小分子配基的,在运行该软件之前就需要先用简单的文本编辑软件将配基删除。最后除去结晶水和小分子配基,同时确保所有必需原子存在,此时的PDB文件中只含蛋白质原子,现在可以运行pdb2gmx命令了。 常用pdb2gmx命令的详细参数: pdb2gmx -f 1AKI.pdb -o 1conf.gro –p conf.top –ignh
GIF动态图制作傻瓜教程:如何截图及转换编辑
GIF动态图制作傻瓜教程:如何截图及转换编辑 一、本教程简要步骤如下: 1、录像操作,主要为ViewletCam v1.7.1 录像软件的基础使用教程,就是怎么使用ViewletCam v1.7.1 把游戏里面的动作录成录像格式; 2、编辑录好的视频操作,主要为在ViewletCam v1.7.1 录像软件里面可以做一些往视频添加字等操作(也可不添加),并导出为AVI格式录像; 3、在Ulead GIF Animator v5.05 GIF图像处理软件里面导入上面做好的AVI 格式录像,并且可以在Ulead GIF Animator v5.05 软件里面进行一些修饰、处理图片操作,当然也可以不处理图片; 4、在Ulead GIF Animator v5.05 里面将最终成型的视频导出为 GIF格式,至此GIF图片制作就完毕了。 二、制作软件: 1、ViewletCam v1.7.1 多国语言版 软件介绍:一个非常好用的屏幕录像软件,可以将电脑屏幕上所有的操作录成AVI视频、SWF、GIF格式的软件。注意:直接使用该软件录成成GIF图片格式,会有非常大的图片颜色失真,不相信的可以自己操作试试。 软件注册信息: 用户名: Qar2003Brace[D.4s] 注册码: 7R68SRGHPBTRNDY2 2、Ulead GIF Animator v5.05 简体中文绿色版 软件介绍:一款常用的GIF图像处理工具,把GIF动态图片分解成一帧一帧动
作,每一帧就是一个静态的图片,可以单独的修改某一帧图片,进而达到修改GIF 效果。同时可以把AVI格式视频转换为GIF格式,从而不会导致图片的颜色失真。 免费软件,不需要注册。在具体的GIF制作前,请先下载并安装好以上两个软件。 三、一个GIF图片详细制作过程: 1、如何使用ViewletCam 录取倩女幽魂游戏中某一段的视频操作: ①打开ViewletCam v1.7.1 软件,界面如下:
全景图制作流程-pixmaker简易教程
PixMaker制作全景图简易教程 1、pixmaker简介 PixMaker是一个简单方便的360度全景图片制作软件,它可以将描写一个环型场景的多个连续图片无缝地接合在一起,形成一个360度“场景”图片。可以在网页上播放,使你的页面更加生动活泼。 2、利用PixMaker制作全景图片注意时项。 (1)使照像机平行于地面并最好固定在三角架上。(2)拍摄每一幅照片时要保持照像机在同一个高度。(3)以一个固定点为圆心,顺时针方向旋转拍摄每一幅照片。 (4)每一幅照片的边界要有30%左右的重叠区域。(5)要注意将移动的物体拍摄在照片的中间,不要让移动物体出现的照片相交错的边界上,以免对以后照片的边界处理带来不便。 (6)根据你使用的照像设备的不同,使用扫描仪等设备将照片处理成JPG格式的图片文件,图片的尺寸可根据你自己的需要决定,文件名最好按字母顺序排列。 3、利用Pixmkaer制作全景图的基本步骤 (1)snap点击snap后可以看到如下对话框
点击ok按钮,打开导入图片的对话框。 可以用shift键进行选择全部的图片进入到Pixmaker 软件。 制作全景图的图片数据导入到pixmaker软件后如图所示:
导入图片后出现一个工具条,如红框所示: 上边有插入、删除、移动图片、图片类型(Type)、镜头选择(Lens)等操作命令图标。其中图片类型选360度,镜头选Auto 即可。 (2)stitch 这一步骤是将前面所抓取的多幅图片无缝地连接在一起,形成一个360度全景的“场景”图。操作非常简单,只需你点击第二个“Stitch”大图标,PixMaker程序会自动地处理图片的边界对齐和连接,在一个弹出的处理窗口中可以动态地看到自动处理图片的全过程。
教你简单制作动画图片的小方法
教你简单制作动画图片的小方法
有朋友问我,如何做作有个性化的动画图?做动画的方法有很多种,我们就从简单学起,介绍如何用Photoshop和Imageready制作动画。 效果图 1)新建文件 点击“文件”—“新建”—“FUPING111”,设定宽度、高度、名称,“背景内容”选透明”,新建一个300(宽)×150(高)象素即可,具体尺寸可以按自己需要设定。 2)制作所需的图片或素材
在photoshop里打开所需的图片,这里只需要一张背景图,再输入您所要显示的文字如图1。鼠标左键点击背景图片,按“ Ctrl+A”,图四周出现流动的虚线框,再 Ctrl+C拷贝该图片。鼠标左键点击刚才新建的“FUPING111”文件,按“Ctrl+V”,就将图片粘贴进“FUPING111”中了。(如果图片不是300×150象素,那么还需要把图片制作成300×150象素) 图1 3)进入动画软件 点击photoshop的工具条最下方的图标。进入Imageready软件,如图2。
图2 如果这个步骤无法完成,表示您安装的是 photoshop迷你版本,请重新下载完整版的photoshop。正常情况下, Imageready在左下角有个“动画窗口”,右下角有“图层窗口”。如果找不到这 2个窗口,请在软件菜单“窗口”菜单中勾选显示出来。
图3 4)动画制作 进入 Imagereadym,在“动画”对话框内默认为一帧。我们初步的设计为文字从左侧进入图片中央,停顿5秒,再从右侧滚出。所以先将第一桢的文字使用移动工具,将文字拖到左侧图框外。点“复制桢”按钮,复制第二桢,在第二桢把文字拖到图框中央,见图4
简单的MG动画如何轻松制作方法教程
MG动画,即图文动画的意思。它融合了平面设计、动画设计和电影语言,表现形式丰富多样,主要应用于电影电视片头、商业广告、MV、产品演示等各个领域。 很多同学可能还不知道MG动画如何制作,今天我们来分享一下其制作方法和教程。 1.构思 在制作MG动画之前,首先你要进行构思,有一个明确具体的制作思路,即你要知道你想表达什么,确定好一个主题并围绕这一主题进行扩张和补充。另外,构思最好有新意,能捉住亮点,吸引受众。
举个例子,比如我们要做一个简易几何图形MG动画片头,首先要考虑到图像的运用,动态效果的设置以及镜头的转换等各个要素,才能更好地进行动画制作制作。 2.准备好素材 素材图片或视频可以上网下载,也可以自己用PS、AI工具进行设计,然后整理在一个文件夹,方便使用。 3.选择MG动画制作工具
常见的动画制作工具有AE。AE是一款专业的图形视频处理软件,它的功能强大,能制作出各种炫酷的电影特效,广告以及动画片头等等。但AE操作界面复杂,刚开始接触动画制作的小伙伴可能不好上手。 这里推荐另一款简单实用的动画制作软件—万彩动画大师,它的操作非常简单,能轻松制作MG动画。 4.制作流程 用万彩动画大师制作动画的方法非常简单,直接套用模板就可以了。 打开软件—>选择你想要套用的模板—>在打开的模板编辑页面中,选中对象(对象可以是文本或图片)—鼠标右键单击对象在时间轴添加的动画效果,便可以对其内容、动画效果等编辑修改。
当然,你还可以为MG动画添加适当的音乐素材,点击左上角音乐图标,“添加音乐”。 最后再进行检查修改,一键生成视频,点击菜单栏中的“发布”—在新弹出窗口
分子动力学模拟I
Gromacs中文教程 淮海一粟 分子动力学(MD)模拟分为三步:首先,要准备好模拟系统;然后,对准备好的系统进行模拟;最后,对模拟结果进行分析。虽然第二步是最耗费计算资源的,有时候需要计算几个月,但是最耗费体力的步骤在于模拟系统准备和结果分析。本教程涉及模拟系统准备、模拟和结果分析。 一、数据格式处理 准备好模拟系统是MD最重要的步骤之一。MD模拟原子尺度的动力学过程,可用于理解实验现象、验证理论假说,或者为一个待验证的新假说提供基础。然而,对于上述各种情形,都需要根据实际情况对模拟过程进行设计;这意味着模拟的时候必须十分小心。 丢失的残基、原子和非标准基团 本教程模拟的是蛋白质。首先需要找到蛋白质序列并选择其起始结构,见前述;然后就要检查这个结构是否包含所有的残基和原子,这些残基和原子有时候也是模拟所必需的。本教程假定不存在缺失,故略去。 另一个需要注意的问题是结构文件中可能包含非标准残基,被修饰过的残基或者配体,这些基团还没有力场参数。如果有这些基团,要么被除去,要么就需要补充力场参数,这牵涉到MD的高级技巧。本教程假定所有的蛋白质不含这类残基。 结构质量 对结构文件进行检查以了解结构文件的质量是一个很好的练习。例如,晶体结构解析过程中,对于谷氨酰胺和天冬酰胺有可能产生不正确的构象;对于组氨酸的质子化状态和侧链构象的解析也可能有问题。为了得到正确的结构,可以利用一些程序和服务器(如 WHATIF)。本教程假定所用的结构没有问题,我们只进行数据格式处理。 二、结构转换和拓扑化 一个分子可以由各个原子的坐标、键接情况与非键相互作用来确定。由于.pdb 结构文件只含有原子坐标,我们首先必须建立拓扑文件,该文件描述了原子类型、电荷、成键情况等信息。拓扑文件对应着一种力场,选择何种力场对于拓扑文件的建立是一个值得仔细考虑的问题。这里我们用的是GROMOS96 53a6连接原子力场,该力场对于氨基酸侧链的自由能预测较好,并且与NMR试验结果较吻合。
动态图制作与编辑的几种简单方法
动态图无处不在,一般我们可以通过搜索与下载获得。但这些远远不能满足我们的需求。今天,灬无言讲些简单实用的动态图制作与编辑的方法。。 一、搜索获取动态图 简单提几个,类似网站还有很多。 1、百度图片 搜索“关键词+动态或gif”,如搜索“可爱动态”或“可爱gif”。 2、通过“soogif”来搜索 搜索“关键词”即可,反正都是动态图了。
3、通过“giphy”搜索 搜索“cute”。 二、动态图制作 1、将一些流媒体在线视频,利用“soogif在线gif工具”来制作动态图。
2、ScreenToGif、ShareX等软件进行电脑屏幕录制。 ScreenToGif录制如下图。 除了录制外,还可以将mp4等格式的视频制作成gif。只需要将视频文件拖入编辑状态的ScreenToGif中。
ShareX录制如下图。 3、PS制作动态图。 最简单的就是添加一行动态的字,当然也可以添加更多特效。 PS打开图片 > 每写一个字添加一帧 > 文件 > 存储为Web所用格式 > 预设选为gif > 存储。然后根据需要更改每一帧的时间。
4、AE制作动态。 简单的动态特效,打关键帧加特效等等。 三、动态图编辑
1、ScreenToGif。只需要将动态图拖入编辑状态的ScreenToGif中。 a)减少帧来减少图片的体积,但相应会降低图片的清晰度。 编辑 > 删除或减少帧计数。减少帧的同时,动态效果会太快而看不清,增加每一帧的时间,让动画变慢。 b)调整动态图大小。 调整动态图的两种方式:一是直接调整图片大小,二是截取图片的部分。
