非编码区和编码区、真核生物的启动子、终止子,真核生物RNA的修饰

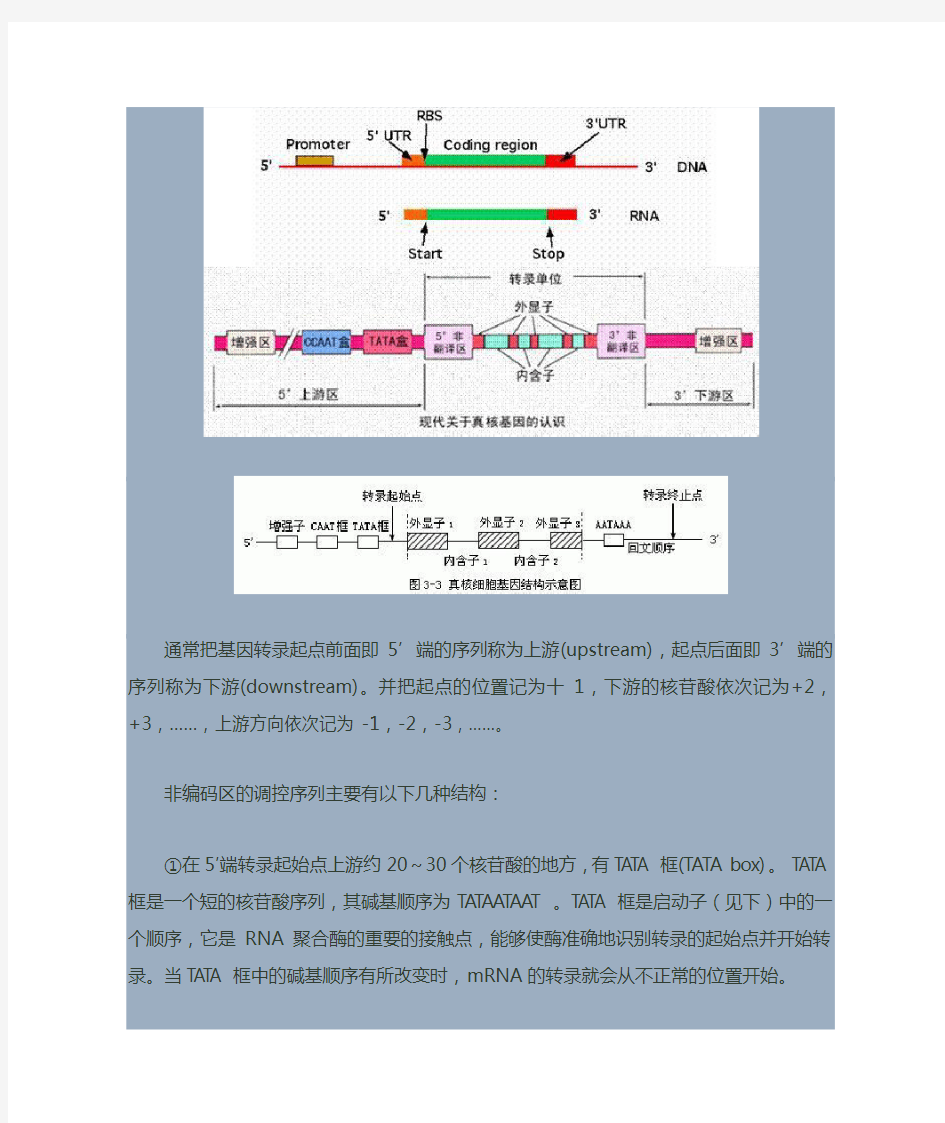
基因是由成千上万个核苷酸对组成。组成基因的核苷酸序列可以分为不同区段。在基因表达的过程中,不同区段所起的作用不同。在遗传学上通常将能编码蛋白质的基因称为结构基因。任何一个基因都包括非编码区和编码区。能够转录为相应信使RNA,进而指导蛋白质合成(也就是能编码蛋白质)的区段叫做编码区。不能转录为信使RNA、不能编码蛋白质的区段叫做非编码区。非编码区位于编码区前后,同属于一个基因,控制基因的表达和强弱。
原核生物的基因
非编码区虽然不能编码蛋白质,但对遗传信息的表达是不可缺少的,因为在它上面由调控遗传信息表达的核苷酸序列,该序列中最重要的是位于编码区上游的RNA聚合酶结合位点。启动子、终止子属于非编码区。因为回文序列的特殊排列,大多都位于非编码区。
原核基因的编码区全部编码蛋白质,真核生物的结构基因是断裂的基因。一个断裂基因能够含有若干段编码序列,可以编码蛋白质的序列称为外显子。在两个外显子之间被一段不编码的间隔序列隔开,这些间隔序列称为内含子。非编码区在每个断裂基因的第一个和最后一个外显子的外侧,有人称其为侧翼序列。在侧翼序列上有一系列调控序列。
真核细胞的基因中编码区特点:间隔的、不连续的。包括:外显子和内含子(位于编码区中的非编码序列)。
通常把基因转录起点前面即5’端的序列称为上游(upstream),起点后面即3’端的序列称为下游(downstream)。并把起点的位置记为十1,下游的核苷酸依次记为+2,+3,……,上游方向依次记为-1,-2,-3,……。
非编码区的调控序列主要有以下几种结构:
①在5′端转录起始点上游约20~30个核苷酸的地方,有TATA框(TATA box)。TATA框是一个短的核苷酸序列,其碱基顺序为TATAATAAT。TATA框是启动子(见下)中的一个顺序,它是RNA聚合酶的重要的接触点,能够使酶准确地识别转录的起始点并开始转录。当TATA框中的碱基顺序有所改变时,mRNA 的转录就会从不正常的位置开始。
②在5′端转录起始点上游约70~80个核苷酸的地方,有CAAT框(CAAT box)。CAAT框是启动子中另一个短的核苷酸序列,其碱基顺序为GGCTCAATCT。CAAT框是RNA聚合酶的另一个结合点,它的作用还不很肯定,但一般认为它控制着转录的起始频率,而不影响转录的起始点。当这段顺序被改变后,mRNA 的形成量会明显减少。
③在5′端转录起始点上游约100个核苷酸以远的位置,有些顺序可以起到增强转录活性的作用,它能使转录活性增强上百倍,因此被称为增强子。当这些顺序不存在时,可大大降低转录水平。研究表明,增强子通常有组织特异性,这是因为不同细胞核有不同的特异因子与增强子结合,从而对不同组织、器官的基因表达有不同的调控作用。例如,人类胰岛素基因5′末端上游约250个核苷酸处有一组织特异性增强子,在胰岛素β细胞中有一种特异性蛋白因子,可以作用于这个区域以增强胰岛素基因的转录。在其他组织细胞中没有这种蛋白因子,所以也就没有此作用。这就是为什么胰岛素基因只有在胰岛素β细胞中才能很好表达的重要原因。
④在3′端终止密码的下游有一个核苷酸顺序为AATAAA,这一顺序可能对mRNA的加尾(mRNA尾部添加多聚A)有重要作用。这个顺序的下游是一个反向重复顺序。这个顺序经转录后可形成一个发卡结构。发卡结构阻碍了RNA聚合酶的移动。发卡结构末尾的一串U与转录模板DNA中的一串A之间,因形成的氢键结合力较弱,使mRNA与DNA杂交部分的结合不稳定,mRNA就会从模板上脱落下来,同时,RNA聚合酶也从DNA上解离下来,转录终止。AATAAA顺序和它下游的反向重复顺序合称为终止子(见下),是转录终止的信号。
启动子和终止子:
启动子(promoter)位于编码区上游的非编码区中。真核生物启动子包括下列几种不同顺序,能促进转录过程:
(1)帽子位点:转录的起始位点。
(2)TATA框(TATA box):又称Hogness框,类似于原核生物的Pribnow框,决定了转录起点的选择。
其一致顺序为TATAATAAT。约在基因转录起始点上游约-30-50bp处,基本上由A-T碱基对组成,为RNA 聚合酶的结合处之一,RNA聚合酶与TATA框牢固结合之后才能开始转录。
(2)CAAT框(CAAT box):其一致顺序为GGGTCAATCT,是真核生物基因常有的调节区,位于转录起始点上游约-80-100bp处,可能也是RNA聚合酶的一个结合处,控制着转录起始的频率。
(3)GC框(GC box):有两个拷贝,位于CAAT框的两侧,由GGCGGG组成,转录因子Sp1能识别GC框并且与之结合,其N端有激活转录的作用。所以,GC框是一个转录调节区,有激活转录的功能。
(4)增强子(enhancer):又称远上游序列(far upstream sequence)。一般都在-1OO以上。增强子的作用主要是对依赖于TATA框的转录和不依赖TATA框的转录都有增强效应,但对前者增强效应高。增强子是通过启动子来增加转录的。有效的增强子可以位于基因的5’端,也可位于基因的3’端,有的还可位于基因的内含子中。增强子的效应很明显,一般能使基因转录频率增加10~200倍,有的甚至可以高达上千倍。
终止子:在一个基因的末端往往有一段特定顺序,它具有转录终止的功能,这段终止信号的顺序称为终止子(termianator)。终止子为反向重复序列,是RNA聚合酶停止工作的信号,反向重复序列转录后,可以形成发夹式结构,并且形成一串U。发夹式结构阻碍了RNA聚合酶的移动,一串U的U与DNA模板中的A结合不稳定,从模板上脱落下来,转录终止。
RNA转录后的加工与修饰
不论原核或真核生物的rRNAs都是以更为复杂的初级转录本形式被合成的,然后再加工成为成熟的RNA 分子。然而绝大多数原核生物转录和翻译是同时进行的,随着mRNA开始的DNA上合成,核蛋白体即附着在mRNA上并以其为模板进行蛋白质的合成,因此原核细胞的mRNA并无特殊的转录后加工过程,相反,真核生物转录和翻译在时间和空间上是分天的,刚转录出来的mRNA是分子很大的前体,即核内不均一RNA。hnRNA分子中大约只有10%的部分转变成成熟的mRNA,其余部分将在转录后的加工过程中被降解掉。
1.在5’端加帽
成熟的真核生物mRNA,其结构的5’端都有一个m7G-PPNmN结构,该结构被称为甲基鸟苷的帽子。如图1所示。鸟苷通过5’-5’焦磷酸键与初级转录物的5’端相连。当鸟苷上第7位碳原子被甲基化形成
m7G-PPNmN时,此时形成的帽子被称为“帽0”,如果附m7G-PPNmN外,这个核糖的第“2”号碳上也甲基化,形成m7G-PPNm,称为“帽1”,如果5’末端N1和N2中的两个核糖均甲基化,成为m7G-PPNmPNm2,称为“帽2”。从真核生物帽子结构形成的复杂可以看出,生物进化程度越高,其帽子结构越复杂。
2.在3’端加尾
大多数的真核mRNA 都有3’端的多聚尾巴(A),多聚(A)尾巴大约为200bp。
多聚(A)屠巴不是由DNA编码的,而是转录后在核内加上去的。受polyA聚合酶催化,该酶能识别,mRNA 的游离3’-OH端,并加上约200个A残基。
近年来已知,在大多数真核基因的3’一端有一个AATAA序列,这个序列是mRNA 3’-端加polyA尾的信号。靠核酸酶在此信号下游10-15碱基外切断磷酸二酯键,在polyA聚合酶催化下,在3’-OH上逐一引入100-200个A碱基。关于polyA尾巴的功能问题尽管经过极其广泛的探索,但还不完全清楚。有人推测polyA可能与mRNA从细胞核转送到细胞质有关,但是相当数量,的没有polyA屠巴的mRNA如组蛋白mRNA,也照样通过核膜进入细胞质。还有人认为这种结构对真核mRNA的翻译效率具有某种作用,并能稳定mRNA 结构,保持一定的生物半衰期。
3.mRNA前体(hnRNA)的拼接
原核生物的结构基因是连续编码序列,而真核生物基因往往是断裂基因,即编码一个蛋白质分子的核苷酸序列被多个插入片断所隔开,一个真核生物结构基因中内含子的数量,往往与这个基因的大小有关,例如胰岛素是一个很小的蛋白质,它结构基因只有两个内含子,而有些很大的蛋白质,它的结构基因中可以有几十个内含子。经过复杂的过程后,切去内元,将有编码意义的核苷酸片段(Extron外元也叫外显子)连接起来(图2)。
常见真核启动子及原核启动子特点
真核启动子 EF1a 常规表达用 mRNA 人延长因子1α来源 的强哺乳动物表达启动子 组成型 表达水平十分稳定,与细胞类型无关 PGK1 (人/小鼠) 常规表达用 mRNA 磷酸甘油酸酯激酶 基因来源的哺乳动物启动子 组成型 广泛表达,但可能因细胞类型而异。由于甲基化或脱乙酰作用,倾向于抵抗启动子下调。 human beta actin 常规表达用 mRNA β-肌动蛋白基因来 源的哺乳动物启动子 组成型 无处不在,鸡的启动子常用与启动子杂交 TRE 常规表达用 mRNA 四环素响应元件启动子 被四环素或 者类似物诱导 通常有本底表达 Ac5 常规表达用 mRNA 果蝇Actin 5c 基因 来源的强昆虫启动子 组成型 果蝇表达系统的常用启动子
CaMKIIa 光遗传学 基因表达 mRNA Ca2+/钙调蛋白依赖 的蛋白激酶II 启动 子 特异的 用于中枢神经系统/神经元表达。受到 钙和钙调蛋白调节。 TEF1 常规表达 用 mRNA 酵母转录延伸因子 启动子 组成型与哺乳动物的EF1a 启动子类似 ADH1 常规表达 用 mRNA 乙醇脱氢酶I的酵母 启动子 被乙醇抑制 全长版本很强,促进高表达。截短启 动子是组成型的,表达较低。 Ubi 常规表达 用 mRNA 玉米泛素基因的植 物启动子 组成型在植物中促进高表达 U6 小RNA 表达 shRNA 来源于人U6小核启 动子 组成型小鼠U6也使用,但效率略差。 常用的原核表达系统启动子
T7lac 高水 平基 因表 达 T7噬菌体来 源的启动子 加上lac操 纵子 几乎没有本底表 达,需要T7 RNA 聚合酶,受到lac 操纵子的控制, 可以被IPTG诱 导。 常用与pET载体,受到lac操纵子的严格调控 araBAD 常规 表达 用 阿拉伯糖代 谢操纵子的 启动子 阿拉伯糖诱导 弱,常用与pBAD载体。适合于快速调控和低的本底表达 lac 常规 表达 用 Lac操纵子 来源的启动 子 可以被IPTG或 者乳糖诱导 在常规的大肠杆菌中,lacI阻遏蛋白表达量不高,仅能满足细 胞自身的lac操纵子,无法应付多拷贝的质粒的需求,导致非 诱导条件下较高地表达,为了让表达系统严谨调控产物表达, 能过量表达lacI阻遏蛋白的lacIq 突变菌株常被选为 Lac/Tac/trc表达系统的表达菌株。现在的Lac/Tac/trc载体上 通常还带有lacIq 基因,以表达更多lacI阻遏蛋白实现严谨的 诱导调控。 pL 高水 平基 因表 达 Lambda噬 菌体来源的 启动子 温度调节通常和温度敏感的cI857 抑制子搭配使用
常见真核启动子及原核启动子特点
真核启动子 EF1a 常规表 达用 mRNA 人延长因子1α来 源的强哺乳动物表达启动子 组成型 表达水平十分稳定,与细胞类型无关 PGK1 (人/小鼠) 常规表 达用 mRNA 磷酸甘油酸酯激酶 基因来源的哺乳动物启动子 组成型 广泛表达,但可能因细胞类型而异。由于甲基化或脱乙酰作用,倾向于抵抗启动子下调。 human beta actin 常规表 达用 mRNA β-肌动蛋白基因来 源的哺乳动物启动子 组成型 无处不在,鸡的启动子常用与启动子 杂交 TRE 常规表 达用 mRNA 四环素响应元件启 动子 被四环素或 者类似物诱导 通常有本底表达
Ac5 常规表 达用mRNA 果蝇Actin 5c 基因 来源的强昆虫启动 子 组成型果蝇表达系统的常用启动子 CaMKIIa 光遗传 学基因 表达 mRNA Ca2+/钙调蛋白依赖 的蛋白激酶 II 启 动子 特异的 用于中枢神经系统/神经元表达。受 到钙和钙调蛋白调节。 TEF1 常规表 达用mRNA 酵母转录延伸因子 启动子 组成型与哺乳动物的EF1a 启动子类似 ADH1 常规表 达用mRNA 乙醇脱氢酶I的酵 母启动子 被乙醇抑制 全长版本很强,促进高表达。截短启 动子是组成型的,表达较低。 Ubi 常规表 达用mRNA 玉米泛素基因的植 物启动子 组成型在植物中促进高表达
U6 小RNA 表达 shRNA 来源于人U6小核启 动子 组成型 小鼠U6也使用,但效率略差。 常用的原核表达系统启动子 T7lac 高水平基因表达 T7噬菌体来源的启动子加上lac 操纵子 几乎没有本底 表达,需要T7 RNA 聚合酶,受 到lac 操纵子的控制,可以被 IPTG 诱导。 常用与pET 载体,受到lac 操纵子的严格调控 Sp6 体外 转录/ 常规 表达 Sp6噬菌体来源的启动子 组成型, 需要SP6 RNA 聚合 酶 当用于体外转录的时候,专路方向有可能是正向的也可能是 反向的,取决于启动子相对于目的基因的方向 araBAD 常规 表达 用 阿拉伯糖代谢操纵 子的启动子 阿拉伯糖诱导 弱,常用与pBAD 载体。适合于快速调控和低的本底表达
真核生物启动子预测相关数据库资源概述
真核生物启动子预测相关数据库资源概述 刘玉瑛1,张江丽2(1.首都师范大学生命科学学院,北京100037;2.廊坊师范学院生命科学学院,河北廊坊065000) 摘要启动子是基因表达调控的重要元件,深入研究启动子的结构和功能,是理解基因转录调控机制和表达模式的关键。随着生物技术和计算机技术的高速发展,应用生物信息学技术对启动子进行预测和分析的方法得到了很大发展。对目前常用的真核生物启动子预测相关数据库和软件资源作了简单介绍。 关键词真核生物;启动子;数据库;预测 中图分类号Q24文献标识码A文章编号0517-6611(2007)24-07418-02 The Databases o f Eukaryo tic Promoters and Related So ftw are Resources LIU Yu2ying et al(Co lleg e of Life Science,Capital N ormal U niv ersity,B eijin g100037) Abstract Eu kary o tic pro mo ters are i mp ortan t elemen ts in reg ulatio n o f the e xpres si on.T o stud y the structu re and functio n o f a p ro m oter deeply,i t is the key to kno w ho w the gene reg ulates its transcri pti on an d starts its exp ression.With the fast d evelo pmen t o f bio log ical and co m puter techno lo gy,sig nifican t ac hiev ements h av e been made in co mp utatio nal predictio n o n Eu kary o tic pro mo ters.In thi s paper mai nly in tro duces the pro g ress made in the datab ases o f predictin g E ukaryo tic p ro mo ters as w ell as the related so ftw are reso urces w as in tro duced. Key w ords Wikipedia;Pro m oter;D atab ase;Predicti on 作为基因表达所必需的重要序列信号和基因转录水平上一种重要的调控元件,真核生物的启动子一直是现代分子生物学的研究热点。用实验的方法分析和鉴定启动子是多年以来进行启动子研究的主要途径。近年来,随着人类基因组测序的完成和根据实验获得的对启动子的序列特征与结构功能的认识,利用生物信息学的方法,通过计算机模拟和计算来预测基因启动子的相关信息获得越来越多的应用。笔者对目前常用的几个启动子预测数据库和相关软件资源作一简单介绍。 1真核生物启动子的基本结构 真核生物的启动子有3种类型,分别由R NA聚合酶?、ò和ó进行转录。典型的真核生物启动子由核心启动子、上游元件和应答元件构成。 核心启动子包括起始子和基本启动子。其中起始子是DN A解链并起始转录的位点。基本启动子序列为中心在-25~-30的7bp保守区,其碱基频率为:T85A97T93A85A2 63A83A50,通常被称为T A T A框或Goldberg2Ho gne ss框,具有选择正确的起始位点,保证精确起始的功能。同时,T A T A框还能影响转录速率。如兔的珠蛋白基因中T A T A框的保守序列A T AAA A人工突变为A TG T AA时,转录效率会下降80%。 上游元件主要包括C AA T框和GC框两种,均具有增强转录活性的功能。其中,C AA T框的保守序列是GGC T2 C A A TC T,一般位于上游-75紧靠-80,与其相互作用的因子有C TF家族的成员C P1、C P2和核因子NF21等;GC框的保守序列是G TGGGC G GG GC AA T,常以多拷贝形式存在-90处,识别该序列的转录激活因子为Sp1。两种上游元件同时存在或者只存在其中之一,但并非所有真核基因的启动子都存在上游启动子元件,有些植物细胞中几乎不存在C AA T框。 应答元件通常位于基因上游,能被转录因子识别和结合,从而调控基因的专一性表达。如热激应答元件、激素应答元件、c A M P应答元件、金属应答元件、糖皮质激素应答元 作者简介刘玉瑛(1982-),女,北京人,硕士研究生,研究方向:生物化学与分子生物学。 收稿日期2007204223件和血清应答元件等。应答元件含有短重复序列,不同基因中应答元件的拷贝数相近。 2真核生物启动子预测相关数据库资源 2.1EPD(Eukaryotic promote r database)[1]EP D数据库(http://w w w.e pd.isb2sib.ch/或者f tp://f tp.e pd.isb2sib.c h/ pub/da taba se s/epd)是一个针对真核R NA聚合酶II型启动子的非冗余数据库。现有启动子序列数据1500多个,按层次组织。关于启动子的描述信息直接摘自科学文献。该数据库中所有的启动子均经过一系列实验证实,如:是否为真核R NA聚合酶ò型启动子、是否在高等真核生物中有生物学活性、是否与数据库中的其他启动子有同源性等。同时,EPD 与其他的相关数据库如EM B L、S WI S S2P RO T、TRA NS FAC等,实现了数据的交叉链接。在其最新版本(第76版)中,EPD 将收集的启动子分为6大类:植物启动子、线虫启动子、拟南芥启动子、软体动物启动子、棘皮类动物启动子和脊椎动物启动子,共2997个条目,其中人类启动子有1871个,约占总数的62%。EPD数据库是目前唯一一个源自实验数据的真核生物启动子数据库,是常用的预测软件测评的手段之一。 2.2PLAC E(Plant cis2ac ting regulatory DNA elements)[2] P LAC E数据库(http://w ww.dna.af frc.go.jp/htdoc s/PL AC E/, F TP服务器为ftp://ftp.dna.a ff rc.go.jp/)是从已发表文献中搜集植物顺式作用元件资料而建立的模体数据库(mo tif data base),始于1991年。目前服务器位于日本农林渔业部。P LAC E数据库中只囊括维管植物的信息,其他与植物顺式作用元件同源的非植物模体也同时被收录。并且所收录信息根据实验最新进展随时得到更新。同时,PL AC E数据库中还包括了对每个模体的描述和在PubM ed中的相关文献编号,以及在DDB J/E MB L/GenB ank的核酸序列数据库的登录号,点击后可阅读相关文献摘要等信息。登陆PL AC E数据库界面,用户可通过关键词、S RS关键词或者同源序列查询顺式作用元件的信息。关键词可以是模体名称、涉及的诱导子或者植物激素、胁迫类型、该基因表达的组织或者器官、原始文献的作者、模体序列、植物种属等。查询结果显示位点(模体)名称、位置、序列和PL ACE登录号,同时,也可以用F AS T A 格式批量上传序列信息。 安徽农业科学,J ou rnal o f Anh ui Ag ri.Sci.2007,35(24):7418-7419责任编辑孙红忠责任校对李洪
原核真核启动子及BL21相关知识
感受态BL21、BL21(DE3)及BL21(DE3)pLysS BL21没有T7 RNA聚合酶基因, 因此不能表达T7启动子控制的目的基因, 但是可以表达大肠杆菌启动子lac、tac、trc及trp控制的基因。 BL21(DE3)是λDE3溶原菌,带有T7 RNA聚合酶,可用于表达T7启动子控制的目的基因,也可以表达大肠杆菌启动子lac、tac、trc及trp控制的基因。 BL21(DE3)pLysS含有pLysS质粒,一种与pET共存的表达T7裂解酶的质粒,可以减少目的基因的本底表达,提供更严紧的控制。 真核原核启动子 原核: 几个常用的启动子和诱导调控表达系统 1.最早应用于的表达系统的是Lac乳糖操纵子,由启动子lacP + 操纵基因lacO + 结构基因组成。其转录受CAP正调控和lacI负调控。 https://www.360docs.net/doc/893824361.html,cUV5突变能够在没有CAP的存在下更有效地起始转录,该启动子在转录水平上只受lacI 的调控,因而随后得到了更广泛采用。lacI产物是一种阻遏蛋白,能结合在操纵基因lacO 上从而阻遏转录起始。乳糖的类似物IPTG可以和lacI产物结合,使其构象改变离开lacO,从而激活转录。这种可诱导的转录调控成为了大肠杆菌表达系统载体构建的常用元件。 3.tac启动子是trp启动子和lacUV5的拼接杂合启动子,且转录水平更高,比lacUV5更优越。 4.trc启动子是trp启动子和lac启动子的拼合启动子,同样具有比trp更高的转录效率和受lacI阻遏蛋白调控的强启动子特性。 5.在常规的大肠杆菌中,lacI阻遏蛋白表达量不高,仅能满足细胞自身的lac操纵子,无法应付多拷贝的质粒的需求,导致非诱导条件下较高地表达,为了让表达系统严谨调控产物表达,能过量表达lacI阻遏蛋白的lacIq 突变菌株常被选为Lac/Tac/trc表达系统的表达菌株。现在的Lac/Tac/trc载体上通常还带有lacIq 基因,以表达更多lacI阻遏蛋白实现严谨的诱导调控。 6.IPTG广泛用于诱导表达系统,但是IPTG有一定毒性,有人认为在制备医疗目的的重组蛋白并不合适,因而也有用乳糖代替IPTG作为诱导物的研究。另外一种研究方向是用lacI 的温度敏感突变体,30℃下抑制转录,42℃开始发挥作用。热诱导不用添加外来的诱导物,成本低,但是由于发酵过程中加热升温比较慢而影响诱导效果,而且热诱导本身会导致大肠杆菌的热休克蛋白激活,一些蛋白酶会影响产物的稳定。 7.以λ噬菌体转录启动子PL、PR 构建的载体也为大家所熟悉。这两个强启动子受控于λ噬菌体cI基因产物。cI基因的温度敏感突变体cI857(ts)常常被用于调控PL、PR启动子的转录。同样也是30℃下阻遏启动子转录,42℃下解除抑制开始转录。同样的,PL、PR表达载体需要以cI857(ts)作为表达菌株,现在更常见的做法是在载体上携带cI857(ts)基因,所以可以有更大的宿主选择范围。另外一种思路是通过严谨调控cI产物来间接调控PL、PR 启动子的转录。比如Invitrogen的PL表达系统,就是将受trp启动子严谨调控的cI基因
[参考实用]常见真核启动子及原核启动子特点
真核启动子 EF1a 常规 表达用 mRNA 人延长因子 1α来源的强哺乳动物表达启动子 组成型 表达水平十分稳定,与细胞类型无关 PGK1(人/小鼠) 常规 表达用 mRNA 磷酸甘油酸 酯激酶基因来源的哺乳动物启动子 组成型 广泛表达,但可能因细胞类型而异。由于甲基化或脱乙酰作用,倾向于抵抗启动子下调。
humanbetaactin 常规 表达 用 mRNA β-肌动蛋白 基因来源的 哺乳动物启 动子 组成型 无处不在,鸡的启动子 常用与启动子杂交 TRE 常规 表达 用 mRNA 四环素响应 元件启动子 被四环 素或者 类似物 诱导 通常有本底表达 Ac5 常规 表达 用 mRNA 果蝇Actin5c 基因来源的 强昆虫启动 子 组成型 果蝇表达系统的常用 启动子
CaMKIIa 光遗 传学 基因 表达 mRNA Ca2+/钙调 蛋白依赖的 蛋白激酶II启 动子 特异的 用于中枢神经系统/神 经元表达。受到钙和钙 调蛋白调节。 TEF1 常规 表达 用 mRNA 酵母转录延 伸因子启动 子 组成型 与哺乳动物的EF1a启 动子类似 ADH1 常规mRNA 乙醇脱氢酶I被乙醇全长版本很强,促进高
表达用 的酵母启动子 抑制 表达。截短启动子是组成型的,表达较低。 Ubi 常规 表达用 mRNA 玉米泛素基 因的植物启 动子 组成型 在植物中促进高表达 U6 小 RNA 表达 shRNA 来源于人U6小核启动子 组成型 小鼠U6也使用,但效率略差。 常用的原核表达系统启动子
T7lac 高 水 平 基 因 表 达 T7噬菌体 来源的启动 子加上lac 操纵子 几乎没有本底表 达,需要T7RNA 聚合酶,受到lac 操纵子的控制, 可以被IPTG诱 导。 常用与pET载体,受到lac操纵子的严 格调控
常见真核启动子及原核启动子特点精编版
常见真核启动子及原核 启动子特点 集团企业公司编码:(LL3698-KKI1269-TM2483-LUI12689-ITT289-
真核 启动子 EF1a 常规 表达用 mRNA 人延长因子 1α来源的强哺乳动物表达启动子 组成型? 表达水平十分稳定, 与细胞类型无关 PGK1(人/小鼠) 常规 表达用 mRNA 磷酸甘油酸 酯激酶基因 来源的哺乳 动物启动子 组成型? 广泛表达,但可能因 细胞类型而异。由于 甲基化或脱乙酰作 用,倾向于抵抗启动 子下调。 humanbetaactin 常规 表达用 mRNA β-肌动蛋白基因来源的哺乳动物启动子 组成型? 无处不在,鸡的启动 子常用与启动子杂交
TRE 常规 表达 用 mRNA 四环素响应 元件启动子 被四环 素或者 类似物 诱导 通常有本底表达 Ac5常规 表达 用 mRNA 果蝇Actin5c 基因来源的 强昆虫启动 子 组成型? 果蝇表达系统的常用 启动子 CaMKIIa 光遗 传学 基因 表达 mRNA? Ca2+/钙调蛋 白依赖的蛋 白激酶II启 动子 特异的 用于中枢神经系统/ 神经元表达。受到钙 和钙调蛋白调节。 GAL1,10常规 表达 用 mRNA 酵母双向启 动子 被半乳 糖诱 导,被 葡萄糖 抑制 可以单独使用或一起 使用。受GAL4和 GAL80调节。 TEF1常规 表达 用 mRNA 酵母转录延 伸因子启动 子 组成型? 与哺乳动物的EF1a 启动子类似
ADH1常规 表达 用 mRNA 乙醇脱氢酶I 的酵母启动 子 被乙醇 抑制 全长版本很强,促进 高表达。截短启动子 是组成型的,表达较 低。 Ubi 常规 表达 用 mRNA 玉米泛素基 因的植物启 动子 组成型?在植物中促进高表达 U6小 RNA 表达 shRNA 来源于人U6 小核启动子 组成型? 小鼠U6也使用,但效 率略差。 常用的原核表达系统启动子 T7lac 高水 平基 因表 达 T7噬菌体 来源的启 动子加上 lac操纵 子 几乎没有本底 表达,需要 T7RNA聚合 酶,受到lac 操纵子的控 制,可以被 常用与pET载体,受到lac操纵子 的严格调控
非编码区和编码区、真核生物的启动子、终止子,真核生物RNA的修饰
基因是由成千上万个核苷酸对组成。组成基因的核苷酸序列可以分为不同区段。在基因表达的过程中,不同区段所起的作用不同。在遗传学上通常将能编码蛋白质的基因称为结构基因。任何一个基因都包括非编码区和编码区。能够转录为相应信使RNA,进而指导蛋白质合成(也就是能编码蛋白质)的区段叫做编码区。不能转录为信使RNA、不能编码蛋白质的区段叫做非编码区。非编码区位于编码区前后,同属于一个基因,控制基因的表达和强弱。 原核生物的基因 非编码区虽然不能编码蛋白质,但对遗传信息的表达是不可缺少的,因为在它上面由调控遗传信息表达的核苷酸序列,该序列中最重要的是位于编码区上游的RNA聚合酶结合位点。启动子、终止子属于非编码区。因为回文序列的特殊排列,大多都位于非编码区。 原核基因的编码区全部编码蛋白质,真核生物的结构基因是断裂的基因。一个断裂基因能够含有若干段编码序列,可以编码蛋白质的序列称为外显子。在两个外显子之间被一段不编码的间隔序列隔开,这些间隔序列称为内含子。非编码区在每个断裂基因的第一个和最后一个外显子的外侧,有人称其为侧翼序列。在侧翼序列上有一系列调控序列。 真核细胞的基因中编码区特点:间隔的、不连续的。包括:外显子和内含子(位于编码区中的非编码序列)。
通常把基因转录起点前面即5’端的序列称为上游(upstream),起点后面即3’端的序列称为下游(downstream)。并把起点的位置记为十1,下游的核苷酸依次记为+2,+3,……,上游方向依次记为-1,-2,-3,……。 非编码区的调控序列主要有以下几种结构: ①在5′端转录起始点上游约20~30个核苷酸的地方,有TATA框(TATA box)。TATA框是一个短的核苷酸序列,其碱基顺序为TATAATAAT。TATA框是启动子(见下)中的一个顺序,它是RNA聚合酶的重要的接触点,能够使酶准确地识别转录的起始点并开始转录。当TATA框中的碱基顺序有所改变时,mRNA 的转录就会从不正常的位置开始。 ②在5′端转录起始点上游约70~80个核苷酸的地方,有CAAT框(CAAT box)。CAAT框是启动子中另一个短的核苷酸序列,其碱基顺序为GGCTCAATCT。CAAT框是RNA聚合酶的另一个结合点,它的作用还不很肯定,但一般认为它控制着转录的起始频率,而不影响转录的起始点。当这段顺序被改变后,mRNA 的形成量会明显减少。 ③在5′端转录起始点上游约100个核苷酸以远的位置,有些顺序可以起到增强转录活性的作用,它能使转录活性增强上百倍,因此被称为增强子。当这些顺序不存在时,可大大降低转录水平。研究表明,增强子通常有组织特异性,这是因为不同细胞核有不同的特异因子与增强子结合,从而对不同组织、器官的基因表达有不同的调控作用。例如,人类胰岛素基因5′末端上游约250个核苷酸处有一组织特异性增强子,在胰岛素β细胞中有一种特异性蛋白因子,可以作用于这个区域以增强胰岛素基因的转录。在其他组织细胞中没有这种蛋白因子,所以也就没有此作用。这就是为什么胰岛素基因只有在胰岛素β细胞中才能很好表达的重要原因。 ④在3′端终止密码的下游有一个核苷酸顺序为AATAAA,这一顺序可能对mRNA的加尾(mRNA尾部添加多聚A)有重要作用。这个顺序的下游是一个反向重复顺序。这个顺序经转录后可形成一个发卡结构。发卡结构阻碍了RNA聚合酶的移动。发卡结构末尾的一串U与转录模板DNA中的一串A之间,因形成的氢键结合力较弱,使mRNA与DNA杂交部分的结合不稳定,mRNA就会从模板上脱落下来,同时,RNA聚合酶也从DNA上解离下来,转录终止。AATAAA顺序和它下游的反向重复顺序合称为终止子(见下),是转录终止的信号。 启动子和终止子: 启动子(promoter)位于编码区上游的非编码区中。真核生物启动子包括下列几种不同顺序,能促进转录过程: (1)帽子位点:转录的起始位点。 (2)TATA框(TATA box):又称Hogness框,类似于原核生物的Pribnow框,决定了转录起点的选择。 其一致顺序为TATAATAAT。约在基因转录起始点上游约-30-50bp处,基本上由A-T碱基对组成,为RNA 聚合酶的结合处之一,RNA聚合酶与TATA框牢固结合之后才能开始转录。 (2)CAAT框(CAAT box):其一致顺序为GGGTCAATCT,是真核生物基因常有的调节区,位于转录起始点上游约-80-100bp处,可能也是RNA聚合酶的一个结合处,控制着转录起始的频率。 (3)GC框(GC box):有两个拷贝,位于CAAT框的两侧,由GGCGGG组成,转录因子Sp1能识别GC框并且与之结合,其N端有激活转录的作用。所以,GC框是一个转录调节区,有激活转录的功能。 (4)增强子(enhancer):又称远上游序列(far upstream sequence)。一般都在-1OO以上。增强子的作用主要是对依赖于TATA框的转录和不依赖TATA框的转录都有增强效应,但对前者增强效应高。增强子是通过启动子来增加转录的。有效的增强子可以位于基因的5’端,也可位于基因的3’端,有的还可位于基因的内含子中。增强子的效应很明显,一般能使基因转录频率增加10~200倍,有的甚至可以高达上千倍。
真核生物启动子的鉴定方法
真核生物启动子的鉴定方法 启动子是RNA聚合酶识别、结合和开始转录的一段DNA序列。它对基因的表达调控起着至关重要的作用。本文对真核生物启动子的鉴定方法作了简要的总结,并对其优缺点进行了比较。 标签:真核;启动子;表达调控;RNA 在原核生物中,RNA多聚酶结合的位置即为启动子[1]。但在真核生物中,启动子一直没有明确的概念,一般认为启动子是位于基因编码区上游,与RNA 聚合酶相互识别、结合并启动转录的DNA序列,它包括在起始位点附近直接活化或抑制转录的DNA序列元件。启动子的定位是研究转录起始调控模式的前提,能否对其精确定位是后续功能性分析成败的关键[2]。本文对真核启动子的鉴定方法进行了简要的综述,并比较了优缺点。 1S1核酸酶法 S1核酸酶法[3]由Arnold Berk和Philip Sharp于1977年创立,近年来关于该方法的优化有许多报道[4-5]。其基本原理是首先构建噬菌体M13衍生型质粒,该质粒包括含有假定转录起始位点和其下游的部分核苷酸的基因区域、限制性酶切位点。将引物和单链噬菌体DNA进行退火,加入Klenow和dNTPs,其中一种dNTP经放射性标记,以产生放射性标记的探针。利用限制性内切酶将DNA 切割成线形,通过变性凝胶电泳分离放射性探针,再将探针和分离到的mRNA 进行杂交。然后用S1核酸酶进行消化,单链RNA和DNA被消化,生成含有部分核苷酸的双链片段。通过变性聚丙烯酰胺凝胶电泳确定放射性标记的抗S1核酸酶的产物大小,最后进行放射自显影或磷酸成像分析。抗性产物的长度应该与从探针的5′末端到转录起始位点的距离相等。这种方法的缺点是杂交分子易发生瞬间解离,产生相当多的背景条带。 2引物延伸方法 引物延伸方法[6]由P.K.Ghosh和S.M.Weissman于1978年提出。首先根据与起始位点5′末端下游大约50~150个核苷酸的mRNA序列设计合成寡核苷酸引物,并将引物的5′末端用32p标记。在根据经验确定的反应条件下,使过量的放射性标记引物与总RNA中特异的RNA分子或经过寡聚dT纯化的mRNA退火。将反转录酶、脱氧核苷三磷酸和适宜的缓冲液加到引物-mRNA杂交分子体系中,催化引物延伸到mRNA的5′末端。用变性聚丙烯酰胺凝胶电泳分析产生的放射性标记的cDNA产物。通过与旁边的标准分子量比较,在凝胶中检测条带的分子量大小。然后计算从合成的寡核苷酸5′末端到mRNA转录起始位点的距离。如果标记的cDNA产物在凝胶的分辨率范围内,转录起始位点的确定可精确到±1个核苷酸。此方法的缺点是很难找到与新基因有效杂交的引物,而且经常出现背景带,另外,有些mRNA 5’末端富含G/C且产生稳定的二级结构,使得反转录
研究真核生物启动子结构与功能的方法
研究真核生物启动子结构与功能的方法 研究启动子结构与功能的方法主要有缺失、点突变和足迹法。在分析得到了启动子的功能序列后,还要弄清与之结合的蛋白质及两者间的相互作用。在研究真核生物启动子的结构与功能时,常采用下列方法。 (1)卵细胞系统(oocyte system)该方法是将DNA直接注射人爪蟾卵细胞的细胞核,分析和观察RNA的转录情况。该方法的局限性在于试验条件受卵细胞内条件的限制。可以用来分析:DNA片段的特性,不能用于分析蛋白质因子与DNA间的结合。 (2)转染系统(transfection system)将外源DNA导人转染的细胞并使之表达。表达可分为瞬时表达(transient expression)和整合表达(integrant expression)。由于转录是在细胞内完成的,可以看成是一种体内试验系统。但外源基因又不是细胞所固有的,和细胞固有基因的表达尚有差别。使用多种宿主细胞,可提高该系统的应用价值。(4)转基因系统(transgenic system)转基因系统将外源基因整合人动物的生殖细胞,使外源基因在部分或全部组织中表达。该系统和转染系统有一些相同的局限性,即外源基因常以多拷贝存在,整合的位置也和内源性基因不同。 (4)体外转录系统(in vitro system)体外转录系统是一种经典的方法。它应用体外转录的方法,结合缺失突变和点突变,来筛选哪些序列是启动子的功能所必需的,哪些序列对启动子的功能有影响,以及
哪些辅助因子对启动子或启动子中的某一片段有何种作用。 启动子研究的第一步是确定启动子的位置及长度。主要方法是用缺失试验来确定启动子的上游边界,即当缺失影响转录始时,说明该处就是启动子的上游边界;用缺失试验结合重组试验来确定下游边界。确定了启动子的位置后,可采用点突变来研究每个碱基在启动子中所起的作用。 研究蛋白质辅助因子与DNA(启动子)的相互作用可采用DNase、足迹法、凝胶阻滞法和硫酸二甲酯方法等。 关于转录调控或基因启动子的研究方法很多,除了较常用的报告基因(Luciferase), CHip, EMSA, pull-down外,还有结合芯片的方法.下面重点介绍五种: 1.Protein Arrays(蛋白芯片) What they are:固定的转录因子阵列,利用标记的基因序列或蛋白质进行探索. What to use them for:用于发现和证实转录因子的结合位点或蛋白-蛋白相互作用. Pros(优点):能提供一种在广泛的因子中检测与特定序列结合的因子.
真核生物三类启动子
真核生物启动子有三类,分别由RNA 聚合酶Ⅰ、Ⅱ和Ⅲ进行转录。 类别Ⅰ(class Ⅰ)启动子: 只控制rRNA 前体基因的转录,转录产物经切割和加工后生成各种成熟rRNA 。 类别Ⅰ启动子由两部分保守序列组成: 核心启动子(core promoter ):位于转录起点附近,从-45至+20; 上游控制元件(upstream control element ,UCE ):位于-180至-107; RNA 聚合酶Ⅰ对其转录需要2种因子参与: UBF1:一条M 为97000的多肽链,结合在上述两部分的富含GC 区; 1个TBP ,即TATA 结合蛋白(TATA-binding protein ,TBP ); SL1:一个四聚体蛋白,含有 3个不同的转录辅助因子TAF Ⅰ; 在SL1因子介导下RNA 聚合酶Ⅰ结合在转录起点上并开始转录。 类别Ⅱ(class Ⅱ)启动子: 类别Ⅱ启动子涉及众多编码蛋白质的基因表达的控制。 该类启动子包含4类控制元件: 基本启动子(basal promoter ):序列为中心在-25至-30左右的7 bp 保守区,TATAAAA/T , 称为TATA 框或Goldberg-Hogness 框。与RNA 聚合酶的定 位有关,DNA 双链在此解开并决定转录的起点位置。失去 TATA 框,转录将在许多位点上开始。 起始子(initiator ):转录起点位置处的一保守序列,共有序列为:P y P y ANT(A)P y P y P y 为嘧啶碱(C 或T ),N 为任意碱基,A 为转录的起点。DNA 在此 解开并起始转录。 上游元件(upstream factor ):普遍存在的上游元件有CAAT 框、GC 框和八聚体(octamer ) 框等。CAAT 框的共有序列是GCCAATCT ,GC 框的共有序 列为GGGCGG 和CCGCCC ,八聚体框含有8bp ,共有序列 为ATGCAAAT ; 应答元件(response element ):诱导调节产生的转录激活因子与靶基因上的应答元件结合。 如热休克效应元件HSE 的共有序列是 CNNGAANNTCCNNG ,可被热休克因子HSF 识别和作用; 血清效应元件SRE 的共有序列CCATATTAGG ,可被血清效 应因子SRF 识别和作用。 +1
真核生物启动子的鉴定方法
龙源期刊网 https://www.360docs.net/doc/893824361.html, 真核生物启动子的鉴定方法 作者:石慧李建远 来源:《中外医学研究》2012年第08期 【摘要】启动子是RNA聚合酶识别、结合和开始转录的一段DNA序列。它对基因的表 达调控起着至关重要的作用。本文对真核生物启动子的鉴定方法作了简要的总结,并对其优缺点进行了比较。 【关键词】真核;启动子;表达调控;RNA 中图分类号R318.04 文献标识码B 文章编号1674-6805(2012)08-0154-02 在原核生物中,RNA多聚酶结合的位置即为启动子[1]。但在真核生物中,启动子一直没有明确的概念,一般认为启动子是位于基因编码区上游,与RNA聚合酶相互识别、结合并启动转录的DNA序列,它包括在起始位点附近直接活化或抑制转录的DNA序列元件。启动子 的定位是研究转录起始调控模式的前提,能否对其精确定位是后续功能性分析成败的关键[2]。本文对真核启动子的鉴定方法进行了简要的综述,并比较了优缺点。 1S1核酸酶法 S1核酸酶法[3]由Arnold Berk和Philip Sharp于1977年创立,近年来关于该方法的优化有许多报道[4-5]。其基本原理是首先构建噬菌体M13衍生型质粒,该质粒包括含有假定转录起始位点和其下游的部分核苷酸的基因区域、限制性酶切位点。将引物和单链噬菌体DNA进行退火,加入Klenow和dNTPs,其中一种dNTP经放射性标记,以产生放射性标记的探针。利用限制性内切酶将DNA切割成线形,通过变性凝胶电泳分离放射性探针,再将探针和分离到的mRNA进行杂交。然后用S1核酸酶进行消化,单链RNA和DNA被消化,生成含有部分核苷酸的双链片段。通过变性聚丙烯酰胺凝胶电泳确定放射性标记的抗S1核酸酶的产物大小,最后进行放射自显影或磷酸成像分析。抗性产物的长度应该与从探针的5′末端到转录起始位点的距离相等。这种方法的缺点是杂交分子易发生瞬间解离,产生相当多的背景条带。 2引物延伸方法 引物延伸方法[6]由P.K.Ghosh和S.M.Weissman于1978年提出。首先根据与起始位点5′末端下游大约50~150个核苷酸的mRNA序列设计合成寡核苷酸引物,并将引物的5′末端用32p 标记。在根据经验确定的反应条件下,使过量的放射性标记引物与总RNA中特异的RNA分子或经过寡聚dT纯化的mRNA退火。将反转录酶、脱氧核苷三磷酸和适宜的缓冲液加到引物-mRNA杂交分子体系中,催化引物延伸到mRNA的5′末端。用变性聚丙烯酰胺凝胶电泳分析产生的放射性标记的cDNA产物。通过与旁边的标准分子量比较,在凝胶中检测条带的分子量
常见真核启动子及原核启动子特点之欧阳歌谷创作
真核启动子 EF1a 常规表达用 mRNA 人延长因子1α 来源的强哺乳动物表达启动子 组成型 表达水平十分稳定,与细 胞类型无关 PGK1 (人/小鼠) 常规表达用 mRNA 磷酸甘油酸酯 激酶基因来源 的哺乳动物启 动子 组成型 广泛表达,但可能因细胞类型而异。由于甲基化或脱乙酰作用,倾向于抵抗启动子下调。 human beta actin 常规表达用 mRNA β-肌动蛋白基 因来源的哺乳动物启动子 组成型 无处不在,鸡的启动子常 用与启动子杂交
TRE 常规表达用 mRNA 四环素响应元 件启动子 被四环素 或者类似物诱导 通常有本底表达 Ac5 常规表达用 mRNA 果蝇Actin 5c 基因来源的强昆虫启动子 组成型 果蝇表达系统的常用启动 子 CaMKIIa 光遗传 学基因表达 mRNA Ca2+/钙调蛋 白依赖的蛋白 激酶 II 启动 子 特异的 用于中枢神经系统/神经元表达。受到钙和钙调蛋白调节。 TEF1 常规表达用 mRNA 酵母转录延伸因子启动子 组成型 与哺乳动物的EF1a 启动 子类似 ADH1 常规表达用 mRNA 乙醇脱氢酶I 的酵母启动子 被乙醇抑 制 全长版本很强,促进高表 达。截短启动子是组成型 的,表达较低。
Ubi 常规表达用 mRNA 玉米泛素基因 的植物启动子 组成型 在植物中促进高表达 U6 小RNA 表达 shRNA 来源于人U6小核启动子 组成型 小鼠U6也使用,但效率略 差。 常用的原核表达系统启动子 T7lac 高水平基因表达 T7噬菌体 来源的启 动子加上 lac 操纵子 几乎没有本底表达,需要T7 RNA 聚合酶, 受到lac 操纵子的控制,可 以被IPTG 诱导。 常用与pET 载体,受到lac 操纵子的严格调控 Sp6 体外 转录/常规表达 Sp6噬菌体来源的启动子 组成型, 需要SP6 RNA 聚合酶 当用于体外转录的时候,专路方向有可能是正向的也可能是反向的,取决于启动子相对于目的基因的方向 araBAD 常规 表达 用 阿拉伯糖代谢操纵 子的启动子 阿拉伯糖诱导 弱,常用与pBAD 载体。适合于快速调控和低的本底表达
DNA启动子概述
启动子概述 启动子是DNA链上一段能与RNA聚合酶结合并能起始mRNA合成的序列,它是基因表达不可缺少的重要调控序列。启动子是一段位于结构基因5’-端上游区的DNA序列,能活化RNA聚合酶,使之与模板DNA准确地结合,并具有转录起始的特异性。基因的特异性转录取决于酶与启动子能否有效地形成二元复合物。启动子分三类:启动子Ⅰ、启动子Ⅱ、启动子Ⅲ.只有启动子Ⅱ指导mRNA的转录。真核生物启动子Ⅱ由两大部分组成:上游元件(upstream element)和启动子核心(core promoter)。上游元件与转录的效率有关;启动子核心包括3部分:TATA 盒、起始子(initinator)及下游元件(downstream element)。TATA盒为转录调控因子包括各种调节蛋白的结合区,与转录起始位点的精确选择及转录有关,起始子是转录起始所必须,下游元件作用尚不清楚。原核生物启动子区范围较小,包括TATAAT区(Pribnow区)及其上游的TTGACA区。 启动子是一段提供RNA聚合酶识别和结合位点的DNA序列,位于基因上游。启动子具有如下特征: 1序列特异性。在启动子的DNA序列中,通常含有几个保守的序列框,序列框中碱基的变化会导致转录启动活性的改变。 2方向性。启动子是一种有方向性的顺式调控元件,有单向启动子和双向启动子两类。 3位置特性。启动子一般位于所启动转录基因的上游或基因内的前端。处于基因的下4种属特异性。原核生物的不同种、属,真核生物的不同组织都具有不同类型的启动 没有启动子,基因就不能转录。原核生物启动子是由两段彼此分开且又高度保守的核苷酸序列组成,对mRNA的合成极为重要。启动子区域:(1)Pribnow盒,位于转录起始位点上游5—10bp,一般由6~8个碱基组成,富含A和T, 故又称为TATA盒或—10区。启动子来源不同,Pribnow盒的碱基顺序稍有变化。(2)—35区,位于转录起始位点上游35bp处,故称—35区,一般由10个碱基组成。 质粒设计时都需要加入启动子序列,以保证目的基因的表达。启动子可分为诱导型启动子和组成型启动子两大类,后者包括CMV,SV40,T7,pMC1,PGK启动子等。一下介绍几个常见的启动子。 (1)U6启动子 U6是二型启动子,一般发现是启动小片段,不带PolyA尾的序列。由Ⅲ类RNA聚合酶启动子U6启动子转录产生shRNA,经剪切后产生成熟siRNA,产生干扰效果。这一类 启动子在腺病毒和慢病毒干扰载体的构建中应用很多。U6更多的是用在shRNA的启动,来达到敲低一个基因的作用。
