载体构建流程v3

DNA重组技术实验原理、方法和步骤
实验原理
1.DNA重组技术
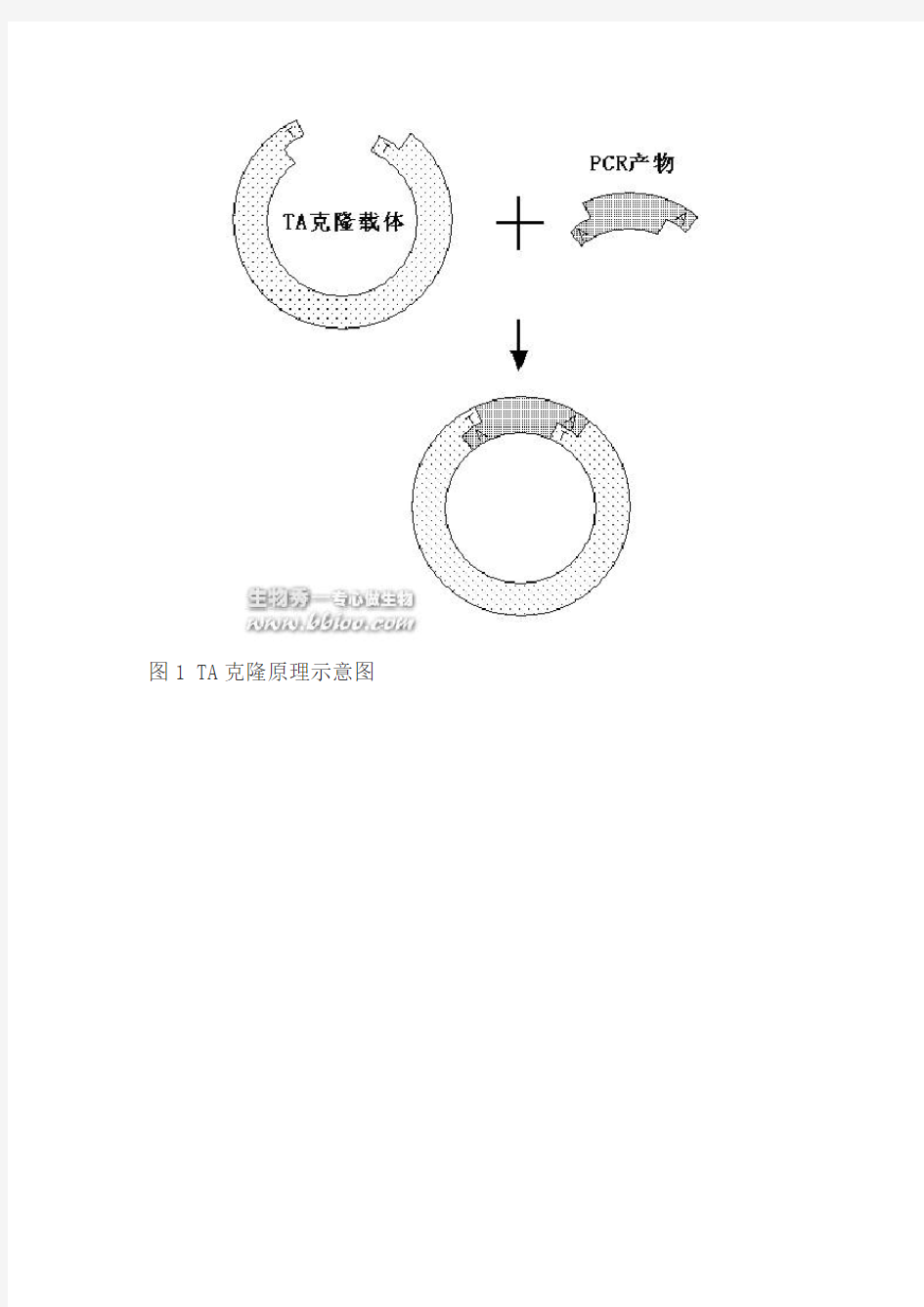
重组DNA(Recombinant DNA)技术是遗传工程的核心技术,也是人类在基因和DNA分子水平进行操作的技术。它包括以下几个步骤:1)重组DNA分子的构建:即将目的基因(DNA或cDNA片段)与载体DNA重组,应用TA克隆方法,将PCR扩增产物快速克隆至质粒载体中。该方法利用了PCR过程中使用的热稳定聚合酶(Taq酶)在所复制的双链分子末端加上单一脱氧腺苷酸(A),从而产生一A-粘性末端的性质,设计一种线性载体,使之含有一T-粘性末端,可直接插入PCR产物,在DNA连接酶作用下,目的DNA和载体DNA连接形成重组DNA分子。2)将重组DNA分子转化宿主细胞。
3)克隆选择增殖:将转化后的液体涂布于琼脂培养基表面,培养基中含有宿主菌敏感的抗生素,重组DNA(TA克隆载体分子)含有相应抗生素的抗性基因,转化的细菌能在培养基上生长形成集落。挑取菌落,置液体培养基中培养,得到大量含重组DNA的细菌。
4)收获扩增后的培养细胞,提取重组DNA分子(质粒),并纯化,获得某一基因或DNA片段的大量拷贝。
图1 TA克隆原理示意图
2.质粒DNA的小量制备
常见的质粒DNA提取方法有简易一步提取法、碱裂解法和煮沸法。本实验采用的是碱裂解法,碱裂解法的原理:在PH 12.0~12.6碱性环境中,线性的大分子量细菌染色体DNA变性,而共价闭环质粒(CC)DNA仍为自然状态。将pH调至中性并有高盐浓度存在的条件下,染色体DNA之间交联形成不溶性网状结构。大部分染色体DNA和蛋白质在去污剂SDS的作用下形成沉淀,而质粒DNA仍为可溶状态。通过离心,将可去除大部分细胞碎片、染色体DNA、RNA及蛋白质,质粒DNA 尚在上清中,再用乙醇沉淀回收质粒DNA。
3.质粒DNA的限制性内切核酸酶(限制酶)切分析
限制酶存在于原核生物体内,根据其结构和功能特性分为:Ⅰ、Ⅱ、
Ⅲ型。Ⅰ型切点不固定,Ⅲ型其切割位点在识别位点之外,只有Ⅱ型其特异性切点位置可以控制和预测,可以产生固定的DNA片段,基因工程中所用的限制酶均为Ⅱ型限制酶。这类酶的特点是具有能够识别双链DNA分子上的特异性核苷酸序列的能力,能在这个特异性核苷酸序列内切断DNA双链,形成一定长度和顺序的DNA片段。选择合适的限制酶对重组的环状质粒进行酶切,将酶切产物进行琼脂糖凝胶电泳检测,用已知相对分子量的线状DNA(DNA分子量标志)及未经酶切的重组环状质粒为对照,可以对重组环状质粒中的目的DNA分子进行初步鉴定
以下所有实验,进行前请认真阅读试剂使用说明书。
一.RNA反转录cDNA
实验原理:RT-PCR是将RNA的反转录(RT)和cDNA的聚合酶链式扩增(PCR)相结合的技术。首先经反转录酶的作用从RNA合成cDNA,再以cDNA为模板,扩增合成目的片段。RT-PCR技术灵敏而且用途广泛,可用于检测细胞中基因表达水平,细胞中RNA病毒的含量和直接克隆特定基因的cDNA序列。作为模板的RNA可以是总RNA、mRNA或体外转录的RNA产物。
实验材料:
试剂盒:Takara PrimeScript? RT reagent Ki t
实验步骤:
实验所用枪头,EP管,水等全部使用RNase free耗材,或者提前使用DEPC处理。处理方式:按照1/1000比例向无菌水中加入DEPC,将所需耗材在含DEPC的水中浸泡过夜;次日早晨高压灭菌处理。
提前调水浴锅至42℃;
按下列组份配制 RT 反应液(反应液配制请在冰上进行) 。
5×PrimeScript? Buffer(for Real Time) 2 μl
PrimeScript? RT Enzyme Mix I 0.5 μl
Oligo dT Primer(50 μM) 0.5 μl
Random 6 mers(100 μM) 0.5 μl
Total RNA 360ng
RNase Free dH2O up to 10 μl
total 10μl
轻柔吹打均匀;
室温放置10min;
42℃水浴锅中放置1h;
冰中冷却2min;
注意事项:
上述反应可根据条件在PCR仪中进行。
当需要使用的基因编码的RNA二级结构较为复杂时,反转录前,将RNA 溶液于65°C加热 5 分钟后迅速置于冰中,利用这种办法可以减弱二级结构的影响,提高逆转录反应效率。
二.目的基因扩增:引物设计原则:
a)特异性:在cDNA中能且只能扩增出目的序列,不会扩增出其
他序列;(通过blast检验)
b)在引物5’端添加酶切位点及3个保护碱基;
c)酶切位点使用所选载体中有的位点,尽量避免使用载体中相邻
的位点,尽量避免使用平末端位点;所选位点在扩增目的片段
中不能出现,可以使用primer premier对序列中的酶切位点
进行分析。
d)设计引物时建议同时设计合成两对或两对以上引物,以免一对
引物扩增失败时,再合成第二对引物浪费时间。
三.PCR反应:
实验原理:PCR技术的基本原理类似于DNA的天然复制过程,其特异性依赖于与靶序列两端互补的寡核苷酸引物。PCR由变性--退火--延伸三个基本反应步骤构成:①模板DNA的变性:模板DNA经加热至93℃左右一定时间后,使模板DNA双链或经PCR 扩增形成的双链DNA解离,使之成为单链,以便它与引物结合,为下轮反应作准备;②模板DNA与引物的退火(复性):模板DNA经加热变性成单链后,温度降至55℃左右,引物与模板DNA 单链的互补序列配对结合;③引物的延伸:DNA模板--引物结合物在TaqDNA聚合酶的作用下,以dNTP为反应原料,靶序列为模板,按碱基配对与半保留复制原理,合成一条新的与模板DNA 链互补的半保留复制链重复循环变性--退火--延伸三过程,就可获得更多的“半保留复制链”,而且这种新链又可成为下次循环的模板。每完成一个循环需2~4分钟, 2~3小时就能将待扩目的基因扩增放大几百万倍。(Plateau)。到达平台期所需循环次数取决于样品中模板的拷贝。
实验材料:dNTP,PCR buffer,DNA聚合酶,上游引物,下游引物,纯水,模板。
操作步骤:
拿到新合成的引物后,按照引物说明书离心,溶解引物至100Mm。
使用时稀释至10mM。将(Tm-5)℃作为退火温度进行PCR反应并
对PCR产物进行琼脂糖凝胶电泳鉴定。如果存在非特异条带,一般会设置退火温度梯度,摸索最适退火温度。(Tm-5)℃不是一个普适的规律,最好不要太依赖。
4k以下的片段使用KOD Plus DNA 聚合酶,4k以上选择KOD FX DNA 聚合酶,按说明书操作即可;如果4k以下的片断用KOD Plus扩增效果不佳,可以换用KOD FX。
四.PCR产物回收:
实验原理:在高盐状态下,纯化树脂专一性地吸附DNA;而在低盐或水溶液状态下,DNA被洗脱下来。
实验材料:OMEGA cycle-pure kit,OMEGA gel extraction kit 取5微升PCR产物进行琼脂糖凝胶电泳鉴定,如果产物为单一条带,且大小符合预期,直接进行PCR产物回收。
如果存在非特异条带,可以考虑进行胶回收,或者继续优化PCR 反应条件。
配制1%琼脂糖凝胶:称取一克琼脂糖,加入100ml TAE溶液中,煮沸至凝胶完全溶解。待温度降至60°C左右时(刚好不烫手)加入10μl 4S green,摇匀,将凝胶溶液倒入槽内,冷凝后备用。
将反转录样品与Loading Buffer混匀,加入凝胶孔内,最右端加Marker,固定电压180V,跑20分钟左右。此时调水浴锅60°C .
操作步骤:
参考意见:500bp以下的片段使用2%琼脂糖凝胶;500bp以上使用1%琼脂糖凝胶。标准并不绝对,可以根据个人经验进行调整。
在紫外灯下切下相应大小的DNA片段,加入EP管内,按照0.1g 凝胶加入100μl的比例加入buffer xp,放入60°C 水浴锅中至胶完全融化。这段时间组装收集管与吸附柱,并在吸附柱上做好
标记。
紫外照射时间不宜过长,避免DNA形成TT二聚体。
用大枪将EP管内溶液吹匀,吸入吸附柱中,12000rpm离心
1min;
倒掉收集管中废液,向吸附柱中加入300μl buffer XP,最高速离心1min,倒掉收集管中废液,向吸附柱中加入700μl wash buffer,最高速离心1min;
重复上一步骤。
最高速空转2min;
取出吸附柱,放入新离心管中,70℃加热2min,待乙醇挥发;
向吸附柱中加入50μl超纯水,70℃加热2min,12000rpm离心1min,离心管内液体即所需回收的DNA溶液。
五.PCR产物与载体的酶切
实验原理:限制性内切酶能特异地结合于一段被称为限制性酶识别序列的DNA序列之内或其附近的特异位点上,并切割双链DNA。它可分为三类:Ⅰ类、Ⅱ类、Ⅲ类。Ⅰ类和Ⅲ类酶在同一蛋白质分子中兼有切割和修饰(甲基化)作用且依赖于ATP的存在。
Ⅰ类酶结合于识别位点并随机的切割识别位点不远处的DNA,而Ⅲ类酶在识别位点上切割DNA分子,然后从底物上解离。Ⅱ类由两种酶组成: 一种为限制性内切核酸酶(限制酶),它切割某一特异的核苷酸序列; 另一种为独立的甲基化酶,它修饰同一识别序列。Ⅱ类中的限制性内切酶在分子克隆中得到了广泛应用,它们是重组DNA的基础。绝大多数Ⅱ类限制酶识别长度为 4至6个核苷酸的回文对称特异核苷酸序列(如EcoRⅠ识别六个核苷酸序
列:5'- G↓AATTC-3'),有少数酶识别更长的序列或简并序列。Ⅱ类酶切割位点在识别序列中,有的在对称轴处切割,产生平末端的DNA段(如Sma Ⅰ:5'-CCC↓GGG-3');有的切割位点在对称轴一侧,产生带有单链突出末端的DNA段称粘性未端,如EcoRⅠ切割识别序列后产生两个互补的粘性末端。
5'…G↓AATTC…3' →5'… G AATTC…3'
3'…CTTAA↑G …5' →3'… CTTAA G…5'
DNA纯度、缓冲液、温度条件及限制性内切酶本身都会影响限制性内切酶的活性。大部分限制性内切酶不受RNA或单链DNA的影响。当微量的污染物进入限制性内切酶贮存液中时,会影响其进一步使用,因此在吸取限制性内切酶时,每次都要用新的吸管头。
如果采用两种限制性内切酶,必须要注意分别提供各自的最适盐浓度。若两者可用同一缓冲液,则可同时水解。若需要不同的盐浓度,则低盐浓度的限制性内切酶必须首先使用,随后调节盐浓度,再用高盐浓度的限制性内切酶水解。也可在第一个酶切反应完成后,用等体积酚/氯仿抽提,加0.1倍体积3mol/L NaAc和2倍体积无水乙醇,混匀后置-70℃低温冰箱30分钟,离心、干燥并重新溶于缓冲液后进行第二个酶切反应。
操作步骤:
使用takara quick cut限制酶或常规限制酶,酶切体系及条件见相应酶的说明书。
Buffer 5μl Buffer 5μl
酶1 1μl 酶1 1μl
酶2 1μl 酶2 1μl
载体1μg 外源片段 1μg
水补至50μl 水补至50μl
温度根据所用不同酶确定,反应时间一般10min。
注意事项:
双酶切时,两种限制酶反应温度不一致时,可以采用两种方法解决:
1.向体系中加入两种限制酶,先在低温下进行反应,再用较高温度下进行反应
2.先加入一种限制酶,使用相应反应温度进行反应,再加入第二种酶,使用第二种酶的温度进行反应
双酶切时,两种限制酶反应buffer不一致时,可以采用两种方法解决:
1.进行两次酶切,两次酶切之间回收PCR产物;
2.先使用盐离子浓度较低的buffer进行第一次酶切,然后加入相应的限制酶和盐离子,继续进行第二次酶切。
六.连接:
实验原理: DNA 连接酶催化相邻的核酸分子间3' -羟基和5'-磷酸基形成磷酸二酯键.
当两条单链的寡核苷酸与同一条靶序列完全互补配对时,DNA 连接酶可催化一条寡核苷酸3' -羟基末端和另一条寡核苷酸5'-磷酸末端通过磷酸二酯键连接;如果一条寡核苷酸3' -羟基末端的碱基出现了突变错配,DNA 连接酶就连接不上,不催化磷酸二酯键形成.
实验材料:NEB DNA ligase
体系中外源片段与载体的分子数比例控制在1:1~1:10之间。对
于长约5K的载体,一般加入100ng,对于长度为nKb的外源片段,加入(20n~200n)ng。体系中其余成分参考相应酶的说明书。七.转化:
实验原理:细菌处于容易吸收外源DNA 的状态叫感受态。转化是指质粒DNA或以它为载体构建的重组子导入细菌的过程。其原理是细菌处于0℃,CaCl2低渗溶液中,菌细胞膨胀成球形。转化混合物中的DNA形成抗DNA酶的羟基-钙磷酸复合物粘附于细胞表面,经42℃短时间热击处理,促进细胞吸收DNA复合物。将细菌放置在非选择性培养基中保温一段时间,促使在转化过程中获得的新的表型(如Ampr等)得到表达,然后将此细菌培养物涂在含有氨苄青霉素的选择性培养基上。
实验材料:
小枪,小枪头,大枪,大枪头,1.5mlEP管,固体LB培养基,无抗性的液体LB培养基
操作步骤:
超净台内放入小枪,小枪头,大枪,大枪头,1.5mlEP管,固体LB培养基,无抗性的液体LB培养基,紫外照射超净台30min,同时调水浴锅至42℃,同时制冰。以下操作如无特别说明,建议均在冰上进行;
从-80℃冰箱内取出感受态细菌,冰上融化(可以室温融化或在手中捂化,不推荐),融化后立即将感受态分装至EP管中,每管约30-50μl,置于冰上;
吸取5-10μl 转化体系,放入感受态中轻柔吹打数次混匀,做好标记;
将EP管置于冰上30min;
将EP管置于42℃水浴锅中加热90s,期间尽量不要扰动EP管;
取出EP管,置于冰上2min;
向EP管中加入400~800μl无抗性LB液态培养基,摇床内100rpm,37℃摇45min;
将EP管内培养基倒到固体LB培养基上,晃动培养基,使菌液在平皿表面分布均匀;
也可采用涂布法和划线法。三种方法各有优势,个人比较习惯第一种方法;
固体培养基有抗性,抗性的选择根据质粒情况而定;
培养箱内37℃倒置培养过夜;
12-16h后,即可看到固体培养基表面长出单个菌落。TOP10 十个小时
八.挑取单克隆摇菌
超净台内放入小枪,小枪头,移液管(可选),大枪,大枪头,大试管,试管架,有抗性的液体LB培养基,紫外照射超净台30min;
向大试管中加入含相应抗性的LB液体培养基适量
一般4ml或其整数倍。
取培养过夜的LB固体培养基,用小枪头挑取单个菌落,将枪头投入大试管中,做好标记,盖好盖子;
挑取时宜选中等大小的菌落,太大太小均不宜挑取;
有时一个大菌落周围会有几个小菌落围绕大菌落而生,此时优先选择这种大菌落,一定不能挑取周围的小菌落;
37℃摇床内振荡过夜,转速220转/min。
一个板子上挑取8-10个克隆。
注意事项:Amp在37摄氏度下过夜培养后失效,不能再用。
九.质粒提取(翻译omega质粒提取试剂盒说明书)实验原理:当菌体在NaOH和 SDS溶液中裂解时,蛋白质与DNA 发生变性,当加入中和液后,质粒DNA分子能够迅速复性,呈溶解状态,离心时留在上清中;蛋白质与染色体DNA不变性而呈絮状,离心时可沉淀下来。
操作步骤:
取振荡过夜的菌液,分装至4ml离心管中,做好标记;
一定一定要做好标记!
室温下,10000g离心1min;
向4ml离心管中加入250μl solution I,吹打均匀,将菌液转移至1.5ml离心管中,吹打均匀。可以在1.5ml离心管中插入牙签,在votex上涡旋振荡
一定要使细菌重悬均匀;
向离心管中加入250μl solution II,轻柔颠倒EP管6-8次,使菌液变澄清裂解时间控制在5min以内;
不可剧烈吹打涡旋等;
向离心管中加入350μl solution III,迅速颠倒混匀;
一定要迅速、彻底混匀;
加入solution III后立即混匀,防止出现局部沉淀;
室温下13200rpm(最高转速)离心10min;此段时间将吸附柱插入收集管,做好标记;
吸取离心后的上清液,加入吸附柱中;
动作要轻柔,不要吸起沉淀;如果上清液中出现沉淀
室温下10000g离心1min,倒掉收集管中液体,将吸附柱放回收集管中;
向吸附柱中加入500μl Buffer HB,室温下10000g离心1min,倒掉收集管中液体,将吸附柱放回收集管中;
向吸附柱中加入700μl Wash Buffer,室温下10000g离心1min,倒掉收集管中液体,将吸附柱放回收集管中;
重复上步操作;
最高速空转2min;
此步骤不可省略;
取出吸附柱放入干净的EP管中,敞盖70℃加热2min;
向吸附柱中加入60μl Elution Buffer(具体体积根据质粒可能的产量调整),闭盖70℃加热2min;
室温下10000g离心1min;
测量质粒浓度与A260/A280值,标记于管壁,-20℃保存。浓度超过1000ng/微升时,将质粒溶液稀释后再测。调零溶液建议使用溶解DNA的溶液
提取质粒后,取约2微升进行电泳鉴定,观察有无细菌基因组DNA 污染。
十.鉴定:
鉴定一般采取酶切的方式。
用构建载体时所用限制酶对提取的质粒进行酶切,电泳。如果载体构建成功,电泳结果会显示两条带,一条与空载体大小相同,另一条与插入片段大小相同。操作与前述相同
可以在插入片断上和载体上分别选择酶切位点进行酶切。
鉴定插入片断正确的质粒,再次转化摇菌,得到的菌液加入50%甘油,1ml分装,-80摄氏度保存。每个载体可以保留5-6管菌种。
表达载体的构建方法及步骤
表达载体的构建方法及步骤 令狐采学 一、载体的选择及如何阅读质粒图谱 目前,载体主要有病毒和非病毒两大类,其中质粒DNA 是一种新的非病毒转基因载体。 一个合格质粒的组成要素: (1)复制起始位点Ori 即控制复制起始的位点。原核生物DNA 分子中只有一个复制起始点。而 真核生物DNA 分子有多个复制起始位点。 (2)抗生素抗性基因可以便于加以检测,如Amp+ ,Kan+ (3)多克隆位点MCS 克隆携带外源基因片段 (4)P/E 启动子/增强子 (5)Terms 终止信号 (6)加poly(A)信号可以起到稳定mRNA 作用 选择载体主要依据构建的目的,同时要考虑载体中应有合适的限制酶切位点。如果构建的目 的是要表达一个特定的基因,则要选择合适的表达载体。 载体选择主要考虑下述3点: 【1】构建DNA 重组体的目的,克隆扩增/基因表达,选择合适的克隆载体/表达载体。 【2】.载体的类型:
(1)克隆载体的克隆能力-据克隆片段大小(大选大,小选小)。如<10kb 选质粒。 (2)表达载体据受体细胞类型-原核/真核/穿梭,E.coli/哺乳类细胞表达载体。 (3)对原核表达载体应该注意:选择合适的启动子及相应的受体菌,用于表达真核蛋白质时注意克服4个困难和阅读框错位;表达天然蛋白质或融合蛋白作为相应载体的参考。 【3】载体MCS 中的酶切位点数与组成方向因载体不同而异,适应目的基因与载体易于链接,不能产生阅读框架错位。 综上所述,选用质粒(最常用)做载体的5点要求: (1)选分子量小的质粒,即小载体(1-1.5kb)→不易损坏,在细菌里面拷贝数也多(也有大载 体); (2)一般使用松弛型质粒在细菌里扩增不受约束,一般10个以上的拷贝,而严谨型质粒<10个。 (3)必需具备一个以上的酶切位点,有选择的余地; (4)必需有易检测的标记,多是抗生素的抗性基因,不特指多位Ampr(试一试)。 (5)满足自己的实验需求,是否需要包装病毒,是否需要加入荧光标记,是否需要加入标签蛋白,是否需要真核抗性(如Puro、G418)等等。 无论选用哪种载体,首先都要获得载体分子,然后采用适当的限制酶将载体DNA 进行切割,获得线性载体分子,以便于与
载体构建流程
载体构建SOP流程: GenBank查询目的基因序列→根据ORF序列利用引物设计软件设计引物→表达目的基因的组织或细胞总RNA提取→RT-PCR获取目的基因→酶切目的基因和载体→分别纯化酶切的目的基因和载体并建立连接反应→转化→初步筛选阳性克隆→阳性克隆测序→测序正确的质粒保种并重提质粒 I.获取目的基因/序列片段 一.获取序列信息 通过GENBANK数据和生物信息的方法设计目的基因或目的片段引物(shRNA、miRNA)。 PCR引物的设计原则: ①引物应用核酸系列保守区内设计并具有特异性。 ②产物不能形成二级结构。 ③引物长度一般在15~30碱基之间。 ④G+C含量在40%~60%之间。 ⑤碱基要随机分布。 ⑥引物自身不能有连续4个碱基的互补。 ⑦引物之间不能有连续4个碱基的互补。 ⑧引物5′端可以修饰。 ⑨引物3′端不可修饰。
⑩避免在引物的3’端使用碱基A。 在实际设计引物中由于ORF两末端序列本身的限制,不能完全按照上述理想的设计原则,但也切记引物不能过长或过短。过长的引物不容易打开其二级结构,与模板结合缓慢,也容易形成引物二聚体,通常不超过35bp(不包括酶切位点和保护碱基)。过短的引物特异性差,扩出其它不相关片段,最终很难得到目的片段,通常不短于18bp(不包括酶切位点和保护碱基)。要将目的基因定向克隆至相应载体,需要在上下游引物两端设计不同的酶切位点,由于酶切位点位于线性末端时酶对其识别切割能力大大降低,需依据NEB目录添加相应保护碱基,酶切时可相应增加时间。 二.制备模板 1.分离高质量RNA:成功的cDNA合成来自高质量的RNA。高质量的RNA至少应保证全长并且不含逆转录酶的抑制剂,如EDTA或SDS。RNA的质量决定了能够转录到cDNA上的序列信息量的最大值。现在实验室通常使用Trizol试剂法提取总RNA,可以从多种组织和细胞中提取高质量的非降解RNA。Trizol试剂法可以从最少100个细胞或1mg组织中提取RNA。在逆转录反应中经常加入RNase抑制剂以增加cDNA合成的长度和产量。RNase抑制剂要在第一链合成反应中,在缓冲液和还原剂(如DTT)存在的条件下加入,因为cDNA合成前的过程会使抑制剂变性,从而释放结合的可
载体构建的基本步骤
载体构建 一.原理 依赖于限制性核酸内切酶,DNA连接酶和其他修饰酶的作用,分别对目的基因和载体DNA进行适当切割和修饰后,将二者连接在一起,再导入宿主细胞,实现目的基因在宿主细胞内的正确表达。 二.操作步骤 1.摇菌 取装有液体培养基的3ml试管两支(依情况而定),每管加40-100μl菌种,过夜摇。2.提质粒 依照提质粒试剂盒中的说明书操作(根据情况最后一步洗脱时可以多洗1-2次)。 3.酶切 按下表加入试剂。 反应所需试剂体积(单位:ul) 质粒10 所需内切酶反应缓冲液 2 所需限制性内切核酸酶 1 H2O 7 将加好的EP管置于37℃保温1-2h。(依照提酶切的具体步骤操作;为了达到最佳酶切的效果,最好根据所选用的酶确定所需要的反应温度) 4.电泳检测 将酶切产物进行琼脂糖凝胶电泳,检测酶切是否成功。 5.连接 如果电泳检测酶切成功的话,则仔细将所需的片段切割下来,将胶体回收(依照胶回收试剂盒说明书操作);之后将回收的片段和载体连接,连接体系如下: 双蒸水5μL 10×T4 DNA连接缓冲液1μL 载体2μL 酶切后的目的基因1μL T4DNA连接酶1μL 总体积10μL 置于温箱,12-16℃,保温8-16h 6.转化 依照转化具体操作步骤做感受态,将上述连接产物进行转化实验,涂板培养,37℃,12-16h。 7.单克隆检测 (1)挑单克隆
先将AMP从冰箱中取出,待融化后,在3ml装有LB液体培养基的试管中加入3μL的AMP,用枪头混匀;取1.5 mlEP管5支(依情况可以多挑几管),给每支管中加500μL上述培养液,然后用接种环(或黄枪头)挑单克隆,挑完后用枪吹打;之后,将挑好的菌摇4-5小时,至混浊即可。 (2)单克隆检测 以每管摇好的菌液为模板,以原有的引物进行PCR,然后将PCR产物跑电泳,观察电泳图像中那几管的条带正确,将正确条带相对应管的菌液再抽取100μL,加到3ml(有LB液体培养液,AMP+)试管中,过夜摇;第二天重摇,将摇好的菌取1ml于1.5mlEP管中送测序,并保种。 注:①挑单克隆时,一定要挑单一圆润的菌落,有卫星斑的不挑。 ②别忘记往培养基中加AMP。 ③用接种环挑菌后,要在酒精灯上反复灼烧,然后再进行下一次挑菌。 三.注意事项 1.连接产物可短时间在-20℃保存,使用时可以取出进行后续实验; 2.在细胞转化时,冰浴和热激要严格控制好时间; 3.连接反应是DNA重组过程中的关键步骤,其成败的重要参数之一就是温度,因此要控制好连接温度。 4.进行黏末端连接时,会产生一定数量的载体自身环化分子,导致转化菌中过高的假阳性克隆背景。针对这一问题,常采用牛小肠碱性磷酸酶(CIP)去掉载体的5’-磷酸以抑制DNA 片段的自身环化。 参考: 1.刘进元,常智杰,赵广荣,等.分子生物学实验指导[M].北京:清华大学出版社,2002 2.周俊宜.分子生物学基本技能和策略[M].北京:科学出版社,2003:117-120 3.李海英,杨峰山,邵淑丽,等.现代分子生物学与基因工程[M].北京:化学工业出版社,2008:138-142 4.朱旭芬.基因工程实验指导[M].北京:高等教育出版社,2006:134-139
慢病毒载体包装构建过程
慢病毒载体包装构建过程 原理:慢病毒载体可以将外源基因或外源的shRNA有效地整合到宿主染色体上,从而达到持久性表达目的序列的效果。在感染能力方面可有效地感染神经元细胞、肝细胞、心肌细胞、肿瘤细胞、内皮细胞、干细胞等多种类型的细胞,从而达到良好的的基因治疗效果。对于一些较难转染的细胞,如原代细胞、干细胞、不分化的细胞等,使用慢病毒载体,能大大提高目的基因或目的shRNA的转导效率,且目的基因或目的shRNA整合到宿主细胞基因组的几率大大增加,能够比较方便快捷地实现目的基因或目的shRNA的长期、稳定表达。 概念:慢病毒载体是指以人类免疫缺陷病毒-1 (H IV-1) 来源的一种病毒载体,慢病毒载体包含了包装、转染、稳定整合所需要的遗传信息,是慢病毒载体系统的主要组成部分。携带有外源基因的慢病毒载体在慢病毒包装质粒、细胞系的辅助下,经过病毒包装成为有感染力的病毒颗粒,通过感染细胞或活体组织,实现外源基因在细胞或活体组织中表达。 辅助成分:慢病毒载体辅助成分包括:慢病毒包装质粒和可产生病毒颗粒的细胞系。 慢病毒载体包含了包装、转染、稳定整合所需要的遗传信息。慢病毒包装质粒可提供所有的转录并包装RNA 到重组的假病毒载体所需要的所有辅助蛋白。为产生高滴度的病毒颗粒,需要利用表达载体和包装质粒同时共转染细胞,在细胞中进行病毒的包装,包装好的假病毒颗粒分泌到细胞外的培养基中,离心取得上清液后,可以直接用于宿主细胞的感染,目的基因进入到宿主细胞之后,经过反转录,整合到基因组,从而高水平的表达效应分子。 基本原理:慢病毒载体系统由两部分组成,即包装成分和载体成分。
包装成分:由HIV-1基因组去除了包装、逆转录和整合所需的顺式作用序列而构建,能够反式提供产生病毒颗粒所必需的蛋白。包装成分通常被分开构建到两个质粒上,一个质粒表达Gag和Pol蛋白,另一个质粒表达Env蛋白,其目的也是降低恢复成野生型病毒的可能。将包装成分与载体成分的3个质粒共转染细胞(如人肾293T细胞),即可在细胞上清中收获只有一次性感染能力而无复制能力的、携带目的基因的HIV-1载体颗粒。 载体成分:与包装成分互补,即含有包装、逆转录和整合所需的HIV顺式作用序列,同时具有异源启动子控制下的多克隆位点及在此位点插入的目的基因。 为降低两种成分同源重组恢复成野生型病毒的可能,需尽量减少二者的同源性,如将包装成分上5′LTR换成巨细胞病毒(CMV)立即早期启动子、3′LTR换成SV40 polyA等。 一、实验流程(1和2为并列步骤) 1.慢病毒过表达质粒载体的构建 设计上下游特异性扩增引物,同时引入酶切位点,PCR(采用高保真KOD酶,3K内突变率为0%)从模板中(CDNA质粒或者文库)调取目的基因CDS区(coding sequence)连入T载体。将CDS区从T载体上切下,装入慢病毒过表达质粒载体。 2.慢病毒干扰质粒载体的构建 合成siRNA对应的DNA颈环结构,退火后连入慢病毒干扰质粒载体 3. 慢病毒载体的包装与浓缩纯化 制备慢病毒穿梭质粒及其辅助包装原件载体质粒,三种质粒载体分别进行高纯度无内毒素抽提,共转染293T细胞,转染后6 h 更换为完全培养基,培养24和48h后,分别收集富含
表达载体的构建方法及步骤
表达载体的构建方法及步骤 一、载体的选择及如何阅读质粒图谱 目前,载体主要有病毒和非病毒两大类,其中质粒DNA 是一种新的非病毒转基因载体。一个合格质粒的组成要素: (1)复制起始位点Ori 即控制复制起始的位点。原核生物DNA 分子中只有一个复制起始点。而 真核生物DNA 分子有多个复制起始位点。 (2)抗生素抗性基因可以便于加以检测,如Amp+ ,Kan+ (3)多克隆位点MCS 克隆携带外源基因片段 (4)P/E 启动子/增强子 (5)Terms 终止信号 (6)加poly(A)信号可以起到稳定mRNA 作用 选择载体主要依据构建的目的,同时要考虑载体中应有合适的限制酶切位点。如果构建的目 的是要表达一个特定的基因,则要选择合适的表达载体。 载体选择主要考虑下述3点: 【1】构建DNA 重组体的目的,克隆扩增/基因表达,选择合适的克隆载体/表达载体。【2】.载体的类型: (1)克隆载体的克隆能力-据克隆片段大小(大选大,小选小)。如<10kb 选质粒。(2)表达载体据受体细胞类型-原核/真核/穿梭,E.coli/哺乳类细胞表达载体。
(3)对原核表达载体应该注意:选择合适的启动子及相应的受体菌,用于表达真核蛋白质时注意克服4个困难和阅读框错位;表达天然蛋白质或融合蛋白作为相应载体的参考。【3】载体MCS 中的酶切位点数与组成方向因载体不同而异,适应目的基因与载体易于链接,不能产生阅读框架错位。 综上所述,选用质粒(最常用)做载体的5点要求: (1)选分子量小的质粒,即小载体(1-1.5kb)→不易损坏,在细菌里面拷贝数也多(也有大载 体); (2)一般使用松弛型质粒在细菌里扩增不受约束,一般10个以上的拷贝,而严谨型质粒<10个。 (3)必需具备一个以上的酶切位点,有选择的余地; (4)必需有易检测的标记,多是抗生素的抗性基因,不特指多位Ampr(试一试)。(5)满足自己的实验需求,是否需要包装病毒,是否需要加入荧光标记,是否需要加入标签蛋白,是否需要真核抗性(如Puro、G418)等等。 无论选用哪种载体,首先都要获得载体分子,然后采用适当的限制酶将载体DNA 进行切割,获得线性载体分子,以便于与目的基因片段进行连接。 如何阅读质粒图谱 第一步:首先看Ori 的位置,了解质粒的类型(原核/真核/穿梭质粒) 第二步:再看筛选标记,如抗性,决定使用什么筛选标记。 (1)Ampr 水解β-内酰胺环,解除氨苄的毒性。 (2)tetr 可以阻止四环素进入细胞。 (3)camr 生成氯霉素羟乙酰基衍生物,使之失去毒性。 (4)neor(kanr)氨基糖苷磷酸转移酶使G418(长那霉素衍生物)失活
质粒构建流程
质粒构建流程 一、引物设计 1)取得目的基因序列,可选用数据库NCBI 2)软件分析目的基因可用酶切位点。使用primer5分析出序列不包含的酶切位点,即为可用没切位点。 3)选择载体。根据转染细胞和实验室资源,选择合适载体。如pcDNA3.1(+), 4)选择酶切位点。对照目的基因可用酶切位点和载体上的酶切位点,选择二者共有的作为备选。载体上两个酶切位点的距离应有几十bp以上,选实验室常用酶切位点。 5)使用primer5设计目的基因引物,目的产物应包含从启动子到终止子全部碱基。 6)根据选择的酶切位点,查找对应的酶切位点保护碱基,将对应片段添加到设计的引物两端,注意酶切位点的前后顺序。一般选择三个保护碱基。 7)引物设计完成,送公司合成。 二、目的片段获取 1. RNA提取 试剂盒:Bioteke 高纯总RNA快速提取试剂盒离心柱型(裂解液RL 4℃、漂洗液RW -20℃保存) 准备:冰盒、4℃预冷离心机、EP管2套、吸附柱RA一套 操作步骤: 1)将1000μl裂解液RL加入细胞中,混合5min。 2)加200μl氯仿混合,震荡15s,室温孵育3min。 3)4℃,12000rpm离心10min。 4)最上层水相转移至新EP管中(体积约550μl) 5)加入1倍体积(550μl)70%乙醇,混匀 6)全部转移到套收集管的吸附柱RA中,4℃,10000rpm离心45s 7)弃废液,重套收集管,加500μl去蛋白液RE,12000rpm离心45s 8)弃废液,重套收集管,加700μl去漂洗液RW,12000rpm离心60s 9)弃废液,重套收集管,加500μl去漂洗液RW,12000rpm离心60s 10)弃废液,重套收集管,12000rpm空离2min 11)吸附柱放入新EP管,加50μl RNase free water于膜上,室温放置2min 12)4℃,12000rpm离心60s 13)点样:5μl RNA+ 1μl 10×buffer,1.5%琼脂糖凝胶电泳,100V,3min,可见3条亮带。14)-20℃保存 2.RNA反转录 试剂盒:TaKaRa primescript RT reagent kit with gDNA eraser(-20℃保存) 准备:冰盒,②④⑤⑥取出解冻,①③为酶不可取出,预冷离心管 操作步骤: 1)基因组DNA去除(10μl体系) ② 5×gDNA eraser buffer 2μl ① gDNA eraser 1μl
载体构建的基本步骤
载体构建的基本步骤 Company Document number:WUUT-WUUY-WBBGB-BWYTT-1982GT
载体构建 一、原理 依赖于限制性核酸内切酶,DNA连接酶和其他修饰酶的作用,分别对目的基因和载体DNA进行适当切割和修饰后,将二者连接在一起,再导入宿主细胞,实现目的基因在宿主细胞内的正确表达。 二、操作步骤 1、摇菌(制作感受态细胞备用) 取装有液体培养基的3ml试管两支(依情况而定),每管加40-100μl菌种,过夜摇。 2、提质粒(也就是载体) 依照提质粒试剂盒中的说明书操作(根据情况最后一步洗脱时可以多洗1-2次)。 3、酶切(双酶切产生粘性末端) 反应所需试剂体积(单位:ul) 质粒 10 所需内切酶反应缓冲液 2 所需限制性内切核酸酶 1 H2O 7 将加好的EP管置于37℃保温1-2h。(依照提酶切的具体步骤操作;为了达到最佳酶切的效果,最好根据所选用的酶确定所需要的反应温度) 4、电泳检测 将酶切产物进行琼脂糖凝胶电泳,检测酶切是否成功。 回收胶:琼脂糖与缓冲液一比一制胶,经过切胶回收目的产物,也有目的产物纯化的功能; 检测胶:琼脂糖与缓冲液一比二制胶,为了检测目的条带与预期是否相符。
切胶回收与产物纯化是差不多的过程,所达到的目的是一样的:切胶回收也是一种纯化过程,它能去除非目的片段,然后用回收试剂盒进行纯化,能将很不纯的DNA溶液纯化;产物纯化是将较纯的DNA溶液进一步除去多余的杂质,用纯化试剂盒,你会发现纯化试剂盒和回收试剂盒的步骤几乎一样。 5、载体与目的基因连接 如果电泳检测酶切成功的话,则仔细将所需的片段切割下来,将胶体回收(依照胶回收试剂盒说明书操作);之后将回收的片段和载体连接。置于温箱,12-16℃,保温8-16h 6、转化(连接产物转化到感受态细胞中) 依照转化具体操作步骤做感受态,将上述连接产物进行转化实验,涂板培养,37℃, 12-16h。 7、单克隆检测 (1)挑单克隆 先将AMP从冰箱中取出,待融化后,在3ml装有LB液体培养基的试管中加入3μL的 AMP,用枪头混匀;取 mlEP管5支(依情况可以多挑几管),给每支管中加500μL上述培养液,然后用接种环(或黄枪头)挑单克隆,挑完后用枪吹打;之后,将挑好的菌摇4-5小时,至混浊即可。 (2)单克隆检测 以每管摇好的菌液为模板,以原有的引物进行PCR,然后将PCR产物跑电泳,观察电泳图像中那几管的条带正确,将正确条带相对应管的菌液再抽取100μ
载体构建
载体构建
载体构建分为以下步骤: 1.载体的选择和引物设计以扩增目标基因 2.目标片段的PCR扩增 3.载体和目标片段的限制性酶切 4.连接转化 5.挑取克隆提质粒 6.单克隆的验证及送样测序。 载体的选择 实验室常用的载体有pUC19(扩繁目标片段),pet28a(原核表达载体Kna+),pGEX-4T-1(原核表达载体Amp+),pCI-NEO(真核表达载体Amp+),pCDNA3.1(真核表达载体Amp+),pLKO.1(shRNA构建Amp+)。根据所要构建的质粒目的的差异以及载体和目标片段的酶切位点分析结果来选择载体。举例如下: 实验目的是将MYC基因插入到载体中,转染进细胞中表达。应选用真核表达载体pCI-NEO 或pCDNA3.1。 引物设计 引物设计的目的是将目标片段扩增出来以便与载体连接,所以在引物的两端应人为加上酶切位点,酶切位点前加上保护碱基。在选择酶切位点是应注意,选择的应该是目的基因片段中没有而载体上有的限制性内切酶,防止目标片段被切不完整,也便于和载体连接。 载体构建之后我们的目的是要表达,所以应该加入标签以便western blot检测蛋白是否表达。常用的标签有Flag标签、6XHis标签、HA标签和Myc标签。 Flag标签: D Y K D D D D K GAT TAC AAG GAT GAC GAC GAT AAG 6XHis标签: H H H H H H CAC CAC CAC CAC CAC CAC HA标签: Y P Y D V P D Y A TAC CCA TAC GAT GTT CCA GAT TAC GCT Myc标签: E Q K L I S E E D L 这些标签被表达成氨基酸后,特定的氨基酸序列会与相应抗体结合,在western blot时可被检测。因此,在设计引物时,这些核酸序列应位于起始密码子ATG之后。
CRISPRCas9基因敲入试验步骤(三)donor载体构建载体构建
CRISPRCas9基因敲入试验步骤(三)donor载体构建载体构 建 1、质粒骨架的选择 CRISPR/Cas9质粒按编辑细胞类型可分为八种,Mammalian 、Bacteria 、Drosophila 、Plant、C. elegans 、Yeast 、Zebrafish 、Xenopus,根据质粒所携带的编辑基因的不同,可分为野生型的cas9、突变型的cas9、cpf1、C2c2(可剪切RNA)。当然,还有一些筛选标记,如puro、GFP、RFP、mcherry、潮霉素等等,我们可以根据自己的需求选择自己合适的质粒。这里故意还掉了一种,最具有争议的Ngago,本楼主也重复过这个试验,也和大多数重复的人一样,GFP验证试验时,看到荧光显著减弱(本人还特意构建了一种Ngago载体,效果比韩老师的效果好很多),但是当时没有验证这个减弱是切割还是敲低,非常遗憾自己想法太简单,以先入为主认为是切割而没有去验证;最终结果是刘东老师使用Ngago发现了有表型(斑马鱼的眼睛上有缺陷),这个就是很强的提示,需要去做下一步,尽管基因组上没有被切割,有表型,那势必会去看该基因表达的数据,最后才有这篇文章。刘东老师运气也很好,knock down 有表型,如果没表型,估计也会放弃。这里就说到这,有不懂的可以留言询问。。具体分类在addgene上有,网址/。
2、酶切位点的选择 插入sgRNA的酶切位点,质粒基本为这两种,BsmbI和Bbsi,具体可以参见该质粒的protocol,后续的连接、转化、验证protocol里面有,我就不啰嗦了。哦还忘了一件事,使用snapgene这个软件可以很好地看质粒图谱,非常方便,强烈推荐!!以慢病毒载体V2为例具体说明3、同源臂的设计 同源臂我们一般都是在切割位点的两端各选一段,长度大约1kb-2kb,效率还可以。当然了,有人也用40-100bp做了也做成功了,但基本原则是越长效率越高。1kb-2kb效率已经很高,所以这个最合适。有相关文献支持,自己Pubmed查看。 4、启动子、目的基因及其终止子的扩增 这里也没什么可说的,启动子在我们选择质粒的时候就决定了,当然我们还可以改造以提高sgRNA的表达效率,这个时候我们可以通过方法筛选出该物种的启动子,替换上去就ok了。 目的基因的话,如果我们要做敲除,基本就是根据基因组,在外显子区域设计sgRNA序列,切割后以非同源性末端接合(Non-homologous end joining, NHEJ),造成变异而使该基因失去功能。当然还有一种修复方式,同源性重组(Homologous recombination,HR)同源性重组修复是利用细胞内的染色体两两对应的特性,若其中一条染色体上的
质粒载体的构建分析
质粒载体的构建 摘要:质粒载体的构建。首先要获得目的DNA。根据其目的基因序列和启动子序列设计引物,为提高目的基因产率,采用两次PCR的方法,即第一次设计引物扩增全序列基因,第二次设计带酶切位点的引物以第一次扩增产物为模板进行扩增,进而加尾连接到T-DNA上,再利用电转化的方法将连接产物转化到带有PCAMBIA1381的DH5α感受态细胞中复制表达。 关键词:质粒DNA PCR 电泳感受态转化 1.引言 质粒(plasmid)是细菌或细胞染色质以外的,能自主复制的,与细菌或细胞共生的遗传成分。其特点如下: ①是染色质外的双链共价闭合环形DNA(cccDNA),可自然形成超螺旋结构,不同质 粒大小在2-300kb之间,<15kb的小质粒比较容易分离纯化,>15kb的大质粒则不易提取。 ②能自主复制,是能独立复制的复制子。一般质粒DNA复制的质粒可随宿主细胞分裂而 传给后代。 ③质粒对宿主生存并不是必需的。某些质粒携带的基因功能有利于宿主细胞的 特定条件下生存,例如,细菌中许多天然的质粒带有抗药性基因,如编码合成能分解破坏四环素、氯霉素、氨芐表霉素等的酶基因,这种质粒称为抗药性质粒,又称R质粒,带有R质粒的细菌就能在相应的抗生素存在生存繁殖。 所以质粒对宿主不是寄生的,而是共生的。现在分子生物学使用的质粒载体
都已不是原来细菌或细胞中天然存在的质粒,而是经过了许多的人工的改造。从不同的实验目的出发,人们设计了各种不同的类型的质粒载体。 质粒载体pBR322是研究得最多,是使用最早且应用最广泛的大肠杆菌质粒载体之一。符号质粒载体pBR322中的“p代表质粒;“BR”代表两位两位研究者Bolivar和Rogigerus姓氏的字首,“322”是实验编号。 质粒载体pBR322的大小为4361bp,相对分子质量较小的是它第一个优点。优点之二是它带有一个复制起始位点,保证了该质粒只在大肠杆菌的细胞中行使复制的功能。具有两种抗生素抗性基因,可供转化子的选择标记是它的第三个优点。 质粒载体pBR322的第四个优点是具有较高的拷贝数,经过氯霉素扩增以后,每个细胞中可累积1000-3000份拷贝,该特性为重组体DNA的制备提供了极大的方便。 构建质粒载体所用的方法基本上是分子克隆技术,是在分子水平上提供一种纯化和扩增特定DNA片段的方法。常含有目的基因,用体外重组方法将它们插入克隆载体,形成重组克隆载体,通过转化与转导的方式,引入适合的寄主体内得到复制与扩增,然后再从筛选的寄主细胞内分离提纯所需的克隆载体,可以得到插入DNA的许多拷贝,从而获得目的基因的扩增。 2. 材料方法 2.1目的DNA的获得 2.1.1 引物设计 第一次引物设计: 正向引物:sinn3F 冰盒标注:P2a 引物序列:5’—AAGCAAAATCTAACCGTGTAATGTA—3’ 引物长度:25bp 反向引物:sinn3R 冰盒标注:P2b
RNA干扰载体的构建的实验流程
RNA干扰载体的构建的实验流程 RNA干扰载体主要用来研究基因表达调控,RNA干扰技术已已被广泛用于基因结构功能研究和传染性疾病及基因治疗领域,进行RNA干扰实验首先是构建RNA干扰载体,本文以pRI 系列载体为例论述了干扰载体的构建的实验流程。 产品技术背景 pRI系列载体是基于III类rna聚合酶启动子:人类H1启动子的专用于哺乳动物细胞RNA干扰的载体。H1启动子在哺乳动物细胞内合成类似siRNA分子的小分子RNA。由于H1启动子有精确的转录起始位点和终止信号,H1启动子转录产物精确生成人工设计的shRNA,shRNA 经过RISC剪切后形成有2个U突出末端的成熟siRNA。由于H1启动子对转录产物长度的严格限制,基本上杜绝了非特异性干扰片段的产生,将载体转染细胞后对其它基因的影响降到最低。 pRI系列载体已经成功用于多种哺乳动物细胞进行基因的RNA干扰。本系列中含有新霉素抗性基因的载体用于稳定表达siRNA,可以在更长时间内对基因表达抑制后的细胞功能和生理现象进行观察和分析。 插入寡核苷酸设计 pRI系列载体的使用需要将人工设计的寡核苷酸片段插入pRI系列载体中特定的酶切位点之间,寡核苷酸片段中包含了针对目标基因的mRNA设计的长度为19nt的干扰片段。 合成时需要化学合成正向和反向两条寡核苷酸。正、反向寡核苷酸退火后与载体连接,插入载体XhoI,BglII位点之间,位于载体上H1启动子下游正确的位置上。连接后的载体转入哺乳动物细胞在H1启动子作用下转录产生shRNA。 1. 选择干扰序列 在RNA干扰实验中,RNA干扰序列的选择会显著影响RNA干扰效果。我们建议您按照以下几点指导原则选择RNA干扰序列: 推荐长度为19 nt,采用21 nt序列也可以取得良好效果。 RNA干扰序列中不包含大于3 nt的连续相同碱基。 RNA干扰序列的GC含量为低到中等水平(推荐GC含量在35%到50%之间)。 不要将RNA干扰序列设计在已知的RNA-蛋白质结合位置附近。 确保RNA干扰序列与其它基因没有较高的同源性。 方向:在编码mRNA的正义链上选择RNA干扰序列。 设计RNA干扰序列是RNAi实验的关键。这里提供的设计原则可以为您设计RNA干扰序列提供帮助。但是,值得注意的是,遵循这些原则不能确保设计的RNA干扰序列对目标基因有好的抑制效果。针对一个目标基因,我们建议您至少设计3条干扰序列并且从中筛选出干扰效果好的序列。 2. 寡核苷酸设计。 (1)设计正义寡核苷酸链(5’-3’方向)。 a) 5’TCGACCC b) 19nt干扰序列正向序列(与目标mRNA一致)。 c) TTCAAGAGA(环状结构)。
载体构建的基本步骤
1、摇菌(制作感受态细胞备用) 上述菌液PCR 有结果的菌液送测,送测结果与 预期相符则该载体构建成功。阳性单克隆筛选 上述转化得到的菌液接种到筛 选培养基中过夜培养挑菌并菌液PCR 转化 感受态细胞制备转化步骤 载体与目的基因连接 质粒与目的基因双酶切 选择内切酶摸索酶切条件(时 间和温度)电泳检测及回收纯化步骤 质粒(载体)提取 质粒选择提取步骤 目的基因克隆 引物设计PCR
取装有液体培养基的3ml试管两支(依情况而定),每管加40-100μl菌种,过夜摇。 2、提质粒(也就是载体) 依照提质粒试剂盒中的说明书操作(根据情况最后一步洗脱时可以多洗1-2次)。3、酶切(双酶切产生粘性末端) 反应所需试剂体积(单位:ul) 质粒10 所需内切酶反应缓冲液 2 所需限制性内切核酸酶 1 H2O 7 将加好的EP管置于37℃保温1-2h。(依照提酶切的具体步骤操作;为了达到最佳酶切的效果,最好根据所选用的酶确定所需要的反应温度) 4、电泳检测 将酶切产物进行琼脂糖凝胶电泳,检测酶切是否成功。 回收胶:琼脂糖与缓冲液一比一制胶,经过切胶回收目的产物,也有目的产物纯化的功能;检测胶:琼脂糖与缓冲液一比二制胶,为了检测目的条带与预期是否相符。 切胶回收与产物纯化是差不多的过程,所达到的目的是一样的:切胶回收也是一种纯化过程,它能去除非目的片段,然后用回收试剂盒进行纯化,能将很不纯的DNA溶液纯化;产物纯化是将较纯的DNA溶液进一步除去多余的杂质,用纯化试剂盒,你会发现纯化试剂盒和回收试剂盒的步骤几乎一样。 5、载体与目的基因连接 如果电泳检测酶切成功的话,则仔细将所需的片段切割下来,将胶体回收(依照胶回收试剂盒说明书操作);之后将回收的片段和载体连接。置于温箱,12-16℃,保温8-16h 6、转化(连接产物转化到感受态细胞中) 依照转化具体操作步骤做感受态,将上述连接产物进行转化实验,涂板培养,37℃,12-16h。 7、单克隆检测 (1)挑单克隆
载体构建
载体构建分为以下步骤: 1.载体的选择和引物设计以扩增目标基因 2.目标片段的PCR扩增 3.载体和目标片段的限制性酶切 4.连接转化 5.挑取克隆提质粒 6.单克隆的验证及送样测序。 载体的选择 实验室常用的载体有pUC19(扩繁目标片段),pet28a(原核表达载体Kna+),pGEX-4T-1(原核表达载体Amp+),pCI-NEO(真核表达载体Amp+),pCDNA3.1(真核表达载体Amp+),pLKO.1(shRNA构建Amp+)。根据所要构建的质粒目的的差异以及载体和目标片段的酶切位点分析结果来选择载体。举例如下: 实验目的是将MYC基因插入到载体中,转染进细胞中表达。应选用真核表达载体pCI-NEO 或pCDNA3.1。 引物设计 引物设计的目的是将目标片段扩增出来以便与载体连接,所以在引物的两端应人为加上酶切位点,酶切位点前加上保护碱基。在选择酶切位点是应注意,选择的应该是目的基因片段中没有而载体上有的限制性内切酶,防止目标片段被切不完整,也便于和载体连接。 载体构建之后我们的目的是要表达,所以应该加入标签以便western blot检测蛋白是否表达。常用的标签有Flag标签、6XHis标签、HA标签和Myc标签。 Flag标签: D Y K D D D D K GAT TAC AAG GAT GAC GAC GAT AAG 6XHis标签: H H H H H H CAC CAC CAC CAC CAC CAC HA标签: Y P Y D V P D Y A TAC CCA TAC GAT GTT CCA GAT TAC GCT Myc标签: E Q K L I S E E D L 这些标签被表达成氨基酸后,特定的氨基酸序列会与相应抗体结合,在western blot时可被检测。因此,在设计引物时,这些核酸序列应位于起始密码子ATG之后。
慢病毒载体构建步骤研究
一、简介 慢病毒(Lentivirus)载体是以HIV-1(人类免疫缺陷I型病毒)为基础发展起来的基因治疗载体。区别一般的逆转录病毒载体,它对分裂细胞和非分裂细胞均具有感染能力。慢病毒载体的研究发展得很快,研究的也非常深入。该载体可以将外源基因有效地整合到宿主染色体上,从而达到持久性表达。 目前慢病毒也被广泛地应用于表达RNAi的研究中。由于有些类型细胞脂质体转染效果差,转移到细胞内的siRNA半衰期短,体外合成siRNA对基因表达的抑制作用通常是短暂的,因而使其应用受到较大的限制。采用事先在体外构建能够表达siRNA的载体, 然后转移到细胞内转录siRNA的策略,不但使脂质体有效转染的细胞种类增加,而且对基因表达抑制效果也不逊色于体外合成siRNA,在长期稳定表达载体的细胞中,甚至可以发挥长期阻断基因表达的作用。慢病毒载体能够产生表达shRNA的高滴度的慢病毒,在周期性和非周期性细胞、干细胞、受精卵以及分化的后代细胞中表达shRNA,实现在多种类型的细胞和转基因小鼠中特异而稳定的基因表达的功能性沉默,为在原代的人和动物细胞组织中快速而高效地研究基因功能,以及产生特定基因表达降低的动物提供了可能性。慢病毒作为siRNA的携带者,不但具备特异性地使基因表达沉默的能力,而且充分发挥了慢病毒载体自身所具备的优势,为基因功能的研究提供了更强有力的工具。 在所构建的siRNA表达载体中,是由RNA聚合酶Ⅲ启动子来指导RNA合成的,这是因为RNA聚合酶Ⅲ有明确的起始和终止序列,而且合成的RNA不会带poly A尾。当RNA 聚合酶Ⅲ遇到连续4个或5个T时,它指导的转录就会停止,在转录产物3’端形成1~4个U。U6和H1 RNA启动子是两种RNA聚合酶Ⅲ依赖的启动子,其特点是启动子自身元素均位于转录区的上游,适合于表达~21ntRNA和~50ntRNA茎环结构(stem loop)。在siRNA表达载体中,构成siRNA的正义与反义链,可由各自的启动子分别转录,然后两条链互补结合形成siRNA;也可由载体直接表达小发卡状RNA(small hairpin RNA, shRNA), 载体包含位于RNA聚合酶Ⅲ启动子和4~5T转录终止位点之间的茎环结构序列,转录后即可折叠成具有1~4 个U 3 ’ 突出端的茎环结构,在细胞内进一步加工成siRNA。构建载体前通常要通过合成siRNA的方法,寻找高效的siRNA,然后从中挑选符合载体要求的序列,将其引入siRNA表达载体(筛选)。 二、实验流程(大致的简单过程) 慢病毒表达载体包含了包装、转染、稳定整合所需要的遗传信息。慢病毒包装质粒可提供所有的转录并包装RNA 到重组的假病毒载体所需要的所有辅助蛋白。为产生高滴度的
micRNA载体构建流程
micRNA载体构建流程: 1.micRNA 片段PCR (1) PCR体系: 5x PrimeSTAR buffer 10.0 μL 2.5 mM dNTPs mixture 4.0 μL Primer1 (10 uM) 1.0 μL Primer2 (10 uM) 1.0 μL PrimeSTAR HS 0.3μL 模板DNA 2μL ddH2O 31.7μL 程序: 98℃ 1min; 98℃ 10s,55℃ 10min,72℃ 1.0min,30 cycles; 72℃ 10min 1.0%琼脂糖胶检测PCR结果:上样5ul 注意:PCR用50μL体系重复两管,回收时,并成一管纯化。回收完后检测纯化产物OD值,纯化产物含量最好高于50ng/ul,若产物浓度低,则再多做几份PCR再回收。 (2)纯化产物补A 上述纯化后,浓度达到要求的产物可进行补A,用于后续连接。 补A体系: 纯化产物:16.8ul 10x PCR buffer:2ul dATP(10mM):1ul (终浓度2 mM) rTaq:0.2 ul 反应条件:72℃15-30min 补A产物放于-20℃备用。 2. CD3-1250载体酶切 做第一步PCR的同时,可先做载体酶切,做好准备。
酶切体系: ddH2O:34ul 10X NEB Buffer2: 5ul Xcm1: 1-1.5ul CD3-1250质粒:10-20 ul 酶切重复6管,胶回收时可将2-3管合并为一管回收。 反应条件:37℃ 3-5小时或过夜。 1.0%琼脂糖胶检测酶切是否完全,若酶切完全,可进行后续回收。上样5ul,检测时140-150V电压,跑胶15-20min即可,勿跑太长时间。 回收:制备一块1.0%琼脂糖新胶(大孔),将酶切产物全部上样,140V-150V跑胶20min 左右。切胶回收大片段载体。 载体片段较大,需用天根胶回收纯化试剂盒回收。 回收完后检测纯化产物OD值,载体纯化产物含量最好高于50ng/ul,若产物浓度低,则再多做几份酶切再切胶回收。 3. 连接反应: 体系:PCR补A产物(约50ng/ul): 1.5ul CD3-1250载体回收产物(约50ng/ul): 1.5ul Soltion I(TAKARA):3ul 条件:16℃ 3-5小时,或过夜连接。 4. 转化DH5α感受态细胞 将上述连接产物全部用于转化,涂布Kan平板,Kan终浓度50ug/ml。倒置培养过夜。 5. 重组子鉴定 (1)菌落PCR鉴定或菌液PCR鉴定阳性克隆。PCR鉴定时别忘了加上阳性对照(col做模板)、阴性对照(不加模板)。鉴定一般用自制TAQ进行。 (2)挑选3-5个阳性克隆摇菌后提取质粒进行酶切鉴定。提质粒时用天根或生工提质粒试剂盒,集菌两次。质粒用BamH1进行酶切鉴定。 BamH1酶切鉴定体系: ddH2O:7ul 10X BamH1 Buffer: 2ul BamH1: 0.5ul 重组质粒:10.5 ul 条件:37℃ 2小时。 1.0%琼脂糖胶检测酶切结果,全部上样或上样10ul检测,检测时140-150V电压,跑胶
载体构建的基本步骤
创作编号: GB8878185555334563BT9125XW 创作者:凤呜大王* 载体构建
一、原理 依赖于限制性核酸内切酶,DNA连接酶和其他修饰酶的作用,分别对目的基因和载体DNA进行适当切割和修饰后,将二者连接在一起,再导入宿主细胞,实现目的基因在宿主细胞内的正确表达。 二、操作步骤 1、摇菌(制作感受态细胞备用) 取装有液体培养基的3ml试管两支(依情况而定),每管加40-100μl菌种,过夜摇。 2、提质粒(也就是载体) 依照提质粒试剂盒中的说明书操作(根据情况最后一步洗脱时可以多洗1-2次)。 3、酶切(双酶切产生粘性末端) 反应所需试剂体积(单位:ul) 质粒10 所需内切酶反应缓冲液 2 所需限制性内切核酸酶 1 H2O 7 将加好的EP管置于37℃保温1-2h。(依照提酶切的具体步骤操作;为了达到最佳酶切的效果,最好根据所选用的酶确定所需要的反应温度) 4、电泳检测 将酶切产物进行琼脂糖凝胶电泳,检测酶切是否成功。 回收胶:琼脂糖与缓冲液一比一制胶,经过切胶回收目的产物,也有目的产物纯化的功能; 检测胶:琼脂糖与缓冲液一比二制胶,为了检测目的条带与预期是否相符。 切胶回收与产物纯化是差不多的过程,所达到的目的是一样的:切胶回收也是一种纯化过程,它能去除非目的片段,然后用回收试剂盒进行纯化,能将很不纯的DNA溶液纯化;产物纯化是将较纯的DNA溶液进一步除去多余的杂质,用纯化试剂盒,你会发现纯化试剂盒和回收试剂盒的步骤几乎一样。
5、载体与目的基因连接 如果电泳检测酶切成功的话,则仔细将所需的片段切割下来,将胶体回收(依照胶回收试剂盒说明书操作);之后将回收的片段和载体连接。置于温箱,12-16℃,保温8-16h 6、转化(连接产物转化到感受态细胞中) 依照转化具体操作步骤做感受态,将上述连接产物进行转化实验,涂板培养,37℃,12-16h。 7、单克隆检测 (1)挑单克隆 先将AMP从冰箱中取出,待融化后,在3ml装有LB液体培养基的试管中加入3μL 的 AMP,用枪头混匀;取1.5 mlEP管5支(依情况可以多挑几管),给每支管中加500μL上述培养液,然后用接种环(或黄枪头)挑单克隆,挑完后用枪吹打;之后,将挑好的菌摇4-5小时,至混浊即可。 (2)单克隆检测 以每管摇好的菌液为模板,以原有的引物进行PCR,然后将PCR产物跑电泳,观察电泳图像中那几管的条带正确,将正确条带相对应管的菌液再抽取100μL,加到3ml(有LB液体培养液,AMP+)试管中,过夜摇;第二天重摇,将摇好的菌取1ml于1.5mlEP 管中送测序,并保种。 注:①挑单克隆时,一定要挑单一圆润的菌落,有卫星斑的不挑。 ②别忘记往培养基中加AMP。 ③用接种环挑菌后,要在酒精灯上反复灼烧,然后再进行下一次挑菌。 三、注意事项 1.连接产物可短时间在-20℃保存,使用时可以取出进行后续实验; 2.在细胞转化时,冰浴和热激要严格控制好时间; 3.连接反应是DNA重组过程中的关键步骤,其成败的重要参数之一就是温度,因此要控 制好连接温度。
载体构建操作流程
载体构建操作流程 一、载体准备 1、质粒准备 电泳检测载体质量 好的质粒为三条带,分别是超螺旋、开环和复制中间体(即没有复制完全的两个质粒连在了一起);也有观点认为是质粒两条链没有断裂的超螺旋结构,开环DNA和线性DNA。超螺旋结构应至少占到90%。 测定质粒浓度 用核酸定量仪测定质粒的浓度,A230、A260和A280值。 2、质粒双酶切 根据需要的酶切位点,选择对应的酶,查找适合的Buffer,进行单酶切。 以TaKaRa的限制酶为例,双酶切的体系如下: 试剂体积 质粒<1 μg Enzyme I 1μL Enzyme II 1μL 10×X Buffer 2μL 灭菌水to 20μL 总体积20μL 将上述体系置于37℃(不同酶可能存在差异)水浴锅中酶切。 根据酶切效率不同确定酶切时间,可以去除2 μl进行电泳检测。 3、回收质粒 凝胶回收 参照TaKaRa MiniBEST Agarose Gel DNA Extraction Kit Ver.3.0试剂盒说明书。 1)称量胶块重量,计算胶块体积。计算胶块体积时,以1mg=1μl进行计向胶 块中加入胶块融化液Buffer GM,我们一般用1%的琼脂糖凝胶电泳,所以一般加入3个凝胶体积量。 2)均匀混合后室温15-25℃融化胶块。此时应间断振荡混合,使胶块充分融化 (约5~10分钟)。 3)将试剂盒中的Spin Column安置于Collection Tube上。将上述操作的溶液转 移至Spin Column 中,12,000 rpm离心1分钟,弃滤液。滤液再加入Spin Column中离心一次,可以提高DNA的回收率。 4)将700μl的Buffer WB加入Spin Column中,室温12,000 rpm离心30秒钟, 弃滤液。 5)重复操作步骤。 6)将Spin Column安置于Collection Tube上,室温12,000 rpm离心1分钟。 7)将Spin Column安置于新的1.5ml的离心管上,在Spin Column膜的中央处 加入30μl的灭菌蒸馏水或Elution Buffer,室温静置1分钟。 8)将灭菌蒸馏水或Elution Buffer加热至60℃使用时有利于提高洗脱效率。 9)室温12,000 rpm离心1分钟洗脱DNA。
