染色体步移试剂盒说明书

DNA Walking步移法加速试剂盒(扩增未知侧翼序列的最佳方法)
| [<<][>>]
品!
科研人员已经发展了几种无需建立cD
NA库或筛选克隆的PCR技术方法,用
于获取与已知序列相连的未知序列。
这些PCR方法包括反向PCR (IPCR),
连接介导PCR (LM-PCR)和随机引物P
CR (RP-PCR)。虽然这些方法有一定的
效果并被不断改进,但是仍然受制于
繁琐的酶切,适配体连接 (adaptor
ligation)和纯化步骤。而且,RP-PC
R的随机引物经常会因为非特异性退
火而产生多余产物。DNA步移法加速
试剂盒通过应用专利的ACP(复性控
制引物)技术,提高了小片断寡聚核
苷酸的特异性,可以克服现有基因步
移法的缺点。
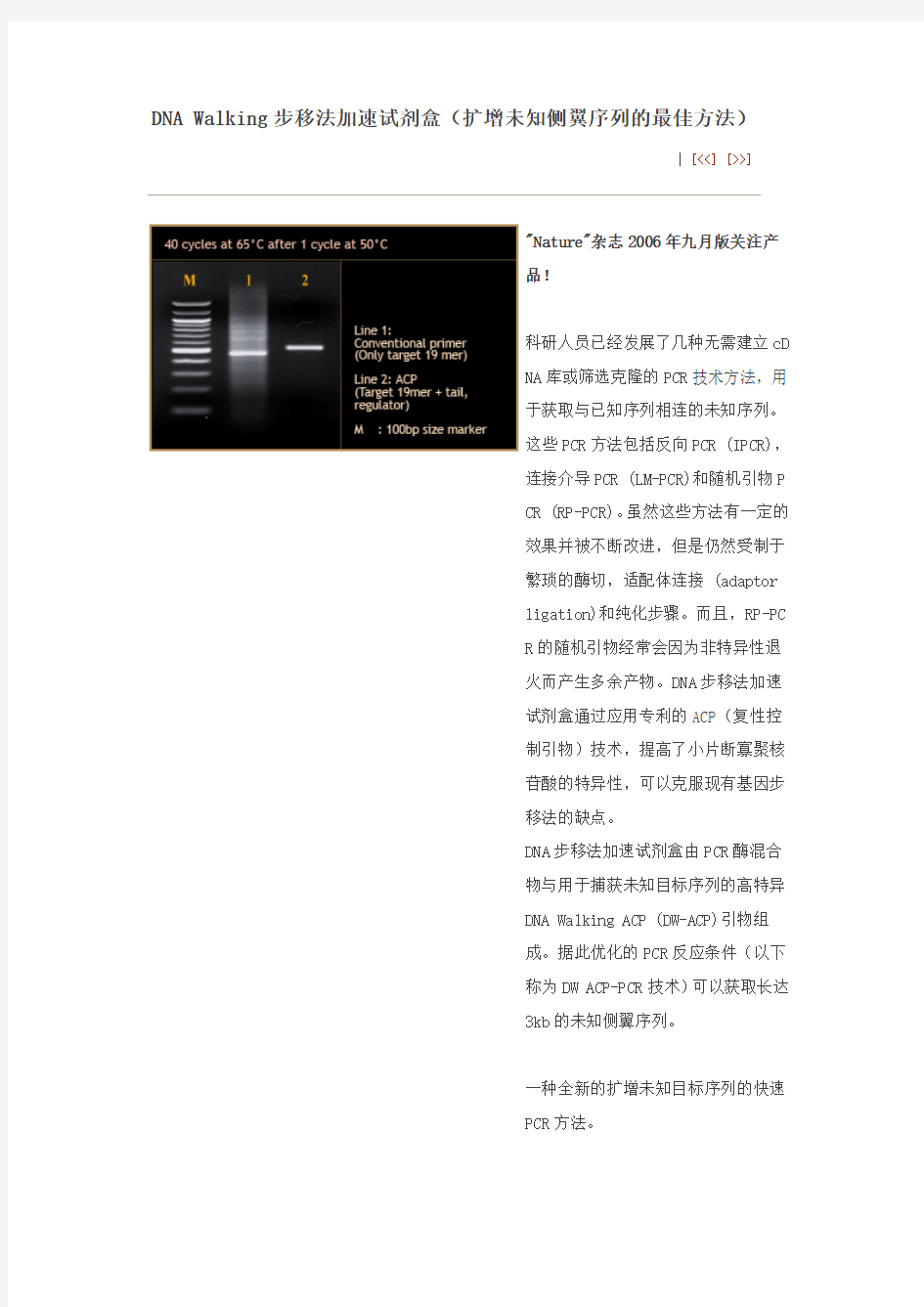
DNA步移法加速试剂盒由PCR酶混合
物与用于捕获未知目标序列的高特异
DNA Walking ACP (DW-ACP)引物组
成。据此优化的PCR反应条件(以下
称为DW ACP-PCR技术)可以获取长达
3kb的未知侧翼序列。
一种全新的扩增未知目标序列的快速
PCR方法。
试剂盒由PCR Master Mix和专为高
特异度捕捉未知靶点的独特DNA步移退火控制引物(DW-ACPs)组成。此最优化的PCR条件,称为 DW ACP-PCR TM 技术,可以获取长达3kb的未知侧区。此试剂盒是直接扩增未知序列的革命性方法。
"Nature 'Product focus-Transgeni cs' September 7, 2006"!
特点:
*最简单
此方法无须建库,无须酶切,无须固定,也无须复杂PCR
*最快捷
你的目标产物可在一天内获得,此方法超越其他现有方法
*最可靠
只获取目标产物
应用范围:
●基因组步移
- 启动子区域的克隆或者测序
- 基因结构(外显子/内含子结合区) - 已知序列上下游侧翼的扩增
- cDNA步移
- 裂隙填补(Gap Filling)
- 序列标签位点(STSs)和表达序列标记(EST)的双向测序
●转基因位点检测
- 在转基因生物如转基因植物,动物,昆虫,鱼和细菌中发现转基因或T-DN A的位置或导向
- 直接扩增并分离出具有转基因或T-DNA的侧翼区域的DNA片断
●缺失/插入/突变体的检测
- 检测基因组DNA或cDNA的缺失,插入,或者突变体
- 剪切分析
●测序
- BAC / PAC DNA的末端测序或者步移
- BACs或者基因组DNA的直接测序
- 基因组DNA,cDNA或质粒DNA (BAC, PAC, YAC)的测序
- 无须亚克隆或者鸟枪克隆即可对BA C或PAC克隆测序
- 长链DNA的快速测序
- 基因组测序计划*基因组步移
-启动子区域的克隆或者测序
-基因结构(外显子/内含子结合区) -裂隙填补(Gap Filling)
客户列表
中国科学院上海生命科学研究院
中国科学院动物所
中国农业科学院
中国热带农业科学院
云南大学
云南农业大学
中国农业大学
浙江大学
南京农业大学
西南农业大学
......
DNA WALKING REFERRENCE
1. Characterization of Mutations in AlHK1 Gene from Alternaria l ongipes: Implication of Limited Function of Two-Component Histid ine Kinase on Conferring Dicarbo ximide Resistance. J. Microbiol. Biotechnol. (2008), 18(1), 15–22
2. Cloning and Expression Analys is of a Chitinase Gene Crchi1 fr om the Mycoparasitic Fungus Clon ostachys rosea. The Journal of M icrobiology, October 2007, p. 42 2-430
3. Cloning and phylogenetic anal ysis of the chitinase gene from the facultative pathogen Paecilo myces lilacinus. Journal of Appl ied Microbiology 103 (2007) 2476
–2488
4. Cloning of the gene Lecanicil lium psalliotae chitinase Lpchi1 and identification of its poten tial role in the biocontrol of r oot-knot nematode Meloidogyne in cognita. Appl Microbiol Biotechn ol (2007) 76:1309–1317
5. New insights into the evoluti on of subtilisin-like serine pro tease genes in Pezizomycotina. B MC Evolutionary Biology 2010, 1 0:68
6. Purification and cloning of a novel serine protease from the nematode-trapping fungus Dactyle llina varietas and its potential roles in infection against nema todes. Appl Microbiol Biotechnol (2007) 75:557–565
7. 刀孢轮枝菌胞外几丁质酶的基因克隆及系统发育分析. 菌物学报, 20 08, 27(3): 368-376
Ki-Bum Park and Suk-Heung Oh(200 7)
Cloning, sequencing and expressi on of a novel glutamate decarbox ylase gene from a newly isolated
lactic acid bacterium, Lactobac illus brevis OPK-3. Bioresource Technology 98(2): 312-319.
Young-Jun Park, Soo Young Choi, and Hee-Bong Lee(2006)
A carboxylesterase from the ther moacidophilic archaeon Sulfolobu s solfataricus P1; purification, characterization, and expressio n.Biochimica et Biophysica (BBA) - General Subjects 1760(5):820-828.
Andrey A. Perelygin, Andrey A. Z harkikh, Svetlana V. Scherbik an d Margo A. Brinton (2006)
The Mammalian 2′-5′ Oligoadeny late Synthetase Gene Family: Evi dence for Concerted Evolution of Paralogous Oas1 Genes in Rodent ia and Artiodactyla. Journal of Molecular Evolution 63(4):562-57 6.
C. Nisbet, I. Butzonitch, M. Col avita, J. Daniels, J. Martin, R. Burns, E. George, M. A. Y. Akho nd, V. Mulholland, and C. J. Jef fries (2006)
Characterization of Potato rough
dwarf virus and Potato virus P: distinct strains of the same vi ral species in the genus Carlavi rus.Plant Pathology 55(6): 803.
Koo BC, Kwon MS, Choi BR, Kim J H, Cho SK, Sohn SH, Cho EJ, Lee HT, Chang W, Jeon I, Park JK, Pa rk JB, Kim T.(2006)
Production of germline transgeni c chickens expressing enhanced g reen fluorescent protein using a MoMLV-based retrovirus vector.F ASEB J. 20(13):2251-60.
Barbet AF, Lundgren AM, Alleman AR, Stuen S, Bjoersdorff A, Brow n RN, Drazenovich NL, Foley JE. (2006)
Structure of the expression site reveals global diversity in MSP 2 (P44) variants in Anaplasma ph agocytophilum. Infect. Immun. 74 (11):6429-37.
Chunhong Yan and Douglas D. Boyd (2006)
Histone H3 Acetylation and H3 K4 Methylation Define Distinct Chr omatin Regions Permissive for Tr ansgene Expression. Molecular an
d Cellular Biology 26(17):6357-6 371.
Alhapel A, Darley DJ, Wagener N, Eckel E, Elsner N, Pierik AJ.(2 006)
Molecular and functional analysi s of nicotinate catabolism in Eu bacterium barkeri.Proc Natl Acad Sci U.S.A. 103(33):12341-6..
Knopf RR and Trebitsh T.(2006) The female-specific Cs-ACS1G gen e of cucumber. A case of gene du plication and recombination betw een the non-sex-specific 1-amino cyclopropane-1-carboxylate synth ase gene and a branched-chain am ino acid transaminase gene. Plan t Cell Physiol. 47(9):1217-28..
Qizhen Shi, David A. Wilcox, Sco t A. Fahs, Hartmut Weiler, Clive W. Wells, Brian C, Cooley, Dras hti Desai, Patricia A. Morateck, Jack Gorski, and Robert R. Mont gomery (2006)
Factor VIII ectopically targeted to platelets is therapeutic in hemophilia A with high-titer inh ibitory antibodies. The Journal
of Clinicel Investigation 116( 7):1974-1982.
Qizhen Shi, David A. Wilcox, Sco t A. Fahs, Hartmut Weiler, Clive W. Wells, Brian C. Cooley, Dras hti Desai, Patricia A. Morateck, Jack Gorski, and Robert R. Mont gomery (2006)
Factor VIII ectopically targeted to platelets is therapeutic in hemophilia A with high-titer inh ibitory antibodies. J. Clin. Inv est. 116(7):1974-1982.
Ahu Altinkut, Olga Raskina, Evia tar Nevo and Alexander Belyayev (2006)
En/Spm-like transposons in Poace ae species: Transposase sequence variability and chromosomal dis tribution. Cellular & Molecular Biology Letters Issue 11(2)214-2 29.
Assaf Distelfield, Cristobal Uau y, Tzion Fahima, and Jorge Dubco vsky (2006)
Physical map of the wheat high-g rain protein content gene Gpc-B1 and development of a high-throu
ghput molecular marker. New Phyt ologist 169:753-763.
Andrey A. Perelygin, Teri L. Lea r, Andrey A. Zharkikh and Margo A. Brinton (2006)
Comparative analysis of vertebra te EIF2AK2 (PKR) genes and assig nment of the equine gene to ECA1 5q24-q25 and the bovine gene to BTA11q12-q15. Genet. Sel. Evol. 38:551-563.
Jung-Wook Kim, Yasuo Yamakoshi, Takanori Iwata, Yuan Yuan Hu, He ngmin Zhang, Jan C.-C. Hu, James P. Simmer (2006)
Porcine dentin matrix protein 1: gene structure, cDNA sequence, and expression in teeth. Europea n Journal Of Oral Sciences114 (1):33-41.
Chengshu Wang and Raymond J. St. Leger(2006)
A collagenous protective coat en ables Metarhiziumanisopliae to e vade insect immune responses. PN AS 103:6647-6652.
EMIKO HAYAMA, SHIN-ICHIRO IMAMUR
A, CUIJIAO WU, MAKOTO NAKAZAWA, RUMIKO MATSUOKA and TOSHIO NAKAN ISHI(2006)
Analysis of Voltage-Gated Potass ium Channel ?1 Subunits in the P orcine Neonatal Ductus Arteriosu s. Pediatric Research 59:167-17 4.
M.-C. Domingo, A. Huletsky, A. B ernal, R. Giroux, D. K. Boudrea u, F. J. Picard and M. G. Berger on(2005)
Characterization of a Tn5382-lik
e transposon containing the vanB
2 gene cluster in a Clostridium strain isolated from human faece s. Journal of Antimicrobial Chem otherapy 55(4):466-474.
Perelygin AA, Lear TL, Zharkikh AA, Brinton MA (2005) Structure of equine 2'-5'oligoad enylate synthetase (OAS) gene fa mily and FISH mapping of OAS gen es to ECA8p15 p14 and BTA17q24q2 5. Cytogenet Genome Res 111(1):5 1-6.
Park, M., H. J. Shin, S. Y. Lee, and T. I. Ahn.(2005)
Characterization of a cDNA of pe roxiredoxin II responding to hyd rogen peroxide and phagocytosis in Amoeba proteus. J Eukaryot Mi crobiol 52:223-30.
中文参考文献:
1)扬子鳄MHC Ⅱ类基因的位点分离与多态性检测
2)扬子鳄基因组BAC文库及MHC Ⅰ类基因BAC克隆重叠群的构建
3)利用转基因小鼠模型进行转基因动物安全评价研究
4)稻瘟菌四个致病相关突变位点的定位及侵染钉缺陷突变体的初步研究5)Bacillus sp. BSD-8肌酐降解酶系的研究
6)一种快速·有效分离T-DNA插入位点的侧翼序列的方法——DW-ACP-PCR 技术
7)刀孢轮枝菌胞外几丁质酶的基因克隆及系统发育分析
8)稻瘟病菌突变体Y34-0453的表型分析和T-DNA插入位点的定位
染色体步移步骤
染色体步移技术(Genome Walking)是一种重要的分子生物学研究技术,使用这种技术可以有效获取与已知序列相邻的未知序列。染色体步移技术主要有以下几方面的应用: 1. 根据已知的基因或分子标记连续步移,获取人、动物和植物的重要调控基因,可以用于研究结构基因的表达调控。如分离克隆启动子并对其功能进行研究; 2. 步查获取新物种中基因的非保守区域,从而获得完整的基因序列; 3. 鉴定T-DNA 或转座子的插入位点,鉴定基因枪转基因法等转基因技术所导致的外源基因的插入位点等; 4. 用于染色体测序工作中的空隙填补,获得完整的基因组序列; 5. 用于人工染色体PAC、YAC 和BAC 的片段搭接。 其主要原理是根据 已知DNA 序列,分别设计三条同向且退火温度较高的特异性引物(SP Primer),与退火温度较低的兼并引物,进行热不对称PCR 反应。 现在有很多Genome Walking Kit,相应的说明书都介绍的很详尽,下面给你发一个原理图:
大致的原理是这样的,依据TaKaRa的试剂盒,具体的操作步骤如下: 1. 基因组DNA 的获取。 基因组DNA 的质量是侧翼序列获取成功与否的关键因素之一。建议不要使用只经过简单处理的基因 组DNA(例如:只进行细胞热处理或蛋白酶处理)作为模板,而要使用经过充分纯化的完整的基因组 DNA。此外,由于本方法灵敏度极高,模板DNA 一定不要污染,所需的DNA 量不要少于3 μg。 2. 已知序列的验证。 在进行PCR 实验之前必须对已知序列进行验证,以确认已知序列的正确性。具体方法为:根据已知序 列设计特异性引物(扩增长度最好不少于500 bp),对模板进行PCR 扩增,然后对PCR 产物进行测 序,再与参考序列比较确认已知序列的正确性。 3. 特异性引物的设计。 根据验证的已知序列,按照前述的特异性引物设计原则设计三条特异性引物,即:SP1、SP2、SP3。 4. 1st PCR 反应。 基因组DNA 经OD 测定准确定量后,取适量作为模板(不同物种的最佳反应DNA 量并不相同,实际 用量参考下面的注*1),以AP Primer(四种中的任意一种,以下以AP1 Primer 为例)作为上游引 物,SP1 Primer 为下游引物,进行1st PCR 反应。
基因组学重点整理
生物五界:动物、植物、真菌、原生生物和原核生物;生物三界:真细菌、古细菌、真核生物 具有催化活性的RNA分子称为核酶(ribozyme)核酶催化的生化反应有:自我剪接、催化切断其它RNA、合成多肽键、催化核苷酸的合成 新基因的产生:基因与基因组加倍1)整个基因组加倍;2)单条或部分染色体加倍;3)单个或成群基因加倍。DNA水平转移:原核生物中的DNA水平转移可通过接合转移,噬菌体转染,外源DNA的摄取等不同途径发生,水平转移的基因大多为非必须基因。动物中由于种间隔离不易进行种间杂交,但其主要来源于真核细胞与原核细胞的内共生。动物种间基因转移主要集中在逆转录病毒及其转座成分。 外显子洗牌与蛋白质创新:产生全新功能蛋白质的方式有二种:功能域加倍,功能域或外显子洗牌 基因冗余:一条染色体上出现一个基因的很多复份(复本)当人们分离到某一新基因时,为了鉴定其生物学功能,常常使其失活,然后观察它们对表型的影响。许多场合,由于第二个重复的功能基因可取代失活的基因而使突变型表型保持正常。这意味着,基因组中有冗余基因存在。看家基因很少重复,它们之间必需保持剂量平衡,因此重复的拷贝很快被淘汰。与个体发育调控相关的基因表达为转录因子,具有多功能域的结构。这类基因重复拷贝变异可使其获得不同的表达控制模式,促使细胞的分化与多样性的产生,并导致复杂形态的建成,具有许多冗余基因。 非编码序列扩张方式:滑序复制、转座因子 模式生物海胆、果蝇、斑马鱼、线虫、蟾蜍、小鼠、酵母、水稻、拟南芥等。模式生物基因组中G+C%含量高, 同时CpG 岛的比例也高。进化程度越高, G+C 含量和CpG 岛的比例就比较低 如果基因之间不存在重叠顺序,也无基因内基因(gene-within-gene),那么ORF阅读出现差错的可能只会发生在非编码区。细菌基因组中缺少内含子,非编码序列仅占11%, 对阅读框的排查干扰较少。细菌基因组的ORF阅读相对比较简单,错误的机率较少。高等真核生物DNA的ORF阅读比较复杂:基因间存在大量非编码序列(人类占70%);绝大多数基因内含有非编码的内含子。高等真核生物多数外显子的长度少于100个密码子 内含子和外显子序列上的差异:内含子的碱基代换很少受自然选择的压力,保留了较多突变。由于碱基突变趋势大多为C-T,故A/T的含量内含子高于外显子。由于终止密码子为TAA\TAG\TGA,如果以内含子作为编码序列,3种读码框有很高比例的终止密码子。 基因注释程序编写的依据:1)信号指令,包括起始密码子,终止密码子,终止信号,剪接受体位和供体位,多聚嘧啶序列,分支点保守序列2)内容指令,密码子偏好,内含子和外显子长短 基因功能的检测:基因失活、基因过表达、RNAi干涉 双链DNA的测序可从一端开始,亦可从两端进行,前者称单向测序,后者称双向测序。 要获得大于50 kb的DNA限制性片段必需采用稀有切点限制酶。 酵母人工染色体(YAC)1)着丝粒在细胞分裂时负责染色体均等分配。2)端粒位于染色体端部的特异DNA序列,保持人工染色体的稳定性3)自主复制起始点(ARS)在细胞中启动染色体的复制 合格的STS要满足2个条件:它应是一段序列已知的片段,可据此设计PCR反应来检测不同的DNA片段中是否存在这一顺序;STS必需在染色体上有独一无二的位置。如果某一STS在基因组中多个位点出现,那么由此得出的作图数据将是含混不清的。 遗传图绘制主要依据由孟德尔描述的遗传学原理,第一条定律为等位基因随机分离,第二条定律为非等位基因自由组合,显隐性规律/不完全显性、共显性、连锁 衡量遗传图谱的水平覆盖程度饱和程度 基因类型:transcribed, translatable gene (蛋白基因) ;transcribed but non-translatable gene ( RNA基因)Non- transcribed, non-translatablegene ( promoter, operator ) rRNA基因,tRNA基因, scRNA基因, snRNA基因, snoRNA基因, microRNA基因 基因组(genome):生物所具有的携带遗传信息的遗传物质总和。 基因组学(genomic):用于概括涉及基因作图、测序和整个基因功能分析的遗传学分支。 染色体组(chromosome set):不同真核生物核基因组均由一定数目的染色体组成,单倍体细胞所含有的全套染色体。 比较基因组学(comparative genomics):比较基因组学是基因组学与生物信息学的一个重要分支。通过模式生物基因组与人类基因组之间的比较与鉴别,为分离重要的候选基因,预测新的基因功能,研究生物进化提供依据。(目标)
基于PCR的染色体步移
1依赖酶切连接的PCR 这类方法首先对基因组DNA酶切之后,然后让酶切产物自我成环或在酶切产物末端加上相应的接头。以已知序列上的特异引物或接头上的锚定引物,扩增已知序列的上下游侧翼序列。 1.1 反向PCR 反向PCR(inversPCR,IPCR)是Triglia等提出的扩增已知序列上游和下游未知序列的方法。该方法的基本原理是对已知序列进行分析,选择已知序列中没有的限制性内切酶位点,酶切基因组DNA后,酶切片段环化自连,然后利用已知序列设计的反向引物,扩增已知序列两侧的未知序列,原理如图1。因此,这种方法包含以下3个步骤:(1)基因组DNA 酶切;(2)线性酶切产物环化;(3)已知序列处设计的反向引物扩增目标片段。在PCR 扩增之前,也可以根据已知序列用合适的内切酶将环化的DNA切割成线性DNA,这样更利于PCR反应的进行。 图1:反向PCR原理图 最初,IPCR用于扩增基因组DNA的侧翼未知序列,后来利用T4聚合酶补平双链cDNA 末端后再环化,可以有效扩增已知cDNA的侧翼未知序列。然而,IPCR存在技术缺点:(1)环化过程难以控制,基因组DNA酶切后,不同的酶切片段连接成线性串联体,致使非特异性扩增;(2)基因组DNA酶切位点随机分布,可能产生过长的酶切片段导致PCR扩增效率下降,成功率降低。Benkel等在此基础上对IPCR的反应体系改进,可以扩增长达40Kb的片段。Kohda等在对基因组DNA酶切之后,环化连接过程中,酶切位点之间加上一段桥连片段,有效扩增侧翼片段,这种方法称为桥连反向PCR(BI-PCR)。BI-PCR扩增的特异性更高,操作更加简便。最近,Tsaftaris等利用改进的滚环复制反向PCR从植物基因组DNA中成功分离出3Kb的启动子序列。以IPCR为代表的连接成环PCR策略是首次利用PCR技术进行的染色体步移技术。以此为基础,对基因组DNA酶切,在酶切末端加上接头或通过相应的方法在酶切末端加上已知序列,获得目标序列的PCR扩增的染色体步移技术大量涌现。 1.2 载体PCR 载体PCR(single specific primePCR,SSP-PCR)是通过把基因组DNA酶切之后,连接到质 粒载体(如SpBluescript KS)上,并用依据已知序列设计的特异引物和载体上的通用引物特异扩增目标片段的一种PCR染色体步移技术。
植物基因克隆
来自dxy 22003luocong 植物基因全长克隆几种方法的比较 基因是遗传物质基本的功能单位,分离和克隆目的基因是研究基因结构、揭示基因功能及表达的基础,因此,克隆某个功能基因是生物工程及分子生物学研究的一个重点。经典克隆未知基因的方法比如通过筛选文库等有个共同的弊病, 即实验操作繁琐, 周期较长、工作量繁重,且不易得到全长序列。又由于在不同植物中目的基因mRNA丰度不同,所以获得目的基因的难易程度又不一样,特别是对于丰度比较低的目的基因即使使用不用的方法也不一定能获得成功。近年来随着PCR技术的快速发展和成熟.已经有多种方法可以获得基因的全长序列, 比如经典的RACE技术,染色体步移法和同源克隆法等,本文主要综述几种重要的克隆方法的原理和运用,并且比较分析这几种方法的优缺点,为你的实验节约时间和成本。 1 RACE技术 1985年由美国PE-Cetus公司的科学家Mulis等[1]发明的PCR技术使生命科学得到了飞跃性的发展。1988年Frohman等[2] 在PCR技术的基础上发明了一项新技术, 即cDNA末端快速扩增技术( rapid amplification of cDNA ends, RACE), 其实质是长距PCR( long distance, PCR)。通过PCR由已知的部分cDNA 序列, 获得5′端和3′端完整的cDNA, 该方法也被称为锚定PCR ( anchored PCR) [3] 和单边PCR( one-sidePCR) [4]。RACE技术又分为3?RACE和5?端RACE。3′RACE 的原理是利用mRNA 的3′端天然的poly(A) 尾巴作为一个引物结合位点进行PCR, 以Oligo( dT) 和一个接头组成的接头引物( adaptor primer, AP)反转录mRNA得到加接头的第一链cDNA。然后用一个正向的基因特异性引物( gene-specific primer, GSP) 和一个含有接头序列的引物分别与已知序列区和poly(A) 尾区退火, 经PCR扩增位于已知序列区域和poly( A) 尾区之间的未知序列,若为了防止非特异性条带的产生, 可采用巢式引物( nested primer) 进行第二轮扩增, 即巢式PCR( nested PCR) [5,6]。5?RACE 跟3?RACE原理基本一样,但是相对于3?RACE来说难度较大。 5'-RACE受到诸多因素的影响而常常不能获取全长,因此研究者都着手改进它。这些措施主要是通过逆转录酶、5'接头引物等的改变来实现的,因此出现了包括基于“模板跳转反转录”的SMART RACE技术( switching mechanism at 5′ end of RNA transcript) [7] , 基于5′脱帽和RNA酶连接技术的RLM-RACE技术(RNA ligase mediated RACE)[8], 利用RNA连接酶为cDNA第一链接上寡聚核苷酸接头的SLC RACE技术(single strand ligation to single-stranded cDNA)[9] , 以及以内部环化的cDNA第一链为模板进行扩增的自连接或环化RACE技术(self-ligation RACE or circular RACE)[10],和通过末端脱氧核苷酸转移酶( TdT)加尾后引入锚定引物的锚定RACE技术( anchored RACE)[11]。 笔者主要介绍两种比较新的RACE技术,基于…模板跳转?的SMART RACE 技术和末端脱氧核苷酸转移酶( TdT)加尾技术。 1.1基于‘模板跳转’的SMART RACE技术[7,12]
应用于染色体步移的PCR扩增技术的研究进展
遗 传HEREDIT AS (Beijing )28(5):587~595,2006 专论与综述 收稿日期:20050907;修回日期:20051109 作者简介:刘 博(1979— ),男,黑龙江人,硕士生,专业方向:植物基因工程。Tel :0411287509077;E 2mail :ppic9k @https://www.360docs.net/doc/d46319530.html, 通讯作者:安利佳(1955— ),男,教授,博士生导师,研究方向:植物基因工程及基因工程药物。Tel :0411284706365;E 2mail :bioeng @https://www.360docs.net/doc/d46319530.html, 应用于染色体步移的PCR 扩增技术的研究进展 刘 博1,2,苏 乔1,汤敏谦2,袁晓东2,安利佳1 (1.大连理工大学环境与生命科学院,大连116023;2.宝生物工程(大连)有限公司,大连116600) 摘 要:各种建立在PCR 基础上染色体步移的方法能够根据已知的基因序列得到侧翼的基因序列。染色体步移技术主要应用于克隆启动子、步查获得新物种中基因的非保守区域、鉴定T 2DNA 或转座子的插入位点、染色体测序工作中的空隙填补,从而获得完整的基因组序列等方面。其方法主要有3种:反向PCR 的方法,连接法介导的PCR 的方法以及特异引物PCR 的方法。文章就各种方法进行举例说明并加以分析比较。关键词:染色体步移;反向PCR ;连接法PCR ;特异引物PCR 中图分类号:Q75 文献标识码:A 文章编号:0253-9772(2006)05-0587-09 Progres s of the PCR Amplification T echniques f or Chromos ome Walking LI U Bo 1,2,SU Qiao 1,T ANG Min 2Qian 2,Y UAN X iao 2Dong 2,AN Li 2Jia 1 (1.School of Environmental and Biological Science and Technology ,Dalian University of Technology ,Dalian 116023,China ;2.Ta KaRa Biotechnology (Dalian )Co.,Ltd.Dalian 116600,China ) Abs t ra ct :Various PCR 2based methods are available for chromosome walking from a known sequence to an unknown region.It is promising in genome 2related re search for the following experiments :promoter cloning ,obtaining non 2con 2servative parts of genes in new species ,identification of T 2DNA or transposon insertion site s and filling in gap s or un 2known chromosome regions in genome sequencing.The se methods consisted of three type s :inverse PCR ,ligation 2me 2diated PCR and specific primer PCR.In this review ,we illustrated and compared the current techniques.Ke y w or ds :chromosome walking ;inverse PCR ;ligation 2mediated PCR ;specific primer PCR 获得与已知序列相邻的未知序列的染色体步移技术是一项重要的分子生物学研究技术。染色体步移技术主要有以下几方面的应用:(1)根据已知的基因或分子标记连续步移获取人、动物和植物的重要调控基因,用于研究结构基因的表达调控,如:分离克隆启动子并对其功能的研究;(2)步查获得新物种中基因的非保守区域,从而获得完整的基因序列;(3)鉴定T 2DNA 或转座子的插入位点,以及鉴定基因枪转基因法等转基因技术所导致的外源基因的插入位点;(4)用于染色体测序工作中的空隙填补,从而获得完整的基因组序列;(5)用于人工染色体PAC 、Y AC 和BAC 的片段搭接。对于基因组测序已 经完成的少数物种(如人、小鼠、线虫、水稻、拟南芥 等)来说,可以轻松地从数据库中找到已知序列的侧翼序列。但是这毕竟只是研究少数模式生物时的情况,对于自然界中种类繁多的其他生物而言,在不知道它们的基因组DNA 序列以前,想要知道一个已知区域两侧的DNA 序列,只能采用染色体步移技术。因而,染色体步移技术在现代分子生物学研究中占有举足轻重的地位,是结构基因组研究以及功能基因组研究的基础。 自从PCR 技术发明以来,人们就在PCR 技术的基础上设计了多种扩增未知序列的染色体步移的方法,这些方法依其原理可分为3类:反向PCR 的
染色体步移技术
染色体步移技术(Genome Walking) 染色体步行是一种常用的克隆已知基因旁侧序列的技术。染色体步行是指由生物基因组或基因组文库中的已知序列出发逐步探知其旁邻的未知序列或与已知序列呈线性关系的目的序列的核苷酸。 现有的染色体步行PCR技术包括反向PCR、锅柄PCR、连接介导PCR、热不对称PCR、SON PCR等染色体步移技术主要有以下几方面的应用: 1. 根据已知的基因或分子标记连续步移,获取人、动物和植物的重要调控基因,可以用于研究结构基因的表达调控。如分离克隆启动子并对其功能进行研究; 2. 步查获取新物种中基因的非保守区域,从而获得完整的基因序列; 3. 鉴定T-DNA或转座子的插入位点,鉴定基因枪转基因法等转基因技术所导致的外源基因的插入位点等; 4. 用于染色体测序工作中的空隙填补,获得完整的基因组序列; 5. 用于人工染色体PAC、Y AC 和BAC 的片段搭接。 其主要原理是根据已知DNA 序列,分别设计三条同向且退火温度较高的特异性引物(SP Primer),与退火温度较低的兼并引物,进行热不对称PCR 反应。 现在有很多Genome Walking Kit,相应的说明书都介绍得很详尽,下面给你发一个原理图: 依据TaKaRa的试剂盒,具体的操作步骤如下: 1. 基因组DNA的获取。 基因组DNA的质量是侧翼序列获取成功与否的关键因素之一。建议不要使用只经过简单
组DNA(例如:只进行细胞热处理或蛋白酶处理)作为模板,而要使用经过充分纯化的完整的基因组 DNA。此外,由于本方法灵敏度极高,模板DNA一定不要污染,所需的DNA量不要少于 3 μg。 2. 已知序列的验证。 在进行PCR 实验之前必须对已知序列进行验证,以确认已知序列的正确性。具体方法为:根据已知序 列设计特异性引物(扩增长度最好不少于500 bp),对模板进行PCR 扩增,然后对PCR 产物进行测 序,再与参考序列比较确认已知序列的正确性。 3. 特异性引物的设计。 根据验证的已知序列,按照前述的特异性引物设计原则设计三条特异性引物,即:SP1、SP2、SP3。 4. 1st PCR 反应。 基因组DNA经OD 测定准确定量后,取适量作为模板(不同物种的最佳反应DNA量并不相同,实际 用量参考下面的注*1),以AP Primer(四种中的任意一种,以下以AP1 Primer 为例)作为上游引 物,SP1 Primer 为下游引物,进行1st PCR 反应。 ①按下列组份配制1st PCR反应液。 Template(基因组DNA)x ng *1 dNTP Mixture(2.5 mΜ each )8 μl 10×LA PCR Buffer II(Mg2+ plus) 5 μl TaKaRa LA Taq ?(5 U/μl)0.5 μl AP1 Primer(100 pmol/μl) 1 μl SP1 Primer(10 pmol/μl) 1 μl dH2O up to 50 μl ②1st PCR 反应条件如下: 94℃ 1 min 98℃ 1 min 94℃30 sec 60~68℃*2 1 min 5 Cycles 72℃2~4 min*3 94℃30 sec;25℃ 3 min;72℃2~4 min*3 94℃30 sec;60~68℃*2 1 min;72℃2~4 min*3 94℃30 sec;60~68℃*2 1 min;72℃2~4 min*3 15 Cycles 94℃30 sec;44℃ 1 min;72℃2~4 min*3 72℃10 min 5. 2nd PCR 反应。 将1st PCR反应液稀释1~1000 倍后,取1 μl作为2nd PCR反应的模板,以AP1 Primer 为上游引 物,SP2 Primer为下游引物,进行2nd
遗传学名词解释(中英对照版)
遗传学名词解释(中英对照版) abortive transduction 流产转导:转导的DNA片段末端掺入到受体的染色体中,在后代中丢失。 acentric chromosome 端着丝粒染色体:染色体的着丝粒在最末端。 Achondroplasia 软骨发育不全:人类的一种常染色体显性遗传病,表型为四肢粗短,鞍鼻,腰椎前凸。 acrocetric chromosome 近端着丝粒染色体:着丝粒位于染色体末端附近。 active site 活性位点:蛋白质结构中具有生物活性的结构域。 adapation 适应:在进化中一些生物的可遗传性状发生改变,使其在一定的环境能更好地生存和繁殖。 adenine 腺嘌呤:在DNA中和胸腺嘧啶配对的碱基。 albino 白化体:一种常染色体隐性遗传突变。动物或人的皮肤及毛发呈白色,主要因为在黑色素合成过程中,控制合成酪氨酸酶的基因发生突变所致。 allele 等位基因:一个座位上的基因所具有的几种不同形式之一。 allelic frequencies (one frequencies)在群体中存在于所有个体中某一个座位上等位基因的频率。 allelic exclusion 等位排斥:杂合状态的免疫球蛋白基因座位中,只有一个基因因重排而得以表达,其等位基因不再重排而无活性。 allopolyploicly 异源多倍体:多倍体的生物中有一套或多套染色体来源于不同物种。 Ames test 埃姆斯测验法:Bruce Ames 于1970年人用鼠伤寒沙门氏菌(大鼠)肝微粒体法来检测某些物质是否有诱变作用。 amino acids 氨基酸:是构成蛋白质的基本单位,自然界中存在20种不同的氨基酸。 aminoacyl-tRNA 氨基酰- tRNA:tRNA的氨基臂上结合有相应的氨基酸,并将氨基酸运转到核糖体上合成蛋白质。 aminoacyl-tRNA synthetase 氨基酰- tRNA合成酶:催化一个特定的tRNA结合到相应的tRNA分子上。因有20种氨基酸,故有20种氨基酰- tRNA合成酶。 amniocentesis 羊膜穿刺术:产前诊断中一种采羊水的方法。 amorph 无效等位基因:一种突变的形式,突变后的基因不能指令合成有功能的蛋白质。 amphiodiploid 双二倍体:即异源四倍体。 amplification 扩增:许多DNA拷贝的产物来自DNA的一个主要区域。 aneuploid 非整倍体:一种染色体数目的变异体,细胞中增加或减少一条或几条染色体。 annealing 退火:即DNA复性,降低温度使两条DNA单链重新互补结合形成双链的过程。 antibody 抗体:一种免疫蛋白分子,由免疫系统产生,可识别特异抗原并与之结合。 anticodon 反密码子:在tRNA的反密码子环上的三个相连的碱基可和mRNA上的密码子 互补结合。 antigen 抗原:任何一种可刺激机体产生抗体,并能与之特异结合的大分之物质。 AP site AP位点:即脱嘧啶脱嘌呤的位点,由DNA上碱基糖苷链的断裂形成,在修复时可供脱嘧啶嘌呤内切酶识别。 ascospore 子囊孢子:某些真菌产生的有性孢子,位于子囊中,为单倍性,常呈线状排列。 attached X 并联X染色体:一对果蝇的X染色体端端相连,作为一个单位进行遗传。
遗传学名词解释
小木虫 遗传学名词解释!!! A 腺嘌呤(adenine) abortive transduction 流产转导:转导的DNA片段末端掺入到受体的染色体中,在 后代中丢失。 acentric chromosome 端着丝粒染色体:染色体的着丝粒在最末端。Achondroplasia 软骨发育不全:人类的一种常染色体显性遗传病,表型为四肢粗短,鞍鼻,腰椎前凸。 acrocetric chromosome 近端着丝粒染色体:着丝粒位于染色体末端附近。 active site 活性位点:蛋白质结构中具有生物活性的结构域。 adapation 适应:在进化中一些生物的可遗传性状发生改变,使其在一定的环境能 更好地生存和繁殖。 adenine 腺嘌呤:在DNA中和胸腺嘧啶配对的碱基。 albino 白化体:一种常染色体隐性遗传突变。动物或人的皮肤及毛发呈白色,主 要因为在黑色素合成过程中,控制合成酪氨酸酶的基因发生突变所致。 allele 等位基因:一个座位上的基因所具有的几种不同形式之一。 allelic frequencies (one frequencies) 在群体中存在于所有个体中某一个座位上等位 基因的频率。 allelic exclusion 等位排斥:杂合状态的免疫球蛋白基因座位中,只有一个基因因 重排而得以表达,其等位基因不再重排而无活性。 allopolyploicly 异源多倍体:多倍体的生物中有一套或多套染色体来源于不同物种。Ames test 埃姆斯测验法: Bruce Ames 于1970年人用鼠伤寒沙门氏菌(大鼠)肝 微粒体法来检测某些物质是否有诱变作用。
染色体步移技术
螺旋讲堂2010年第4期总第23期 染色体步移 内容概览: 一、染色体步移技术概述 二、常见的染色体步移技术介绍 1、结合基因组文库的染色体步移技术 1) 物理剪切法构建亚克隆文库2) 限制性内切酶法构建亚克隆文库2、基于PCR 技术的染色体步移技术 1) 连接成环PCR 2)外源接头介导PCR (1)连接载体的PCR (2)连接单链接头的PCR (3)连接双链接头PCR 3)半随机引物PCR 策略 (1)新Alu-PCR (2)DW-ACP TM PCR (3)热不对称交错PCR (TAIL-PCR ) 三、染色体步移技术之TAIL-PCR 1、TAIL-PCR 的原理 2、TAIL-PCR 的优点以及技术难点 3、TAIL-PCR 的操作流程 4、TAIL-PCR 中的注意事项 5、常见问题分析 生物人的网上家园 plum 螺旋网HelixNet 染色体步移技术是一种常用的克隆已知片段旁侧序列的技术。本文中主要总结了近年来染色体步移技术的发展情况,介绍了结合基因组文库的染色体步移技术和 基于PCR 的染色体步移技术,并且比较了他们之间的优缺点,最后着重与大家一起 讨论一下TAIL-PCR 的相关内容,希望能对大家有所帮助。
一、染色体步移技术概述 染色体步移(Chromosome walking)又称为基因组步移(Genome walking)是指由生物基因组或基因组文库中的已知序列出发,逐步探知其旁邻的未知序列或与已知序列呈线性关系的目标序列的方法。对于已经完成基因组测序的模式生物种(如人、小鼠、线虫、水稻、拟南芥等),可直接从数据库中轻松的找到已知序列的侧翼序列。但是目前为止,自然界中大多数生物基因组DNA序列仍未知,要想知道一个已知区域两侧的DNA序列,染色体步移无疑是一种非常有效的方法。因此,染色体步移技术在现代分子生物学研究中较为重要,本文主要就染色体步移的相关方法,不同方法之间的比较,着重以TAIL-PCR的原理,实验操作及注意事项等进行讨论。 染色体步移技术的主要应用可归结为以下5个方面: (1)鉴定T—DNA或转座子的插入位点,鉴定转基因技术所导致的外源基因的插入位点; (2)根据基因的已知片段、EST或插入的转座子序列克隆目的基因,分离基因的启动子及调控元件; (3)用于人工染色体PAC、YAC和BAC的片段搭接; (4)构建图位克隆中的重叠群; (5)转化标记辅助育种中的STS或SCAR标记等。 二、常见的染色体步移技术介绍 目前,分离侧翼序列的染色体步移方法主要有两种,一是结合基因组文库为主要手段的染色体步移技术,构建基因组文库进行染色体步移尽管步骤比较繁琐,但是适于长距离步移,可以获得代表某一特定染色体的较长连续区段的重叠基因组克隆群。随着亚克隆文库条件构建条件的优化及测序技术的进步,这种方法也将更加快捷,准确。另一个是基于PCR扩增为主要手段的染色体步移技术。基于PCR扩增为主要手段的染色体步移技术步移距离相对较短,但是操作比较简单,尤其适合于已知一段核苷酸序列的情况下进行的染色体步移。在此基础上人们相继发明了十几种侧翼序列克隆的方法,依据其技术原理,可以将这些方法分为3类:连接成环PCR、外源接头介导PCR和半随机引物PCR。 1、结合基因组文库的染色体步移技术 基因组文库是指生物体全部DNA,经过合适的核酸内切酶消化或机械切割以后,克隆到适当的载体分子中,构成重组体分子群体,然后转化给诸如大肠杆菌这样的寄主菌株进行复制繁殖,如此构建的理论上含有生物体整个基因组全部遗传信息的克隆集合体,称作基因组文库,它是进行基因组学研究的重要技术平台,在基因组物理图谱的构建以及基因的图位克隆中发挥了重要作用。基因组文库的种类主要有入噬菌体文库、酵母人工染色体文库(yeast artificial chromosome,YAC)、P1噬菌体文库和细菌人工染色体文库(bacterial artificial chromosome,BAC)等。其中,BAC文库因其插入片段大、嵌合率低、遗传稳定性好、 易于操作等优点而备受青睐,近年来,小麦、棉花、水稻等越来越多的物种构建了BAC文库。 在基因克隆中,该方法用于鉴定一系列彼此重叠的DNA限制片段,分离序列及表达产物未知的基因,特别 是发育相关的基因。利用基因组文库克隆目的基因,首先要有一个根据目的基因建立起来的遗传分离群体, 找到与目的基因紧密连锁的分子标记,然后用遗传作图将目的基因定位在染色体的特定位置,找到与目的 基因紧密连锁的分子标记。构建含有插人大片段DNA的基因组文库(BAC或YAC),以与目的基因连锁的 分子标记为探针筛选基因组文库,鉴定出分子标记所在的大片段克隆,再以该克隆为染色体步移的起点, 用外侧克隆末端作为探针筛选基因组文库,分离新的重叠克隆用获得的阳性克隆,多次重复上述步骤,逐 渐逼近目的基因,构建目的基因区域的重叠群。随后通过亚克隆文库获得含有目的基因的小片段克隆,最 后通过遗传转化和功能互补验证最终确定目的基因的碱基序列。 目前构建亚克隆文库比较常用的DNA片段化方法主要有两种:物理剪切法和限制性内切酶法。
分子生物学考试大题
1、Each allele has a different phenotype ,why(illustrate by example). 位于染色体同一位置的分别控制两种不同性状的基因是等位基因,等位基因之间具有多种关系。比如,果蝇中white位点的存在对红眼的形成是必要的。这个位点是根据无义突变命名的,该突变使果蝇在突变杂合子中具有白色的眼睛。表述野生型和突变型基因时,通常在野生型基因后面加上+。 当等位基因存在时,一个动物可能是携带两种突变基因的杂合子。这种杂合子的表型依赖于每一个突变所遗留下的活性。从本质上讲,两个突变基因间的关系与野生型和突变型间没有区别,一个突变可能都是显性的,也可能部分是显性的。在任何一个遗传位点上并非一定要有一个野生型基因。人类学行系统的比较就提供了一个例子:缺失功能有空白型表示,即O型;但是功能性的A型和B 型是共显性的,并且对O型表现出显性。 2、Conservation of exons and its application. 外显子的保守性可以做为鉴定编码区的基础,即通过确认那些在许多生物体中都存在的序列片段。对于含有这样基因的区域,即在许多生物中这些基因的功能长期被保留下来,这个序列所代表的蛋白质应当有两个特性:它必须有一个开放的阅读框(ORF);在其它生物中很可能存在与它相关的序列。 物种杂交可作为鉴定基因的第一条标准,将来自一定区域的短片段作为探针,通过Southern杂交检测来自不同物种的相关DNA,如果我们发现几个物种中的杂交片段与某一探针相关(探针通常来源于人的DNA),则这个探针就可成为一条基因外显子的候补片段。将这些候补片段进行测序,如果它们有阅读框,则它们就可被用来分离周围的基因组区域;如果它们是一个外显子的一部分,则可以用它来鉴定整条基因,分离相应的cDNA和mRNA,从而最终分离出蛋白质。3、Chromosome walking and its application. 从第一个重组克隆插入片段的一端分离出一个片段作为探针从文库中筛选第二个重组克隆,该克隆插入片段含有与探针重叠的顺序和染色体的其它顺序。从第二个重组克隆的插入片段再分离出末端小片段筛选第三个重组克隆,如此重复,得到一个相邻片段,等于在染色体上移了一步,称为染色体步移。 染色体步移技术是一种重要的分子生物学研究技术,使用这种技术可以有效
反向PCR技术原理和应用
反向PCR技术原理和应用 摘要:PCR只能扩增两端序列已知的基因片段,反向PCR可扩增中间一段已知序列,而两端序列未知的基因片段不扩增。反向PCR的目的在于扩增一段已知序列旁侧的DNA,也就是说这一反应体系不是在一对引物之间而是在引物外侧合成DNA。随着现代医学和分子生物学的飞速发展,该技术在各个领域都发挥着重要的作用。 关键词:反向PCR;原理;不足之处;应用 反向PCR是一种多聚合酶链式反应(PCR)应用的方法,可使已知序列的核心区边侧的未知DNA成几何级数扩增。用适当的限制性内切裂解含核心区的DNA,以产生适合于P CR扩增大小的片段,然后片段的末端再连接形成环状分子。PCR的引物同源于环上核心区的末端序列,但其方向可使链的延长经过环上的未知区而不是分开引物的核心区。这种反向PCR方法可用于扩增本来就在核心区旁边的序列,还可应用于制备未知序列探针或测定边侧区域本身的上、下游序列。 用传统的缓冲液和其他提供者推荐的条件裂解DNA。反向PCR所扩增的片段的大小由PCR扩增片段的大小决定,目前,PCR扩增的实际上限为3-4 kb。在许多情况下,首先需要进行Southern杂交来确定内切酶用以产生大小适于环化及反向PCR的片段的末端片段。能裂解核心区的内切酶使反向PCR只能扩增引物所定模板(依赖于引物)的上游或上游区,而不裂解核心区的酶则使两上边侧序列都扩增,并带有由内切酶和环化类型决定的接点(例如,互补突头连接与钝头连接)。对于扩增左翼或右翼序列,初试时最好靠近识别多个碱基位点的酶,并已知在核心区有其方便的裂解位点。如果用反向PCR从含有大量不同的克隆片段的同一载体中探测杂交探针,建议事先在载体中引入合适的酶切位点。 用T4连接酶在稀DNA浓度下环化更容易形成单环。在一些实验中,为产生对反向PC R大小适当的DNA片段需要两种内切酶,但这样所产生的片段末端则不适于连接,环化前需用Klenow或噬菌体T4 DNA聚合酶修理(钝化)。连接前,需用酚或热变性使内切酶失活。 聚合酶链反应条件与经典所用的相同,例如,94℃-30秒变性,58℃-30秒引物退火,T aq聚合酶70℃延伸3分钟,进行30个循环。可改变PCR条件以生产特异产物。将反向P CR用于测序时,与核心区末端后部结合的扩增引物更为有用,它使测序引物扩增部分的核心序列与未知边侧序列间的接点更近,减少了扩增引物的干扰。 1 反向PCR技术原理 反向PCR可用于研究与已知DNA区段相连接的未知染色体序列,因此又可称为染色体缓移或染色体步移。这时选择的引物虽然与核心DNA区两末端序列互补,但两引物3’端是相互反向的。扩增前先用限制性内切酶酶切样品DNA,然后用DNA连接酶连接成一个环状DNA分子,通过反向PCR扩增引物的上游片段和下游片段;现已制备了酵母人工染色体(YAC)大的线状DNA片段的杂交探针,这对于转座子插入序列的确定和基因库染色体上DNA片段序列的识别十分重要。 2 反向PCR技术不足之处 该方法的不足是:①需要从许多酶中选择限制酶,或者说必须选择一种合适的酶
基因组步移技术概述
基因组步移技术概述 高俊平(山东省潍坊科技学院贾思勰农学院潍坊262700) 摘要基因组步移技术是一种常用的克隆已知片段旁侧序列的技术。本文介绍了近年来出现的几种主流基因组步移技术,以期为相关研究提供方法借鉴。 关键词基因组步移基因组酶切连接PCR 基因组步移(genomic walking)是指从第一个重组克隆插入片段的一端分离出一个片段作为探针,从文库中筛选第二个重组克隆,该克隆含有与探针相重叠的序列。从第二个重组克隆再分离出末端小片段作为探针筛选第三个重组克隆,如此重复,得到一个相邻的片段。它是一种重要的分子生物学研究技术,可以有效克隆已知片段侧翼的未知序列。 迄今为止,基因组步移主要有两种方法:一是结合基因组文库的基因组步移技术,此方法适于长距离步移,可以获得某一特定染色体较长连续区段的重叠基因组克隆群;另一个是以聚合酶链式反应(polymerase chain reaction,PCR)扩增为主要手段的基因组步移技术,该方法产生的步移距离相对较短,但是操作简单,尤其适合于寻求已知片段旁侧的未知序列。1基于基因组文库的基因组步移技术 基因组文库是指生物体的全部DNA经过合适的核酸内切酶消化或机械切割以后,克隆到合适的载体分子中,所有这些插入了基因组DNA片段的载体分子的集合体,就包含了这个生物体的整个基因组信息,也就成为了此生物体的基因组文库。它是基因组学研究的重要技术平台,目前构建基因组文库的方法主要有两种:物理剪切法和限制性内切酶法[1]。 以基因组文库为基础进行基因组步移的步骤大体如下:①用与目的基因连锁的分子标记为探针筛选整个基因组文库,鉴定出分子标记所在的大片段克隆; ②再以该克隆为基因组步移的起点,用外侧克隆末端作为探针筛选基因组文库,分离新的重叠克隆(阳性克隆);③多次重复上述步骤,逐渐逼近目的基因,构建目的基因区域的重叠群;④通过亚克隆文库获得含有目的基因的小片段克隆,并对其进行遗传转化和功能互补验证,从而最终确定目的基因的核酸序列。 2基于PCR技术的基因组步移技术 随着PCR技术的发明,以该技术为基础发展了多种基因组步移方法,这些方法按原理可分为两类:一是依赖酶切连接介导的PCR法;二是不需要酶切连接的PCR法。 2.1依赖酶切连接的PCR此类方法首先对基因组DNA进行酶切,然后把酶切产物连接成环或在酶切产物末端加上相应的接头。根据已知序列设计的特异引物和接头上的锚定引物分别作为PCR反应的正反向引物,以此来扩增已知序列的上下游侧翼序列。2.1.1反向PCR反向PCR(inverse PCR,IPCR)的原理是对已知序列进行限制性内切酶位点分析,然后选取已知序列中没有位点的限制性内切酶,对基因组DNA进行酶切并将酶切片段环化自连,然后根据已知序列设计反向引物,扩增已知序列两侧的未知序列。IPCR的优点是操作简单;缺点是环化过程难以控制,这是因为酶切位点随机分布,有可能产生较长的片段,从而导致PCR扩增效率下降。鉴于此,科研人员在此基础上发展了桥连IPCR:环化的过程中,在酶切位点之间特意加上一段桥连片段,从而能更有效地扩增侧翼片段。Chen等[2]通过IPCR技术克隆了小麦花粉特异性基因TaPSG719的启动子序列。尽管IPCR应用不多,但它开创了利用PCR技术进行基因组步移的先河。以此为基础,大量应用PCR技术进行基因组步移的方法相继涌现。 2.1.2载体PCR载体PCR(single specific primer PCR,SSP-PCR)是指把基因组DNA酶切片段连接到质粒载体上,并用已知序列设计的特异引物和载体上的通用引物特异扩增目标片段的一种PCR技术。SSP-PCR实验设计简单,尤其适合于保存有cDNA或基因组文库的实验室,但是其操作较繁琐,特异性较低,产物仍需做进一步的鉴定。Gowik等[3]运用SSP-PCR技术成功克隆出了一种黄顶菊基因(ppcA1)的启动子序列。2.1.3连接单链接头的PCR单链接头PCR(panhandle PCR,P-PCR)是连接单链接头PCR的典型代表,因其关键环节形成一个锅柄状结构,故也称锅柄PCR。其原理为:基因组DNA先用限制性内切酶酶切,然后在酶切片段末端连接上含有自由端的单链寡核苷酸链(3'端与已知序列中部分片段完全反向互补),这样含有连接片段的单链DNA在合适的温度下会自身退火形成一个类似锅柄结构的环,接下来用在已知序列内呈线性排列的3个单引物进行3次PCR扩增,即得到目的片段。P-PCR的优点是特异性较高,但由于单接头连接效率低,会限制锅柄结构的形成。在此基础上,科研人员对其进行了改进,即在形成锅柄之前在3'末 · 77 · 生物学教学2019年(第44卷)第3期
基因组学-名词解释
Chromosome walking,染色体步移,通过鉴定克隆DNA的重叠部分来构建克隆重叠群的一种方法 Contig,(重叠群)一组连续的重叠DNA序列 C-value paradox,(C值悖论)在每一种生物中其单倍体基因组的DNA总量是特异的,被称为C值 (C Value)。C值和生物结构或组成的复杂性不一致的现象称为C值悖论 CpG island,(CPG岛)人类基因组中大约56%的基因上游富含GC的DNA区域Physical gap,(物理间隙)指构建基因组文库时被丢失的DNA序列,它们从已有的克隆群体中永久性地消失 Restriction mapping,限制性酶切图谱,通过分析限制性酶切片段的大小确定DNA分子中限制性酶切位点 Scaffold,骨架序列,序列间隙分开的一系列序列重叠群 Genomics, 基因组学,研究生物基因组和如何利用基因的一门学问。用于概括涉及基因作图、测序和整个基因组功能分析的遗传学分支。 Proteomics, 蛋白质组学,用来研究蛋白质组的各种技术 Histone code,组蛋白密码,组蛋白化学修饰的模式影响各种细胞活性的假说Map-based cloning,又称定位克隆(positional cloning),用该方法分离基因是根据目的基因在染色体上的位置进行的,无需预先知道基因的DNA 顺序,也无需预先知道其表达产物的有关信息 Restriction fragment length polymorphism (RFLP),限制性片段长度多态性,因为在其一端或两端存在多态性限制位点而产生的长度各异的限制性片段Epigenetics,表观遗传学,是研究基因的核苷酸序列不发生改变的情况下,基因表达的可遗传的变化的一门遗传学分支学科 pseudogene, (假基因)一个失活,即无功能性的基因拷贝 nucleoid,(拟核)原核生物中的含DNA区域 fluorescent in situ hybriduzation, 荧光原位杂交,一种通过观察荧光标记在染色体的位置而确定标记物的技术 sequence tagged site mapping,序列标签位点,基因组中唯一的一段DNA序列mapping reagent,作图试剂,在STS作图中使用的一种分布于单个染色体或整个基因组的DNA片段集合 SSLP, 简单序列长度多态图,一系列表现长度多态性的重复序列 variable number of tandem repeat, 可变数串联重复,由十几个核苷酸的重复序列拷贝组成的简单序列长度多态图,也叫小卫星 short tandem repea t,短序列重复,由二,三或四核甘酸重复单位顺序排列组成的一种简单序列多态性,也叫微卫星 l ong(or short) interspersed nuclear element,长散布重复片段,一种基因组范围的重复序列,常常具有转座活性 RNA world, 进化早期所有生化反应以RNA为中心的时期 genetic mapping, 遗传作图,采用遗传学技术构建基因组图谱 physical mapping(物理图)采用分子生物学技术构建基因组图谱的方法
