a new mathematical model for relative quantification real-time rt-PCR

?2001Oxford University Press Nucleic Acids Research,2001,Vol.29,No.900 A new mathematical model for relative quantification in real-time RT–PCR
Michael W.Pfaffl*
Institute of Physiology,FML-Weihenstephan,Center of Life and Food Sciences,Technical University of Munich, Germany
Received December18,2000;Revised February21,2001;Accepted March14,2001
ABSTRACT
Use of the real-time polymerase chain reaction(PCR) to amplify cDNA products reverse transcribed from mRNA is on the way to becoming a routine tool in molecular biology to study low abundance gene expression.Real-time PCR is easy to perform, provides the necessary accuracy and produces reli-able as well as rapid quantification results.But accu-rate quantification of nucleic acids requires a reproducible methodology and an adequate mathe-matical model for data analysis.This study enters into the particular topics of the relative quantification in real-time RT–PCR of a target gene transcript in comparison to a reference gene transcript.There-fore,a new mathematical model is presented.The relative expression ratio is calculated only from the real-time PCR efficiencies and the crossing point deviation of an unknown sample versus a control. This model needs no calibration curve.Control levels were included in the model to standardise each reac-tion run with respect to RNA integrity,sample loading and inter-PCR variations.High accuracy and reproducibility(<2.5%variation)were reached in LightCycler PCR using the established mathematical model.
INTRODUCTION
Reverse transcription(RT)followed by the polymerase chain reaction(PCR)is the technique of choice to analyse mRNA expression derived from various sources.Real-time RT–PCR is highly sensitive and allows quantification of rare transcripts and small changes in gene expression.As well as this,it is easy to perform,provides the necessary accuracy and produces reliable as well as rapid quantification results.The simplest detection technique for newly synthesised PCR products in real-time PCR uses SYBR Green I fluorescence dye that binds specifically to the minor groove double-stranded DNA(1).The quantification method of choice depends on the target sequence,the expected range of mRNA amount present in the tissue,the degree of accuracy required and whether quantifica-tion needs to be relative or absolute(2).Generally two quanti-fication types in real-time RT-PCR are possible.(i)A relative quantification based on the relative expression of a target gene versus a reference gene.To investigate the physiological changes in gene expression,the relative expression ratio is adequate for the most purposes.(ii)An absolute quantification, based either on an internal or an external calibration curve (1,3).Using such a calibration curve,the methodology has to be highly validated and the identical LightCycler PCR amplifi-cation efficiencies for standard material and target cDNA must be confirmed(4–6).Nevertheless,the generation of stable and reliable standard material,either recombinant DNA or recom-binant RNA,is very time consuming and it must be precisely quantified(2,7,8).Furthermore,a normalisation of the target gene with an endogenous standard is recommended.Therefore, mainly non-regulated reference genes or housekeeping genes like glyceraldehyde-3-phosphate dehydrogenase(G3PDH or GAPDH),albumin,actins,tubulins,cyclophilin,18S rRNA or 28S rRNA(9)were applicable.Housekeeping genes are present in all nucleated cell types since they are necessary for basis cell survival.The mRNA synthesis of these genes is considered to be stable and secure in various tissues,even under experimental treatments(9–11).But numerous studies have already shown that the housekeeping genes are regulated and vary under experimental conditions(12–15).To circum-vent the high expenditure of design and production of standard material,as well as optimisation and validation of a calibration curve based quantification model,and finally the need for normalisation of the target transcripts to an endogenous house-keeping transcript,a reliable and accurate relative quantifica-tion model in real-time RT–PCR is needed.
This study enters into the particular topics of the relative quantification of a target gene in comparison to a reference gene.A new and simple mathematical model for data analysis was established,the application of the new model was tested and compared with available mathematical calculation models. Derived reproducibility,based on intra-and inter-test variation of this relative quantification and accuracy of the model will be discussed.
MATERIALS AND METHODS
RNA source,total RNA extraction and RT
RNA extraction was performed as described previously(16)in bacterial Escherichia coli culture grown either in M9minimal
*To whom correspondence should be addressed at present address:Institut für Physiologie,Weihenstephaner Berg3,85354Freising,Weihenstephan,Germany. Tel:+498161713511;Fax:+498161714204;Email:pfaffl@weihenstephan.de
Nucleic Acids Research,2001,Vol.29,No.900
P AGE 2003OF 2007
media (sample preparation)or LB rich media (control prepara-tion),both with 0.4%glucose concentration (17).RNA integrity was electrophoretically verified by ethidium bromide staining and by OD 260/OD 280nm absorption ratio >1.95.Escherichia coli total RNA (1μg)was reverse transcribed with 100U of Superscript II Plus RNase H –Reverse Trancriptase (Gibco BRL Life Technologies,Gaithersburg,MD)using 100μM random hexamer primers (Pharmacia Biotech,Uppsala,Sweden)according to the manufacturer’s instructions.Optimisation of RT–PCR
Highly purified salt-free primer for target gene1(TyrA,tryptophan operon:forward primer,AAG CGT CTG GAA CTG GTT GC;reverse primer,AAA CGC TGT GCG TAA TCG CC),target gene 2(PyrB,aspartate transcarbamylase:forward primer,GCT CCA ACC AAC ATC CGA;reverse primer,TTC ACG TTG GCG TAC TCG G)and reference gene (Gst,glutathione transferase:forward primer,CTT TGC CGT TAA CCC TAA GGG;reverse primer,GCT GCA ATG TGC TCT AAC CC)were generated (MWG Biotech,Ebersberg,Germany)and optimised to an equal annealing temperature of 60°C.Condi-tions for all PCRs were optimised in a gradient cycler (Master-cycler Gradient,Eppendorf,Germany)with regard to Taq DNA polymerase (Roche Diagnostics,Basel,Switzerland),forward and reverse primers,MgCl 2concentrations (Roche Diagnostics),dNTP concentrations (Roche Diagnostics)and various annealing temperatures (55–65°C).RT–PCR amplification products were separated on a 4%high resolution NuSieve agarose (FMC Bio Products,Rockland,ME)gel electro-phoresis and analysed with the Image Master system (Pharmacia Biotech).Optimised results were transferred on the following LightCycler PCR protocol.LightCycler real-time PCR
For LightCycler reaction a mastermix of the following reaction components was prepared to the indicated end-concentration:13μl water,2.4μl MgCl 2(4mM),0.8μl forward primer (0.4μM),0.8μl reverse primer (0.4μM)and 2.0μl LightCyler (Fast Start DNA Master SYBR Green I;Roche Diagnostics).LightCycler mastermix (19μl)was filled in the LightCycler glass capillaries and 1μl cDNA (3.2,4.0,4.8,16,20or 24ng reverse transcribed total RNA)was added as PCR template.Capillaries were closed,centrifuged and placed into the Light-
Cycler rotor.The following LightCycler experimental run protocol was used:denaturation program (95°C for 10min),amplification and quantification program repeated 40times (95°C for 15s,60°C for 10s,72°C for 60s with a single fluorescence measurement),melting curve program (60–95°C with a heating rate of 0.1°C per second and a continuous fluo-rescence measurement)and finally a cooling step to 40°C.For the mathematical model it is necessary to determine the crossing points (CP)for each transcript.CP is defined as the point at which the fluorescence rises appreciably above the background fluorescence.‘Fit Point Method’must be performed in the LightCycler software 3.3(Roche Diagnostics),at which CP will be measured at constant fluorescence level (18).RESULTS
Confirmation of primer specificity
Specificity of RT–PCR products was documented with high resolution gel electrophoresis and resulted in a single product with the desired length (TyrA,978bp;PyrB,530bp;and Gst,
Table 1.Intra-assay (test precision)and inter-assay variation (test variability)of LightCycler real-time RT–PCR
Determination of variation was done in 20ng reverse transcribed total RNA.Test variation is based on CP variation and expressed as mean CP with CV.
Intra-assay variation (n =3)Inter-assay variation (n =3)Mean CP
CV Mean CP CV TyrA (sample)15.0200.06%15.311 3.58%TyrA (control)20.3030.09%20.426 2.98%PyrB (sample)16.031 2.16%16.289 3.91%PyrB (control)11.720 1.32%12.011 3.18%Gst (sample)14.5330.65%14.371 2.26%Gst (control)
14.277
0.11%
13.997
2.62%
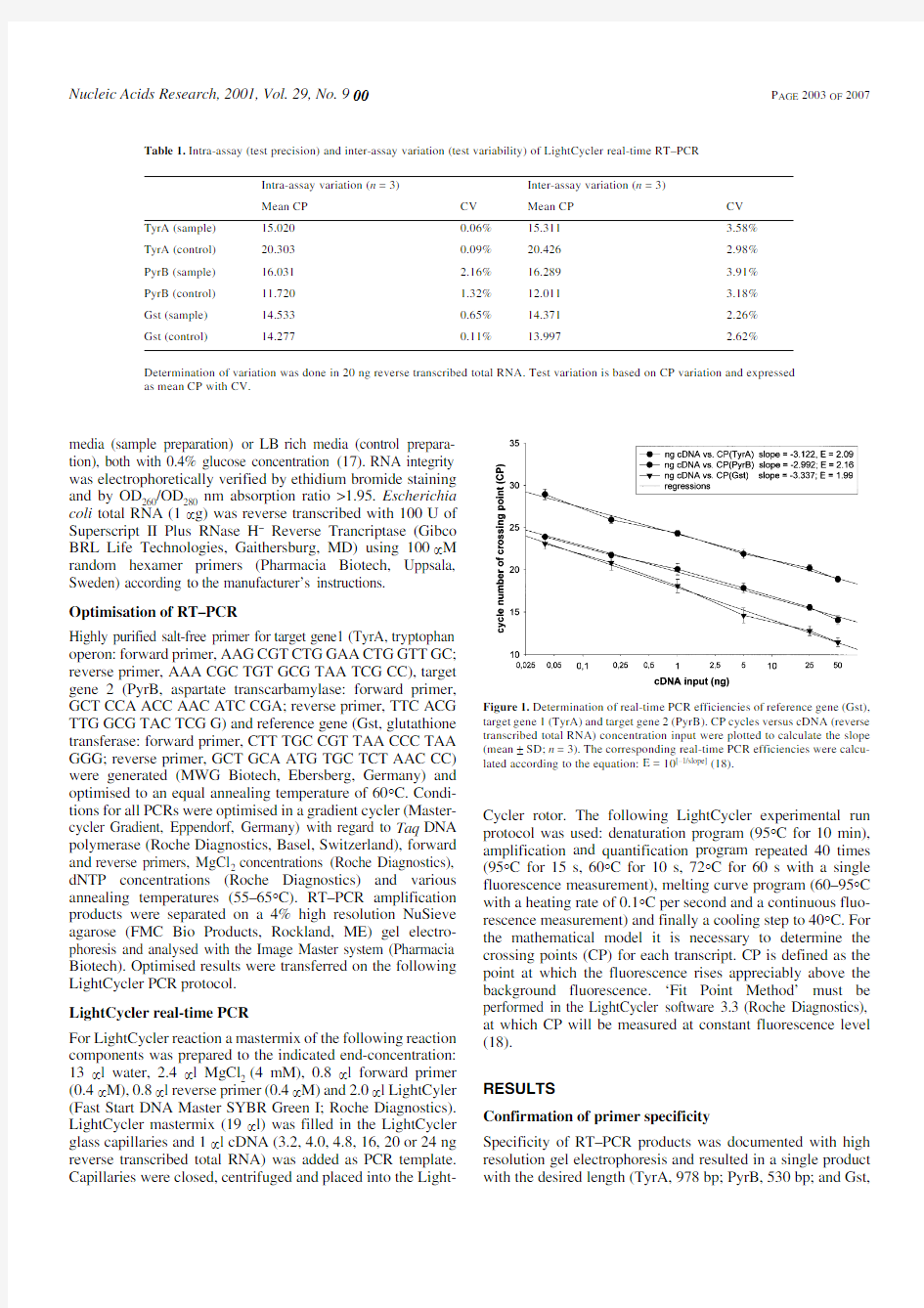
Figure 1.Determination of real-time PCR efficiencies of reference gene (Gst),target gene 1(TyrA)and target gene 2(PyrB).CP cycles versus cDNA (reverse transcribed total RNA)concentration input were plotted to calculate the slope (mean ±SD;n =3).The corresponding real-time PCR efficiencies were calcu-lated according to the equation:E =10[–1/slope](18).
P AGE2004OF200700Nucleic Acids Research,2001,Vol.29,No.9 402bp).In addition a LightCycler melting curve analysis was
performed which resulted in single product specific melting
temperatures as follows:TyrA,89.6°C;PyrB,88.5°C;and Gst,
88.3°C.No primer-dimers were generated during the applied40
real-time PCR amplification cycles.
Real-time PCR amplification efficiencies and linearity
Real-time PCR efficiencies were calculated from the given
slopes in LightCycler software.The corresponding real-time
PCR efficiency(E)of one cycle in the exponential phase was
calculated according to the equation:E=10[–1/slope](Fig.1)(18).
Investigated transcripts showed high real-time PCR efficiency
rates;for TyrA,2.09;PyrB,2.16;and Gst,1.99in the investi-
gated range from0.40to50ng cDNA input(n=3)with high
linearity(Pearson correlation coefficient r>0.95).
Intra-and inter-assay variation
To confirm accuracy and reproducibility of real-time PCR the
intra-assay precision was determined in three repeats within
one LightCycler run.Inter-assay variation was investigated in
three different experimental runs performed on3days using
three different premix cups of LightCycler,Fast Start DNA Master SYBR Green I kit(Roche Diagnostics).Determination of variation was done in20ng transcribed total RNA(Table1). Test reproducibility for all investigated transcripts was low in inter-test experiments(<3.91%)and even lower in intra-test experiments(<2.16%).The calculation of test precision and test variability is based on the CP variation from the CP mean value.
Mathematical model for relative quantification in real-time PCR
A new mathematical model was presented to determine the relative quantification of a target gene in comparison to a refer-ence gene.The relative expression ratio(R)of a target gene is calculated based on E and the CP deviation of an unknown sample versus a control,and expressed in comparison to a reference gene.
1 Equation1shows a mathematical model of relative expression ratio in real-time PCR.The ratio of a target gene is expressed in a sample versus a control in comparison to a reference gene.
E target is the real-time PCR efficiency of target gene transcript;
E ref is the real-time PCR efficiency of a reference gene tran-script;?CP target is the CP deviation of control–sample of the target gene transcript;?CP ref=CP deviation of control–sample of reference gene transcript.The reference gene could be a stable and secure unregulated transcript,e.g.a house-keeping gene transcript.For the calculation of R,the individual real-time PCR efficiencies and the CD deviation(?CP)of the investigated transcripts must be known.Real-time PCR efficiencies were calculated,according to E=10[–1/slope](18),as shown in Figure1.CP deviations of control cDNA minus sample of the target gene and reference genes were calculated according to the derived CP values.Mean CP,variation of CP and?CP values between control and sample of investigated transcripts are listed in Table2.The influence of differing cDNA input concentrations on?CP are also shown.Intended cDNA input concentration variation of control and sample were compared at different levels(low level,3.2,4.0,4.8ng cDNA;high level,16,20and24ng cDNA).They resulted in stable and constant?CP cycle numbers.In Table3the corre-sponding ratios of target genes in comparison to the reference gene were calculated,through to the established mathematical model(equation1).The expression ratios of target genes remain stable,even under intended±20%cDNA variation and low and high cDNA input levels,performed in two runs.A minimal coefficient of variation(CV)of2.50and1.74%was observed,respectively.
Regulation of investigated gene transcripts
All investigated transcript expressions were regulated diver-gently(Table3).The expression of Gst was constant,inde-pendent of media conditions,and therefore was chosen as endogenous standard or reference gene transcript Fig.2.TyrA mRNA expression,measured in20ng cDNA,was up-regulated 49.1-fold(2.095.283)in M9minimal compared to LB rich medium under high cDNA input conditions.Under the considera-tion of the reference gene expression the real up-regulation ratio was,on average,58.5-fold.PyrB mRNA expression was down-regulated under M9minimal medium conditions by a factor of27.6(2.164.311).With the normalisation of the endog-enous standard transcript,the exact relative expression ratio can be calculated to23.2.
DISCUSSION
RT followed by PCR is the most powerful tool to amplify small amounts of mRNA(19).Because of its high ramping rates,limited annealing and elongation time,the rapid cycle PCR in the LightCycler system offers stringent reaction condi-tions to all PCR components and leads to a primer sensitive and template specific PCR(20).The application of fluo-rescence techniques to real-time PCR combines the PCR amplification,product detection and quantification of newly synthesised DNA,as well as verification in the melting curve analysis.This led to the development of new kinetic RT–PCR
ratio=
E target
()?CP target control sample
–
()
E ref
()?CP ref control sample
–
()
-------------------------------------------------------------------
-
Figure2.Real-time RT–PCR SYBR Green I fluorescence history versus cycle
number of target gene1(TyrA),target gene2(PyrB)and reference gene(Gst)
in sample cDNA and control cDNA.CP determination was done at fluores-
cence level1.
Nucleic Acids Research,2001,Vol.29,No.900
P AGE 2005OF 2007
methodologies that are revolutionising the possibilities of mRNA quantification (21).
In this paper,we focused on the relative quantification of target gene transcripts in comparison to a reference gene tran-script.A new mathematical model for data analysis was presented to calculate the relative expression ratio on the basis of the PCR efficiency and crossing point deviation of the investigated transcripts (equation 1).The concept of threshold fluorescence is the basis of an accurate and reproducible quan-tification using fluorescence-based RT–PCR methods (22).Threshold fluorescence is defined as the point at which the fluorescence rises appreciably above the background fluores-cence.In the Fit Point Method,the threshold fluorescence and therefore the DNA amount in the capillaries is identical for all samples.CP determination with the ‘Second Derivative Maximum Method’is not adequate for our mathematical model,because quantification is done at the point of most effi-cient real-time PCR where the second derivative is at its maximum (18).
A linear relationship between the CP,crossing the threshold fluorescence,and the log of the start molecules input in the reaction is given (18,23).Therefore,quantification will always occur during the exponential phase,and it will not be affected by any reaction components becoming limited in the plateau phase (7).In the established model the relative expression ratio
of a target gene is normalised with the expression of an endo-genous desirable unregulated reference gene transcript to compensate inter-PCR variations between the runs.The CP of the chosen reference gene is the same in the control and the sample (?CP =0).Stable and constant reference gene mRNA levels are given.Under these considerations of an unregulated reference gene transcript,no normalisation is needed and equa-tion 1can be shortened to equation 2.
2
Equation 2shows a mathematical model of relative expression ratio in real-time PCR under constant reference gene expression.CP values in the sample and control are equal and represent ideal housekeeping conditions (?CP ref =0,E ref =1).
Two other mathematical models are available for the relative quantification during real-time PCR.The ‘efficiency calibrated mathematical method for the relative expression ratio in real-time PCR’is presented by Roche Diagnostics in a truncated form in an internal publication (24).The complete equation is,
Table 2.Mean CP and CV of target gene 1(TyrA)and target gene 2(PyrB)in comparison to intended
concentration variation of reference gene (Gst)low level (3.2,4.0and 4.8ng per capillary)and high level cDNA input (16,20and 24ng)
cDNA input (ng)
Mean CP (n =3)
CV (%)
?CP (cycles)
High cDNA input level TyrA (sample)2015.0200.06+5.283
TyrA (control)2020.3030.09PyrB (sample)2016.031 2.16–4.311PyrB (control)2011.720 1.32Gst (sample)1614.2900.73–0.277Gst (control)1614.0130.39Gst (sample)2014.5330.65–0.256Gst (control)2014.2770.11Gst (sample)2414.957 1.29–0.227Gst (control)24
14.730
1.14
Low cDNA input level TyrA (sample) 4.017.353 2.16+5.487
TyrA (control) 4.022.840 1.26PyrB (sample) 4.017.6670.75–4.277PyrB (control) 4.013.3900.12Gst (sample) 3.216.9270.643–0.050Gst (control) 3.216.877 1.81Gst (sample) 4.016.750 2.31–0.127Gst (control) 4.016.623 1.04Gst (sample) 4.816.297 1.68–0.077Gst (control)
4.8
16.220
0.69
ratio =E target ()
?CP target control -sample )
(E ref ()0
------------------------------------------------------------------ratio =E target ()
?CP target control -sample ()
P AGE 2006OF 2007
00Nucleic Acids Research,2001,Vol.29,No.9
in principle,the same and the results are in identical relative expression ratio like our model (equation 3).
3
Efficiency calibrated mathematical method for the relative
expression ratio in real-time PCR presented by Soong et al.(24).But the method of calculation in the described mathematical model is hard to understand.The second model available,the ‘Delta–delta method’for comparing relative expression results between treatments in real-time PCR (equation 4)is presented by PE Applied Biosystems (Perkin Elmer,Forster City,CA).4
Equation 4shows a mathematical delta–delta method for comparing relative expression results between treatments in real-time PCR developed by PE Applied Biosystems (Perkin Elmer).Optimal and identical real-time amplification efficiencies of target and reference gene of E =2are presumed.The delta–delta method is only applicable for a quick estima-tion of the relative expression ratio.For such a quick estima-tion,equation 1can be shortened and transferred into equation 4,under the condition that E target =E ref =2.Our presented formula combines both models in order to better understand the mode of CP data analysis and for a more reliable and exact relative gene expression.
Relative quantification is always based on a reference tran-script.Normalisation of the target gene with an endogenous standard was done via the reference gene expression,to compensate inter-PCR variations.Beside this further control levels were included in the mathematical model to standardise each reaction run with respect to RNA integrity,RT efficiency or cDNA sample loading variations.The reproducibility of the RT step varies greatly between tissues,the applied RT isola-tion methodology (25)and the RT enzymes used (26).Different cDNA input concentrations were tested on low and high cDNA input ranges,to mimic different RT efficiencies (±20%)at different quantification levels.In the applied two-step RT–PCR,using random hexamer primers,all possible interferences during RT will influence all target transcripts as well as the internal reference transcript in parallel.Occurring background interferences retrieved from extracted tissue components,like enzyme inhibitors,and cDNA synthesis efficiency were related to target and reference similarly.All products underwent identical reaction conditions during RT and variations only disappear during real-time PCR.Any source of error during RT will be compensated through the model itself.Widely distributed single-step RT–PCR models are not applicable,because in each reaction set-up and for each investigated factor individual and slightly different RT condi-tions will occur.Therefore,the variation in a two-step RT–PCR will always be lower,and the reproducibility of the assay will be higher,that in a single-step RT–PCR (8).Reproducibility of
the developed mathematical model was dependent on the exact determination of real-time amplification efficiencies and on the given low LightCycler CP variability.In our mathematical model the necessary reliability and reproducibility was given,which was confirmed by high accuracy and a relative error of <2.5%using low and high template concentration input.CONCLUSION
LightCycler real-time PCR using SYBR Green I fluorescence dye is a rapid and sensitive method to detect low amounts of mRNA molecules and therefore offers important physiological insights on mRNA expression level.The established mathe-matical model is presented in order to better understand the mode of analysis in relative quantification in real-time RT–PCR.It is only dependent on ?CP and amplification efficiency of the transcripts.No additional artificial nucleic acids,like recombinant nucleic acid constructs in external calibration curve models,are needed.Reproducibility of LightCycler RT–PCR in general and the minimal error rate of the model allows for an accurate determination of the relative expression ratio.Even different cDNA input resulted in minor variations.Rela-tive expression is adequate for the most relevant physiological expression changes.In future it is not necessary to establish more complex and time consuming quantification models based on calibration curves.For the differential display of mRNA the relative expression ratio is an ideal and simple tool for the verification of RNA or DNA array chip technology results.
ACKNOWLEDGEMENTS
The author thanks D.Schmidt for technical assistance.Primers,primer sequences and samples were kindly donated by Drs
ratio =E ref ()Sample
CP
E target ()Sample CP ----------------------------------÷E ref ()
Calibrator
CP
E target ()
Calibrator
CP
--------------------------------------ratio =2?CP sample -?CP control []
–ratio =2
??CP
–Table 3.Influence of variation in cDNA input (±20%)of control and sample (high and low level)on the variation in relative expression ratio based on equation 1and the error of mathematical model,expressed as CV of R Gst
TyrA PyrB E =1.99
E =2.09
E =2.16
High cDNA input level ?CP =+5.283?CP =–4.31180%cDNA input ?CP =–0.277R =59.476R =0.04375100%cDNA input ?CP =–0.256R =58.625R =0.04312120%cDNA input ?CP =–0.227R =57.459R =0.04226Mean of R 58.459000.04304Error/CV of R 1.73%
1.74%
Low cDNA input level ?CP =+5.487?CP =–4.27716%cDNA input ?CP =–0.050R =59.104R =0.0384420%cDNA input ?CP =–0.127R =62.102R =0.0403124%cDNA input DCP =–0.077R =60.208R =0.03914Mean of R 60.4710.03930Error/CV of R
2.50%
2.49%
Nucleic Acids Research,2001,Vol.29,No.900P AGE2007OF2007
S.Wegener and W.Mann in collaboration with the BioChip division of MWG Biotech in Ebersberg,Germany. REFERENCES
1.Morrison,T.,Weis,J.J.and Wittwer,C.T.(1998)Quantification of low-
copy transcripts by continuous SYBR Green I monitoring during
amplification.Biotechniques,24,954–962.
2.Freeman,W.M.,Walker,S.J.and Vrana,K.E.(1999)Quantitative RT–
PCR:pitfalls and potential.Biotechniques,26,112–122.
3.Pfaffl,M.W.(2001)Development and validation of an externally
standardised quantitative insulin like growth factor-1(IGF-1)RT–PCR using LightCycler SYBR?Green I technology.In Meuer,S.,Wittwer,C.
and Nakagawara,K.(eds),Rapid Cycle Real-time PCR,Methods and
Applications.Springer Press,Heidelberg,Germany pp.281–291.
4.Ferré,F.(1992)Quantitative or semi-quantitative PCR:reality versus
myth.PCR Methods Appl.,2,1–9.
5.Souazé,F.,Ntodou-Thomé,A.,Tran,C.Y.,Rostene,W.and Forgez,P.(1996)
Quantitative RT–PCR:limits and accuracy.Biotechniques,21,280–285.
6.Overbergh,L.,Valckx,D.,Waer,M.and Mathieu,C.(1999)Quantification
of murine cytokine mRNAs using real time quantitative reverse
transcriptase PCR.Cytokines,11,305–312.
7.Bustin,S.A.(2000)Absolute quantification of mRNA using real-time
reverse transcription polymerase chain reaction assays.J.Mol.
Endocrinol.,25,169–193.
8.Pfaffl,M.W.and Hageleit,M.(2001)Validities of mRNA quantification
using recombinant RNA and recombinant DNA external calibration
curves in real-time RT–PCR.Biotechnol.Lett.,23,275–282.
9.Marten,N.W.,Burke,E.J.,Hayden,J.M.and Straus,D.S.(1994)Effect of
amino acid limitation on the expression of19genes in rat hepatoma cells.
FASEB J.,8,538–544.
10.Foss,D.L.,Baarsch,M.J.and Murtaugh,M.P.(1998)Regulation of
hypoxanthine phosphoribosyltransferase,glyceraldehyde-3-phosphate
dehydrogenase andβ-actin mRNA expression in porcine immune cells and tissues.Anim.Biotechnol.,9,67–78.
11.Thellin,O.,Zorzi,W.,Lakaye,B.,De Borman,B.,Coumans,B.,Hennen,G.,
Grisar,T.,Igout,A.and Heinen,E.(1999)Housekeeping genes as internal standards:use and limits.J Biotechnol.,75,291–295.
12.Bhatia,P.,Taylor,W.R.,Greenberg,A.H.and Wright,J.A.(1994)Comparison
of glyceraldehyde-3-phosphate dehydrogenase and28S-ribosomal RNA gene expression as RNA loading controls for northern blot analysis of cell lines of varying malignant potential.Anal.Biochem.,216,223–226.13.Bereta,J.and Bereta,M.(1995)Stimulation of glyceraldehyde-3-phosphate
dehydrogenase mRNA levels by endogenous nitric oxide in cytokine-activated https://www.360docs.net/doc/f63134412.html,mun.,217,363–369.
14.Chang,T.J.,Juan,C.C.,Yin,P.H.,Chi,C.W.and Tsay,H.J.(1998)Up-
regulation ofβ-actin,cyclophilin and GAPDH in N1S1rat hepatoma.
Oncol.Rep.,5,469–471.
15.Zhang,J.and Snyder,S.H.(1992)Nitric oxide stimulates auto-ADP-
ribosylation of glyceraldehydes3phosphate dehydrogenase.Proc.Natl https://www.360docs.net/doc/f63134412.html,A,89,9382–9385.
16.Pfaffl,M.W.,Meyer,H.H.D.and Sauerwein,H.(1998)Quantification of
the insulin like growth factor-1mRNA:development and validation of an internally standardised competitive reverse transcription-polymerase
chain reaction.Exp.Clin.Endocrinol.Diabetes,106,502–512.
17.Maniatis,T.,Fritsch,E.F.and Sambrook,J.(1982)Molecular Cloning:A
Laboratory Manual.Cold Spring Harbor Laboratory Press,Cold Spring Harbor,NY.
18.Rasmussen,R.(2001)Quantification on the LightCycler.In Meuer,S.,
Wittwer,C.and Nakagawara,K.(eds),Rapid Cycle Real-time PCR,
Methods and Applications.Springer Press,Heidelberg,pp.21–34.
19.Wang,A.M.,Doyle,M.V.and Mark,D.F.(1989)Quantitation of mRNA by the
polymerase chain reaction.Proc.Natl https://www.360docs.net/doc/f63134412.html,A,86,9717–9721.
20.Wittwer,C.T.,Ririe,K.M.,Andrew,R.V.,David,D.A.,Gundry,R.A.and
Balis,U.J.(1997)The LightCycler:a microvolume multisample
fluorimeter with rapid temperature control.Biotechniques,22,176–181.
21.Orlando,C.,Pinzani,P.and Pazzagli,M.(1998)Developments in
quantitative https://www.360docs.net/doc/f63134412.html,b.Med.,36,255–269.
22.Higuchi,R.,Fockler,C.,Dollinger,G.and Watson,R.(1993)Kinetic PCR
analysis:real-time monitoring of DNA amplification reactions.
Biotechnology,11,1026–1030.
23.Gibson,U.E.,Heid,C.A.and Williams,P.M.(1996)A novel method for
real time quantitative RT–PCR.Genome Res.,6,995–1001.
24.Soong,R.,Ruschoff,J.and Tabiti,K.(2000)Detection of colorectal
micrometastasis by quantitative RT–PCR of cytokeratin20mRNA.
Roche Diagnostics internal publication.
25.Mannhalter,C.,Koizar,D.and Mitterbauer,G.(2000)Evaluation of RNA
isolation methods and reference genes for RT–PCR analyses of rare target https://www.360docs.net/doc/f63134412.html,b.Med.,38,171–177.
26.Wong,L.,Pearson,H.,Fletcher,A.,Marquis,C.P.and Mahler,S.(1998)
Comparison of the efficiency of M-MuLV reverse transcriptase,RNase H–M-MuLV reverse transcriptase and AMV reverse transcriptase for the amplification of human immunglobulin genes.Biotechnol.Techniques, 12,485–489.
中考必会几何模型:8字模型与飞镖模型
8字模型与飞镖模型模型1:角的8字模型 如图所示,AC 、BD 相交于点O ,连接AD 、BC . 结论:∠A +∠D =∠B +∠C . O D C B A 模型分析 证法一: ∵∠AOB 是△AOD 的外角,∴∠A +∠D =∠AOB .∵∠AOB 是△BOC 的外角, ∴∠B +∠C =∠AOB .∴∠A +∠D =∠B +∠C . 证法二: ∵∠A +∠D +∠AOD =180°,∴∠A +∠D =180°-∠AOD .∵∠B +∠C +∠BOC =180°, ∴∠B +∠C =180°-∠BOC .又∵∠AOD =∠BOC ,∴∠A +∠D =∠B +∠C . (1)因为这个图形像数字8,所以我们往往把这个模型称为8字模型. (2)8字模型往往在几何综合题目中推导角度时用到. 模型实例 观察下列图形,计算角度: (1)如图①,∠A +∠B +∠C +∠D +∠E =________; 图图① F D C B A E E B C D A 图③ 2 1O A B 图④ G F 12 A B E 解法一:利用角的8字模型.如图③,连接CD .∵∠BOC 是△BOE 的外角, ∴∠B +∠E =∠BOC .∵∠BOC 是△COD 的外角,∴∠1+∠2=∠BOC . ∴∠B +∠E =∠1+∠2.(角的8字模型),∴∠A +∠B +∠ACE +∠ADB +∠E =∠A +∠ACE +∠ADB +∠1+∠2=∠A +∠ACD +∠ADC =180°. 解法二:如图④,利用三角形外角和定理.∵∠1是△FCE 的外角,∴∠1=∠C +∠E .
∵∠2是△GBD 的外角,∴∠2=∠B +∠D . ∴∠A +∠B +∠C +∠D +∠E =∠A +∠1+∠2=180°. (2)如图②,∠A +∠B +∠C +∠D +∠E +∠F =________. 图② F D C B A E 312图⑤ P O Q A B F C D 图⑥ 2 1 E D C F O B A (2)解法一: 如图⑤,利用角的8字模型.∵∠AOP 是△AOB 的外角,∴∠A +∠B =∠AOP . ∵∠AOP 是△OPQ 的外角,∴∠1+∠3=∠AOP .∴∠A +∠B =∠1+∠3.①(角的8字模型),同理可证:∠C +∠D =∠1+∠2.② ,∠E +∠F =∠2+∠3.③ 由①+②+③得:∠A +∠B +∠C +∠D +∠E +∠F =2(∠1+∠2+∠3)=360°. 解法二:利用角的8字模型.如图⑥,连接DE .∵∠AOE 是△AOB 的外角, ∴∠A +∠B =∠AOE .∵∠AOE 是△OED 的外角,∴∠1+∠2=∠AOE . ∴∠A +∠B =∠1+∠2.(角的8字模型) ∴∠A +∠B +∠C +∠ADC +∠FEB +∠F =∠1+∠2+∠C +∠ADC +∠FEB +∠F =360°.(四边形内角和为360°) 练习: 1.(1)如图①,求:∠CAD +∠B +∠C +∠D +∠E = ; 图 图① O O E E D D C C B B A A 解:如图,∵∠1=∠B+∠D ,∠2=∠C+∠CAD , ∴∠CAD+∠B+∠C+∠D+∠E=∠1+∠2+∠E=180°. 故答案为:180° 解法二:
软件开发模型介绍与对比分析
常用的软件开发模型 软件开发模型(Software Development Model)是指软件开发全部过程、活动和任务的结构框架。软件开发包括需求、设计、编码和测试等阶段,有时也包括维护阶段。 软件开发模型能清晰、直观地表达软件开发全过程,明确规定了要完成的主要活动和任务,用来作为软件项目工作的基础。对于不同的软件系统,可以采用不同的开发方法、使用不同的程序设计语言以及各种不同技能的人员参与工作、运用不同的管理方法和手段等,以及允许采用不同的软件工具和不同的软件工程环境。 1. 瀑布模型-最早出现的软件开发模型 1970年温斯顿?罗伊斯(Winston Royce)提出了著名的“瀑布模型”,直到80年代早期,它一直是唯一被广泛采用的软件开发模型。 瀑布模型核心思想是按工序将问题化简,将功能的实现与设计分开,便于分工协作,即采用结构化的分析与设计方法将逻辑实现与物理实现分开。将软件生命周期划分为制定计划、需求分析、软件设计、程序编写、软件测试和运行维护等六个基本活动,并且规定了它们自上而下、相互衔接的固定次序,如同瀑布流水,逐级下落。从本质来讲,它是一个软件开发架构,开发过程是通过一系列阶段顺序展开的,从系统需求分析开始直到产品发布和维护,每个阶段都会产生循环反馈,因此,如果有信息未被覆盖或者发现了问题,那么最好“返回”上一个阶段并进行适当的修改,开发进程从一个阶段“流动”到下一个阶段,这也是瀑布开发名称的由来。 瀑布模型是最早出现的软件开发模型,在软件工程中占有重要的地位,它提供了软件开发的基本框架。其过程是从上一项活动接收该项活动的工作对象作为输入,利用这一输入实施该项活动应完成的内容给出该项活动的工作成果,并作为输出传给下一项活动。同时评审该项活动的实施,若确认,则继续下一项活动;否则返回前面,甚至更前面的活动。对于经常变化的项目而言,瀑布模型毫无价值。(采用瀑布模型的软件过程如图所示)
美国常青藤名校的由来
美国常青藤名校的由来 以哈佛、耶鲁为代表的“常青藤联盟”是美国大学中的佼佼者,在美国的3000多所大学中,“常青藤联盟”尽管只是其中的极少数,仍是许多美国学生梦想进入的高等学府。 常青藤盟校(lvy League)是由美国的8所大学和一所学院组成的一个大学联合会。它们是:马萨诸塞州的哈佛大学,康涅狄克州的耶鲁大学,纽约州的哥伦比亚大学,新泽西州的普林斯顿大学,罗德岛的布朗大学,纽约州的康奈尔大学,新罕布什尔州的达特茅斯学院和宾夕法尼亚州的宾夕法尼亚大学。这8所大学都是美国首屈一指的大学,历史悠久,治学严谨,许多著名的科学家、政界要人、商贾巨子都毕业于此。在美国,常青藤学院被作为顶尖名校的代名词。 常青藤盟校的说法来源于上世纪的50年代。上述学校早在19世纪末期就有社会及运动方面的竞赛,盟校的构想酝酿于1956年,各校订立运动竞赛规则时进而订立了常青藤盟校的规章,选出盟校校长、体育主任和一些行政主管,定期聚会讨论各校间共同的有关入学、财务、援助及行政方面的问题。早期的常青藤学院只有哈佛、耶鲁、哥伦比亚和普林斯顿4所大学。4的罗马数字为“IV”,加上一个词尾Y,就成了“IVY”,英文的意思就是常青藤,所以又称为常青藤盟校,后来这4所大学的联合会又扩展到8所,成为现在享有盛誉的常青藤盟校。 这些名校都有严格的入学标准,能够入校就读的学生,自然是品学兼优的好学生。学校很早就去各个高中挑选合适的人选,许多得到全国优秀学生奖并有各种特长的学生都是他们网罗的对象。不过学习成绩并不是学校录取的惟一因素,学生是否具有独立精神并且能否快速适应紧张而有压力的大一新生生活也是他们考虑的重要因素。学生的能力和特长是衡量学生综合素质的重要一关,高中老师的推荐信和评语对于学生的入学也起到重要的作用。学校财力雄厚,招生办公室可以完全根据考生本人的情况录取,而不必顾虑这个学生家庭支付学费的能力,许多家境贫困的优秀子弟因而受益。有钱人家的子女,即使家财万贯,也不能因此被录取。这也许就是常青藤学院历经数百年而保持“常青”的原因。 布朗大学(Brown University) 1754年由浸信会教友所创,现在是私立非教会大学,是全美第七个最古老大学。现有学生7000多人,其中研究生近1500人。 该校治学严谨、学风纯正,各科系的教学和科研素质都极好。学校有很多科研单位,如生物医学中心,计算机中心、地理科学中心、化学研究中心、材料研究实验室、Woods Hole 海洋地理研究所海洋生物实验室、Rhode 1s1and反应堆中心等等。设立研究生课程较多的系有应用数学系、生物和医学系、工程系等,其中数学系海外研究生占研究生名额一半以上。 布朗大学的古书及1800年之前的美国文物收藏十分有名。 哥伦比亚大学(Columbia University) 私立综合性大学,位于纽约市。该校前身是创于1754年的King’s College,独立战争期间一度关闭,1784年改名力哥伦比亚学院,1912年改用现名。
1第一章 8字模型与飞镖模型(1)
O D C B A 图12图E A B C D E F D C B A O O 图12图E A B C D E D C B A H G E F D C B A 第一章 8字模型与飞镖模型 模型1 角的“8”字模型 如图所示,AB 、CD 相交于点O , 连接AD 、BC 。 结论:∠A+∠D=∠B+∠C 。 模型分析 8字模型往往在几何综合 题目中推导角度时用到。 模型实例 观察下列图形,计算角度: (1)如图①,∠A+∠B+∠C+∠D+∠E= ; (2)如图②,∠A+∠B+∠C+∠D+∠E+∠F= 。 热搜精练 1.(1)如图①,求∠CAD+∠B+∠C+∠D+∠E= ; (2)如图②,求∠CAD+∠B+∠ACE+∠D+∠E= 。 2.如图,求∠A+∠B+∠C+∠D+∠E+∠F+∠G+∠H= 。
D C B A M D C B A O 135E F D C B A 105O O 120 D C B A 模型2 角的飞镖模型 如图所示,有结论: ∠D=∠A+∠B+∠C 。 模型分析 飞镖模型往往在几何综合 题目中推导角度时用到。 模型实例 如图,在四边形ABCD 中,AM 、CM 分别平分∠DAB 和∠DCB ,AM 与CM 交于M 。探究∠AMC 与∠B 、∠D 间的数量关系。 热搜精练 1.如图,求∠A+∠B+∠C+∠D+∠E+∠F= ; 2.如图,求∠A+∠B+∠C+∠D = 。
O D C B A O D C B A O C B A 模型3 边的“8”字模型 如图所示,AC 、BD 相交于点O ,连接AD 、BC 。 结论:AC+BD>AD+BC 。 模型实例 如图,四边形ABCD 的对角线AC 、BD 相交于点O 。 求证:(1)AB+BC+CD+AD>AC+BD ; (2)AB+BC+CD+AD<2AC+2BD. 模型4 边的飞镖模型 如图所示有结论: AB+AC>BD+CD 。
新整理描写常青藤优美句段 写常青藤作文散文句子
描写常青藤优美句段写常青藤作文散文句子 描写常青藤优美句段写常青藤作文散文句子第1段: 1.睁开朦胧的泪眼,我猛然发觉那株濒临枯萎的常春藤已然绿意青葱,虽然仍旧瘦小,却顽强挣扎,嫩绿的枝条攀附着窗格向着阳光奋力伸展。 2.常春藤是一种常见的植物,我家也种了两盆。可能它对于很多人来说都不足为奇,但是却给我留下了美好的印象。常春藤属于五加科常绿藤本灌木,翠绿的叶子就像火红的枫叶一样,是可爱的小金鱼的尾巴。常春藤的叶子的长约5厘米,小的则约有2厘米,但都是小巧玲珑的,十分可爱。叶子外圈是白色的,中间是翠绿的,好像有人在叶子上涂了一层白色的颜料。从叶子反面看,可以清清楚楚地看见那凸出来的,一根根淡绿色的茎。 3.渴望到森林里探险,清晨,薄薄的轻雾笼罩在树林里,抬头一看,依然是参天古木,绕着树干一直落到地上的常春藤,高高低低的灌木丛在小径旁张牙舞爪。 4.我们就像马蹄莲,永不分开,如青春的常春藤,紧紧缠绕。 5.我喜欢那里的情调,常春藤爬满了整个屋顶,门把手是旧的,但带着旧上海的味道,槐树花和梧桐树那样美到凋谢,这是我的上海,这是爱情的上海。 6.当我离别的时候,却没有你的身影;想轻轻地说声再见,已是人去楼空。顿时,失落和惆怅涌上心头,泪水也不觉悄悄滑落我伫立很久很久,凝望每一条小路,细数每一串脚印,寻找你
的微笑,倾听你的歌声――一阵风吹过,身旁的小树发出窸窸窣窣的声音,像在倾诉,似在安慰。小树长高了,还有它旁边的那棵常春藤,叶子依然翠绿翠绿,一如昨天。我心头不觉一动,哦,这棵常春藤陪伴我几个春秋,今天才惊讶于它的可爱,它的难舍,好似那便是我的生命。我蹲下身去。轻轻地挖起它的一个小芽,带着它回到了故乡,种在了我的窗前。 7.常春藤属于五加科常绿藤本灌木,翠绿的叶子就像火红的枫叶一样,是可爱的小金鱼的尾巴。常春藤的叶子的长约5厘米,小的则约有2厘米,但都是小巧玲珑的,十分可爱。叶子外圈是白色的,中间是翠绿的,好像有人在叶子上涂了一层白色的颜料。从叶子反面看,可以清清楚楚地看见那凸出来的,一根根淡绿色的茎。 8.常春藤是多么朴素,多么不引人注目,但是它的品质是多么的高尚,不畏寒冷。春天,它萌发出嫩绿的新叶;夏天,它郁郁葱葱;秋天,它在瑟瑟的秋风中跳起了欢快的舞蹈;冬天,它毫不畏惧呼呼作响的北风,和雪松做伴常春藤,我心中的绿色精灵。 9.可是对我而言,回头看到的只是雾茫茫的一片,就宛如窗前那株瘦弱的即将枯死的常春藤,毫无生机,早已失去希望。之所以叫常春藤,可能是因为它一年四季都像春天一样碧绿,充满了活力吧。也许,正是因为如此,我才喜欢上了这常春藤。而且,常春藤还有许多作用呢!知道吗?一盆常春藤能消灭8至10平
软件开发工程师招聘试题
附录一 附录一【软件开发工程师招聘试题一】 考试时间:60分钟:______成绩:______ 一、单选题(共9题,每题2分) 1.对象b 最早在以下哪个选项前被垃圾回收?() public class Test5 { static String f(){ String a="hello"; String b="bye"; String c=b+"!"; //lineA String d=b; b=a; //lineB d=a; //lineC return c; //lineD } public static void main(String[] args) { String msg=f(); System.out.println(msg); } } A.lineA B.lineB C.lineC D.lineD 2.2.运行下列代码,结果如何?() class Example { int milesPerGallon; int index; Example(){} Example(int mpg){ milesPerGallon=mpg;
index=0; } public static void main(String[] args) { int index; Example e = new Example(25); if(args.length>0){ if(args[index].equals("Hiway")){ https://www.360docs.net/doc/f63134412.html,esPerGallon=2; } System.out.println("mpg:"+https://www.360docs.net/doc/f63134412.html,esPerGallon); } } } 这段代码通过编译,并且如果命令行输入”Hiway”则显示”mpg:50”,如果输入不是”Hiway”则显示”mpg:25”; 这段代码通过编译,并且如果命令行输入”Hiway”则显示”mpg:50”,如果输入不是”Hiway”则抛出ArrayIndexOutputBoundsException异常。 这段代码不能通过编译,因为自动变量index没有被初始化。 这段代码不能通过编译,因为milesPerGallon没有被初始化。 见例子Example.java 3.3.当编译如下代码时,会显示什么?() int i=1; switch(i){ case 0: System.out.println("zero"); case 1: System.out.println("one"); case 2: System.out.println("two"); default: System.out.println("default"); } One B. one,default C. one,two,default D.default 见例子:Test3.java 4.4.当编译运行如下代码时会发生什么现象?() public class MyClass { public static void main(String arguments[] ) { amethod(arguments); } public void amethod(String []arguments){
软件工程复习题及答案
2006-2007-2软件工程复习 一、单项选择题(20选10) 1. 结构化分析的主要描述手段有( B )。 A. 系统流程图和模块图 B. DFD图、数据词典、加工说明 C. 软件结构图、加工说明 D. 功能结构图、加工说明 2. 用于表示模块间的调用关系的图叫( D )。 A.PAD B.SC C.N-S D.HIPO 3. 在( B )模型中是采用用例驱动和架构优先的策略,使用迭代增量建造方法,软件“逐渐”被开发出来的。 A.快速原型 B. 统一过程 C.瀑布模型 D. 螺旋模型 4. 常用的软件开发方法有面向对象方法、面向( A )方法和面向数据方法。 A. 过程 B. 内容 C. 用户 D. 流程 5 从工程管理的角度来看,软件设计分两步完成( D )。 A. ①系统分析②模块设计 B. ①详细设计②概要设计 C. ①模块设计②详细设计 D. ①概要设计②详细设计 6. 程序的三种基本结构是( B )。 A. 过程、子程序、分程序 B.顺序、条件、循环 C.递归、堆栈、队列 D.调用、返回、转移 7. 程序的三种基本结构是( B )。 A. 过程、子程序、分程序 B.顺序、条件、循环 C.递归、堆栈、队列 D.调用、返回、转移 8. SD方法衡量模块结构质量的目标是( C )。 A. 模块间联系紧密,模块内联系紧密 B. 模块间联系紧密,模块内联系松散 C. 模块间联系松散,模块内联系紧密 D. 模块间联系松散,模块内联系松散 9.为提高软件测试的效率,应该( C )。 A.随机地选取测试数据 B.取一切可能的输入数据作为测试数据 C.在完成编码后制定软件测试计划 D.选择发现错误可能性大的数据作为测试数据 10.( D )测试用例发现错误的能力较大。 A.路径覆盖 B.条件覆盖 C.判断覆盖 D.条件组合覆盖 11.软件需求分析应确定的是用户对软件的( A )。 A. 功能需求和非功能需求 B. 性能需求 C. 非功能需求 D. 功能需求 12.下列各种图可用于动态建模的有( C )。 A.用例图 B. 类图 C. 序列图 D. 包图 13.软件过程模型有瀑布模型、( B )、增量模型等。 A. 概念模型 B. 原型模型 C. 逻辑模型 D. 物理模型 14.面向对象的分析方法主要是建立三类模型,即( D )。 A. 系统模型、ER模型、应用模型 B. 对象模型、动态模型、应用模型 C. E-R模型、对象模型、功能模型 D. 对象模型、动态模型、功能模型 15.测试的分析方法是通过分析程序( B )来设计测试用例的方法。 A.应用范围 B.内部逻辑 C.功能 D.输入数据 16. 软件工程是研究软件( B )的一门工程学科。 A. 数学 B. 开发与管理 C. 运筹学 D. 工具 17. 需求分析可以使用许多工具,但( C )是不适合使用的。 A.数据流图 B.判定表 C.PAD图 D.数据字典 18.划分模块时,一个模块内聚性最好的是( A )。 A. 功能内聚 B. 过程内聚 C. 信息内聚 D. 逻辑内聚 19.软件可移植性是用来衡量软件的( D )的重要尺度之一。 A.效率 B. 质量 C. 人机关系 D. 通用性 20.软件配置管理是在软件的整个生存周期内管理( D )的一组活动。 A.程序 B.文档 C.变更 D.数据 二、判定题(20选10) 1统一过程是一种以用户需求为动力,以对象作为驱动的模型,适合于面向对象的开发方法。(×) 2当模块中所有成分结合起来完成一项任务,该模块的内聚是偶然内聚。(×) 3SD方法衡量模块结构质量的目标是模块间联系松散,模块内联系紧密(√) 4当模块中所有成分结合起来完成一项任务,该模块的内聚是功能内聚。(√) 5在进行需求分析时,就应该同时考虑软件的可维护性问题。(√) 6需求分析可以使用许多工具,但数据流图是不适合使用的。(×)
关于美国常青藤
一、常青藤大学 目录 联盟概述 联盟成员 名称来历 常春藤联盟(The Ivy League)是指美国东北部八所院校组成的体育赛事联盟。这八所院校包括:布朗大学、哥伦比亚大学、康奈尔大学、达特茅斯学院、哈佛大学、宾夕法尼亚大学、普林斯顿大学及耶鲁大学。美国著名的体育联盟还有太平洋十二校联盟(Pacific 12 Conference)和大十联盟(Big Ten Conference)。常春藤联盟的体育水平在美国大学联合会中居中等偏下水平,远不如太平洋十校联盟和大十联盟。 联盟概述 常春藤盟校(Ivy League)指的是由美国东北部地区的八所大学组成的体育赛事联盟(参见NCAA词条)。它们全部是美国一流名校、也是美国产生最多罗德奖学金得主的大学联盟。此外,建校时间长,八所学校中的七所是在英国殖民时期建立的。 美国八所常春藤盟校都是私立大学,和公立大学一样,它们同时接受联邦政府资助和私人捐赠,用于学术研究。由于美国公立大学享有联邦政府的巨额拨款,私立大学的财政支出和研究经费要低于公立大学。 常青藤盟校的说法来源于上世纪的50年代。上述学校早在19世纪末期就有社会及运动方面的竞赛,盟校的构想酝酿于1956年,各校订立运动竞赛规则时进而订立了常青藤盟校的规章,选出盟校校长、体育主任和一些行政主管,定期聚会讨论各校间共同的有关入学、财务、援助及行政方面的问题。早期的常青藤学院只有哈佛、耶鲁、哥伦比亚和普林斯顿4所大学。4的罗马数字为"IV",加上一个词尾Y,就成了"IVY",英文的意思就是常青藤,所以又称为常青藤盟校,后来这4所大学的联合会又扩展到8所,成为如今享有盛誉的常青藤盟校。 这些名校都有严格的入学标准,能够入校就读的学生,必须是品学兼优的好学生。学校很早就去各个高中挑选合适的人选,许多得到全国优秀学生奖并有各种特长的学生都是他们网罗的对象。不过学习成绩并不是学校录取的惟一因素,学生是否具有独立精神并且能否快速适应紧张而有压力的大一新生生活也是他们考虑的重要因素。学生的能力和特长是衡量学生综合素质的重要一关,高中老师的推荐信和评语对于学生的入学也起到重要的作用。学校财力雄厚,招生办公室可以完全根据考生本人的情况录取,而不必顾虑这个学生家庭支付学费的能力,许多家境贫困的优秀子弟因而受益。有钱人家的子女,即使家财万贯,也不能因
常见的软件开发模型
常见的软件开发模型 软件开发模型是软件开发全部过程、活动和任务的结构框架。 1.软件开发模型是对软件过程的建模,即用一定的流程将各个环节连接起来,并可用规范的方式操作全过程,好比工厂的流水线。 2.软件开发模型能清晰、直观地表达软件开发全部过程,明确规定要完成的主要活动和任务,它用来作为软件项目工作的基础。 3.软件开发模型应该是稳定和普遍适用的 软件开发模型的选择应根据: 1.项目和应用的特点 2.采用的方法和工具 3.需要控制和交付的特点 软件工程之软件开发模型类型 1.边做边改模型 2.瀑布模型 3.快速原型模型 4.增量模型 5.螺旋模型 6.喷泉模型 边做边改模型(Build-and-Fix Model) 国内许多软件公司都是使用"边做边改"模型来开发的。在这种模型中,既没有规格说明,也没有经过设计,软件随着客户的需要一次又一次地不断被修改. 在这个模型中,开发人员拿到项目立即根据需求编写程序,调试通过后生成软件的第一个版本。在提供给用户使用后,如果程序出现错误,或者用户提出新的要求,开发人员重新修改代码,直到用户满意为止。 这是一种类似作坊的开发方式,对编写几百行的小程序来说还不错,但这种方法对任何规模的开发来说都是不能令人满意的,其主要问题在于:(1)缺少规划和设计环节,软件的结构随着不断的修改越来越糟,导致无法继续修改; (2)忽略需求环节,给软件开发带来很大的风险; (3)没有考虑测试和程序的可维护性,也没有任何文档,软件的维护十分困难。
瀑布模型(Waterfall Model) 1970年Winston Royce提出了著名的"瀑布模型",直到80年代早期,它一直是唯一被广泛采用的软件开发模型。瀑布模型将软件生命周期划分为制定计划、需求分析、软件设计、程序编写、软件测试和运行维护等六个基本活动,并且规定了它们自上而下、相互衔接的固定次序,如同瀑布流水,逐级下落。 在瀑布模型中,软件开发的各项活动严格按照线性方式进行,当前活动接受上一项活动的工作结果,实施完成所需的工作内容。当前活动的工作结果需要进行验证,如果验证通过,则该结果作为下一项活动的输入,继续进行下一项活动,否则返回修改。 瀑布模型强调文档的作用,并要求每个阶段都要仔细验证。但是,这种模型的线性过程太理想化,已不再适合现代的软件开发模式,几乎被业界抛弃,其主要问题在于: (1)各个阶段的划分完全固定,阶段之间产生大量的文档,极大地增加了工作量; (2)由于开发模型是线性的,用户只有等到整个过程的末期才能见到开发成果,从而增加了开发的风险; (3)早期的错误可能要等到开发后期的测试阶段才能发现,进而带来严重的后果。 我们应该认识到,"线性"是人们最容易掌握并能熟练应用的思想方法。当人们碰到一个复杂的"非线性"问题时,总是千方百计地将其分解或转化为一系列简单的线性问题,然后逐个解决。一个软件系统的整体可能是复杂的,而单个子程序总是简单的,可以用线性的方式来实现,否则干活就太累了。线性是一种简洁,简洁就是美。当我们领会了线性的精神,就不要再呆板地套用线性模型的外表,而应该用活它。例如增量模型实质就是分段的线性模型,螺旋模型则是接连的弯曲了的线性模型,在其它模型中也能够找到线性模型的影子. 快速原型模型(Rapid Prototype Model) 快速原型模型的第一步是建造一个快速原型,实现客户或未来的用户与系统的交互,用户或客户对原型进行评价,进一步细化待开发软件的需求。通过逐步调整原型使其满足客户的要求,开发人员可以确定客户的真正需求是什么;第二步则在第一步的基础上开发客户满意的软件产品。 显然,快速原型方法可以克服瀑布模型的缺点,减少由于软件需求不明确带来的开发风险,具有显著的效果。 快速原型的关键在于尽可能快速地建造出软件原型,一旦确定了客户的真正需求,所建造的原型将被丢弃。因此,原型系统的内部结构并不重要,重要的是必须迅速建立原型,随之迅速修改原型,以反映客户的需求。 增量模型(Incremental Model) 又称演化模型。与建造大厦相同,软件也是一步一步建造起来的。在增量模型中,软件被作为一系列的增量构件来设计、实现、集成和测试,每一个构件是由多种相互作用的模块所形成的提供特定功能的代码片段构成. 增量模型在各
什么是美国常青藤大学
https://www.360docs.net/doc/f63134412.html, 有意向申请美国大学的学生,大部分听过一个名字,常青藤大学联盟。那么美国常青藤大学盟校到底是怎么一回事,又是由哪些大大学组成的呢?下面为大家介绍一下美国常青藤大学联盟。 立思辰留学360介绍,常青藤盟校(lvy League)是由美国的七所大学和一所学院组成的一个大学联合会。它们是:马萨诸塞州的哈佛大学,康涅狄克州的耶鲁大学,纽约州的哥伦比亚大学,新泽西州的普林斯顿大学,罗德岛的布朗大学,纽约州的康奈尔大学,新罕布什尔州的达特茅斯学院和宾夕法尼亚州的宾夕法尼亚大学。这8所大学都是美国首屈一指的大学,历史悠久,治学严谨,许多著名的科学家、政界要人、商贾巨子都毕业于此。在美国,常青藤学院被作为顶尖名校的代名词。 常青藤由来 立思辰留学介绍,常青藤盟校的说法来源于上世纪的50年代。上述学校早在19世纪末期就有社会及运动方面的竞赛,盟校的构想酝酿于1956年,各校订立运动竞赛规则时进而订立了常青藤盟校的规章,选出盟校校长、体育主任和一些行政主管,定期聚会讨论各校间共同的有关入学、财务、援助及行政方面的问题。早期的常青藤学院只有哈佛、耶鲁、哥伦比亚和普林斯顿4所大学。4的罗马数字为“IV”,加上一个词尾Y,就成了“IVY”,英文的意思就是常青藤,所以又称为常青藤盟校,后来这4所大学的联合会又扩展到8所,成为现在享有盛誉的常青藤盟校。 这些名校都有严格的入学标准,能够入校就读的学生,自然是品学兼优的好学生。学校很早就去各个高中挑选合适的人选,许多得到全国优秀学生奖并有各种特长的学生都是他们网罗的对象。不过学习成绩并不是学校录取的惟一因素,学生是否具有独立精神并且能否快速适应紧张而有压力的大一新生生活也是他们考虑的重要因素。学生的能力和特长是衡量学生综合素质的重要一关,高中老师的推荐信和评语对于学生的入学也起到重要的作用。学校财力雄厚,招生办公室可以完全根据考生本人的情况录取,而不必顾虑这个学生家庭支付学费的能力,许多家境贫困的优秀子弟因而受益。有钱人家的子女,即使家财万贯,也不能因此被录取。这也许就是常青藤学院历经数百年而保持“常青”的原因。
软件开发工程师招聘试题
专业技术资料 附录一 附录一【软件开发工程师招聘试题一】 考试时间:60分钟姓名:______成绩:______ 一、单选题(共9题,每题2分) 1.对象b 最早在以下哪个选项前被垃圾回收?() public class Test5 { static String f(){ String a="hello"; String b="bye"; String c=b+"!"; //lineA String d=b; b=a; //lineB d=a; //lineC return c; //lineD } public static void main(String[] args) { String msg=f(); System.out.println(msg); } } A.lineA B.lineB C.lineC D.lineD 2.2.运行下列代码,结果如何?() class Example { int milesPerGallon; int index; Example(){} Example(int mpg){ milesPerGallon=mpg;
index=0; } public static void main(String[] args) { int index; Example e = new Example(25); if(args.length>0){ if(args[index].equals("Hiway")){ https://www.360docs.net/doc/f63134412.html,esPerGallon=2; } System.out.println("mpg:"+https://www.360docs.net/doc/f63134412.html,esPerGallon); } } } 这段代码通过编译,并且如果命令行输入”Hiway”则显示”mpg:50” ,如果输入不是”Hiway”则显示”mpg:25”; 这段代码通过编译,并且如果命令行输入”Hiway”则显示”mpg:50” ,如果输入不是”Hiway”则抛出ArrayIndexOutputBoundsException异常。 这段代码不能通过编译,因为自动变量index没有被初始化。 这段代码不能通过编译,因为milesPerGallon没有被初始化。 见例子Example.java 3.3.当编译如下代码时,会显示什么?() int i=1; switch(i){ case 0: System.out.println("zero"); case 1: System.out.println("one"); case 2: System.out.println("two"); default: System.out.println("default"); } One B. one,default C. one,two,default D.default 见例子:Test3.java 4.4.当编译运行如下代码时会发生什么现象?() public class MyClass { public static void main(String arguments[] ) { amethod(arguments); } public void amethod(String []arguments){
常用软件开发模型比较分析
常用软件开发模型比较分析 2007-09-26 20:21 正如任何事物一样,软件也有其孕育、诞生、成长、成熟和衰亡的生存过程,一般称其为“软件生命周期”。软件生命周期一般分为6个阶段,即制定计划、需求分析、设计、编码、测试、运行和维护。软件开发的各个阶段之间的关系不可能是顺序且线性的,而应该是带有反馈的迭代过程。在软件工程中,这个复杂的过程用软件开发模型来描述和表示。 软件开发模型是跨越整个软件生存周期的系统开发、运行和维护所实施的全部工作和任务的结构框架,它给出了软件开发活动各阶段之间的关系。目前,常见的软件开发模型大致可分为如下3种类型。 ① 以软件需求完全确定为前提的瀑布模型(Waterfall Model)。 ② 在软件开发初始阶段只能提供基本需求时采用的渐进式开发模型,如螺旋模型(Spiral Model)。 ③ 以形式化开发方法为基础的变换模型(T ransformational Model)。 本节将简单地比较并分析瀑布模型、螺旋模型和变换模型等软件开发模型。 1.2.1 瀑布模型瀑布模型即生存周期模型,其核心思想是按工序将问题化简,将功能的实现与设计分开,便于分工协作,即采用结构化的分析与设计方法将逻辑实现与物理实现分开。瀑布模型将软件生命周期划分为软件计划、需求分析和定义、软件设计、软件实现、软件测试、软件运行和维护这6个阶段,规定了它们自上而下、相互衔接的固定次序,如同瀑布流水逐级下落。采用瀑布模型的软件过程如图1-3所示。
图1-3 采用瀑布模型的软件过程 瀑布模型是最早出现的软件开发模型,在软件工程中占有重要的地位,它提供了软件开发的基本框架。瀑布模型的本质是一次通过,即每个活动只执行一次,最后得到软件产品,也称为“线性顺序模型”或者“传统生命周期”。其过程是从上一项活动接收该项活动的工作对象作为输入,利用这一输入实施该项活动应完成的内容给出该项活动的工作成果,并作为输出传给下一项活动。同时评审该项活动的实施,若确认,则继续下一项活动;否则返回前面,甚至更前面的活动。瀑布模型有利于大型软件开发过程中人员的组织及管理,有利于软件开发方法和工具的研究与使用,从而提高了大型软件项目开发的质量和效率。然而软件开发的实践表明,上述各项活动之间并非完全是自上而下且呈线性图式的,因此瀑布模型存在严重的缺陷。 ① 由于开发模型呈线性,所以当开发成果尚未经过测试时,用户无法看到软件的效果。这样软件与用户见面的时间间隔较长,也增加了一定的风险。 ② 在软件开发前期末发现的错误传到后面的开发活动中时,可能会扩散,进而可能会造成整个软件项目开发失败。 ③ 在软件需求分析阶段,完全确定用户的所有需求是比较困难的,甚至可以说是不太可能的。 1.2.2 螺旋模型螺旋模型将瀑布和演化模型(Evolution Model)结合起来,它不仅体现了两个模型的优点,而且还强调了其他模型均忽略了的风险分析。这
2019年美国常春藤八所名校排名
2019年美国常春藤八所名校排名享有盛名的常春藤盟校现在是什么情况呢?接下来就来为您介绍一下!以下常春藤盟校排名是根据2019年美国最佳大学进行的。接下来我们就来看看各个学校的状态以及真实生活。 完整的常春藤盟校名单包括耶鲁大学、哈佛大学、宾夕法尼亚大学、布朗大学、普林斯顿大学、哥伦比亚大学、达特茅斯学院和康奈尔大学。 同时我们也看看常春藤盟校是怎么样的?也许不是你所想的那样。 2019年Niche排名 3 录取率5% 美国高考分数范围1430-1600 财政援助:“学校选择美国最优秀的学生,想要他们来学校读书。如果你被录取,哈佛会确保你能读得起。如果你选择不去入学的话,那一定不是因为经济方面的原因。”---哈佛大三学生2019年Niche排名 4 录取率6% 美国高考分数范围1420-1600 学生宿舍:“不可思议!忘记那些其他学校的学生宿舍吧。在耶鲁,你可以住在一个豪华套房,它更像是一个公寓。一个公寓有许多人一起住,包括一个公共休息室、洗手间和多个卧室。我再不能要求任何更好的条件了。这个套房很大,很干净,还时常翻修。因为学校的宿舍深受大家喜爱,现在有90%的学生都住在学校!”---耶鲁大二学生
2019年Niche排名 5 录取率7% 美国高考分数范围1400-1590 综合体验:“跟任何其他学校一样,普林斯顿大学有利有弊。这个学校最大的好处也是我选择这个学校的主要原因之一就是它的财政援助体系,任何学生想要完成的计划,它都会提供相应的财政支持。”---普林斯顿大二学生 2019年Niche排名 6 录取率9% 美国高考分数范围1380-1570 自我关心:“如果你喜欢城市的话,宾夕法尼亚大学是个不错的选择。这里对于独立的人来说也是一个好地方,因为在这里你必须学会自己发展。要确保进行一些心理健康的训练,因为这里的人通常会过量工作。如果你努力工作并且玩得很嗨,二者都会使你精疲力尽,所以给自己留出点儿时间休息。”---宾夕法尼亚大一学生 2019年Niche排名7 录取率7% 美国高考分数范围1410-1590 综合体验:“学校的每个人都很关心学生,包括我们的身体状况和学业成绩。在这里,你可以遇到来自世界各地的多种多样的学生。他们在学校进行的安全防范教育让我感觉受到保护。宿舍生活非常精彩,你会感觉跟室友们就像家人一样。总之,能成为学校的一员我觉得很棒,也倍感荣幸!”---哥伦比亚大二学生2019年Niche排名9 录取率9% 美国高考分数范围1370-1570 学术点评:“新的课程培养学术探索能力,在过去的两年中我
(四)应用程序模型
XAF是重量型框架,确实够重量的,方方面面都做得规规矩矩。 如果看了前面三节,可能会认为,这N多的Attribute到底都是从哪里来的?到底有多少这样的Attribute?如果不够用了怎么办?等着官方开发吗? 好吧,我不是为了解决上面的这些问题的,从另一个角度来看一下我们的应用程序吧! 我们回过头来看看解决方案中的项目都是做些什么用途吧: XAF的默认项目结构中,XCRMDemo.Module中写了代码,就会同时生成了web和win项目。 也就是说,上图中的模块是从上到下的继承关系。 但实事上,做过开发的朋友都知道,web中显示的textbox和win中显示的textbox是完全不同的控件,一个是html支持的,一个是winform中的控件。 XAF只是把他们做成了同一个名称的控件,让类型与控件有了对应关系,但有些时候,Web 下面和Win下面可能并没有一个同样功能的控件,比如我使用了第三方的控件Excel编辑控件,但我只找到了Win版本,没有Web版本,这时,我们只能把控件扩展写到XCRMDemo.Win.Module中去。
再来看看应用程序模型,展开XCRMDemo.Module项目,双击Model.DesignedDiffs.xafml 文件,可以看到: 如果你是从上面章节中下载的源码,请编译一次ctrl+shift+B 可以看到,这里可以控制着应用程序中的方方面面功能。 其中,ActionDesign,是对按钮进行设置的,比如之前开发中使用过的New按钮,Save,SaveAndClose等等。 假如我们想要开发的系统仅有中文,也可以直接在这里修改进行汉化。 为了展示效果,我做个简单的修改设置: 如上图所示,我将Cancel的标题,即为在界面上显示的文字修改为“取消” 并用同样的方法将,Delete,New,Save等几个按钮都做修改。
软件开发模型的优缺点和适用范围
软件开发模型的优缺点和适用范围 软件开发模型大体上可以分为三种类型。第一种是以软件需求完全确定为前提的瀑布模型;第二种是在软件开发初始阶段只能提供基本需求时采用的渐进式开发模型,如原型模型、 螺旋模型等;第三种是以形式化开发方法为基础的的变换模型。时间中经常将几种模型组合使用, 以便充分利用各种模型的优点。 1. 瀑布模型 瀑布模型也称软件生存周期模型。它在软件工程中占有重要地位,它提供了软件开发的基本框架,这比依靠“个人技艺”开发软件好得多。它有利于大型软件开发过程中人员的组织、管理,有利于软件开发方法和工具的研究与使用,从而提高了大型软件项目开发的质量和效率。 瀑布模型的缺点:一是个阶段的划分完全固定,阶段之间产生大量的文档,极大地增加了工作量;二是由于开发模型是线性的用户只有等到整个过程的末期才能见到开发成果,从而卡增加了开发的风险;三是早期的错误可能要等到开发后期的测试阶段才能发现,进而带来严重后果。 2. 原型模型 原型模型的主要思想:先借用已有系统作为原型模型,通过“样品”不断改进, 使得最后的产品就是用户所需要的。原型模型通过向用户提供原型获取用户的反 馈,使开发出的软件能够真正反映用户的需求。 原型模型的特点:开发人员和用户在“原型”上达成一致。这样一来,可以减少设计中的错误和开发中的风险,也减少了对用户培训的时间,而提高了系统的实用、正确性以及用户的满意程度。缩短了开发周期,加快了工程进度。降低成本。 原型模型的缺点:当告诉用户,还必须重新生产该产品时,用户是很难接受的。 这往往给工程继续开展带来不利因素。不宜利用原型系统作为最终产品。 3. 螺旋模型 螺旋模型采用一种周期性的方法来进行系统开发。这会导致开发出众多的中间版 本。 螺旋模型的优点: 1)设计上的灵活性,可以在项目的各个阶段进行变更。 2)以小的分段来构建大型系统,使成本计算变得简单容易。 3)客户始终参与每个阶段的开发,保证了项目不偏离正确方向及项目的可控性。 4)随着项目推进,客户始终掌握项目的最新信息,从而他或她能够和管理层有效地交互。 5)客户认可这种公司内部的开发方式带来的良好的沟通和高质量的产品。
美国常青藤大学研究生申请条件都有哪些
我国很多学子都想前往美国的常青藤大学就读于研究生,所以美国常青藤大学研究生申请条件都有哪些? 美国常青藤大学研究生申请条件: 1、高中或本科平均成绩(GPA)高于3.8分,通常最高分是4分,平均分越高越好; 2、学术能力评估测试I(SAT I,阅读+数学)高于1400分,学术能力评估测试II(SAT II,阅读+数学+写作)高于2000分; 3、托福考试成绩100分以上,雅思考试成绩不低于7分; 4、美内国研究生入学考试(GRE)成绩1400分以上,经企管理研究生入学考试(GMAT)成绩700分以上。 大学先修课程(AP)考试成绩并非申请美国大学所必需,但由于大学先修课程考试对于高中生来说有一定的挑战性及难度,美国大学也比较欢迎申请者提交大学先修课程考试的成绩,作为入学参考标准。
有艺术、体育、数学、社区服务等特长者优先考容虑。获得国际竞赛、辩论和科学奖等奖项者优先考虑,有过巴拿马国际发明大赛的得主被破例录取的例子。中国中学生在奥林匹克数、理、化、生物比赛中获奖也有很大帮助。 常春藤八所院校包括:哈佛大学、宾夕法尼亚大学、耶鲁大学、普林斯顿大学、哥伦比亚大学、达特茅斯学院、布朗大学及康奈尔大学。 新常春藤包括:加州大学洛杉矶分校、北卡罗来纳大学、埃默里大学、圣母大学、华盛顿大学圣路易斯分校、波士顿学院、塔夫茨大学、伦斯勒理工学院、卡内基梅隆大学、范德比尔特大学、弗吉尼亚大学、密歇根大学、肯阳学院、罗彻斯特大学、莱斯大学。 纽约大学、戴维森学院、科尔盖特大学、科尔比学院、瑞德大学、鲍登学院、富兰克林欧林工程学院、斯基德莫尔学院、玛卡莱斯特学院、克莱蒙特·麦肯纳学院联盟。 小常春藤包括:威廉姆斯学院、艾姆赫斯特学院、卫斯理大学、斯沃斯莫尔学院、明德学院、鲍登学院、科尔比学院、贝茨学院、汉密尔顿学院、哈弗福德学院等。
