bioconductor系列教程之一分析基因芯片上
?bioconductor系列教程之一分析基因芯片上
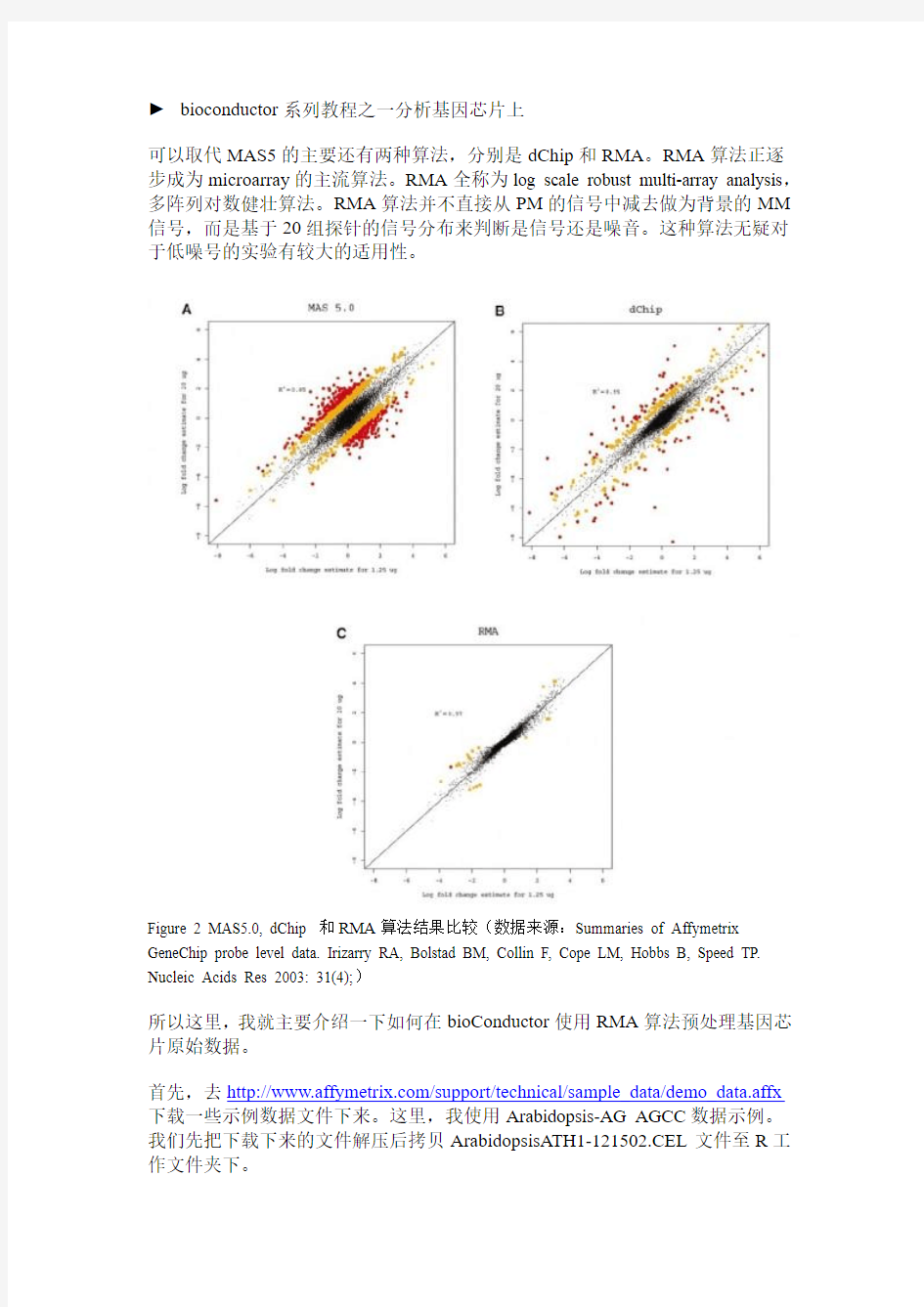
可以取代MAS5的主要还有两种算法,分别是dChip和RMA。RMA算法正逐步成为microarray的主流算法。RMA全称为log scale robust multi-array analysis,多阵列对数健壮算法。RMA算法并不直接从PM的信号中减去做为背景的MM 信号,而是基于20组探针的信号分布来判断是信号还是噪音。这种算法无疑对于低噪号的实验有较大的适用性。
Figure 2 MAS5.0, dChip 和RMA算法结果比较(数据来源:Summaries of Affymetrix GeneChip probe level data. Irizarry RA, Bolstad BM, Collin F, Cope LM, Hobbs B, Speed TP. Nucleic Acids Res 2003: 31(4);)
所以这里,我就主要介绍一下如何在bioConductor使用RMA算法预处理基因芯片原始数据。
首先,去https://www.360docs.net/doc/0a15995929.html,/support/technical/sample_data/demo_data.affx 下载一些示例数据文件下来。这里,我使用Arabidopsis-AG AGCC数据示例。我们先把下载下来的文件解压后拷贝ArabidopsisATH1-121502.CEL文件至R工作文件夹下。
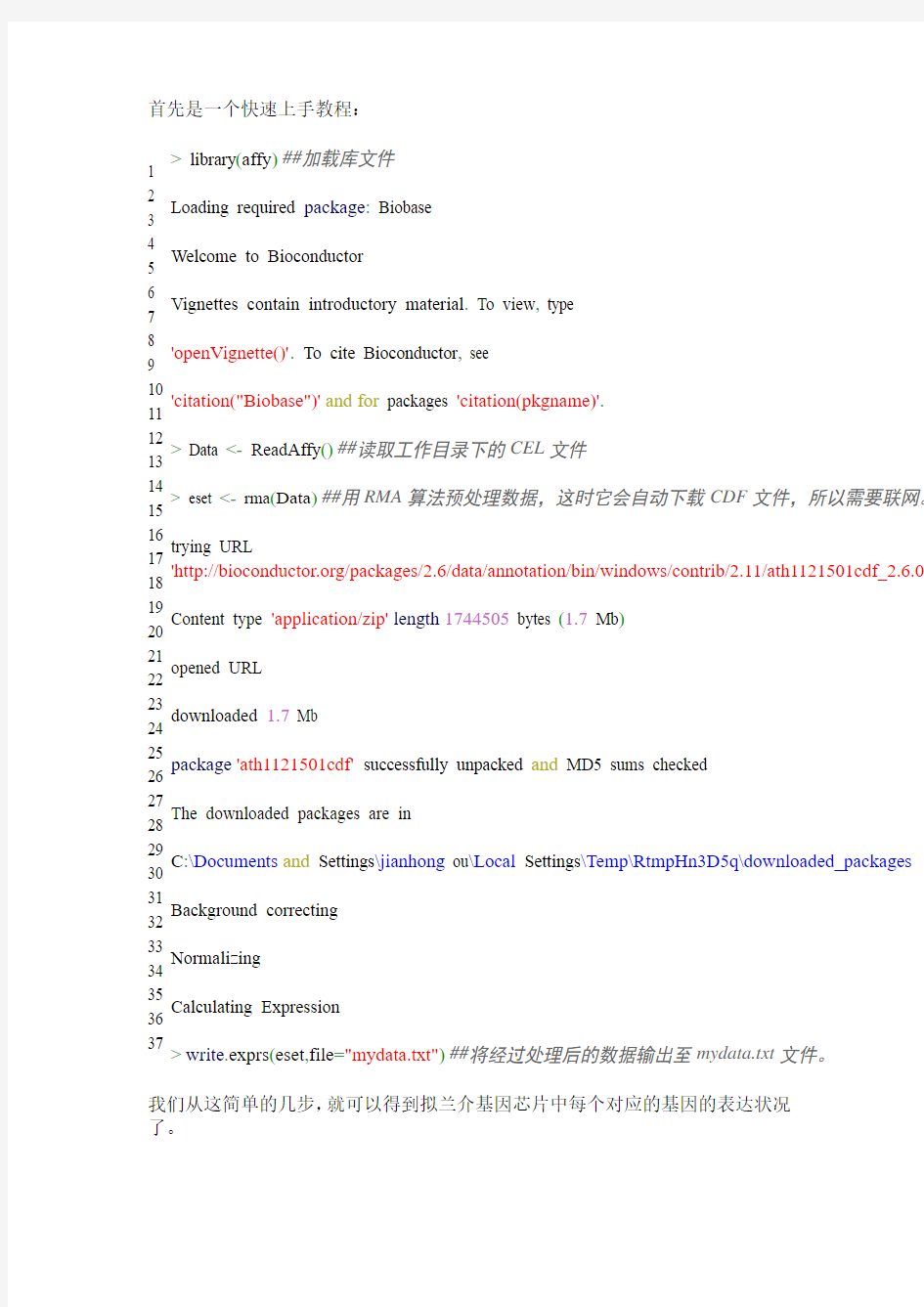
首先是一个快速上手教程:
1 2 3 4 5 6 7 8 9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37 > library(affy)##加载库文件
Loading required package: Biobase
Welcome to Bioconductor
Vignettes contain introductory material. To view, type
'openVignette()'. To cite Bioconductor, see
'citation("Biobase")'and for packages 'citation(pkgname)'.
> Data <- ReadAffy()##读取工作目录下的CEL文件
> eset <- rma(Data)##用RMA算法预处理数据,这时它会自动下载CDF文件,所以需要联网。trying URL
'https://www.360docs.net/doc/0a15995929.html,/packages/2.6/data/annotation/bin/windows/contrib/2.11/ath1121501cdf_2.6.0. Content type 'application/zip'length1744505 bytes (1.7 Mb)
opened URL
downloaded 1.7 Mb
package'ath1121501cdf' successfully unpacked and MD5 sums checked
The downloaded packages are in
C:\Documents and Settings\jianhong ou\Local Settings\Temp\RtmpHn3D5q\downloaded_packages Background correcting
Normalizing
Calculating Expression
>write.exprs(eset,file="mydata.txt")##将经过处理后的数据输出至mydata.txt文件。
我们从这简单的几步,就可以得到拟兰介基因芯片中每个对应的基因的表达状况了。
?bioconductor系列教程之一分析基因芯片中(质量控制)
上一节,我们了解了分析基因芯片的预处理的基本知识。其实那只是一个热身。这一节,我们来学习拿到基因芯片数据时更基本的操作:质量控制。只有通过质量检测合格的芯片数据才会真正地进入数据分析的步骤。本节将学习以下内容:
背景
MAS5 标准化
Affymetrix公司制定的内参
教程数据下载
质量控制总览图及报告
使用FitPLM生成权重,残差及NUSE图像
RNA降解曲线及MVA线图
PCA分析
总结
背景
通过上一节的介绍,我们了解到Affymetrix基因芯片中的探针都是由25个碱基组成的寡聚核苷酸序列。每个芯片上可能包含上百万的探针,它们被整齐有序的印刷在芯片上。而探针的排序以组为单位,随机排列。而每一组,都由20对探针组成。这一组探针被称为探针组(probeset)。每一对探针都由perfect match(PM)和mismatch(MM)组成,称为探针对(probe pair)(figure 1)。MM与PM维一的不同,就是正中央的那个碱基不同,其余的都一致。人们期待MM不会象PM 那样与RNA或者DNA有特异性配对,有的只是非特异性配对。而事实上,我们都知道,这是不可能的。在后面的教程中,会可能提及一些这方面的分析。
而每一个探针组都均匀包含了目标基因3’至5’不同区段特异序列。这种设计一方面可以通过均衡它们结果的方式来获取目标基因的表达强度(这一过程被称为总结步骤(summarization step)),另一方面,它也可以提供mRNA降解的程度信息。我们知道一般mRNA都是按5’端至3’端的顺序来降解的,而这些探针组应该能体现这一趋势。
上一节我们谈到过标准化的问题。这一节并不会深入探讨这个问题,但是我们会简单地应用上一节提到过了两个标准化方法MAS5和RMA方法。使用它们只是作为一种示例来表达如何通过试用不同的标准化方法来获得最佳的结果。
Affymetrix公司在指导手册上就已经提出了用于判断基因芯片质量的多种标准。这些标准大多都是依照该公司的MAS5算法而提出的,所以我们还是得重新提及一下MAS5算法。
提取差异表达的基因
从基因芯片当中提取生物学的信息需要合理的统计学方法。人们已经为优化传统统计学方法在基因芯片方面的应用做出了多年的努力。但是直到现在,最主要的努力依然还是依据实验设计的差别,用统计学方法提取出差异表达的基因,然后再转回使用实验的方法去验证这个结果。
在提取差异表达的基因时,人们总是会有这两种考虑,一是不可漏过一个,二是不能错杀过多(在英语里称为false discovery rate(FDR)错误发现率)。常见的手段是使用多种统计学方法来分析同样一个结果,尽可能多的得到差异表达的基因,而排除那些假的信号。然而学习和使用多种统计分析手段并不一定对于每一个生物学工作者都是非常容易的,这需要付出时间和努力。在这里,我们尽量多介绍几种常用的统计分析手段,并给出实践中人们常常使用的组合,来帮助你更好的分析自己的数据。
现在常用的分析手段主要有:significance analysis of microarrays(SAM),CyberT 和Rank products(RP)三种手段。其中CyberT是bioconductor当中最为常用的分析手段,因为它的算法完整地被limma库实现。但有研究指出,使用SAM和RP算法相结合可能是最佳的方案。其实任何一种算法都是有局限性的,我们需要从根本上对算法有所了解,然后才能有针对性地选择合适的算法。
SAM:Tusher VG, Tibshirani R, Chu G. Significance analysis of microarrays applied to the ionizing radiation response. Proc Natl Acad Sci USA 2001; 98:5116-21 CyberT: Baldi P, Long AD. A Bayesian framework for the analysis of microarray expression data: regularized t-test and statistical inferences of gene changes. Bioinformatics 2001; 17:509-19
RP: Breitling R, Armengaud P, Amtmann A, et al. Rank products: a simple, yet powerful, new method to detect differentially regulated genes in replicated microarray experiments. FEBS lett 2004;573:83-92
生物芯片的市场分析
生物芯片的市场分析 全球市场总额很小 企业收入增长缓慢 全球的市场有多大?国内的市场又有多大?前景如何?现在国内没有公开的文章回答这些问题。国内的市场小,人们对生物芯片的技术和应用还没有普遍的认识。介绍生物芯片技术的论文、报告和新闻唾手可得,前几年投资炒作的文章也能找到几篇大作,但关于生物芯片的市场,现在国内还看不到一篇专题文章,也没有一家芯片公司或咨询公司做过有意义的市场调查;曾有公司在网上做过消费者调查,响应者却寥寥无几。我从网上找到了3家国际知名市场研究公司的公开数据,翻译过来,列举如下:2003年7月24日,国际知名的市场研究和数据分析公司Research and Markets公司发布了定价998美元的159页的报告《美国生物芯片和设备的市场和业务》,这份报告认为,2002年的全球生物芯片市场规模是11亿美元,将以19.5%的年平均增长率增长,2007年将达到27亿美元。2003年底,雷曼兄弟(Lehman Brother)公司发布的分析报告指出,全球芯片市场约有8亿美元的规模。2004年3月30日,英国伦敦的大型国际咨询公司Frost & Sullivan公司出版了价值4,950美元的关于全球芯片市场的分析报告:《世界DNA芯片市场的战略分析》。报告认为,全球DNA生物芯片市场每年平均增长6.7%,2003年的市场总值是5.96亿美元,2010年将达到9.37亿美元。 比较这3家公司估计的2003年生物芯片市场的市场规模:Frost & Sullivan公司仅考虑了生物芯片市场中的DNA芯片市场,为6亿美元;雷曼兄弟估计为8亿美,Research and Markets公司估计为13亿美元,我们发现,这3家单位估计的全球生物芯片市场总额的数据相差不远,在8-13亿美元,他们估计的数据体现了这个产业的客观市场规模应该在这个范围内。台湾生物芯片协会估计的市场是2003年为2.2亿美元,其中医疗芯片销售额6,500万美元,研究芯片销售额1.55亿美元,数额偏低,估计没有包括生物芯片仪器市场。 全球生物芯片霸主是以医药个体化为目标的Affymetrix公司,今年继续在全球市场上领先,很多专家估计其市场份额占全球1/3至1/2。如果我们清楚了Affymetrix公司的市场情况,也就知道了全球一半的市场。根据Affymetrix公司《2003年年度报告》披露的信息,我们能看到这个霸主的一些市场业绩。假设市场份额正如专家们所估计的那样,Affymetrix公司占了全球1/2至1/3的市场,按Affymetrix公司的营业额估算,2003年全球市场也就6-9亿美元左右。如果最近5年的市场增长速度保持下去,今后5年的全球市场增长2倍,至2008年,全球市
基因芯片的数据分析
基因表达谱芯片的数据分析 基因芯片数据分析就是对从基因芯片高密度杂交点阵图中提取的杂交点荧光强度信号进行的定量分析,通过有效数据的筛选和相关基因表达谱的聚类,最终整合杂交点的生物学信息,发现基因的表达谱与功能可能存在的联系。然而每次实验都产生海量数据,如何解读芯片上成千上万个基因点的杂交信息,将无机的信息数据与有机的生命活动联系起来,阐释生命特征和规律以及基因的功能,是生物信息学研究的重要课题[1]。基因芯片的数据分析方法从机器学习的角度可分为监督分析和非监督分析,假如分类还没有形成,非监督分析和聚类方法是恰当的分析方法;假如分类已经存在,则监督分析和判别方法就比非监督分析和聚类方法更有效率。根据研究目的的不同[2,3],我们对基因芯片数据分析方法分类如下。(1)差异基因表达分析:基因芯片可用于监测基因在不同组织样品中的表达差异,例如在正常细胞和肿瘤细胞中;(2)聚类分析:分析基因或样本之间的相互关系,使用的统计方法主要是聚类分析;(3)判别分析:以某些在不同样品中表达差异显著的基因作为模版,通过判别分析就可建立有效的疾病诊断方法。 1 差异基因表达分析(difference expression, DE) 对于使用参照实验设计进行的重复实验,可以对2样本的基因表达数据进行差异基因表达分析,具体方法包括倍数分析、t检验、方差分析等。 1.1倍数变化(fold change, FC) 倍数分析是最早应用于基因芯片数据分析的方法[4],该方法是通过对基因芯片的ratio值从大到小排序,ratio 是cy3/cy5的比值,又称R/G值。一般0.5-2.0范围内的基因不存在显著表达差异,该范围之外则认为基因的表达出现显著改变。由于实验条件的不同,此阈值范围会根据可信区间应有所调整[5,6]。处理后得到的信息再根据不同要求以各种形式输出,如柱形图、饼形图、点图等。该方法的优点是需要的芯片少,节约研究成本;缺点是结论过于简单,很难发现更高层次功能的线索;除了有非常显著的倍数变化的基因外,其它变化小的基因的可靠性就值得怀疑了;这种方法对于预实验或实验初筛是可行的[7]。此外倍数取值是任意的,而且可能是不恰当的,例如,假如以2倍为标准筛选差异表达基因,有可能没有1条入选,结果敏感性为0,同样也可能出现很多差异表达基因,结果使人认为倍数筛选法是在盲目的推测[8,9]。 1.2 t检验(t-test) 差异基因表达分析的另一种方法是t检验[10],当t超过根据可信度选择的标准时,比较
IT前沿技术讲座总结
关于IT前沿技术讲座报告 ——罗瑞13级计算机联合 班 在开学后的第三个月,华南理工大学计算机科学与工程学院给学生安排了关于IT前沿技术的一系列讲座,听了这些由学院资历深厚的老师所授的讲座后,我的收获颇丰。几位主讲老师针对信息技术的不同领域给予了不同高度的讲解,并且和学院热心的同学们积极交流,传授知识,以及人生道路上的经验。 几位主讲老师主要选择了一下几个话题进行讲解: 1、科学与技术研究之科学; 2、智能计算机; 3、机器人发展机遇与挑战; 4、大数据时代的高性能计算。 自从来到大学并且学习了计算机科学与技术这个专业后,我慢慢发现,我的学习与生活与电脑的关系越来越密切,对信息技术的应用也越来越熟悉与广泛。印象最深的是每次的实验课,在机房完成当天的作业后,都会借助快速便捷的网络发送到自己的邮箱里,回到宿舍后再在自己的邮箱里下载到电脑上。这样,无需借助任何实体媒介,作业就以数据形式传送到我的电脑了,似乎有个人一直在操纵、管理着,可是实际上,这一切都是信息技术发展的必然结果,那个操纵者就是人们探索出的信息技术。
我是一个理工科的学生,但是经过主讲老师对科学进行阐述的讲座,我才真正明白了科学的含义,科学是一种不断升华的思维艺术。具体来讲,它是指由权威人、组织和机构经过实践、论证所得出的具有普遍性、必然性的数据,并通过一系列技术完善、确认、推荐、宣传、传授和捍卫的一种广泛领域的思维学术。科学是严谨的,是需要人们不断探索、证实,并且公诸于世,从而去推动社会发展。正如远古时代出现的具有里程碑意义的火一样,如果无人理睬,便会熄灭,社会也将会停止前进的步伐,但是一旦有人用心探索,就会照亮一片新的绚丽天地。 肖南峰老师给我们带来的第五代计算机—智能计算机,介绍了第五代计算机是把信息采集、存储、处理、通信同人工智能结合在一起的智能计算机系统。它能进行数值计算或处理一般的信息,主要能面向知识处理,具有形式化推理、联想知识.人-机之间可以直接通过自然语言(声音、文字)或图形图象交换信息。智能计算机是指能存储大量信息和知识,会推理,具有学习功能,能以自然语言、文字、声音、图形、图像和人交流信息和知识的非冯诺依曼结构的通用高速并行处理计算机。虽然目前智能计算机的技术还不够成熟,不能广泛使用。但是照其定义看来,如果将来有一天人们在智能计算机领域得以拥有成熟的技术和完善的设备并且广泛投入生产,大规模使用的话,拥有如此多极高能力的计算机似乎已经可以代替人类的存在,但是,我们需要明白,我们研究发明任何高科技的智能产品,其目的都不是想要
生物芯片的基本原理
第二章生物芯片的基本原理 § 2.1 生物芯片的基本概念 一般而言,我们所指的芯片是以硅晶体为材料制造的用来存储信息、进行科学计算等用途的半导体器件,如各种计算机芯片。硅芯片是通过电路高低电平来表示逻辑1或逻辑0,不同的0,1组合可以代表自然界的一切信息,从而方便存储。生物电子芯片与硅芯片有很大的相似之处。20世纪70年代,人们发现脱氧核糖核酸(DNA, Deoxyribonucleic acid)处于不同的状态可以代表信息的存在或没有信息。这一发现引起科学家们的极大兴趣,科学家们立即投身到生物电子元件这一研究领域[1]。 80年代初,国际上提出了“生物芯片”这一概念,形象地把微电子集成电路技术与生物活性分子功能结合,提出构建具有生物活性的能够获取存储信息并进行处理和传输的微生物构件(微功能单元),以达到仿生信息处理的目的。在此基础上诞生了“分子电子学”。 90年代以来,在美国硅谷又兴起了研究和开发“生物芯片”的热潮[1][2]。这一“生物芯片”的概念是指运用大规模集成电路的光刻技术以及生物分子的自组装技术,在一微小芯片上组装成千上万个不同生物分子(DNA,蛋白质,多肽,细胞等)微阵列,实现生物分子信息的快速、并行、大规模检测[1][3]。 芯片分析的实质是在面积不大的基片表面上有序地点阵排列了一系列固定于一定位置的可寻址的识别分子。结合或反应在相同条件下进行。反应结果用同位素、化学荧光法、化学发光法或酶标法显示,然后用精密的扫描仪或CCD摄像技术记录。通过计算机软件分析,综合成可读的IC总信息[3][4][5]。 芯片分析实际上也是传感器分析的组合。芯片点阵中的每一个单元微点都是一个传感器的探头[6]。所以传感器技术的精髓往往都被应用于芯片的发展。阵列检测可以大大提高检测效率,减少工作量,增加可比性。所以芯片技术也是传感器技术的发展。 生物芯片的概念来自计算机芯片,但是到90年代初以后,在人类基因组计划的推动下,才得以迅速发展起来。
基因芯片数据功能分析
生物信息学在基因芯片数据功能分析中的应用 2009-4-29 随着人类基因组计划(Human Genome Project)即全部核苷酸测序的即将完成,人类基因组研究的重心逐渐进入后基因组时代(Postgenome Era),向基因的功能及基因的多样性倾斜。通过对个体在不同生长发育阶段或不同生理状态下大量基因表达的平行分析,研究相应基因在生物体内的功能,阐明不同层次多基因协同作用的机理,进而在人类重大疾病如癌症、心血管疾病的发病机理、诊断治疗、药物开发等方面的研究发挥巨大的作用。它将大大推动人类结构基因组及功能基因组的各项基因组研究计划。生物信息学在基因组学中发挥着重大的作用, 而另一项崭新的技术——基因芯片已经成为大规模探索和提取生物分子信息的强有力手段,将在后基因组研究中发挥突出的作用。基因芯片与生物信息学是相辅相成的,基因芯片技术本身是为了解决如何快速获得庞大遗传信息而发展起来的,可以为生物信息学研究提供必需的数据库,同时基因芯片的数据分析也极大地依赖于生物信息学,因此两者的结合给分子生物学研究提供了一条快捷通道。 本文介绍了几种常用的基因功能分析方法和工具: 一、GO基因本体论分类法 最先出现的芯片数据基因功能分析法是GO分类法。Gene Ontology(GO,即基因本体论)数据库是一个较大的公开的生物分类学网络资源的一部分,它包含38675 个Entrez Gene注释基因中的17348个,并把它们的功能分为三类:分子功能,生物学过程和细胞组分。在每一个分类中,都提供一个描述功能信息的分级结构。这样,GO中每一个分类术语都以一种被称为定向非循环图表(DAGs)的结构组织起来。研究者可以通过GO分类号和各种GO数据库相关分析工具将分类与具体基因联系起来,从而对这个基因的功能进行描述。在芯片的数据分析中,研究者可以找出哪些变化基因属于一个共同的GO功能分支,并用统计学方法检定结果是否具有统计学意义,从而得出变化基因主要参与了哪些生物功能。 EASE(Expressing Analysis Systematic Explorer)是比较早的用于芯片功能分析的网络平台。由美国国立卫生研究院(NIH)的研究人员开发。研究者可以用多种不同的格式将芯片中得到的基因导入EASE 进行分析,EASE会找出这一系列的基因都存在于哪些GO分类中。其最主要特点是提供了一些统计学选项以判断得到的GO分类是否符合统计学标准。EASE 能进行的统计学检验主要包括Fisher 精确概率检验,或是对Fisher精确概率检验进行了修饰的EASE 得分(EASE score)。 由于进行统计学检验的GO分类的数量很多,所以EASE采取了一系列方法对“多重检验”的结果进行校正。这些方法包括弗朗尼校正法(Bonferroni),本杰明假阳性率法(Benjamini falsediscovery rate)和靴带法(bootstraping)。同年出现的基于GO分类的芯片基因功能分析平台还有底特律韦恩大学开发的Onto-Express。2002年,挪威大学和乌普萨拉大学联合推出的Rosetta 系统将GO分类与基因表达数据相联系,引入了“最小决定法则”(minimal decision rules)的概念。它的基本思想是在对多张芯片结果进行聚类分析之后,与表达模式
学术讲座心得体会集萃
学术讲座心得体会集萃 利用小学期时间,学校和学院给我们安排了一系列讲座,在我 看来,旨在丰富小学期生活,积累专业知识,拓宽视野。这些讲座与 我们专业知识紧密相关,但是却不单一,涉及不同课题观摩聆听名师 讲座,名师神采飞扬,听者亦有心得。一千个读者的心中有一千个哈姆 雷特。而面对着鲜活的教学对象,智慧的教师必然没有相同的课堂。 第一次讲座专由我校信管业的王璇老师主持,内容是信息与科技。谈到了信息技术发展的必然,从古至今,信息的发展经历了结绳记事、账簿、计算机,最后到因特网。所谓的信息技术,是能够延长或扩展 人的信息水平的各种技术的总称,是对声音、图像、文字等信息实行 收集、加工、存储、传递和利用的技术。战略资源的定义则是,任何 一种社会的经济活动都是以若干种资源为依托的,在这些资源中,最 基本最重要的资源就被称为战略资源。仅仅这些定义就能够引起我们 的思考,当今社会什么最重要,精准快速的信息以及先进高等的科技。 第二次讲座的老师——沈凤武,据他自己说是第一次做讲座,所 讲内容是管理缺失下的垃圾危机问题研究,涉及垃圾的危害,主要包 括生活垃圾对人类的影响以及垃圾堆土地资源的耗费,同时提出了对 生活垃圾的处理方法,即焚烧发电、填埋处理以及堆肥。基于国土资 源的垃圾危机治理,我们每个公民有义务为此做贡献。 第三次和第四次讲座的主题不离经济贸易,有谈到讲师的研究方向,也有宏观分析当前形势。当今社会,对外贸易在国家或者地区的 经济发展中扮演着越来越重要的角色。一国要获得经济快速的经济发展,必须学会利用国际国内两个市场。通过对外贸易,实行物产的互 通有无,从而实现资源的优化配置。对贸易行为的分析通常分为总量 分析和结构分析,总量分析是从量的角度分析问题,而结构分析更注 重从质的角度考察贸易行为。而对外贸易结构是一国或地区经济技术 发展水平、产业结构状况、商品国际竞争水平、在国际分工和国际贸 易中的地位等的综合反映,而商品结构和区域结构是对外贸易结构的
基因芯片数据处理流程与分析介绍
基因芯片数据处理流程与分析介绍 关键词:基因芯片数据处理 当人类基因体定序计划的重要里程碑完成之后,生命科学正式迈入了一个后基因体时代,基因芯片(microarray) 的出现让研究人员得以宏观的视野来探讨分子机转。不过分析是相当复杂的学问,正因为基因芯片成千上万的信息使得分析数据量庞大,更需要应用到生物统计与生物信息相关软件的协助。要取得一完整的数据结果,除了前端的实验设计与操作的无暇外,如何以精确的分析取得可信数据,运筹帷幄于方寸之间,更是画龙点睛的关键。 基因芯片的应用 基因芯片可以同时针对生物体内数以千计的基因进行表现量分析,对于科学研究者而言,不论是细胞的生命周期、生化调控路径、蛋白质交互作用关系等等研究,或是药物研发中对于药物作用目标基因的筛选,到临床的疾病诊断预测,都为基因芯片可以发挥功用的范畴。 基因表现图谱抓取了时间点当下所有的动态基因表现情形,将所有的探针所代表的基因与荧光强度转换成基本数据(raw data) 后,仿如尚未解密前的达文西密码,隐藏的奥秘由丝丝的线索串联绵延,有待专家抽丝剥茧,如剥洋葱般从外而内层层解析出数千数万数据下的隐晦含义。 要获得有意义的分析结果,恐怕不能如泼墨画般洒脱随兴所致。从raw data 取得后,需要一连贯的分析流程(图一),经过许多统计方法,才能条清理明的将raw data 整理出一初步的分析数据,当处理到取得实验组除以对照组的对数值后(log2 ratio),大约完成初步的统计工作,可进展到下一步的进阶分析阶段。
图一、整体分析流程。基本上raw data 取得后,将经过从最上到下的一连串分析流程。(1) Rosetta 软件会透过统计的model,给予不同的权重来评估数据的可信度,譬如一些实验操作的误差或是样品制备与处理上的瑕疵等,可已经过Rosetta error model 的修正而提高数据的可信值;(2) 移除重复出现的探针数据;(3) 移除flagged 数据,并以中位数对荧光强度的数据进行标准化(Normalized) 的校正;(4) Pearson correlation coefficient (得到R 值) 目的在比较技术性重复下的相似性,R 值越高表示两芯片结果越近似。当R 值超过0.975,我们才将此次的实验结果视为可信,才继续后面的分析流程;(5) 将技术性重复芯片间的数据进行平均,取得一平均之后的数据;(6) 将实验组除以对照组的荧光表现强度差异数据,取对数值(log2 ratio) 进行计算。 找寻差异表现基因 实验组与对照组比较后的数据,最重要的就是要找出显著的差异表现基因,因为这些正是条件改变后而受到调控的目标基因,透过差异表现基因的加以分析,背后所隐藏的生物意义才能如拨云见日般的被发掘出来。 一般根据以下两种条件来筛选出差异表现基因:(i) 荧光表现强度差异达2 倍变化(fold change 增加2 倍或减少2倍) 的基因。而我们通常会取对数(log2) 来做fold change 数值的转换,所以看的是log2 ≧1 或≦-1 的差异表现基因;(ii) 显著值低于0.05 (p 值< 0.05) 的基因。当这两种条件都符合的情况下所交集出来的基因群,才是显著性高且稳定的差异表现基因。
生物芯片阅读仪
调研报告 生物芯片阅读仪
【概述】 生物芯片,是指利用微细加工技术并结合有关的化学合成技术,将大量探针分子固定于载体即微小的基片(如玻璃、硅片、有机材料薄膜等)上,然后与标记的样品分子进行杂交,通过检测杂交信号的强弱,对靶分子的序列和数量进行分析检验的微型器件。 生物芯片阅读仪是生物芯片能否得到广泛应用的重要仪器。通过生物芯片阅读仪可以将芯片上测定的结果转变成可供分析处理的图像数据,正确、有效的获取芯片上的生物信息。目前的生物芯片阅读仪主要有两种:CCD系统生物芯片阅读仪和激光共聚焦生物芯片阅读仪。前者具有结构简单、体积小、检测速度快、成本低等优点,对于点阵相对较低的生物芯片的检测有明显的优势;后者以激光作光源,采用共聚焦探测光路,结合高速X向扫描和Y向步进,实现了对生物芯片的扫读和分析。激光共聚焦扫描仪具有检测灵敏度高、动态范围宽、信噪比好、测量精度高等优点,可望成为今后的主流机型。【基本原理与设备组成框图】 一、CCD系统生物芯片阅读仪 CCD系统生物芯片阅读仪有三种,即它激式荧光检测、化学荧光检测和对用同位素曝光的胶片进行检测,本文主要以它激式荧光检测生物芯片阅读仪为例来介绍。该仪器适用于化学自发光、多种激发荧光等生物芯片弱光样片的检测和分析。主要由冷却型科学零级CCD、光学物镜、氙灯光源、均匀照明系统、暗箱、电机驱动选择的发射窄带干涉滤光片和激发窄带干涉滤光片、图像采集卡等部分组成。组成
框图如图1所示。 图1 CCD系统生物芯片阅读仪设备组成框图 主要组成部分的功能及作用: ①光源:CCD系统生物芯片阅读仪采用高压汞灯作为光源,结构比较简单。工作时,用均匀化处理的特殊波长的光去激发生物芯片上的荧光。 ②激发滤光片:经激发窄带滤光片可去除其他波长的光,降低检测背景。 ③发射滤光片、CCD和计算机:生物芯片上标记有荧光染料的靶分子在单色光激发下产生荧光,再经发射窄带干涉滤光片由摄像镜头捕获,成像在CCD相面上,再传至图像采集卡,将信号转化成数字信号进
电子信息新技术系列讲座报告 (7)
电子信息新技术系列讲座报告 班级 学号 姓名 2014年12月
物联网技术 定义: 物联网(Internet of Things)指的是将无处不在(Ubiquitous)的末端设备(Devices)和设施(Facilities),包括具备“内在智能”的传感器、移动终端、工业系统、数控系统、家庭智能设施、视频监控系统等、和“外在使 能”(Enabled)的,如贴上RFID的各种资产(Assets)、携带无线终端的个人与车辆等等“智能化物件或动物”或“智能尘埃”(Mote),通过各种无线和/或有线的长距离和/或短距离通讯网络实现互联互通(M2M)、应用大集成(Grand Integration)、以及基于云计算的SaaS营运等模式,在内网(Intranet)、专网(Extranet)、和/或互联网(Internet)环境下,采用适当的信息安全保障机制,提供安全可控乃至个性化的实时在线监测、定位追溯、报警联动、调度指挥、预案管理、远程控制、安全防范、远程维保、在线升级、统计报表、决策支持、领导桌面(集中展示的Cockpit Dashboard)等管理和服务功能,实现对“万物”的“高效、节能、安全、环保”的“管、控、营”一体化。[1] 诞生发展 1999年诞生,2005年普及,2009年大发展。 物联网(Internet of Things)这个词,国内外普遍公认的是MIT Auto-ID中心Ashton教授1999年在研究RFID时最早提出来的。在2005年国际电信联盟(ITU)发布的同名报告中,物联网的定义和范围已经发生了变化,覆盖范围有了较大的拓展,不再只是指基于RFID技术的物联网。 自2009年8月温家宝总理提出“感知中国”以来,物联网被正式列为国家五大新兴战略性产业之一,写入“政府工作报告”,物联网在中国受到了全社会极大的关注,其受关注程度是在美国、欧盟、以及其他各国不可比拟的。 物联网的概念与其说是一个外来概念,不如说它已经是一个“中国制造”的概念,他的覆盖范围与时俱进,已经超越了1999年Ashton教授和2005年ITU 报告所指的范围,物联网已被贴上“中国式”标签。 关键 简单讲,物联网是物与物、人与物之间的信息传递与控制。在物联网应用中有三项关键技术。 1、传感器技术,这也是计算机应用中的关键技术。大家都知道,到目前为止绝大部分计算机处理的都是数字信号。自从有计算机以来就需要传感器把模拟信号转换成数字信号计算机才能处理。[2]
基因芯片绝版复习总结
第一章生物芯片概述生物芯片概念 生物芯片是将大量生物分子按预先设计的排列固定于一种载体表面,利用生物分 子的特异性亲和反应,来分析各种生物分子存在的量的一种技术。 生物芯片的分类 根据生物芯片的结构特点 根据用途不同: 二、生物芯片的研究概况 生物芯片的发展 最初级的生物芯片 DNA芯片 1991 寡核苷酸芯片 1994 测序芯片 1995 cDNA芯片 其他生物芯片 生物芯片技术研究存在的问题 重复性(稳定性)提高 灵敏度增强 标准化实现 设备及软件完善 操作过程简化 三、生物芯片技术的基础知识 生物芯片技术工作的总流程 生物芯片的制备 生物芯片技术主要包括四个基本技术环节: 芯片制备、样品制备、生物分子反应、信号的检测与分析 生物芯片的制备步骤有哪些?分别有什么目的? 基片处理、点样、固定、封闭 第二章核酸芯片
一、核酸芯片简介 概念:核酸芯片是指采用一定的技术将许多特定的DNA序列排列固定于固相支持物表面,然后与标记的样品进行杂交,通过检测杂交信号来实现对生物样品的快速、并行和高效的检测和分析。 二、核酸芯片的载体 载体概念:用于连接、吸附或包埋各种生物分子使其以水不溶性状态行使功能的固相材料统称为载体。 如何选择载体? 载体表面必须具有可进行化学反应的活性基团,以便于生物分子进行偶联。 使单位载体上结合的生物分子达到最佳容量 载体应当是惰性的和有足够的稳定性 载体具有良好的生物兼容性,以利于制作不同种类的芯片。 载体类型:玻片、硅片、硝酸纤维素膜、尼龙膜、塑料等 三、寡核苷酸芯片技术oligonucleotide microarray oligochip概念:寡核苷酸芯片是把寡核苷酸固定在玻片上,与荧光标记的待检序列在一定条件下杂交,经洗涤后扫描获得检测信息。 制作技术与原理 原位合成原理(略) 合成后微点样原理 利用手工或自动点样装置将预先合成和纯化的寡核苷酸点在经特殊处理的载体上即可。 包括接触式与非接触式两种,主要用于中低密度芯片制备 点样方式及点样针 比较三种针的优缺点? 使用裂缝针时,如果看到玻片上某些点没有点上,分析可能的原因? 点印完以后,含有斑点的区域必须加以行列标志,为什么?如何保存? 点样的后期处理 目的:为了使探针能与载体表面牢固结合,同时,避免在杂交过程中非特异性的吸附对实验结果(特别是背景)造成影响。 小结: 寡核苷酸芯片的基本概念 寡核苷酸芯片的制备原理 光引导原位合成 点样针及点样过程 四、cDNA微阵列芯片 cDNA是与mRNA互补的DNA分子,长约0.2-5.0kb。 cDNA微阵列芯片是由固定于基质材料上的cDNA片段组成的微阵列,待测样品标记后与芯片上的探针分子杂交,通过荧光强度的检测对杂交结果进行分析。主要内容: cDNA文库的构建 提高cDNA文库构建的效率 cDNA基因文库构建的步骤 细胞总RNA的提取和mRNA的分离 第一条cDNA合成 双链cDNA合成 双链cDNA克隆进质粒或噬菌体载体并导入宿主中繁殖 cDNA文库构建效率 cDNA文库构建的效率低的表现?
电子与通信工程前沿技术系列讲座结课论文
电子与通信工程前沿技术系列讲座结课论文 姓名:XXX 学号:XXXXXX 院系:XXXXXX 指导老师:XXXXXX
电子与通信工程前沿技术系列讲座结课论文 第一讲先进信号处理理论及在无线通信、多媒体等领域中的应用 这次报告主要讲了四方面的内容:分数阶傅里叶变换、压缩感知理论框架、无线通信系统信号处理领域和多媒体信号与信息处理领域。陈老师结合分数阶傅里叶变换理论及压缩感知理论,介绍了这些先进信号处理理论的发展研究状况,并通过实例给出了相关理论在无线通信和多媒体领域中的应用研究。接着,他讲述了自己主持的国家自然科学基金以及郑州大学与北京理工大学等院校联合在研的国家自然科学基金重点项目的研究进展。 第二讲未来通信技术——认知无线电与协作通信 穆晓敏讲课的主要内容有:当前频谱利用现状、静态频谱分配的瓶颈及解决方案以及当前遇到的问题,同时还向我们介绍了互联网+、智慧城市、人工智能(AI)、工业 4.0、DT 时代等相关内容。 认知无线电技术已经向“网络与系统”的框架转变,为增强认知能力、降低认知成本,协作手段成为必然。物理层链路技术面临进一步提升性能的“瓶颈”,通过不同网络元素间的多维度协作提高系统整体性能是下一阶段移动通信系统增强的主要途径。在这一过程中,对环境背景信息和用户业务特征的广泛感知是智能化协作与联合资源管理的重要基础。认知无线电与多维度协作通信的结合将成为技术发展的必然趋势。 第三讲智能可穿戴设备概念、基于纺织纤维的可穿戴式产品 文老师主要向我们介绍了智能可穿戴设备的概念以及文老师所创建公司研发的基于纺织纤维的可穿戴产品。 智能可穿戴设备是应用穿戴式技术对日常穿戴进行智能化设计、开发出可以穿戴的设备的总称,如眼镜、手套、手表、服饰及鞋等。最早的可穿戴设备用于军事、户外运动、人体检测等。苹果手表、微软手环和谷歌眼镜是当前最热门的智能穿戴设备,国内也涌现出大量的可穿戴智能设备厂商,像小米手环等。 在不久的将来,智能可穿戴设备将成为人体的一部分,就像皮肤、手臂一样。在更远的未来,手机可能只需向人体植入芯片,而Siri将能直接通过对话帮你打电话,帮你订餐馆,了解你的一切隐私,跟你的亲密程度甚至超过你的家人——可能谷歌眼镜和苹果手表都不再是植入人体的芯片了,他们已经成为人体基因的一部分,可以参与人类的繁衍和进化。第四讲嵌入式系统的开发
Bioconductor基因芯片数据分析系列(一):数据的读取
Bioconductor基因芯片数据分析系列(一):R包中数据的读取 R软件的Bioconductor包是分析芯片数据的神器,今天小编打算推出芯片数据的系列教程。首先讲数据读取,以CLL数据包中的数据为例。 打开R studio。 #安装所需的R包以及CLL包,注意大小写,一般函数都是小写的 source("https://www.360docs.net/doc/0a15995929.html,/biocLite.R"); biocLite(“CLL”) 图1.显示已经安装好Bioconductor了,版本为3.4 #打开CLL包 library(CLL)
图2.显示打开CLL成功
图3.右侧栏内可见看到目前载入的程序包 data(CLLbatch) #调用RMA算法对数据预处理 CLLrma<-rma(CLLbatch) #读取处理后所有样品的基因表达值 e<- exprs(CLLrma) #查看数据 e 我们可以看到,CLL数据集中共有24个样品(CLL10.CEL, CLL11.CEL, CLL12.CEL, 等),此数据集的病人分为两组:稳定组和进展组,采用的设计为两组之间的对照试验(Control Test)。从上面的结果可知,Bioconductor具有强大的数据预处理能力和调用能力,仅仅用了6行代码就完成了数据的读取及预处理。
Bioconductor基因芯片数据分析系列(二):GEO下载数据CEL的读取首先得下载一个数据,读取GEO的CEL文件采用如下命令: 登陆pubmed,找到一个你感兴趣的数据库
在底下栏目下载CEL文件 打开R软件 #安装所需的R包以及CLL包,注意大小写,一般函数都是小写的 source("https://www.360docs.net/doc/0a15995929.html,/biocLite.R"); biocLite(“CLL”) >library(affy) >affybatch<- ReadAffy(celfile.path = "GSE36376_RAW") 请注意目录的路径,在window下,反斜杠‘\’要用转义字符“\\”表示。 然后可以使用RMA或者MAS5等方法对数据进行background.correction, normaliztion, pm.correct等等一系列处理。如果你一切用默认参数,则可以使用如下命令: >eset<- rma(affybatch),or eset<- mas5(affybatch) >exp<- exprs(eset) exp就是数字化的表达谱矩阵了 请注意,rma只使用匹配探针(PM)信号,exp数据已经进行log2处理。mas5综合考虑PM和错配探针(MM)信号,exp数据没有取对数。 下一期就得等到2017年春节期间啦,敬请期待~ 另外一种是直接利用GEO上面的GEO2R按钮里面的R script下载文件: # Version info: R 3.2.3, Biobase 2.30.0, GEOquery 2.40.0, limma 3.26.8 # R scripts generated Mon Dec 26 06:54:42 EST 2016 Server: https://www.360docs.net/doc/0a15995929.html, Query: acc=GSE36376&platform=GPL10558&type=txt&groups=&color s=&selection=XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXX&padj=fdr&logtransform=auto&col umns=ID&columns=adj.P.Val&columns=P.Value&columns=F&c
股票投资分析讲座心得体会
优质文档在您身边/双击可除股票投资分析讲座心得体会 股票投资分析讲座心得体会 我们作为一名金融专业的大学生,即将踏入社会,但是,我们缺少丰富的市场分析及理财常识,导致很多在校大学生不敢投资,或是盲目投资,无形中造成了资源的浪费。颜老师通过五次的讲课,为我们深刻剖析了如何在股票市场上规避风险,取得先机,传授了我们股票投资分析的一些技巧和方法。使我们受益匪浅。直到活动结束,同学们都还意犹未尽。下面是我从这五次讲座中所学到的知识及心得体会。 现在的社会是市场经济的社会,受社会发展形势的影响,人们的思想也逐步发生转变,尤其是在财富的创造方面,现在的人们不再像以前那样只懂得把钱攥在手里,而是逐渐趋向于投资,用财富去创造财富。 投资是调动产业机构和产品机构、优化资源配置的基本途径,也是投资者实现经济利益的重要手段。随着国民经济的发展和经济市场化程度的不断提高,投资范围和投资规模将不断扩大,投资主体和投资渠道将逐渐多元化,影响投资的因素也将日益复杂多样。投资渠道和投资方式不同,投资者所承担的风险和所获得的投资收益也将有所不同。投资者只有掌握科学的投资分析理论和分析方法,提高投资决策的科学性,才能降低投资风险,实现最佳投资收益。所以投资分析越来越重要。 股票投资分析顾名思义是指对股票投资方面的分析。要想了解股票投资分析就要先了解什么是股份公司、股票。 股份公司在国外已经有好几百年历史了。而而在中国,也就只有几十年的历史,还有好多人感到生疏。其实我们经常说的一些世界有名的大公司,例如美国的IBM、可口可乐公司、德国的西门子公司、日本的索尼公司、松下公司,无一不是股份公司。国内许多股份公司的产品也早就家喻户晓,比如长虹电视机,春兰空调,嘉**摩托车等等。为什么要采用股份公司的形式那?股份公司有其他企业所没有的优越性一方面他可以筹集到大量的资金,满足社会大生产的需要,另一方面他将风险分散到整个社会极大地增强了企业抵抗风险的能力。 股份公司的股份分别由不同的人持有,每人持有的数量有多有少,而不论多少,凡是持有股份的人就是股东,持有的凭证就是股票。股票的几个特性,无期性、权责性、流通性、风险性。 股票分析包括基本分析和技术分析两部分。基本分析研究影响股票供给和需求关系变化的因素,它的主要内容是分析国家的宏观经济环境、股市政策、上市公司的各种情况以及能够影响股市变化的其它信息,作为股市投资的参考,以帮助投
分子机制-核酸检测-基因芯片
主题:基因芯片 概述: 基因芯片(又称DNA芯片、生物芯片)技术系指将大量(通常每平方厘米点 阵密度高于400)探针分子固定于支持物上后与标记的样品分子进行杂交,通过检测每个探针分子的杂交信号强度进而获取样品分子的数量和序列信息。通俗地说,就是通过微加工技术,将数以万计、乃至百万计的特定序列的DNA 片段(基因探针),有规律地排列固定于2cm2的硅片、玻片等支持物上,构成的一个二维DNA探针阵列,与计算机的电子芯片十分相似,所以被称为基 因芯片。基因芯片主要用于基因检测工作。 基因芯片又称为DNA微阵列(DNA microarray),可分为三种主要类型:1) 固定在聚合物基片(尼龙膜,硝酸纤维膜等)表面上的核酸探针或cDNA片段,通常用同位素标记的靶基因与其杂交,通过放射显影技术进行检测。这种方法的优点是所需检测设备与目前分子生物学所用的放射显影技术相一致,相对比较成熟。但芯片上探针密度不高,样品和试剂的需求量大,定量检测存在较多问题。2)用点样法固定在玻璃板上的DNA探针阵列,通过与荧光标记的靶基因杂交进行检测。这种方法点阵密度可有较大的提高,各个探针在表面上的结合量也比较一致,但在标准化和批量化生产方面仍有不易克服的困难。3)在玻璃等硬质表面上直接合成的寡核苷酸探针阵列,与荧光标记的靶基因杂交进行检测。该方法把微电子光刻技术与DNA化学合成技术相结合,可以使基因芯 片的探针密度大大提高,减少试剂的用量,实现标准化和批量化大规模生产,有着十分重要的发展潜力。 目的: 基因功能的研究。通过基因芯片可以大规模,高通量地对成千上万个基因进行同时研究。 原理:
基因芯片的测序原理是杂交测序方法,即通过与一组已知序列的核酸探针杂交进行核酸序列测定的方法。在一块基片表面固定了序列已知的八核苷酸的探针,当溶液中带有荧光标记的核酸序列,例如TATGCAATCTAG,与基因芯片上对应位置的核酸探针产生互补匹配时,通过确定荧光强度最强的探针位置,获得一组序列完全互补的探针序列。据此可重组出靶核酸的序列。 基因芯片的测序原理图 步骤: 1.样品的准备:由于灵敏度所限,多数方法需要在标记和分析前对样品进行适当的扩增。 2.杂交检测,显色和分析测定方法主要为荧光法,其重复性较好,但灵敏度较低。目前正在发展的方法有质谱法、化学发光法、光导纤维法等。以荧光法为例,当前主要的检测手段是激光共聚焦显微扫描技术,以便于对高密度探针阵列每个位点的荧光强度进行定量分析。 3.结果分析: 3.1.差异表达基因分析和聚类分析 基因芯片数据标准化、上调基因/下调基因、t-检验(P值)、方差分析(F-检验)、基因聚类(Cluster)、热图(Heatmap)、多样本分子标志物聚类(如肿瘤分子标志物聚类分析)。可分析mRNA表达谱、miRNA表达谱和甲基化基因芯片数据表达谱差异。 3.2.基因数据相关性分析 分析内容:基因芯片数据质量控制和分析(结果形式:相关性分析结果表格)
生物芯片现状及展望
生物芯片的现状和展望 (2001-01-02) >>>欢迎进入网易实时个性化股票系统 想的崭新世纪。在这个充满期冀的世纪,人类以往的许许多多遐想 有希望美梦成真,而生物芯片正是实现这些美梦最有希望的技术之 一。目前从事这一行业的人是一群名副其实的二十一世纪追梦人, 他们明天可能取得的成就无疑会造福于人类,极大地方便人们的生 活。目前的生物芯片技术既不象一些人说的无所不能,更不像另外 一些人说的仅仅是概念和骗人的把戏。以上两种人仅仅凭着自己的一知半解就对生物芯片发 表盲人摸象的看法,十分不利于它的健康发展。其实只要稍稍抱有深入了解的想法,就不难 发现生物芯片正在飞速成长,正从粗糙的原型变为精致的产品,正从实验室走向社会,迟早 有一天会和现在的电脑CPU一样普遍,深入到千家万户。也不过就3、4年光景,我们已经 很难想象当初在实验室用三种温度的水浴锅做PCR的情景,当初谁又能料到PCR技术今天 的应用是如此普及和便利。 生物芯片(Biochip或Bioarray)概念虽然来源于计算机芯片,但其实和CPU有着天壤之别, 唯一相似的地方就是它们都具有集成化微型化特征。生物芯片是根据生物分子间特异相互作 用的原理,将生化分析过程集成于芯片表面,从而实现对DNA、RNA、多肽、蛋白质以及 其它生物成分的高通量快速检测。生物芯片诞生至今不过十年光景,但无论是具体形式还是 应用领域都有很大发展。目前生物芯片总的发展趋势是微型化、集成化和多样化,在技术的 更新换代速度上会越来越快,一些已有的产品或技术很有可能被后来不断涌现的新技术和新 产品部分替代甚至完全替代,与此同时,新技术和新产品也带来更广泛的应用领域和更大的 市场空间。 狭义的生物芯片概念已经众所周知,主要是指通过不同方法将生物分子(寡核苷酸、cDNA、genomic DNA、多肽、抗体、抗原等)固着于硅片、玻璃片(珠)、塑料片(珠)、 凝胶、尼龙膜等固相介质上形成的生物分子点阵。其实这一类芯片可以界定为Microarray类 型的芯片(BioArray)。生物芯片在此类芯片的基础上又发展出微流体芯片(Mirofluidics Chip, 亦称微电子芯片Microelectronic Chip),也就是我们所说的缩微实验室芯片(Lab-on-Chip)。 目前我们国内也有人在开展这方面的研究,但离微流体芯片的真正实用可能还有很长一段路 要走,估计至少要再过五年,才会有较为成熟的产品出来。虽然微流体芯片对我们来说还比 较远,但我们应该看到这种芯片可能最接近生物芯片概念的核心理念,一旦成熟,市场空间 不可限量。而且目前已经有几家公司在推出这类芯片的原始模型,比如日本公司Nanogen,今 年就在中国市场推出可以进行简单杂交反应的微流体芯片产品NanoChipTM Cartridge,只有 扑克牌大小。另外一家从惠普公司脱离出来的小公司则推出了和电脑CPU非常相似的微流体 芯片产品,可以进行蛋白或核酸的电泳分离。这些原始产品功能相对简单,但已经让我们看 到了未来成熟产品的身影。目前美国的圣地亚哥(San Diega)是生物芯片技术创新的重要源泉 之一。几家在技术上取得领先的公司都在那里设有研发中心,如Illumina, Inc.和Nanogen AVIVA Biosciences corp.等公司。根据Nanogen公司自己的介绍,该公司开发的100-test site 的微流体芯片已在Mayo Clinic和University of Texas Southwestern Medical Centre用于遗传病
微型计算机概述
返回首页
一片集成电路芯片上,显然该芯片是整个微机系统的核心,称为中央处理器CPU, 微处理器是微机系统的核心部分,自70年代初出现第一片微处理器芯片以来,微几乎每两年翻一番,其发展速度大大超过了前几代计算机。 微机系统及相关技术的发展,主要涉及到以下几个方面:CPU、主频、缓存、新技术、3D NOW!技术等)。 一、微机的发展 微机系统的核心部件为CPU,因此我们主要以CPU的发展、演变过程为 线索,来介绍微机系统的发展过程,主要以Intel公司的CPU为主线。 第一代:4位及低档8位微处理器 ?1971年,Intel公司推出第一片4位微处理器Intel4004,以其为核心组成了随后出现的Intel4040,是第一片通用的4位微处理器。 ?1972年,Intel8008,8位,集成度约2000管/片,时钟频率1MHz。 第二代:中、低档8位微处理器 ?1973年~1974年,Intel8008、M6800、Rockwell6502,8位,集成度5000管/ 这一时期,微处理器的设计和生产技术已经相当成熟,组成微机系统的其它部件也着提高集成度、提高功能与速度,减少组成系统所需的芯片数量的方向发展。 第三代:高、中档8位微处理器 ?1975年~1976年,Z-80,Intel8085,8位,时钟频率2~4MHz,集成度约 一系列单片机。 第四代:16及低档32位微处理器 ?1978年,Intel首次推出16位处理器8086(时钟频率达到4~8MHz),808线都是16位,地址总线为20位,可直接访问1MB内
存单元。 ?1979年,Intel又推出8086的姊妹芯片8088(时钟频率达到48MHz),集成片。它与8086不同的是外部数据总线为8位(地址线为20位)。 ?1982年,Intel推出了80286(时钟频率为10MHz),该芯片仍然为16位结24位,可访问16MB内存,其工作频率也较8086提高了许多。80286向后兼容808(实模式),并增加了部分新指令和一种新的工作模式——保护模式。 ?1985年,Intel又推出了32位处理器80386(时钟频率为20MHZ),该芯片总线都是32位,可访问4GB内存,并支持分页机制。除了实模式和保护模式外,8拟8086”的工作模式,可以在操作系统控制下模拟多个8086同时工作。 ?1989年推出了80486(时钟频率为30~40MHz),集成度达到15万~50万管至上百万管/片,因此被称为超级微型机。早期的80486相当于把80386和完成浮80387以及8kB的高速缓存集成到一起,这种片内高速缓存称为一级(L1)缓存,二级(L2)缓存。后期推出的80486 DX2首次引入了倍频的概念,有效缓解了外部上CPU主频发展速度的矛盾。 第五代:高档32位微处理器 ?1993年,Intel公司推出了新一代高性能处理器Pentium(奔腾),Pentium 标量结构(支持在一个时钟周期内执行一至多条指令),且一级缓存的容量增加到大提升了CPU的性能,使得Pentium的速度比80486快数倍。除此之外,Pentium 能,把一个低主频CPU当作高主频CPU来使用,使得花费较低的代价可获得较高的 ●AMD和Cyrix推出了与Pentium兼容的处理器K5和6x86,但是由于这些产品的超频性能不强,且主频始终跟在Intel后面跑,因此只获得了少部分的市场份额。 ?1996年,Intel公司推出了Pentium Pro(高能奔腾),该芯片具有两大特色CPU同频运行的256kB或512kB二级缓存;二是支持动态预测执行,可以打乱程序优化顺序同时执行多条指令,这两项改进使得Pentium Pro的性能又有了质的飞跃 ?1997年初,Intel发布了Pentium的改进型号——Pentium MMX(多能奔腾)32kB,同时增加了57条MMX(多媒体扩展)指令,有效地增强了CPU处理音频、用的能力。
