胚胎发育相关基因stk40的蛋白表达和生物信息学分析
生命科学数据分析的方法与应用

生命科学数据分析的方法与应用近年来,生命科学领域的迅速发展,使得大量数据积累在了科研工作者的手中。
而数据的分析与应用,已经成为生命科学研究的必然趋势。
而针对如此多的数据,如何进行高效的挖掘,成为了生命科学研究领域中重要的问题之一。
本文将结合实际案例,探讨生命科学数据分析的方法与应用。
一、SNP数据分析与应用SNP(sing nucleotide polymorphisms)是指单个核苷酸的多态性,以其多变性为基础进行基因千变万化地研究。
SNP数据如何进行分析和应用呢?首先,进行SNP芯片的数据分析。
通过芯片中的探针将样本DNA与芯片固定的单核苷酸进行匹配并输出,然后对输出结果进行标准化和归一化处理。
接着进行基因型的分析,根据样本中每个SNP位点的基因型分析, 确定每个个体所携带的所有SNP位点的基因型。
其次,进行SNP数据的应用。
SNP 基因组分型技术的应用包括疾病风险预测和药物治疗反应性预测。
例如,针对遗传性疾病,可以通过SNP与疾病之间的关联,估计个体发病风险。
针对癌症治疗,可以根据药物代谢 SNP 和靶标 SNP 的情况,预测药物在患者体内的代谢效率和治疗效果。
二、生物成像数据分析与应用生物成像技术已经成为了生命科学研究中不可或缺的工具,如何进行生物成像数据的分析与应用呢?首先,进行生物成像数据的处理。
对于不同的生物成像技术,数据处理方法和软件有所不同,例如,对于荧光显微镜下的图像,需要进行图像处理和去噪,以得到清晰的细胞图像。
其次,进行生物成像数据的分析。
对生物成像样品的图像进行分析,可以获得样品中各种特征如面积、位置、大小、形态等,并可以对生命过程进行动态跟踪。
例如,在活体成像中,可以利用时间序列数据分析细胞轨迹并找出细胞的运动及各个时间点的强度。
最后,进行生物成像数据的应用。
生物成像技术的应用广泛,例如,在肿瘤病理学中,可以通过活体成像技术来实时观察肿瘤细胞的生长、扩散和药物疗效;在生物学研究中,可以通过高分辨率显微镜图像,在细胞水平上探究细胞器的组成和功能。
生物信息学分析在蛋白质组学中的应用

生物信息学分析在蛋白质组学中的应用近年来,生物信息学分析在蛋白质组学中的应用已成为研究热点。
蛋白质组学是研究生物样品(如细胞、组织、生物体)中所有蛋白质的形态、结构、功能和相互作用的技术和方法学。
生物信息学是利用计算机和生物学知识,研究生物信息的学科。
生物信息学的分析方法包括序列分析、结构分析、功能分析等。
该分析方法在蛋白质组学中的应用,可以帮助我们更好的理解蛋白质的功能和相互作用,从而在疾病预防和治疗方面做出更好的决策。
一、蛋白质组学中的生物信息学分析方法1. 序列分析序列分析是生物信息学中最基本的分析方法。
它通过比较蛋白质序列中氨基酸的差异,揭示蛋白质的结构和功能。
序列分析包括多序列比对、同源性搜索和序列分类。
多序列比对法将多条相似序列按某种规则进行比对,从而找出相同的部分,判断它们之间的差异和相似度,进而预测蛋白质的结构和功能。
同源性搜索是指利用已知的蛋白质序列“搜索”数据库中的序列,以寻找和已知蛋白质相似的新序列。
序列分类是将蛋白质序列进行分类,以便对新蛋白质序列进行分析和预测。
2. 结构分析结构分析是通过对蛋白质的结构进行分析,揭示蛋白质的功能、相互作用和调控机制等信息。
结构分析方法主要包括蛋白质结构预测、结构比对和蛋白质互作分析等。
蛋白质结构预测是利用已知的蛋白质结构数据,预测新的蛋白质结构。
结构比对是将蛋白质结构与数据库中的已知蛋白质结构进行比对,以发现蛋白质之间的差异和相似性。
蛋白质互作分析是研究生物大分子之间相互作用的过程,揭示蛋白质的通讯机制、信号传递和调控机制等。
3. 功能分析功能分析是通过生物信息学分析方法揭示蛋白质在生物体内的功能和调控机制。
功能分析方法包括蛋白质功能注释、基因本体论和通路分析等。
蛋白质功能注释是通过对蛋白质序列、结构和相互作用等进行分析,明确蛋白质的功能和生物学作用。
基因本体论是一种分类方法,将蛋白质的功能按照一定的规则进行分类,以便对新的蛋白质进行预测和注释。
生物信息学中的蛋白质分析技术

生物信息学中的蛋白质分析技术蛋白质是生物体中不可或缺的重要分子,其功能包括酶催化、信号传递、结构支持等多种生命活动。
蛋白质分析是生物信息学研究中的重要领域之一,目的是从生物样品中获取有关蛋白质的信息。
这项技术不仅可以揭示蛋白质的结构和功能,还可以为医学诊断和药物研发提供重要的参考。
一、蛋白质分析的基本流程蛋白质分析的基本流程包括蛋白质提取、分离纯化、分析鉴定等几个步骤。
蛋白质提取是将目标蛋白从生物样品中提取出来,一般采用机械破碎、化学分解、超声波等方法。
分离纯化是将目标蛋白与其他蛋白分离开来,可以采用电泳、层析、过滤等方法。
分析鉴定则是对分离得到的蛋白进行化学、物理和生物学的分析,如质谱分析、核酸测序、免疫学检测等方法。
二、质谱分析技术的应用质谱分析是一种可以同时检测多种蛋白质组成和结构的方法,其技术基础是将蛋白质分离并进行离子化后进行质量分析。
这种方法被广泛地应用于蛋白质组学和蛋白质互作等领域。
在蛋白质组学中,将样品中的所有蛋白质分离并进行质谱分析,可以获得大量的信息,如蛋白质的数量、种类、分布和修饰状态等。
质谱分析技术的应用还包括蛋白质互作的研究。
蛋白质互作通常是指两个或多个蛋白质之间的相互作用,这在生物活动中非常重要。
质谱分析可以用来鉴定已知的蛋白质互作或发现新的蛋白质互作,这对于深入理解生物活动机理具有重要意义。
三、结构生物学的应用结构生物学是研究蛋白质三维结构的一种技术,其目的是探究蛋白质结构与功能之间的关系。
现有的结构生物学技术主要包括X射线晶体学、核磁共振和电子显微镜。
通过这些技术,可以确定单个蛋白质的原子结构,也可以确定蛋白质的超分子结构,如蛋白质-DNA复合物和蛋白质-蛋白质复合物等。
在药物研发方面,结构生物学的应用也非常广泛。
通过了解蛋白质的结构,可以设计出针对特定靶标的药物,并对药物与靶标之间的相互作用进行优化和改良。
四、生物信息学的应用生物信息学是将计算机和数学等方法应用于生物学研究的一种学科。
【江苏省自然科学基金】_信息学软件_期刊发文热词逐年推荐_20140816
推荐指数 2 2 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
2010年 序号 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20
科研热词 错义突变 结肠直肠肿瘤 生物信息学分析 生物信息学 灰飞虱 植物双元表达载体 抗病毒蛋白bcturso 家族性 可视化 可扩展矢量图形(svg) 分子伴侣 共生菌 交互 不结球白菜 wolbachia web val384asp pse-bio hmlh1基因 groel
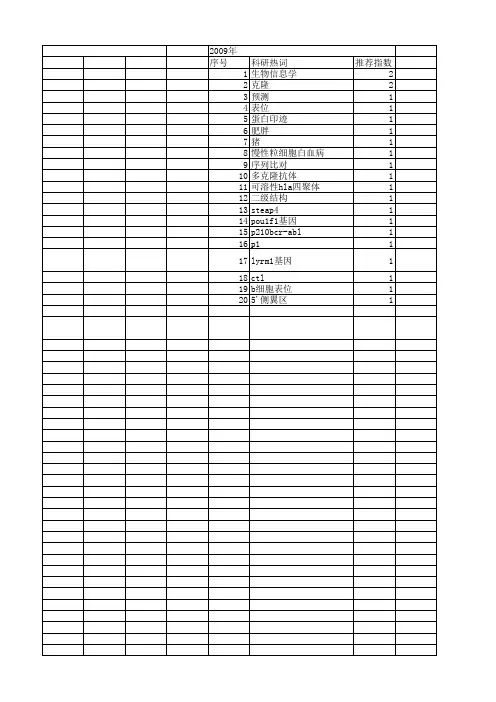
2009年 序号 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20
科研热词 生物信息学 克隆 预测 表位 蛋白印迹 肥胖 猪 慢性粒细胞白血病 序列比对 多克隆抗体 可溶性hla四聚体 二级结构 steap4 pou1f1基因 p210bcr-abl p1 lyrm1基因 ctl b细胞表位 5'侧翼区
2012年 序号 1 2 3 4 5 6 7 8 9 ቤተ መጻሕፍቲ ባይዱ0 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25
2013年 科研热词 推荐指数 序号 转录因子结合位点 2 1 生物信息学分析 2 2 p73启动子区 2 3 dna甲基化 2 4 随机引物pcr 1 5 进化树分析 1 6 血清多肽 1 7 荧光素酶报告质粒 1 8 肝受体类似物-1(lrh-1) 1 9 组织表达 1 10 磁珠富集 1 11 生物信息学 1 12 湖羊 1 时间飞行质谱 1 序列分析 1 幽门螺杆菌 1 干扰素调节因子-1(irf-1) 1 山羊痘病毒 1 启动子 1 原核表达 1 卵巢癌 1 x染色体连锁的凋亡抑制蛋白相关因子1(xaf1) 1 race 1 p32基因 1 nadh脱氢酶 1
小鼠早期胚胎发育过程中细胞凋亡及凋亡基因表达的检测

A :A A G G T G G A A G A G T G G T G A G T
Bax
外引物
S :G A C A G G G G C C T T T T T G T T A C
A :A A A G A A T T C G C T A G C A A A G
内引物
S :C C A G G A T G C G T C C A C C A A
见表 1 。 1.8 免疫组化
取处于不同发育时期的形态正常胚胎,4% 多 聚甲醛固定 30 min, PBS 洗涤 3 次,每次 5 min, 37 ℃,10% 马血清封闭 1 h;加入兔抗鼠多抗(Bcl- 2、Bax、Bak),4 ℃温育过夜。PBS 洗涤 3 次, 加入生物素化标记的山羊抗兔二抗,室温温育 3 0 min。P B S 洗涤 3 次,加入碱性磷酸酶标记的卵白
关键词 早期胚胎发育;凋亡;基因表达
在哺乳动物中,大约有 30% ̄70% 的胚胎在发 育过程中夭折,其中绝大部分发生在胚胎发育的早 期。胚胎死亡的原因很复杂,可能与遗传、环境等 因素有关,胚胎细胞凋亡是胚胎夭折的主要方式[1]。 诱发胚胎细胞凋亡的原因很多,受精过程的异常[2]、 胚胎发育过程中温度的不稳定[3]、不适宜的生长因 子水平[ 4 ] 、细胞内基因的非正常表达[ 5  ̄ 7 ] 等,都可 能诱发胚胎细胞发生凋亡。
Oct 4和Caspase 6在牦牛卵母细胞和早期胚胎发育过程中的表达及LIF对其在卵母细胞中表达的

Oct 4和Caspase 6在牦牛卵母细胞和早期胚胎发育过程中的表达及LIF对其在卵母细胞中表达的影响引言:牦牛作为高寒地区重要的畜牧业资源,其繁殖力对于其养殖业的进步具有重要意义。
卵母细胞的健康和早期胚胎发育能力直接影响到牦牛的繁殖能力。
Oct 4和Caspase 6是两个与细胞命运和凋亡相关的基因,在早期胚胎发育中扮演着重要的角色。
本文旨在探讨Oct 4和Caspase 6在牦牛卵母细胞和早期胚胎发育过程中的表达状况,并探究白介素-6(Leukemia Inhibitory Factor,LIF)对其在卵母细胞中表达的影响。
方法与材料:本探究选取健康的牦牛为探究对象,采集其卵巢组织样本和早期胚胎组织样本。
起首,使用免疫组化技术检测Oct 4和Caspase 6在牦牛卵母细胞和早期胚胎中的表达状况。
其次,通过荧光定量PCR技术检测Oct 4和Caspase 6的mRNA水平,并与其蛋白表达状况进行比较分析。
最后,接受细胞培育技术,探究LIF对牦牛卵母细胞中Oct 4和Caspase 6的表达影响。
结果:免疫组化结果显示,Oct 4在牦牛卵母细胞和早期胚胎中的表达较为明显,而Caspase 6的表达相对较低。
荧光定量PCR结果显示,Oct 4的mRNA水平在卵母细胞和早期胚胎中均较高,而Caspase 6的mRNA水平则相对较低。
细胞培育试验结果显示,添加LIF后,牦牛卵母细胞中Oct 4 mRNA和蛋白表达水平显著上调,而Caspase 6的表达则下调。
谈论:Oct 4是一种转录因子,能够调控胚胎发育中的细胞命运,增进干细胞的自我更新。
在牦牛卵母细胞和早期胚胎中,Oct 4的高表达可能与其干细胞特性相关,有助于维持卵母细胞的健康和早期胚胎的正常发育。
Caspase 6则是一种凋亡相关蛋白,其相对低表达可能是为了维持早期胚胎的存活。
此外,本探究发现LIF可影响牦牛卵母细胞中Oct 4和Caspase 6的表达,这表明LIF可能参与调控牦牛卵母细胞和早期胚胎发育过程中的细胞命运和凋亡。
生物信息学及其在蛋白质组学中的应用
蛋白质组数据库是蛋白质组学研究的主要内容之一 。 通过构建不同环境条件下组织或细胞全部蛋白质的数据库 来研究蛋白质表达的差异情况 [4] 。与其他数据库相比 ,目前 大部分蛋白质组数据库都有以下几个方面的特点 : (1) 由于 蛋白质相关数据的种类繁多 ,蛋白质组数据库的种类也多种 多样 ,如双向电泳数据库 、基于蛋白序列的数据库 、蛋白质一 级或高级结构数据库 、蛋白质相互作用数据库等等 ; (2) 数据 更新速度快 ,网络上的蛋白质组数据库的数据几乎每天都在 更新 ; (3) 网络共享程度高 ,越来越多的数据库资源与互联网 相互配合 ,使得蛋白质相关数据的利用率空前的提高 。蛋白
2. 1 基于双向电泳图谱的数据库 双向电泳技术是蛋白质组学研究中最重要的实验技术
之一 ,所以基于双向电泳图片的数据库也成了蛋白质组学研 究中主要内容 。这些数据库有以下几个特点 : (1) 数据直观 。 以蛋白质双向电泳图片为索引 ,将图片放在互联网上 ,每一 个蛋白点的信息 (等电点 、分子量等等) 都可以通过点击图片 上相应位置的蛋白点得到 ; (2) 以蛋白质双向电泳图片为基 础 ,并与其他数据 (蛋白质序列 、结构和功能等信息) 进行整 合 。目前 , 主要有水稻蛋 白 质 组 数 据 库 ( The Rice Proteome Database) [5] 、SWISS - 2DPAGE[6] 、大肠杆菌双向电泳数据库 ( ECO - 2DBASE) [7] 、酵母蛋白质组数据库 ( YPD) [8] 、造血干 细胞 蛋 白 质 组 数 据 库 ( HSC - 2DPAGE) [9] 、SIENA - 2DPA2 GE[10] 、PHCI - 2DPAGE[11] 等等 。 2. 1. 1 水稻蛋白质组数据库
【江苏省自然科学基金】_内含子_期刊发文热词逐年推荐_20140820
2012年 序号 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35
科研热词 推荐指数 序列分析 2 鲤 1 靶基因 1 转基因组合植株 1 质型多角体病毒 1 表达特征 1 血清类黏蛋白1样蛋白3基因 1 簇毛麦 1 生物信息学 1 淋球菌 1 液泡膜水通道蛋白 1 水稻品种 1 抗性 1 慢病毒载体 1 微小rna 1 序列提取 1 差异表达 1 家蚕 1 大豆全基因组 1 基因表达 1 哮喘 1 启动特性 1 启动子 1 功能 1 内含子剪接 1 内含子 1 人cd46基因启动子 1 xip 1 perl程序 1 ospgip1基因 1 igf2b 1 hsa-mir-26b 1 egfp 1 dvblt101基因 1 3-羟酰辅酶a脱氢酶 1
推荐指数 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
2010年 序号 1 2 3 4 5 6 7 8 9 10 11 12 13
科研热词 单核苷酸多态性 质型多角体病毒 胰岛素样生长因子2 相关性 清远麻鸡 感染应答假定蛋白 家蚕 增重 基因型 吉富罗非鱼 全长cdna 体型 capn1基因
2008年 序号 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26
科研热词 推荐指数 绵羊 2 snps 2 黏附 1 鹅 1 转录因子ap-4 1 荧光原位杂交 1 肌肉生长抑制索基因 1 生长发育性状 1 生物信息学 1 淋病奈瑟菌 1 小麦-簇毛麦易位系 1 家蚕 1 奥利亚罗非鱼 1 基因胚抗原相关细胞黏附因子1 1 leptin基因 1 leptin 1 hv-s/tpk基因 1 gene 1 ef-1α 1 cos-1细胞 1 cdna末端快速扩增 1
牛ANGPTL4基因结构和功能的生物信息学分析
个跨膜结构 区域 。通过 分子进化 树研 究发现牛 的A G T4基因在人 、 鼠 、 鼠、 、 N PL 小 大 猪 黑猩猩 、 、 狗 鸡等物种 中 ,
( . o eeo i c n e X na gN r l nvr t, iyn 6 0 0 C ia 2 T eFr e i d 1 C l g f f S i c , iyn o iesy X n ag4 4 0 , hn ; . h i t no Mi— l Le e ma U i sS r
d e Sc o lo n y u t Zh m a a 63 0 Ch n 3. le e o i lS in e a d Te h l g l h o fPig u Co n y, u din 4 3 0, i a; Col g fAn ma c e c n c noo y,No t — rh
ti s a ay e n l d n t tu t r en wa n lz d i c u i g i sr cu e,p y io—c e c lp o e y,sg lp p i e,ta s mb a e sr c s h sc h mi a r p r t ina e td r n me r n tu —
与 猪 的亲 缘 关 系最 近 。
关键词 : ; 牛 血管生成素样蛋 白 一 ( NG T4 ; 4 A P L ) 序列分析 ; 生物信息学
中 图 分 类 号 :8 3 2 ¥2 . 文献标识码 : A 文 章 编 号 :0 0— 2 6 2 1 ) 1— 15— 6 10 2 8 (0 0 0 0 3 0
小麦TaHKT家族基因的生物信息学分析
麦类作物学报 2024,44(2):147-157J o u r n a l o fT r i t i c e a eC r o ps d o i :10.7606/j.i s s n .1009-1041.2024.02.02网络出版时间:2023-12-13网络出版地址:h t t ps ://l i n k .c n k i .n e t /u r l i d /61.1359.S .20231212.1500.008小麦T a H K T 家族基因的生物信息学分析收稿日期:2023-03-06 修回日期:2023-04-25基金项目:山东省重点研发计划项目(2022L Z G 001)第一作者E -m a i l :960412723@q q .c o m (苏瑞平)通讯作者E -m a i l :c h m _q i n y x @u jn .e d u .c n (秦余香)苏瑞平1,张宝1,王玉宁1,张淑娟2,秦余香1(1.济南大学生物科学与技术学院,山东济南250022;2.山东省农业科学院作物研究所,山东济南250131)摘 要:高亲和性钾离子转运蛋白(h i g h -a f f i n i t y K +t r a n s po r t e r ,H K T )是植物体内一种非常重要的离子转运蛋白,具有运输N a +/K +的能力,在植物响应盐胁迫过程中发挥重要作用㊂为系统了解小麦T a H K T 家族基因,挖掘有效的小麦耐盐基因,本研究采用生物信息学的方法,对小麦T a H K T 家族基因进行了全基因组分析,鉴定了家族成员,构建了系统进化树,并对其跨膜结构域㊁染色体定位㊁基因结构㊁M o t i f ㊁上游顺式作用元件㊁共线性以及表达谱等进行了分析㊂结果鉴定出23个T a H K T 基因,其编码蛋白质长度为443~590a a,等电点范围8.2~10.4,跨膜结构域为6~8个㊂23个基因分布于小麦的2㊁4㊁6㊁7号染色体上,根据亲缘关系可将其分为3个亚族,同一亚族的成员具有较为相似的M o t i f 组成和基因结构㊂23个基因中,发现了4对串联重复基因,10对大片段复制基因,具有良好的共线性㊂顺式作用元件分析发现,大部分T a H K T 成员含有盐胁迫响应元件MY B ㊁G -b o x ㊁A B R E 和D R E ㊂表达谱分析发现,T a H K T 基因在小麦的16种组织中均有表达,在根㊁茎中的表达量较高,其中T r a e s C S 4D 02G 361300(I D ,下同)在根中的表达量最高㊂在盐胁迫处理后,不同成员对盐胁迫响应程度不同,T r a e s C S 7B 02G 318400在盐胁迫处理后表达量逐渐降低;T r a e s C S 2B 02G 451400和T r a e s C S 2D 02G 428200在盐胁迫处理后的表达量也明显降低;T r a e s C S 2B 02G 451800在根部几乎不表达,但在受到盐胁迫处理后表达量逐渐提高,推测它们在小麦抵御盐胁迫过程中发挥不同作用㊂关键词:小麦;T a H K T ;生物信息学;基因家族分析;盐胁迫中图分类号:S 512.1;S 330 文献标识码:A 文章编号:1009-1041(2024)02-0147-11B i o i n f o r m a t i cA n a l y s i s o f t h eT a H K TG e n eF a m i l yi n W h e a t S UR u i p i n g 1,Z H A N GB a o 1,W A N GY u n i n g 1,Z H A N GS h u j u a n 2,Q I NY u x i a n g1(1.S c h o o l o fB i o l o g i c a l S c i e n c e a n dT e c h n o l o g y ,U n i v e r s i t y o f J i n a n ,J i n a n ,S h a n g d o n g 250022,C h i n a ;2.C r o p Re s e a r c h I n s t i t u t e ,S h a n d o n g A c a d e m y o fA g r i c u l t u r a l S c i e n c e s ,J i n a n ,S h a n d o n g 250131,C h i n a )A b s t r a c t :T h eh i g h -a f f i n i t y K +t r a n s p o r t e r (H K T )i sav e r y i m p o r t a n ti o nt r a n s po r t e r p r o t e i ni n p l a n t s ,w i t h t h e a b i l i t y t o t r a n s p o r tN a +/K +,a n d p l a y s a c r u c i a l r o l e i n p l a n t r e s po n s e t o s a l t s t r e s s .I no r d e r t os y s t e m a t i c a l l y u n d e r s t a n dt h eT a H K T g e n e f a m i l y i n w h e a t a n de x pl o r ee f f e c t i v ew h e a t s a l t t o l e r a n c e r e l a t e d g e n e s ,i n t h i s s t u d y ,w e c o n d u c t e d a g e n o m e -w i d e a n a l y s i s o f t h i s g e n e f a m i l y ,i d e n t i f i e d t h em e m b e r s ,c o n s t r u c t e d a p h y l o g e n e t i c t r e e ,a n d a n a l yz e d t h e i r t r a n s m e m b r a n e s t r u c t u r a l d o m a i n s ,c h r o m o s o m a l l o c a l i z a t i o n ,g e n e s t r u c t u r e ,m o t i f ,u p s t r e a mc i s -a c t i n g el e m e n t s ,c o v a r i a n c e a n d e x p r e s s i o n p r o f i l e s a n a l y s i s .A s a r e s u l t ,23T a H K T g e n e f a m i l y me m b e r sh a v eb e e n i d e n t if i e d ,a l l o fw h i c h e n c o d e d p r o t e i n s r a ng i n g f r o m443t o 590a m i n o a c i d s i n l e n g th ,wi t h 8.2t o 10.4i s o e l e c -t r i c p o i n t s ,a n d 6t o 8t r a n s m e m b r a n ed o m a i n s .T a H K T g e n e sw e r ed i s t r i b u t e do nc h r o m o s o m e s 2,4,6a n d 7o fw h e a t a n d c o u l db e d i v i d e d i n t o t h r e e s u b f a m i l i e s a c c o r d i n g t o t h e i r g e n e t i c p h y l o g e n y,a n dm e m b e r s o f t h e s a m e s u b f a m i l y h a d r e l a t i v e l y s i m i l a rm o t i f c o m p o s i t i o n a n d g e n e s t r u c t u r e .F o u r p a i r s o f t a n d e mr e p e a t g e n e sa n d10p a i r so f l a r g e f r a g m e n td u pl i c a t i o n g e n e sw i t h g o o dc o v a r i a n c ew e r e f o u n da m o n g t h e23g e n e f a m i l y m e m b e r s.A n a l y s i so f c i s-a c t i n g e l e m e n t s r e v e a l e dt h a tm o s t g e n e f a m i l y m e m b e r s c o n t a i n e ds a l t s t r e s s r e s p o n s i v ec i s-e l e m e n t s:MY B,G-b o x,A B R Ea n dD R E.E x p r e s s i o n p r o f i l e a n a l y s i s r e v e a l e d t h a tT a H K T g e n e sw e r e e x p r e s s e d i n a l l16t i s s u e s o fw h e a t,e s-p e c i a l l y i n r o o t s a n ds t e m s.T r a e s C S4D02G361300s h o w e d t h eh i g h e s t e x p r e s s i o n i nt h e r o o t.A f t e r s a l t s t r e s s t r e a t m e n t,t h e e x p r e s s i o n p a t t e r n s o f d i f f e r e n t f a m i l y m e m b e r s d i f f e r e d.T h e e x p r e s s i o n o f T r a e s C S7B02G318400w a s g r a d u a l l y d e c r e a s e d;T r a e s C S2B02G451400a n d T r a e s C S2D02G428200 w e r e a l s os i g n i f i c a n t l y d e c r e a s e d;T r a e s C S2B02G451800w a sh a r d l y e x p r e s s e d i nr o o t s,b u t t h ee x-p r e s s i o n g r a d u a l l y i n c r e a s e d,i n d i c a t i n g t h e y m a y p l a y e s s e n t i a l r o l e s i nw h e a t t o l e r a n c e t o s a l t s t r e s s. K e y w o r d s:W h e a t;T a H K T;B i o i n f o r m a t i c s;G e n e f a m i l y a n a l y s i s;S a l t s t r e s s土壤盐渍化已经成为全球重大的农业问题,威胁着农业㊁粮食和资源安全[1-2]㊂盐胁迫会造成植物离子失衡和渗透胁迫,影响其光合作用㊁蛋白质㊁脂质合成等代谢系统,过度的盐碱化甚至导致植株死亡,显著降低作物产量[3-7]㊂小麦(T r i t i c-u ma e s t i v u m L.)是世界上近三分之一人口的主食,盐胁迫严重影响其产量㊂研究小麦耐盐相关基因,了解其响应盐胁迫的分子调控机制,对小麦耐盐优良种质资源的筛选与利用具有重要的意义㊂高盐度通常以高N a+含量为主,当N a+浓度达到某个阈值时,会引发离子毒性[8]㊂K+可以调节钠离子的转移和运输,细胞和整个植株的N a+和K+比例与植物耐盐性关系密切[9]㊂高亲和性钾离子转运蛋白(h i g h-a f f i n i t y K+t r a n s p o r t e r, H K T)是植物体内非常重要的离子转运蛋白,在植物体应对盐胁迫的过程中具有关键作用[10]㊂H K T蛋白属于钾转运蛋白T r K超家族成员,具有典型的T r k H保守结构域[11]㊂H K T蛋白一般都含有8个跨膜结构域和4个保守孔状P-L o o p,每2个跨膜结构域和1个P-L o o p组成1个M P M跨膜基序,因此H K T蛋白共含有4个M P M跨膜基序[12]㊂H K T蛋白可以分为H K T1和H K T2两类,因为结构不同其功能有一定差异[13]㊂H K T1只介导转运N a+;H K T2既可以进行N a+-K+的协同转运,也可以进行N a+或K+的单向转运[9,14]㊂植物中的H K T1蛋白主要定位于根中柱木质部薄壁细胞的质膜[15],可以使N a+从木质部转运至其周围的薄壁细胞中,限制N a+向地上部分运输,以此来调节植物体内的N a+/K+比,使植物的地上部分在盐胁迫下也能维持低钠高钾,从而保证植物光合作用等生理活动的正常进行[9,16]㊂目前,在小麦中已发现3个耐盐主效Q T L,分别为N a x1㊁N a x2和K n a1[17],N a x1和N a x2为来源于一粒小麦中的T mHK T1;4和T mHK T1;5,K n a1为T a HK T1;5-D(编码H K T8),但T mHK T1;5定位于5A L染色体组, T a HK T1;5定位在4D L染色体组[14,18]㊂它们均可将N a+从木质部转运到周围薄壁细胞中,以减少小麦地上部N a+的含量,使叶片中保持低N a+/K+比㊂研究发现,在敲除小麦基因T a H-K T2;1后,其组织细胞内的N a+浓度明显降低,说明降低H K T2;1表达量可以减少N a+的摄入量[19]㊂上述研究结果表明,H K T在小麦抵御盐胁迫过程中发挥了至关重要的作用㊂H K T广泛存在于高等植物中㊂目前,只对棉花和水稻的H K T基因家族进行了全基因组水平的系统分析[20],而在小麦中除了T a HK T2;1和T a HK T1;5外,鲜有对H K T家族基因中其他成员的报道,更没有对小麦H K T家族基因在全基因组水平上的系统分析㊂本研究拟采用生物信息学的方法,在全基因组水平对小麦T a H K T家族基因进行系统分析,为进一步研究小麦T a H K T 家族基因的功能及筛选小麦耐盐T a H K T基因提供参考㊂1材料与方法1.1数据来源从E n s e m b l P l a n t s数据库中下载小麦(T r i t i c u ma e s t i v u m)㊁水稻(O r y z as a t i v a)㊁玉米(Z e am a y s)㊁拟南芥(A r a b i d o p s i s t h a l i a n a)㊁葡萄(V i t i s v i n i f e r a)㊁蓖麻(R i c i n u s c o mm u n i s)㊁甘薯(I p o m o e ab a t a t a s)和木龙葵(S o l a n u m s c a-b r u m)的相关数据,主要包括全基因组序列㊁C D S 序列㊁全蛋白质序列以及基因组注释信息㊂结合所查阅的文献在N C B I上搜索小麦H K T8蛋白的氨基酸序列;将搜索到的序列提交到HMM数㊃841㊃麦类作物学报第44卷据库中,得到H K T家族的P f a m号:P F02386;在P f a m数据库中下载其隐马尔可夫模型㊂1.2小麦T a H K T家族基因的鉴定根据H K T家族的隐马尔可夫模型进行HMM搜索,并进行C l u s t a l W多序列比对,创建小麦T a H K T家族基因特异性的隐马尔可夫模型,进行二次HMM搜索,筛选出E值<0.001的基因,将其对应的蛋白序列提交至P f a m㊁N C B I 和S MA R T三大数据库中进行再次确认,去除不含H K T结构域或可信度较低的成员,得到最终确认的小麦T a H K T基因家族成员㊂1.3H K T家族成员系统进化分析利用MA G A7软件中的C l u s t a l W对小麦㊁水稻㊁玉米㊁拟南芥㊁葡萄㊁蓖麻㊁甘薯及木龙葵8个物种H K T蛋白序列进行多序列比对;选择邻位归并法(N e i g h b o r-J o i n i n g)构建系统进化树, B o o t s t r a p校验参数为1000次,通过在线网站e v o l v i e w对进化树进行美化[21]㊂1.4小麦T a H K T家族基因序列分析使用M E M E-v4.12.0进行M o t i f分析,利用T B t o o l s软件进行绘图;提取1.2中得到的家族成员外显子㊁内含子㊁C D S和U T R的位置信息,使用G S D S9绘制基因结构图[22]㊂使用p e r l脚本对小麦中T a HK T蛋白的长度㊁等电点及分子量进行预测;在网站E x P A S y-P r o t P a r a m对其不稳定指数㊁脂肪系数及总亲水性指数进行预测[23];在网站C E L L O进行亚细胞定位预测;在D e e p T MHMM进行跨膜结构域预测㊂1.5小麦T a H K T基因染色体定位及共线性分析提取小麦T a H K T基因在染色体上的位置信息和小麦染色体的长度信息,保存为f a i格式文件,将整理好的数据提交到MA P C h a r t绘制T a H K T基因定位到染色体上的图㊂利用M C S-c a n X,在默认参数下分析T a H K T在小麦基因组中的串联重复基因;利用T B t o o l s分析小麦基因组内及小麦与粗山羊草㊁四倍体硬粒小麦㊁水稻和玉米之间的共线性并用C i r c o s绘制共线性图谱㊂提取上述串联重复基因对的C D S序列,进行C l u s t a l W比对,并计算每一对串联重复基因的非同义替换率(K a)和同义替换率(K s)比值进行选择压力分析[24],K a/K s>1㊁=1和<1分别表示正向选择㊁中性选择和纯化选择㊂1.6小麦T a H K T基因上游顺式作用元件分析从小麦基因组序列中提取T a H K T基因上游1500b p的启动子序列,利用在线网站P l a n t-C A R E分析顺式作用元件[25],再用G S D S9在线网站进行绘图㊂1.7小麦T a H K T基因在不同组织及盐胁迫下的表达分析在E x p V I P网站得到小麦T a HK T基因在不同组织中的表达情况;在G e n ev e s t i g a t o r软件中添加已确定的小麦T a H K T基因的I D,并对数据进行S a l t S t r e s s筛选;查找中国春(C h i n e s e S p r i n g)和青麦6号(Q i n g m a i6)H K T基因在盐胁迫下的表达情况,在T B t o o l s中绘制表达热图㊂2结果与分析2.1小麦T a H K T家族基因分析在小麦基因组中共鉴定到了23个T a H K T 基因家族成员,23个T a H K T蛋白序列长度为443~590a a;相对分子量为48.5~64.6K D a;理论等电点为8.20~10.40,即23个T a H K T蛋白均为碱性;总亲水性指数为0.120~0.496,表明均是疏水性蛋白;不稳定指数为25.74~45.29,其中不稳定指数小于40的蛋白有9个,属于稳定蛋白(表1)㊂亚细胞定位预测分析发现,23个T a H K T蛋白全部定位于质膜上,说明其发挥作用的部位可能为细胞质膜㊂跨膜结构域分析表明(图1),除了T r a e s C S2B02G451700(基因I D号,下同)编码的蛋白具有6个跨膜结构域㊁T r a e s C S2D02G428300等4个基因编码的蛋白具有7个跨膜结构域外, T r a e s C S2B02G451800等大多数基因编码的蛋白具有8个跨膜结构域,符合H K T蛋白的典型特征㊂2.2H K T家族成员系统进化分析采用邻位归并法构建了小麦与水稻㊁玉米㊁拟南芥㊁葡萄㊁蓖麻㊁甘薯及木龙葵8个物种H K T 基因家族的系统进化树(图2)㊂将8个物种共计42个H K T基因分为5个亚家族,基因T r a e s C S2B02G451700和T r a e s C S2D02G428500位于b r a n c h5,二者间的自展值为100,表明它们的同源性较高;基因T r a e s C S7D02G411300㊁T r a e s C S7A02G418600和T r a e s C S7B02G318800都位于b r a n c h1的末端,表明它们在进化中的亲缘关系较近,二者间的自展值为100,说明该分支的可信度也极高㊂在b r a n c h5中小麦基因T r a e s-C S6B02G182600㊁T r a e s C S6D02G144500与水稻基因O s02t0175000位于同一分支,表明二者之㊃941㊃第2期苏瑞平等:小麦T a H K T家族基因的生物信息学分析表1小麦T a H K T基因家族成员的基本信息T a b l e1B a s i c i n f o r m a t i o no fw h e a t T a H K T g e n e f a m i l y基因号G e n e I D染色体C h r o m o s o m e 基因位置G e n ep o s i t i o n蛋白长度P r o t e i nl e n g t h/a a分子量M o l e c u l a rw e i g h t/k D a等电点p I不稳定指数I n s t a b i l i t yi n d e x脂肪系数A l i p h a t i ci n d e x总亲水性指数G R A V Y亚细胞定位S u b c e l l u l a rl o c a t i o nT r a e s C S2A02G4306002A683567466~68357850958264.610.2441.9199.020.186P T r a e s C S2B02G4513002B644549026~64455350356362.410.0843.94100.090.222P T r a e s C S2B02G4514002B644844360~64484821157863.39.8943.1396.710.198P T r a e s C S2B02G4516002B645302238~64530603359064.410.0242.3496.390.179P T r a e s C S2B02G4517002B645307235~64531041048154.19.7045.2997.920.120P T r a e s C S2B02G4518002B645470305~64547446955661.110.1141.20101.730.272P T r a e s C S2D02G4282002D540062509~54006691056362.210.4044.34102.380.269P T r a e s C S2D02G4283002D540161984~54016607844348.69.1841.68103.660.298P T r a e s C S2D02G4284002D540190217~54019638244548.58.2040.3296.850.218P T r a e s C S2D02G4285002D540197396~54020083745751.39.2342.3096.240.139P T r a e s C S4B02G3708004B656815048~65681744451857.59.2232.89102.660.409P T r a e s C S4B02G3760004B659664756~65966689350456.28.3034.40103.210.386P T r a e s C S4D02G3613004D507965542~50796782251657.38.9129.75103.620.353P T r a e s C S6B02G1826006B204450429~20445266953259.99.1625.74103.680.269P T r a e s C S6D02G1445006D115043730~11504610053259.98.8129.26103.700.289P T r a e s C S7A02G4182007A609549041~60955080545750.38.3239.52109.370.416P T r a e s C S7A02G4185007A610433701~61043620954460.19.0339.91112.960.490P T r a e s C S7A02G4186007A610438735~61044110750856.08.9642.35109.780.449P T r a e s C S7B02G3184007B568488183~56849059854460.28.8340.37111.580.496P T r a e s C S7B02G3187007B568645776~56864779954460.29.0740.05112.650.492P T r a e s C S7B02G3188007B568650957~56865282150856.19.2941.52114.350.490P T r a e s C S7D02G4112007D530102460~53010515553158.88.9137.92111.900.496P T r a e s C S7D02G4113007D530108294~53011021850856.08.8738.93111.100.453P G R A V Y:G r a n d a v e r a g e o f h y d r o p a t h i c i t y;P:P l a s m am e m b r a n e.间的亲缘关系极近,可能由共同祖先进化而来㊂B r a n c h3中包括拟南芥㊁葡萄㊁甘薯㊁蓖麻和木龙葵的HK T基因,它们单独作为一个分支,说明其与小麦㊁玉米和水稻的亲缘关系较远㊂2.3小麦T a H K T基因序列分析T a H K T基因结构分析表明,除T r a e s C S2B-02G451800和T r a e s C S2B02G451700分别具有2个和4个外显子外,其余基因均有3个外显子(图3A)㊂对T a H K T蛋白进行保守基序分析,共发现10个M o t i f(图3B),M o t i f6㊁M o t i f4和M o t i f 7以三联体形式出现在所有蛋白中,极为保守㊂T r a e s C S2A02G430600㊁T r a e s C S2D02G428200㊁T r a e s C S2B02G451400和T r a e s C S2B02G451300具有完全相同的M o t i f,推测其具有相同功能㊂2.4小麦T a H K T基因染色体定位及共线性分析23个T a H K T基因分布于小麦的第2㊁4㊁6㊁7同源群染色体上(图4),其中第2同源群染色体上的数量最多(10个),在2B㊁2D等染色体上还发现了由多个基因聚集形成的基因簇,它们可能来自共同的祖先,并具有相同或相似的功能㊂除基因T r a e s C S6B02G182600和T r a e s C S6D02G144500位于染色体6B和6D的短臂,其他基因均位于染色体长臂靠近染色体末端的位置㊂小麦T a H K T基因间共线性分析显示(图5),有10对共线性基因(大片段复制基因),且在2号和7号染色体上共线性较好,其中位于2号染色体上的T r a e s C S2A02G430600㊁T r a e s C S2D02G428200㊁T r a e s-C S2B02G451300是三联体基因㊂此外,还发现了4对串联重复基因,K a/K s值均小于1,推测受纯化选择作用(表2)㊂为了解T a H K T基因的进化关系,分别分析了二倍体粗山羊草和四倍体硬粒小麦与六倍体小㊃051㊃麦类作物学报第44卷A :T r a e s C S 2B 02G 451700;B :T r a e sC S 2D 02G 428300;C :T r a e s C S 2B 02G 451800.图1 T a H K T 部分基因编码蛋白跨膜结构域预测分析F i g .1 A n a l y s i s o f t h e t r a n s m e m b r a n e d o m a i n p r e d i c t i o no f t h eT a H K T g e n e -e n c o d i n gpr o t e i ns A :小麦T a H K T 家族基因进化树;B :8个物种H K T 家族基因进化树;T r a e s :小麦;O s :水稻;Z m :玉米;A T :拟南芥;AMY :葡萄;B A S :甘薯;A X A :蓖麻;A L O :木龙葵㊂A :P h y l o g e n e t i c t r e e o fT a H K T g e n e f a m i l y ;B :P h y l o g e n e t i c t r e e o f t h eH K T g e n e f a m i l y f r o me i g h t s pe c i e s ;T r a e s :T r i t i c u ma e s -t i v u m ;O s :O r y z a s a t i v a ;Z m :Z e am a y s ;A T :A r a b i d o p s i s t h a l i a n a ;AMY :V i t i s v i n if e r a ;B A S :I po m o e ab a t a t a s ;A X A :R i c i n u s c o mm u n i s ;A L O :S o l a n u ms c a b r u m .图2 H K T 基因家族系统进化树F i g .2 P h y l o g e n e t i c t r e e o fH K T g e n e f a m i l y㊃151㊃第2期苏瑞平等:小麦T a H K T 家族基因的生物信息学分析A :基因结构分析;B :M o t i f 分析㊂A :G e n e s t r u c t u r e a n a l y s i s ;B :M o t i f a n a l ys i s .图3 T a H K T 基因结构和M o t i f 分析F i g .3G e n e s t r u c t u r e a n dm o t i f a n a l ys i s o fT a H KT 图4 小麦T a H K T 基因染色体定位F i g.4 C h r o m o s o m e l o c a t i o no fT a H K T g e n e s i nw h e a t 麦共线性关系及小麦与水稻和玉米之间的共线性关系(图6)㊂分别在二倍体粗山羊草㊁四倍体硬粒小麦和六倍体小麦中发现6㊁15和23个H K T 基因,且在2㊁4㊁6㊁7同源群染色体上具有较好的共线性㊂相比玉米,小麦与水稻间的共线性关系更好,说明二者的同源性较高㊂2.5 小麦T a H K T 基因上游顺式作用元件分析对小麦T a H K T 基因启动子序列分析表明,其1.5k b 上游区域含有多种顺式作用元件㊂对其与盐胁迫相关的顺式作用元件进行分析发现(图7),基因T r a e s C S 2A 02G 430600㊁T r a e s C S 2B 02G 451400和T r a e s C S 4B 02G 370800同时具有盐胁迫相关的㊃251㊃麦 类 作 物 学 报 第44卷图5小麦T a H K T基因的共线性F i g.5C o l l i n e a r i t y o fT a H K T g e n e s i nw h e a t表2T a H K T串联复制基因T a b l e2T a n d e md u p l i c a t i o n g e n e s o fT a H K T串联重复基因T a n d e md u p l i c a t i o n g e n e s K a K s K a/K s T r a e s C S2B02G451400&T r a e s C S2B02G4513000.0508890.2759130.184439 T r a e s C S2D02G428300&T r a e s C S2D02G4282000.0381070.2317270.164448 T r a e s C S7A02G418500&T r a e s C S7A02G4186000.3607361.4552100.247892 T r a e s C S7D02G411300&T r a e s C S7D02G4112000.5808252.0790100.2793764种顺式作用元件:受高盐㊁干旱胁迫诱导的顺式作用元件MY B和D R E,参与光反应和盐胁迫的顺式调节元件G-b o x,参与脱落酸反应的顺式作用元件A B R E;基因T r a e s C S2B02G451600和T r a e s C S2D02G428400除具有MY B㊁G-b o x㊁A B R E外还含有参与防御和应激反应的顺式作用元件T C-r i c h㊂顺式作用元件MY B㊁G-b o x和A B R E往往同时出现在同一基因上,推测这些基因对盐胁迫有响应㊂2.6小麦T a H K T基因在不同组织及盐胁迫下的表达分析23个小麦T a HK T基因在所选的16种组织中均有表达(图8),但表达量存在明显差异,表明这些成员在功能上存在一定分化㊂基因T r a e s C S4D02G361300主要在根(r o o t s)㊁叶轴(r a c h i s)㊁花梗(p e d u n c l e)和胚根(r a d i c l e)中表达,在根中的表达量最高;基因T r a e s C S7B02G318400主要在根(r o o t s)㊁叶鞘(l e a f s h e a t h)和胚根(r a d i c l e)中表达;基因T r a e s C S2B02G451800在各个组织中的表达量均较低;基因T r a e s C S2B02G451700在各个组织中均有较高表达,在根中的表达量最低㊂利用鉴定到的小麦T a H K T基因的I D在G e n e v e s t i g a t o r软件中得到了中国春和青麦6号在盐胁迫(150mm o l㊃L-1N a C l)处理不同时间下T a H K T基因在根组织中的表达热图(图9)㊂在盐胁迫下,T a H K T基因在两个小麦品种根部的表达模式相似,但青麦6号总体表达量略高于中国春㊂基因T r a e s C S7B02G318400在盐胁迫处理后表达量逐渐降低;基因T r a e s C S2B02G451400和㊃351㊃第2期苏瑞平等:小麦T a H K T家族基因的生物信息学分析T T :二倍体粗山羊草;T A :六倍体小麦;A E G :四倍体硬粒小麦;Z M :玉米;O S:水稻㊂T T :A o g i l o p s t a u s c h i i ;T A :T t i t i c u ma e s t i v u m ;A E G :T r i t i c u md u r u m D e s f .;Z M :Z e am a y s ;O S :O r yz a s a t i v a .图6 小麦T a H K T 基因与其他物种之间的共线性关系F i g .6 C o l l i n e a r i t y b e t w e e nw h e a t T a H K T g e n e s a n do t h e r s pe c i es 顺式作用元件用不同的彩色方框表示㊂D i f f e r e n t c o l o r e db o x e s i n d i c a t e d i f f e r e n t c i s -a c t i n g el e m e n t s i n t h e s c a l e a t b o t t o m.图7 小麦T a H K T 基因启动子区域与盐胁迫相关的顺式作用元件F i g .7 C i s -a c t i n g e l e m e n t s r e l a t e d t o s a l t s t r e s s i n t h e p r o m o t e r r e gi o no fw h e a t T a H K T g e n e s T r a e s C S 2D 02G 428200在盐胁迫处理后的表达量也有明显降低;基因T r a e s C S 2B 02G 451800在根部几乎不表达,但在受到盐胁迫处理后表达量逐渐提高,且在24h 时达到峰值㊂这些受到盐胁迫处理后表达量发生明显变化的基因,可能在小麦对盐胁迫的响应中发挥重要作用㊂3 讨论本研究在小麦基因组中鉴定出23个T a H -K T 基因,与G a r c i a d e b l ás 等[26]根据水稻HK T ㊃451㊃麦 类 作 物 学 报 第44卷R a d :胚根;C o l :胚芽鞘;S a :茎轴;R :根;L e :叶舌;P :花梗;S p :小穗;A :芒;G :颖片;L :叶;S c :种皮;F :旗叶;S h :幼苗;S t :茎;R a :叶轴;L s:叶鞘㊂R a d :R a d i c l e ;C o l :C o l e o p t i l e ;S a :S t e ma x i s ;R :R o o t ;L e :L e a f l i g u l e ;P :P e d u n c l e ;S p :S pi i k e l e t s ;A :A w n s ;G :G l u m e s ;L :L e a f ;S c :S e e d c o a t ;F :F l a gl e a f ;S h :S h o o t s ;S t :S t e m ;R a :R a c h i s ;L s :L e a f s h e a t h .图8 小麦T a H K T 基因家族成员在16种组织中的表达热图F i g .8 R e l a t i v e e x pr e s s i o n p r o f i l e s o fT a H K T g e n e s i n16t i s s u es C S :中国春;QM :青麦6号;c o n :对照;N a C l :N a C l 胁迫㊂C S :C h i n e s eS p r i n g ;QM :Q i n gm a i 6;c o n :C o n t r o l ;N a C l :N a C l s t r e s s .图9 T a H K T 基因在盐胁迫下小麦根中的表达模式F i g .9 R e l a t i v e e x pr e s s i o n p a t t e r n s o fT a H K T g e n e s i nw h e a t r o o t s u n d e r s a l t s t r e s s 基因家族推测的小麦中可能含有18个甚至更多个H K T 基因相符㊂亚细胞定位和跨膜结构域分析表明,小麦H K T 蛋白均定位在质膜上,表明该蛋白在质膜上起作用,且大多数H K T 蛋白具有8个跨膜结构域,符合H K T 蛋白的典型特征㊂对T a H K T 蛋白进行保守基序分析发现,M o t i f 6㊁M o t i f 4和M o t i f 7以及M o t i f 1和M o t i f 3在所有基因家族成员中有规律地出现,它们可能是与该家族基因功能密切相关的结构;系统进化树分析发现,在进化树上分支较近的家族成员具有相同或相似的M o t i f 组成和基因结构,推测分支较近的成员之间具有相同或相似的功能㊂㊃551㊃第2期苏瑞平等:小麦T a H K T 家族基因的生物信息学分析通过共线性分析,在基因家族成员中找到了10对共线性基因,4对串联重复基因,其K a/K s 值均小于1,表明均受纯化选择作用㊂在小麦与粗山羊草和四倍体硬粒小麦的共线性分析时发现,该基因家族在其2㊁4㊁6㊁7号染色体上共线性较好;在水稻中发现8个H K T基因,在玉米中发现3个H K T基因,且都与小麦中的H K T基因具有共线性,推测它们可能由共同的祖先进化而来㊂启动子区域的顺式作用元件在调控应激相关基因的表达中起着重要作用,且能够增加植物对非生物和生物胁迫的耐受性㊂转录因子通过与基因启动子区域中的顺式作用元件的特异性结合参与多种植物过程,包括生长㊁发育和胁迫信号传导㊂MY B转录因子结合顺式作用元件参与植物对高盐和干旱胁迫的应答;A B R E是介导A B A 依赖性信号传导的典型顺式作用元件;D R E是受高盐㊁低温和干旱胁迫诱导的顺式作用元件;G-b o x为参与光反应和盐胁迫的顺式调节元件[27-29]㊂通过分析T a H K T基因的顺式作用元件,可以进一步了解其调控机理及其可能参与的生理过程㊂本研究发现,小麦T a H K T基因启动子区域含有多种顺式作用元件,包括MY B㊁A B R E㊁D R E等响应激素和逆境胁迫元件(高盐㊁干旱),表明小麦T a H K T基因在参与逆境胁迫中可能发挥重要作用,也说明T a H K T基因的表达受多种因素调控㊂对不同小麦品种㊁不同组织部位中T a H K T 基因表达模式进行分析,发现基因T r a e s C S4D02G361300在根中表达量最高,且含有响应盐胁迫的顺式作用元件MY B㊁G-b o x和A B R E,说明其在根中的高表达可能与抵御盐胁迫有关㊂不过,在盐胁迫处理后,其表达量没有明显变化,表明其可能只是组织特异性基因而非诱导表达型基因㊂将其氨基酸序列提交到N C B I比对,发现T r a e s C S4D02G361300为已在小麦中鉴定到的T a HK T8(T a HK T1;5-D)[14,30]㊂基因T r a e s C S2B02G451800在各个组织中的表达量均较低,但受N a C l诱导表达,同源比对分析发现,其是已克隆的小麦T a HK T7(T a HK T1;4)㊂T a HK T7和T a HK T8是小麦中的两个主效耐盐基因,可在盐渍化条件下降低小麦叶片中N a+的积累[31],在盐胁迫条件下提高硬粒小麦产量[32]㊂T r a e s C S7B02G318400在根和叶鞘中表达量较高,而在叶中表达量较低,受到盐胁迫后表达量降低,经比对发现,该基因为从小麦中发现的第一个H K T基因T a HK T1(T a HK T2;1)[33],是根系中的高亲和N a+转运系统㊂这些研究结果表明,通过全基因组水平的基因家族鉴定及表达谱分析,可以很好地筛选响应某种环境条件的靶基因,预测基因的功能㊂基因T r a e s C S2B02G451700在各个组织中均有较高表达,在根中的表达量较低, T r a e s C S2B02G451700等其他家族成员在N C B I 上的记录多为根据基因组序列自动计算分析预测得到的,有待进一步实验证实㊂总之,本研究结果可为进一步研究小麦T a HK T基因的功能和作用机制提供参考㊂参考文献:[1]MU K HO P A D H Y A Y R,S A R K A RB,J A T HS,e t a l.S o i l s a-l i n i t y u n d e r c l i m a t e c h a n g e:C h a l l e n g e s f o r s u s t a i n a b l e a g r i c u l-t u r e a n d f o o ds e c u r i t y[J].J o u r n a l o f E n v i r o n m e n t a lM a n-a g e m e n t,2021,280:111736.[2]L I JG,P U LJ,H A N M F,e t a l.S o i l s a l i n i z a t i o nr e s e a r c h i nC h i n a:A d v a n c e s a n d p r o s p e c t s[J].J o u r n a l o f G e o g r a p h i c a l S c i e n c e s,2014,24:943[3]胡涛,张鸽香,郑福超,等.植物盐胁迫响应的研究进展[J].分子植物育种,2018,16(9):3006.HU T,Z H A N GGX,Z H E N GFC,e t a l.R e s e a r c h p r o g r e s s i n p l a n ts a l ts t r e s sr e s p o n s e[J].M o l e c u l a r P l a n t B r e e d i n g, 2018,16(9):3006.[4]L I A N G W J,MA X L,WA N P,e ta l.P l a n ts a l t-t o l e r a n c e m e c h a n i s m:Ar e v i e w[J].B i o c h e m i c a la n d B i o p h y s i c a lR e-s e a r c hC o mm u n i c a t i o n s,2018,495(1):287.[5]K A T I Y A R-A G A RWA LS,Z HUJ,K I M K,e t a l.T h e p l a s m a m e m b r a n eN a+/H+a n t i p o r t e r S O S1i n t e r a c t sw i t hR C D1a n d f u n c t i o n s i no x i d a t i v es t r e s st o l e r a n c ei n A r a b i d o p s i s[J].P r o c e e d i n g s o f t h eN a t i o n a lA c a d e m y o f S c i e n c e s o f t h eU-n i t e dS t a t e s o f Am e r i c a,2006,103(49):18816.[6]Z H A OSS,Z H A N GQK,L I U M Y,e t a l.R e g u l a t i o n o f p l a n t r e s p o n s e s t o s a l t s t r e s s[J].I n t e r n a t i o n a l J o u r n a l o f M o l e c u-l a rS c i e n c e s,2021,22(9):4609.[7]T A N V E E R M,S H A H A N.A n i n s i g h t i n t o s a l t s t r e s s t o l e r-a n c em e c h a n i s m s o f C h e n o p o d i u m a lb u m[J].E n v i r o n m e n t a l Sc i e n c e a n dP o l l u t i o nR e s e a r c h,2017,24(19):16531. [8]H A U S E R F,H O R I E T.Ac o n s e r v ed p r i m a r y s a l t t o le r a n c e m e c h a n i s m m e d i a t e db y H K Tt r a n s p o r t e r s:A m e c h a n i s mf o r s o d i u me x c l u s i o n a n dm a i n t e n a n c e o f h ig hK(+)/N a(+)r a-t i o i n l e a v e sd u r i n g s a l i n i t y s t r e s s[J].P l a n t,C e l l&E n v i-r o n m e n t,2010,33(4):553.[9]HAMAMO T OS,H O R I E T,H A U S E RF,e t a l.H K Tt r a n s-p o r t e r sm e d i a t e s a l t s t r e s s r e s i s t a n c e i n p l a n t s:F r o ms t r u c t u r e a n d f u n c t i o n t o t h e f i e l d[J].C u r r e n tO p i n i o n i nB i o t e c h n o l o-g y,2015,32:113.[10]M I A N A,O OM E N RJFJ,I S A Y E N K O VS,e t a l.O v e r-e x-㊃651㊃麦类作物学报第44卷p r e s s i o no f a n N a+-a n dK+-p e r m e a b l e H K Tt r a n s p o r t e r i n b a r l e y i m p r o v e s s a l t t o l e r a n c e[J].T h eP l a n tJ o u r n a l:f o rC e l l a n d M o l e c u l a rB i o l o g y,2011,68(3):469.[11]C O R R A T GÉ-F A I L L I E C,J A B N O U N E M,Z I MM E R-MA N NS,e ta l.P o t a s s i u m a n ds o d i u mt r a n s p o r t i nn o n-a n i m a l c e l l s:T h eT r k/K t r/H K Tt r a n s p o r t e r f a m i l y[J].C e l l u l a r a n d M o l e c u l a rL i f eS c i e n c e s,2010,67(15):2530.[12]王甜甜,郝怀庆,冯雪,等.植物H K T蛋白耐盐机制研究进展[J].植物学报,2018,53(5):710.WA N G T T,HA O H Q,F E N G X,e t a l.R e s e a r c ha d v a n c e s i n t h ef u n c t i o no f t h eh i g h-a f f i n i t y K+t r a n s p o r t e r(H K T) p r o t e i n s a n d p l a n ts a l tt o l e r a n c e[J].C h i n e s e B u l l e t i no fB o t a n y,2018,53(5):710.[13]R I E D E L S B E R G E R J,M I L L E R J K,V A L D E B E N I T O-MA T U R A N A B,e ta l.P l a n t H K Tc h a n n e l s:A nu p d a t e d v i e wo ns t r u c t u r e,f u n c t i o na n d g e n e r e g u l a t i o n[J].I n t e r-n a t i o n a l J o u r n a l o f M o l e c u l a r S c i e n c e s,2021,22(4):1892.[14]B Y R TCS,X UB,K R I S HN A N M,e t a l.T h eN a(+)t r a n s-p o r t e r,T a H K T1;5-D,l i m i t ss h o o tN a(+)a c c u m u l a t i o n i n b r e a dw h e a t[J].T h eP l a n tJ o u r n a l:f o rC e l la n d M o l e c u-l a rB i o l o g y,2014,80(3):517.[15]Z HA N GSC,T O N GYX,L IYJ,e t a l.G e n o m e-w i d e i d e n t i-f i c a t i o no f t h e HK Tg e n e s i nf i v eR o s a c e a es p e c i e sa n de x-p r e s s i o na n a l y s i s o f HK T g e n e s i n r e s p o n s e t o s a l t-s t r e s s i nF r a g a r i av e s c a[J].G e n e s&G e n o m i c s,2019,41(3):326.[16]MU N N S R,T E S T E R M.M e c h a n i s m so fs a l i n i t y t o l e r a n c e [J].A n n u a lR e v i e wo f P l a n tB i o l o g y,2008,59:651.[17]L IH Y,X U GZ,Y A N GC,e t a l.G e n o m e-w i d e i d e n t i f i c a t i o na n de x p r e s s i o na n a l y s i so f H K Tt r a n s c r i p t i o nf a c t o ru n d e r s a l t s t r e s s i n n i n e p l a n t s p e c i e s[J].E c o t o x i c o l o g y a n dE n v i-r o n m e n t a l S a f e t y,2019,171:435.[18]D A V E A,A G A RWA L P,A G A RWA L P K.M e c h a n i s m o fh i g ha f f i n i t y p o t a s s i u m t r a n s p o r t e r(H K T)t o w a r d si m-p r o v e d c r o pp r o d u c t i v i t y i ns a l i n ea g r i c u l t u r a l l a n d s[J].3B i o t e c h,2022,12(2):51.[19]A R I Y A R A T HN A H C K,U L-H A Q T,C O L M E R T D,e ta l.C h a r a c t e r i z a t i o no f t h em u l t i g e n e f a m i l y T a H K T2;1i nb r e a dw h e a t a n d t h e r o l e o f g e n em e m b e r s i n p l a n tN a+a n d K+s t a t u s[J].B M CP l a n tB i o l o g y,2014,14(1):159.[20]M I S H R AS,S I N G HB,P A N D AK,e t a l.A s s o c i a t i o n o f S N Ph a p l o t y p e s o fH K T f a m i l y g e n e sw i t h s a l t t o l e r a n c e i n I n d i a n w i l d r i c e g e r m p l a s m[J].R i c e,2016,9(1):15. [21]Z HA N GJ,Q IY,WA N G L,e t a l.G e n o m i c c o m p a r i s o na n d p o p u l a t i o nd i v e r s i t y a n a l y s i s p r o v i d e i n s i g h t s i n t o t h e d o m e s-t i c a t i o n a n d i m p r o v e m e n t o f f l a x[J].i S c i e n c e,2020,23(4): 100967.[22]HU B,J I NJ,G U O A Y,e t a l.G S D S2.0:A nu p g r a d e d g e n ef e a t u r ev i s u a l i z a t i o n s e r v e r[J].B i o i n f o r m a t i c s,2015,31(8):1296.[23]W I L K I N S M R,G A S T E I G E R E,B A I R O C H A,e ta l.P r o-t e i n i d e n t i f i c a t i o na n da n a l y s i st o o l s i nt h eE x P A S y s e r v e r [J].M e t h o d s i nM o l e c u l a rB i o l o g y,1999,112:536. [24]Z HA N GZ,L I J,Z HA O X Q,e t a l.K a K s_C a l c u l a t o r:C a l c u-l a t i n g k a a n d k s t h r o u g hm o d e l s e l e c t i o n a n dm o d e l a v e r a g i n g [J].G e n o m i c s,P r o t e o m i c s&B i o i n f o r m a t i c s,2006,4(4): 259.[25]L E S C O T M,DÉH A I SP,T H I J SG,e t a l.P l a n t C A R E,ad a-t a b a s e o f p l a n t c i s-a c t i n g r e g u l a t o r y e l e m e n t s a n d a p o r t a l t o t o o l s f o r i n s i l i c o a n a l y s i s o f p r o m o t e r s e q u e n c e s[J].N u c l e i cA c i d sR e s e a r c h,2002,30(1):326.[26]G A R C I A D E B LÁSB,S E N N M E,B AÑU E L O S M A,e t a l. S o d i u m t r a n s p o r ta n d H K T t r a n s p o r t e r s:T h er i c e m o d e l [J].T h eP l a n t J o u r n a l,2003,34(6):789.[27]E R P E NL,D E V IHS,G R O S S E RJW,e t a l.P o t e n t i a l u s e o f t h eD R E B/E R F,MY B,N A Ca n d WR K Yt r a n s c r i p t i o nf a c-t o r s t o i m p r o v e a b i o t i c a n db i o t i c s t r e s s i nt r a n s g e n i c p l a n t s [J].P l a n t C e l l,T i s s u ea n d O r g a n C u l t u r e(P C T O C), 2018,132(1):1.[28]X UZS,N IZ Y,L I U L,e ta l.C h a r a c t e r i z a t i o no f t h eT a-A I D F a g e n e e n c o d i n g aC R T/D R E-b i n d i n g f a c t o r r e s p o n s i v e t o d r o u g h t,h i g h-s a l t,a n d c o l d s t r e s s i nw h e a t[J].M o l e c u l a rG e n e t i c s a n dG e n o m i c s,2008,280(6):507.[29]MU K H E R J E E K,C H O U D HU R Y A R,G U P T A B,e ta l.A nAB R E-b i n d i n g f a c t o r,O S B Z8,i sh i g h l y e x p r e s s e d i ns a l t t o l e r a n t c u l t i v a r s t h a n i n s a l t s e n s i t i v e c u l t i v a r s o f i n d i c a r i c e [J].B M CP l a n tB i o l o g y,2006,6:18.[30]S C H A C H T MA N D P,S C H R O E D E R JI.S t r u c t u r ea n d t r a n s p o r t m e c h a n i s m o fa h i g h-a f f i n i t y p o t a s s i u m u p t a k e t r a n s p o r t e r f r o mh i g h e r p l a n t s[J].N a t u r e,1994,370:656.[31]X U B,WA T E R SS,B Y R TCS,e t a l.S t r u c t u r a l v a r i a t i o n s i n w h e a tH K T1;5u n d e r p i n d i f f e r e n c e s i nN a+t r a n s p o r t c a p a c-i t y[J].C e l l u l a ra n d M o l e c u l a rL i f eS c i e n c e s,2018,75(6): 1135.[32]J AM E SR A,B L A K E C,B Y R T CS,e ta l.M a j o r g e n e s f o r N a+e x c l u s i o n,N a x1a n dN a x2(w h e a tH K T1;4a n dH K T1;5),d e c r e a s eN a+a c c u m u l a t i o n i nb r e a dw h e a t l e a v e su n d e r s a l i n e a n dw a t e r l o g g e dc o n d i t i o n s[J].J o u r n a lo f E x p e r i-m e n t a lB o t a n y,2011,62(8):2940.[33]MU N N SR,J AM E SR A,X U B,e t a l.W h e a t g r a i n y i e l do n s a l i n e s o i l s i s i m p r o v e d b y a n a n c e s t r a lN a+t r a n s p o r t e r g e n e [J].N a t u r eB i o t e c h n o l o g y,2012,30:363.㊃751㊃第2期苏瑞平等:小麦T a H K T家族基因的生物信息学分析。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
论著胚胎发育相关基因stk40的蛋白表达和生物信息学分析崔骥 张军强 陈洁 朱丹丹 郭锡熔 童国庆摘要 目的 检测胚胎发育相关基因stk40的表达,并对其进行生物信息学分析。
方法 取小鼠早期各发育阶段的胚胎样本,用Western blot 方法检测stk40的蛋白表达。
用生物信息学软件或数据库分析预测stk40基因及其编码蛋白的基因结构、染色体定位、蛋白质理化性质、二级结构、疏水性/亲水性及结构域。
结果 证实stk40蛋白在小鼠8细胞发育阻滞胚胎中的表达显著低于发育正常的早期胚胎。
生物信息学分析显示,stk40基因mR NA 全长3877bp,开放阅读框长1350bp,编码449个氨基酸,相对分子质量50563 9;定位于染色体4D2 2区域,含13个外显子和12个内含子。
蛋白结构域分析提示,stk40基因编码蛋白存在一ST Y K c 结构域,可能与细胞有机体的发生有关。
结论 st k40基因编码蛋白的成功检测及生物信息学分析为进一步研究该基因奠定了基础。
关键词 胚胎发育;stk40基因中图分类号 Q132 文献标识码 A 文章编号 0253 3685(2011)05 0500 03Protein expression and bioinformatics analysis of stk40gene related to embryo development CU I Ji ,ZHANG Junqiang ,CHEN Jie ,et al.Fo rth Schoo l of Clinica l Medicine ,Nanjing Medical U niversity ,Nanjing 210004,CHI NAAbstract Objective T o detcet the pro tein ex pr essio n of stk40and analyze the bio info rmat ics of embry o development r elated g ene.Methods W estern blo t w as perfo rmed to detcet the protein ex pression of stk40in the ear ly developmental embr yo s of mo use.A n initial bioinfo rmatics ana lysis was perfor med o n its gene str ucture,g eno me lo ca lizatio n,the physical and chemical characterist ics of its coding pro tein,secondar y str ucture,hydrophobicity /hydr ophilicity,st ructur al domain and so on.Results I t was demonstrated that the prot ein ex pression o f stk40w as low er in t he development arr ested 8 cell embryo s than that in t he no rmal ones.Bio informat ics analysis sho wed that stk40g ene was a 3877bp mRN A,containing 1350nucleotides o f an o pen r eading fr ame predicting 449amino acids with a molecular mass o f 50563.9.NCBI M ap View er ana lysis rev ea led that the stk40gene w as located on chromo so me 4D2 2and w as composed o f 13exo ns and 12intro ns.T he stk40had a ST Y K c do main related to emerg ence of cellular o rganisms.Conclusion T he detcetion and analysis of stk40gene may pro vide foundatio n and no vel info rmation for the furt her st udy.Key words Embryo dev elo pment;stk40Gene[Jia ngsu M ed J ,Ma rch 2011,37(5):500 502.]基金项目:江苏省自然科学基金项目(BK2009047);南京医科大学科技发展基金重点项目(07NM U Z026);南京市卫生局项目(ZKX08006)作者单位:210004 南京医科大学第四临床医学院(崔骥、朱丹丹);南京医科大学附属南京妇幼保健院(张军强、陈洁、郭锡熔、童国庆)责任作者:童国庆 E mail:tongguoqing@发育阻滞现象是哺乳动物早期胚胎在体外培养过程中表现出的重要特征之一。
大约有不到50%的胚胎在体外受精后可以发育到囊胚阶段[1],而这些发育到囊胚阶段的胚胎在移植入子宫后大部分不能继续种植发育[2]。
胚胎发育过程与多种因素有关,包括氧自由基的损害[3]和培养液成分的平衡等外界因素,胚胎自身的基因调控等内在因素[4],而胚胎自身基因调控的研究近年来备受关注。
迄今,有关早期胚胎发育调控的关键转录因子已有一些报道,如Oct4、So x2和Nanog 可共同组成调控网络,调控胚胎的早期发育[5]。
然而,目前发现的基因不能完全诠释胚胎发育阻滞的机制,因此,继续寻找与发现新的、调控早期胚胎发育的关键转录因子,并对其调控机制进行研究仍十分必要。
本研究试图从小鼠早期胚胎中检测出该基因的蛋白表达,并对该基因进行生物信息学分析,为其功能研究提供有用的线索和依据。
材料与方法一、实验材料ICR 小鼠购自上海斯莱克公司,孕马血清(PM SG)和绒毛膜促性腺激素(H CG)购自宁波激素厂;Western blot 所用试剂购自上海生工生物工程有限公司;基因生物技术信息学分析软件,源于美国国立生物技术信息中心(NCBI)核酸及蛋白数据库和网上其他生物数据库。
二、实验方法1 小鼠超排与收集胚胎 挑选雌性小鼠于下午16:30~18:00间,腹腔注射PMSG(10IU /只),48~50h 后腹腔注射H CG(10IU/只),当晚将雌鼠与成年雄鼠1 1合笼交配,次日晨8:00看阴栓。
有阴栓的雌鼠于受精后1 5、2 5、4 5d 处死,将2细胞、4~8细胞和囊胚取出,-80 保存待用;另取部分1 5d 的2细胞胚胎,在体外进行培养,将发育阻滞的各细胞期胚胎收集,-80 保存待用。
2 Western blo t 分析 (1)凝胶制备:安装电泳槽装置,根据需要量配置分离胶和浓缩胶,将加样梳放入,室温30~60min 待胶凝固后上样电泳;(2)上样:将蛋白质标本中加入2 lo ading Buffer 煮沸5~10min,12000r/min 离心后加样;(3)电泳:浓缩胶10m A 恒流电泳至交界处,分离胶20mA 恒流电泳至溴酚蓝到达凝胶下缘;(4)其他:半干转膜仪转膜;PBS 洗2遍,封闭1h;将硝纤膜置于抗体稀释液中,4 摇床孵育过夜;PBS 洗3遍,加入二抗稀释液,室温孵育2h;PBS 洗3遍,化学发光试剂显示条带,FluorChem 5500,Alpha Innotech 捕获图像。
3 生物信息学分析 利用Spidey 、m eg aBlast 等工具在线分析stk40基因核苷酸序列的基本特征;分析蛋白质理化性质采用ProtPar am 在线工具;运用ProtScale 工具完成疏水性/亲水性的预测;应用SOPMA 在线工具完成蛋白质二级结构的预测;利用SM ART 工具进行结构域分析。
结果一、stk40在小鼠早期胚胎各阶段中的表达取小鼠各个阶段的早期胚胎,用stk40蛋白抗体进行Western blo t 实验。
结果证实stk40蛋白在8细胞发育阻滞胚胎中的表达显著低于发育正常的早期胚胎(图1)。
二、生物信息学分析stk40基因在GenBank 中有2个转录本(NM _001145827 1、N M_028800 3),转录本2在5 端非1 发育阻滞2 正常对照图1 s tk40低表达于发育阻滞的8细胞胚胎中编码区与转录本1不同,缺少一段5 端编码的区域,并且使用下游翻译启动子。
2个转录本编码两个不同的蛋白(NP_001139299 1和NP_083076 3),其中转录本1(NM _001145827 1)最为常见,mRNA 长3877bp,开放阅读框511~1860bp,编码449个氨基酸。
应用Spidey 工具,将stk40基因mRNA 序列与小鼠基因组序列进行比对,结果显示stk40基因含13个外显子和12个内含子,内含子均以GT 开始,以AG 结束,遵守GT AG 规律。
检索NCBI SNP 数据库发现stk40基因共有2个SNP 位点已被鉴定,N CBI 登陆号分别为rs27528762、rs27528755,均为同义突变,其临床意义均未见报道。
运用ProtPar am 工具对stk40基因的蛋白序列进行分析,结果显示其物理、化学参数如下:其编码蛋白理论相对分子质量50563 9;理论等电点8 30;氨基酸组成Ala 6 9%,Ar g 7 3%,A rg 7 3%,Asp 6 0%,Cys 1 8%,Gln 5 6%,Glu 6 7%,Gly 6 2%,H is 2 9%,Ile 4 7%,Leu 11 6%,Ly s 6 0%,Met 2 7%,Phe 2 7%,Pro 4 2%,Ser 7 6%,Thr 4 5%,Trp 0 7%,Tyr 2 9%,Val 6 7%;原子组成C 2221H 3582N 640O 667S 20;半衰期30h;不稳定系数48 82;总平均疏水性-0 385。
应用NCBI 在线megaBlast 分析工具,将stk40基因mRNA 序列与小鼠基因组数据库进行序列比对分析,结果发现stk40基因定位于染色体4D2 2区域。
蛋白质的亲、疏水性的相互作用是维持蛋白质三级结构最重要的作用力之一。
运用Pro tScale 预测stk40氨基酸序列的疏水性/亲水性,结果表明多肽链第97位的Glu 具有最低的分值-3 011,第155位的Leu 具有最高的分值2 678。
