怎样查找一个基因的启动子区域
确定启动子位置的方法(一)

确定启动子位置的方法(一)确定启动子位置引言启动子是指基因的一个特殊区域,它在基因转录过程中起着重要的作用。
确定启动子位置是基因组学研究中的一项重要任务,对于深入理解基因的调控机制有着非常重要的意义。
本文将介绍几种常见的方法来确定启动子位置。
1.实验方法5’ RACE5’ RACE (Rapid Amplification of cDNA Ends) 是一种常用的实验方法,用于确定基因的启动子位置。
该方法通过引物扩增方法,在未知启动子区域的5’端合成一条cDNA链,并通过PCR扩增获得启动子序列。
5’ RACE在启动子区域进行测序,可获得启动子的精确位置。
Chromatin Immunoprecipitation (ChIP)ChIP是一种通过抗体和染色质上的特定蛋白结合来确定启动子位置的方法。
该方法首先通过交联和剪切处理来固定染色质上的蛋白质-DNA复合物,然后使用特定的抗体来免疫沉淀(IP)所要分析的蛋白质,最后通过PCR或测序来检测与启动子相关的DNA序列。
2.计算方法基于序列保守性的方法基于序列保守性的方法通过比对物种间的基因组序列来确定启动子位置。
这种方法假设启动子处的序列在不同物种间具有高度的保守性,因此可以通过比对序列中的保守区域来确定启动子的位置。
基于转录因子结合位点的方法许多转录因子结合在启动子区域,因此基于转录因子结合位点的方法可以帮助确定启动子位置。
通过分析转录因子结合位点的分布情况,并结合表观遗传学修饰等信息,可以预测启动子的位置。
基于表达谱和转录本结构的方法基于表达谱和转录本结构的方法可以通过分析基因的表达谱和转录本结构来确定启动子位置。
这种方法假设在基因的表达谱和转录本结构中存在着与启动子相关的特征,通过分析这些特征可以推断出启动子的位置。
总结确定启动子位置是基因组学研究中的一项重要任务。
本文介绍了几种常见的方法,包括实验方法和计算方法。
实验方法包括5’ RACE 和ChIP等,而计算方法则包括基于序列保守性、转录因子结合位点和表达谱转录本结构等方法。
干货7个步骤教你找到启动子

干货7个步骤教你找到启动子
作者:解螺旋·子非鱼
如需转载请注明来源:解螺旋·医生科研助手
导语
看到一大串密码一样的序列,要怎么找出启动子呢?其实很简单,也就7步,跟着小鱼做就行。
师弟对着电脑上的一大串序列发呆,小鱼问道,“师弟,你这是在格物致知吗?”
“没有啦,我在想怎么把这个基因启动子找出来。
”
“试试用Map viewer吧!”
下面小鱼就以人的K-RAS基因为例讲述一下找基因启动子序列的具体操作步骤:
1.打开NCBI的Map viewer页面,
/mapview/index.html
2.点击“GO”出现如下页面:
3.出现下图,RAS参考序列给出了两个,序列有微小的差异,但总体来说基本相同。
现在普遍采用的是“reference”那个序列。
4.点击上述两条序列第一条序列(即12 reference)对应的“Genes seq”,出现新的页面,点击下图出现的“Download/ViewSequence/Evidence ”,即可下载查看序列等功能。
5.出现的页面提示K-ras基因在染色体上的位置:
6.因为启动子一般在-2000~+200区域,把页面中的参数修改一下:
7.那么就得到K-ras的启动子区域,如下图:。
一文教会你查找基因的启动子、UTR、TSS等区域以及预测转录因子结合位点

一文教会你查找基因的启动子、UTR、TSS等区域以及预测转录因子结合位点基础知识首先我们了解一些基础知识(注:文中图片皆可点击放大查看!):启动子(promoter):与RNA聚合酶结合并能起始mRNA合成的序列。
做生信分析时,一般选择上游1 kb,下游 500 nt,也有选上下游各1 kb的。
如果关注核心启动子,可见生信宝典之前发布的Jaspar数据库介绍。
获取正链或负链的启动子序列时要注意方向。
之前awk的教程中有些提及。
转录起始点(TSS):转录时,mRNA链第一个核苷酸相对应DNA链上的碱基,通常为一个嘌呤。
UTR(Untranslated Regions):即非翻译区,是信使RNA (mRNA)分子编码区(CDS)两端的非编码片段。
5’-UTR从mRNA起点的甲基化鸟嘌呤核苷酸帽延伸至AUG起始密码子,3’-UTR从编码区末端的终止密码子延伸至多聚A尾巴(Poly-A)的末端。
生信老司机以中心法则为主线讲解组学技术的应用和生信分析心得- 限时免费中讲述了如何基于高通量数据对这些区域的调节变化进行分析,可配合此文观看。
1. 查找基因的启动子区域-NCBI1. 打开PubMed:/pubmed2. 选择Gene,输入IL17A,点击search,结果如下图,点击第一个:3. 下拉到下图位置,可以看到该基因的以下信息:点击Tools,选择Sequence Text View:还可以看到如下序列信息:4. 以上只是该基因的一些信息,可以用于查找相应的UTR等区域,下面进入正题,寻找promoter区域。
还是拉到如下图位置,点击FASTA:5. 基因位置信息如下图:6. 一般认为基因上游2 kb区域为该基因的promoter区域,所以将基因上游2 kb序列调出来:7. 复制上述序列就是基因的启动子序列了。
2. 查找基因的启动子区域-UCSC1. 打开UCSC:/,点击Table Browser:2. 按照下图所示填好基因相关信息,点击get output:3.选择genomic:4. 勾选Promoter/Upstream by选项,并将其改为2000 bases,然后点击get sequence:5. 得到下面的序列信息,开头直到第一个大写字母前面的所有小写字母序列即为该基因的promoter序列,你可以跟NCBI上得到的序列比对一下,看看是不是一样的呢?3. 转录因子结合位点的预测1.后面的预测步骤是改版前的Jaspar,可见上一篇介绍Jaspar的文章学习在新版Jaspar中怎么预测启动子区域的转录因子结合位点。
如何找一个基因的启动子序列呢

如何找一个基因的启动子序列呢一个基因的启动子序列是一个基因组区域,位于基因的上游,并能够识别和结合转录因子,调控基因的转录活性。
寻找一个基因的启动子序列可以通过多种方法和技术来进行。
1. 基因组数据挖掘:最简单的方法是使用公开的基因组数据库,例如Ensembl、NCBI等,使用基因名或序列信息目标基因,并获取其序列信息。
这些数据库通常会提供基因的起始位置和上游区域的信息。
2.序列比对和多序列比较:如果基因组数据库中没有目标基因的启动子序列信息,可以通过对已知相关物种的基因组进行序列比对来获取启动子序列。
过去研究或其他相关文献中可能已经报道了该基因位点的启动子信息,可以通过多序列比较来找到高度保守的区域进行分析。
3.实验方法:寻找基因的启动子序列也可以通过实验方法来进行。
以下是几个常用的实验方法:-基因克隆:通过PCR扩增目标基因的上游区域,然后将PCR产物克隆到适当的载体中进行测序。
从测序结果中截取相应的序列作为启动子序列。
- 5' RACE(Rapid Amplification of cDNA Ends):通过5' RACE技术,可以找到目标基因的转录起始位点,从而确定启动子序列。
这种方法从mRNA上游端引导逆转录聚合酶链式反应(RT-PCR),然后再通过测序获取启动子序列。
-转录组学方法:RNA测序和转录组学方法可以检测到基因的转录产物,从而很大程度上能够帮助确定启动子序列。
RNA测序可以生成从基因的5'端到3'端的转录产物的序列信息,因此可以利用这些数据来识别基因的启动子区域。
4. 计算方法:计算方法可以利用一些生物学特征或机器学习算法来预测基因的启动子序列。
例如,启动子序列通常富含一些特定的DNA序列模式,如TATA box、CAAT box和GC box等。
利用这些DNA序列模式的分布和相互作用关系,可以预测和确定基因的启动子区域。
在寻找基因的启动子序列时,需要根据研究目的选择适当的方法。
如何找一个基因的启动子序列呢?
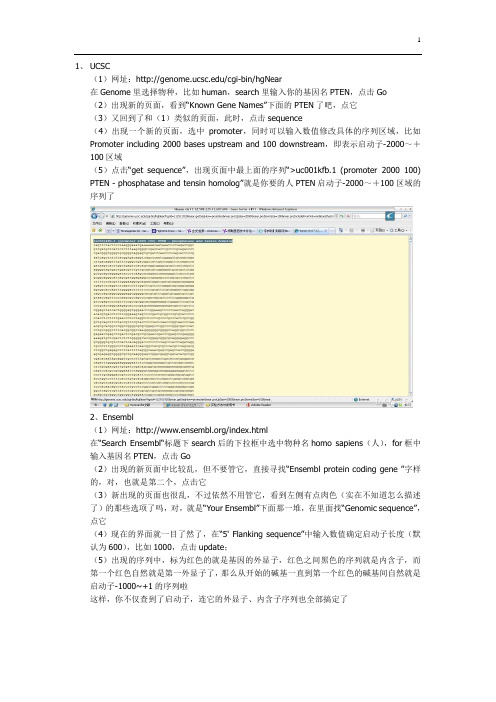
1、UCSC(1)网址:/cgi-bin/hgNear在Genome里选择物种,比如human,search里输入你的基因名PTEN,点击Go(2)出现新的页面,看到“Known Gene Names”下面的PTEN了吧,点它(3)又回到了和(1)类似的页面,此时,点击sequence(4)出现一个新的页面,选中promoter,同时可以输入数值修改具体的序列区域,比如Promoter including 2000 bases upstream and 100 downstream,即表示启动子-2000~+100区域(5)点击“get sequence”,出现页面中最上面的序列“>uc001kfb.1 (promoter 2000 100) PTEN - phosphatase and tensin homolog”就是你要的人PTEN启动子-2000~+100区域的序列了2、Ensembl(1)网址:/index.html在“Search Ensembl“标题下search后的下拉框中选中物种名homo sapiens(人),for框中输入基因名PTEN,点击Go(2)出现的新页面中比较乱,但不要管它,直接寻找“Ensembl protein coding gene ”字样的,对,也就是第二个,点击它(3)新出现的页面也很乱,不过依然不用管它,看到左侧有点肉色(实在不知道怎么描述了)的那些选项了吗,对,就是“Your Ensembl”下面那一堆,在里面找“Genomic sequence”,点它(4)现在的界面就一目了然了,在“5' Flanking sequence”中输入数值确定启动子长度(默认为600),比如1000,点击update;(5)出现的序列中,标为红色的就是基因的外显子,红色之间黑色的序列就是内含子,而第一个红色自然就是第一外显子了,那么从开始的碱基一直到第一个红色的碱基间自然就是启动子-1000~+1的序列啦这样,你不仅查到了启动子,连它的外显子、内含子序列也全部搞定了3、SIB-EPD(1)网址:http://www.epd.isb-sib.ch/(2)具体使用方法大同小异,就是输入物种名、基因名,限定启动子序列区域不过有了前两个,我想已经足够用了,个人感觉SIB-EPD的库容量太小,很多基因查不到我以前回的贴,总结一下ensembl一般也和NCBI的一致,你的情况可能例外。
使用UCSC查找基因启动子区域方法

应用UCSC查找基因启动子(promoter)、内含子、外显子序列(2015)本文原文来源于网络,因网站相关网页有所变化,特改写原文,望对同同仁有所帮助。
启动子的甲基化,转录因子与启动子的结合调控基因的表达等研究领域一直较为热门。
本文图文形式讲解了启动子的概念,利用UCSC如何查找一个基因的启动子序列,以及外显子和内含子序列的显示。
在讲述某个基因的启动子查询之间,我们有必要对基础知识进行一下复习和总结。
先看一下中心法则:启动子是在DNA转录为RNA这一步过程中发挥作用的,在此要与DNA自身复制起始点(称作复制子)和由mRNA翻译为蛋白质时的翻译起始点(以起始密码子ATG为标志)区别开来。
定义:启动子是参与特定基因转录及其调控的DNA序列。
包含核心启动子区域和调控区域。
核心启动子区域产生基础水平的转录,调控区域能够对不同的环境条件作出应答,对基因的表达水平做出相应的调节。
启动子是RNA聚合酶特异性识别和结合的部位。
启动子方向性,位于转录起始点上游,本身并不被转录。
DNA链上与RNA链的第一个核苷酸对应的碱基标记为+1(如下图),由此碱基向上游(5’端)数的碱基顺序数为负(-1,-2,……),向下游(3’端)数的碱基为正(+2,+3,……)区域:启动子的范围非常大,可以包含转录起始位点上游2000bp,有些特定基因的转录区内部也存在着转录因子的结合位点,因此也属于启动子范围。
总结起来,也就是说启动子约在与mRNA所对应的DNA序列之前约2000个左右的碱基。
明白了启动子的含义之后,我们以人(Human)的NDRG4(N-MycDownstream- Regulated Gene 4)为例,应用UCSC基因组浏览器开始查找该基因的启动子序列。
网址/index.html。
进入UCSC的主页后,在其左侧(如上图)点击第一项GenomeBrowser,进入基因组浏览器入口,如下图按照上图中的下拉选项,选择Mammal下的Human,assembly选择最新,search term 处填写需要查找的基因名称。
如何查找一个基因的启动子序列

如何查找一个基因的启动子序列发表者:刘小丰 (访问人次:6102)刘小丰收集整理定义:启动子是参与特定基因转录及其调控的DNA序列。
包含核心启动子区域和调控区域。
核心启动子区域产生基础水平的转录,调控区域能够对不同的环境条件作出应答,对基因的表达水平做出相应的调节。
区域:启动子的范围非常大,可以包含转录起始位点上游2000bp,有些特定基因的转录区内部也存在着转录因子的结合位点,因此也属于启动子范围。
这项搜寻要从UCSC基因组浏览器开始,网址为/cgi-bin/hgGateway。
以编码pendrin (PDS)的基因为例来说明上述问题。
PDS与耳蜗的异常发育、感觉神经性听力下降以及弥散性甲状腺增大(甲状腺肿)有关。
进入UCSC的主页后,在Organism的下拉菜单中选择Human,然后点击Browser。
使用者现在到了人类基因组浏览器入口。
本例的搜寻很简单:在assembly的下拉菜单中选择Dec. 2001,在position框中键入pendrin,然后点击Submit。
返回的页面结果显示一个已知的基因和两个mRNA序列。
继续点击mRNA序列的登录号AF030880,出现包含这个mRNA区域的图解概要。
为了获得这个区域更清晰的图像,点击紧靠zoom out的1.5X按钮。
最后点击页面中部的reset all按钮,使各个路径的设置恢复默认状态。
然而,对于本例的搜寻目的来说,默认设置不是理想的设置。
按照视图利用页面底部的Track Controls按纽,将一些路径设置为hide模式(即不显示),其他设置为dense模式(所有资料密集在一条直线上);另一些路径设置为full模式(每个特征有一个分开的线条,最多达300)。
在考虑这些路径内究竟存在那些资料之前,对这些路径的内容和表现做一个简要的讨论是必要的,许多这些讨论是由外界提供给UCSC的。
下面是对基因预测方法的更进一步讨论,这些信息也可以在其他地方找到。
NCBI查基因 编码区 启动子区 外显子
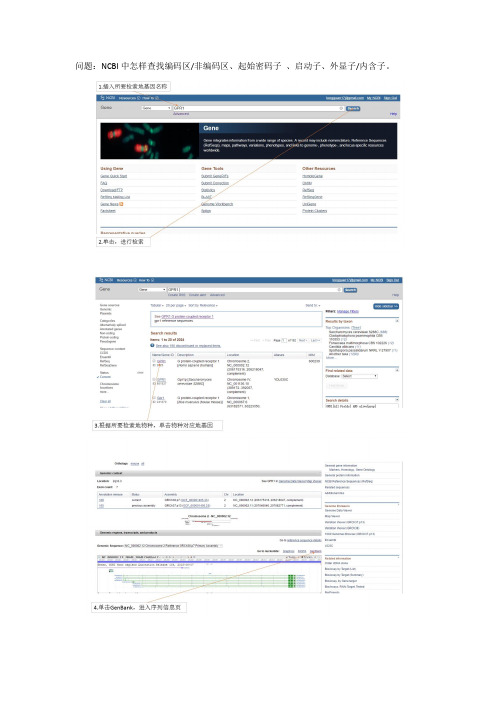
问题:NCBI中怎样查找编码区/非编码区、起始密码子、启动子、外显子/内含子。
启动子
一般定义启动子,都是upstream 1000bp,downstream1000bp的那段序列。
或者根据你的实验。
你去ensemble,输入基因,找到exon,点开,在configuration里面选好flank多少bp 的序列,选好之后自动刷新,就出来了。
在序列里面,ensemble用不同的颜色标出来不同区域,5‘UTR之类的,还有exon,intron,转录起始位点等等,flank的区域就是你选的promoter 了。
开放阅读框
在分子生物学中,开放阅读框(Open Reading Frame, ORF)从起始密码子开始,是DNA序列中具有编码蛋白质潜能,一段无终止密码子打断的碱基序列。
