Mega的使用以及进化树的绘制

1.MEGA构建系统进化树的步骤
2.CLUSTALX进行序列比对
1.MEGA构建系统进化树的步骤
1. 将要用于构建系统进化树的所有序列合并到同一个fasta格式文件,注意:所有序列的方向都要保持一致( 5’-3’)。如图:
2. 打开MEGA软件,选择"Alignment" - "Alignment Explorer/CLUSTAL",在对话框中选择Retrieve sequences from a file, 然后点OK,找到准备好的序列文件并打开,如图:
。
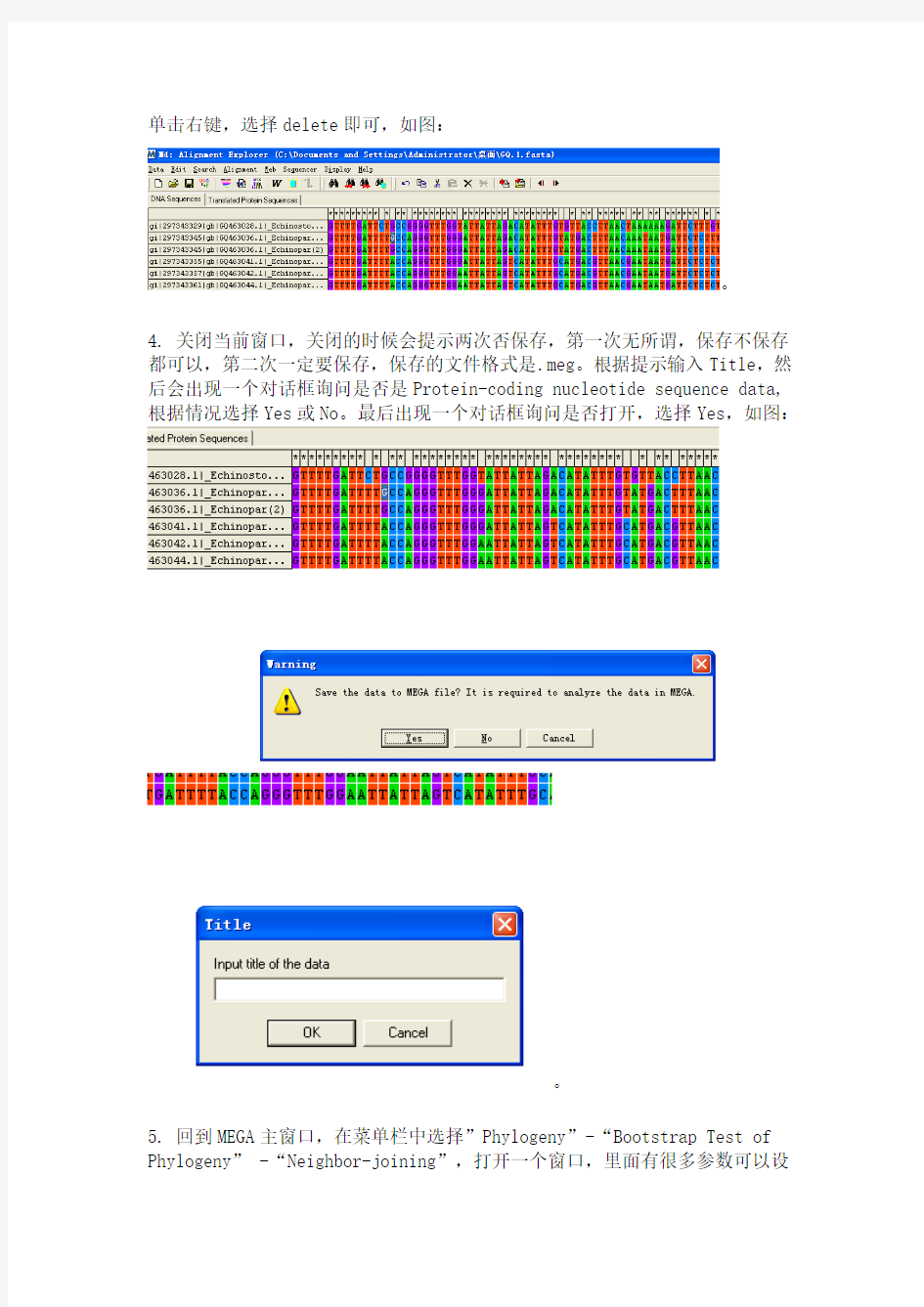
3. 在打开的窗口中选择”Alignment”-“Align by ClustalX” 进行对齐,对齐过程需要一段时间,对齐完成后,最好将序列两端切齐,选择两端不齐的部分,
单击右键,选择delete即可,如图:
。
4. 关闭当前窗口,关闭的时候会提示两次否保存,第一次无所谓,保存不保存都可以,第二次一定要保存,保存的文件格式是.meg。根据提示输入Title,然后会出现一个对话框询问是否是Protein-coding nucleotide sequence data, 根据情况选择Yes或No。最后出现一个对话框询问是否打开,选择Yes,如图:
。
5. 回到MEGA主窗口,在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” -“Neighbor-joining”,打开一个窗口,里面有很多参数可以设
置,如何设置这些参数请参考详细的MEGA说明书,不会设置就暂且使用默认值,不要修改,点击下面的Compute按钮,系统进化树就画出来了,如图:
在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“Minimun-evolution”,如图:
在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“Maximun-parsimony”,如图:
在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“UPGMA”,
如图:
6. 最后,使用TreeExplorer窗口中提供的一些功能可以对生成的系统进化树进行调整和美化。
CLUSTALX进行序列比对
1.将下载的序列放入一个Text文本文档中,序列按一定的格式,
>pig AGAGACGGCCGCATCTTCTTGTGCAGTGCCAGCCTCGTCCCGTA GACAAAATGGTGAAGGTCGGTGTGAACGGATTTGGCCGTATTG GGCGCCTGGTCACCAGGGCTGCCATTTGCAGTGGCAAAGTGGA GATTGTTGCCATCAA的格式复制过来,放在一个文本文档里。
新建文本文档.txt
注意:CLustal 1.83分析出了全序列比对,彩色比对区上门的*越多,表示这段序列越保守
2.在File--load sequence载入序列,如图所示
4.点击align
5.
在桌面上即可生成aln格式的文本文档(用于下面Mega5.02进行进化树构建)
Mega5.02序列比对及建进化树
序列比对
1.用文本格式的序列数据进行比对
2.Align---edit alignment即新建一个数据---0k---DNA
3.
Edit--insert sequence from file ---选择文本文档
4.Alignent---Align by ciust W--ok---关闭序列比对--并保存在桌面上---Phylogeny--MAX
5.两种不同的方法最大释然和邻近法
邻近法更准确
建进化树1.File--convert file format to mega
2.
3.点击文件夹从桌面载入aln格式的文本文档
4.
5.点击OK,再命名
6.
7.
8.关闭窗口
9.点击OK
10.phylogen--text neighbor -joining tree -或者Maximum的方式进行构建--从桌面上选择刚命名的文件
11.
点击打开
12.
13.
14.点击Yes
15.
16.
17.将Test phylogery中的None改成如图
18.
19.
20.进化树就构建成功--点击横线进行细节修改
21.
22.点击左边第五个蓝色的图标
进行细节修改
23.可以双击分类后的名称,进行名称修改,如
Primer 5设计引物
1.File---New--DNA sequence
2.将序列复制过来(序列的格式必须是文本格式)--as is --OK
3.点击Primer
4.点击S--File-perferences
Length设置为20
点击OK
6.Search
7.type--both-PCR size 100-1000--primer length 25-5--OK
、
点击OK
Mega的使用以及进化树的绘制
1.MEGA构建系统进化树的步骤 2.CLUSTALX进行序列比对 1.MEGA构建系统进化树的步骤 1. 将要用于构建系统进化树的所有序列合并到同一个fasta格式文件,注意:所有序列的方向都要保持一致( 5’-3’)。如图: 2. 打开MEGA软件,选择"Alignment" - "Alignment Explorer/CLUSTAL",在对话框中选择Retrieve sequences from a file, 然后点OK,找到准备好的序列文件并打开,如图: 。 3. 在打开的窗口中选择”Alignment”-“Align by ClustalX” 进行对齐,对齐过程需要一段时间,对齐完成后,最好将序列两端切齐,选择两端不齐的部分,
单击右键,选择delete即可,如图: 。 4. 关闭当前窗口,关闭的时候会提示两次否保存,第一次无所谓,保存不保存都可以,第二次一定要保存,保存的文件格式是.meg。根据提示输入Title,然后会出现一个对话框询问是否是Protein-coding nucleotide sequence data, 根据情况选择Yes或No。最后出现一个对话框询问是否打开,选择Yes,如图: 。 5. 回到MEGA主窗口,在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” -“Neighbor-joining”,打开一个窗口,里面有很多参数可以设
置,如何设置这些参数请参考详细的MEGA说明书,不会设置就暂且使用默认值,不要修改,点击下面的Compute按钮,系统进化树就画出来了,如图: 在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“Minimun-evolution”,如图: 在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“Maximun-parsimony”,如图: 在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“UPGMA”,
运用mega5构建系统发生进化树.
1.准备序列文件 准备fasta格式序列文件(fasta格式:大于号>后紧跟序列名,换行后是序列。举例如下)。每条序列可以单独为一个文件,也可以把所有序列放在同一文件内。 核酸序列: >sequence1_name CCTGGCTCAGGATGAACGCT 氨基酸序列: >sequence2_name MQSPINSFKKALAEGRTQIGF 2.多序列比对 打开MEGA 5,点击Align,选择Edit/Build Alignment,选择Create a new alignment,点击OK。
这时需要选择序列类型,核酸(DNA)或氨基酸(Protein)。 选择之后,在弹出的窗口中直接Ctrl + V粘贴序列(如果所有序列在同一个文件中,即可全选序列,复制)。也可以:点击Edit,选择Insert Sequence From File,选择序列文件(可多选)。
序列文件加载之后,呈蓝色背景(为选中状态)。点击按钮,选择Align DNA (如果是氨基酸序列,则会出现Align Protein)。弹出的窗口中设置比对参数,一般都是采用默认参数即可。点击OK,开始多序列比对。
比对完成后,呈现以下状态。 这时需要截齐两端含有---的序列:选中含有---的序列,按键Delete删除(注意:两端都需要截齐)。截齐之后,保存文件为:filename.mas
3.构建系统进化树 多序列比对窗口,点击Data,选择Phylogenetic Analysis,弹出窗口询问:所用序列是否编码蛋白质,根据实际情况选择Yes或No。此时,多序列比对文件就激活了,可以返回MEGA 5主界面建树了。
进化树软件MEGA最新6.06说明书
第一步:打开软件 下面介绍菜单的使用: Data菜单: Creat a new :创建一个新的数据比对文件,也就是说当我们比对完一组后,想接着比对另一组,那么使用它就可以不用退出直接把数据文件导入; Open :打开先前已经比对并保存好的文件,它包含两个子菜单:retive sequence from file 和saved aligment session ; Close: 关闭当前的比对数据文件;
Save session :保存当前比对结果,可以给比对的结果一个文件名; Export alignment :将当前的序列比对结果输出到指定文件,有两种输入格式可供选 择:MGTA 和FASTA. DNA sequence :使用它来选择输入的数据DNA 序列,这里需要说明的是如果你输入的数据是氨基酸序列的话,比对窗口只显示一个标签,若是DNA 序列的话则显示两个标签,一个是DNA 序列的,另一个是氨基酸序列的。 Protein sequences :选择输入的氨基酸序列,选择后,所以的位点就被当作氨基酸残 基位点来对待。 Translate/untranslate :只有比对的序列是编码蛋白的DNA序列的时候才可用。它可以根据指定的遗传密码表将DNA 序列翻译成特定的氨基酸序列。 Select genetic code table :使用它将编码蛋白的DNA 翻译成特定的蛋白序列。 R everse complement :将选择的一整行的DNA 序列变为与之互补配对碱基序列。Exit alignment explorer :退出序列比对的资源管理窗口 Edit 菜单: 使用这个菜单可以对我们的比对序列进行想要的一些编辑工作具体为 Undo:撤销上一步操作; Copy:复制;Cut:剪切;Paste:粘贴;这三个操作都可以只针对一个碱基或 氨基酸残基也可以是一段甚至是整个序列; Delete:从比对表格中删除一段序列; Delete gaps:去掉序列中的空缺; Insert blank sequence:重新插入一空行;标签和序列都是空的; Insert sequence from file :从已保存的文件中插入新的序列;
MEGA构建系统进化树的步骤(以MEGA7为例)
MEGA构建系统进化树的步骤(以MEGA7为例) 本文是看中国慕课山东大学生物信息学课程总结出来的 分子进化的研究对象是核酸和蛋白质序列。研究某个基因的进化,是用它的DNA序列,还是翻译后的蛋白质序列呢?序列的选取要遵循以下原则:1)如果DNA序列的两两间的一致度≥70%,选用DNA 序列。因为,如果DNA序列都如此相似,它的蛋白质会相似到看不出区别,这对构建系统发生树是不利的。所以这种情况下应该选用DNA序列,而不选蛋白质序列。2)如果DNA序列的两两间的一致度≤70%,DNA序列和蛋白质序列都可以选用。 1. 将要用于构建系统进化树的所有序列合并到同一个fasta格式文件,注意:所有序列的方向都要保持一致( 5’-3’)。 想要做系统发生树先要做多序列比对,然后把多序列比对的结果提交给建树软件进行建树,所以在用MEGA建树时可以输入一个已经比对好的多序列比对,也可以输入一条原始序列,让MEGA先来做多序列比对,再建树(一般我们都是原始序列)。所以我们以后者为例。 2.打开MEGA软件,选择主窗口的”File”→“Open A File”→找到并打开fasta文件,这时会询问以何种方式打开,我们是原始序列,需要先进行多序列比对,所以选择“Align”。如果是比对好的多序列比对可以直接选择“Analyze”。 3.在打开的Alignment Explorer窗口中选择”Alignment”-“Align by ClustalW”进行多序列比对(MEGA提供了ClustalW和Muscle两种多序列比对方法,这里选择熟悉的ClustalW),弹出窗口询问“Nothing selected for alignment,Select all?”选择“OK”。 4. 之后,弹出多序列比对参数设置窗口。这个窗口和EMBL在线多序列比对一样,可以设置替换记分矩阵、不同的空位罚分(罚分填写的是正数,计算时按负数计算)等参数。MEGA的所有默认参数都是经过反复考量设置的,这保证了MEGA傻瓜机全自动档的品质,所以当你无从下手,或者没有什么特别要求的时候,直接点击“OK”,接受这些默认参数,开始多序列比对。
用MEGA构建进化树
如何用MEGA构建进化树 MEGA3、1就是一个关于序列分析以及比较统计得工具包,其中包括有距离建树法与MP建树法;可自动或手动进行序列比对,推断进化树,估算分子进化率,进行进化假设测验,还能联机得Web数据库检索。下载后可直接使用,主要包括几个方面得功能软件:i)DNA与蛋白质序列数据得分析软件。ii)序列数据转变成距离数据后,对距离数据分析得软件。iii)对基因频率与连续得元素分析得软件。iv)把序列得每个碱基/氨基酸独立瞧待(碱基/氨基酸只有0与1得状态)时,对序列进行分析得软件。v)绘制与修改进化树得软件,进行网上blast搜索。 用MEGA构建进化树有以下步骤: 1、16S rDNA测序与参考序列选取 从环境中分离到单克隆,去重复后扩增16S rDNA序列并测序,然后与数据库比对,找到相似度最高得几个序列,确定一下您分离得细菌大约属于哪个科哪个属,如果相似度达到百分之百那基本可以确定您分离得到得就就是Blast到得那个,然后找一到两个同科得,再找一到两个同目得,再找一到两个同纲得细菌,把序列全部下下来,以FSATA形式整合在TXT文档中,如 >TS1 GCAGTCGAACGATGAAGCCCAGCTTGCTGGGTGGA TTAGTGGCGAACGGGTGAGTAACACGTGGGTGATCTGCCCTGCACTTCGGGATAAGCCTGGGAAACTGGGTCTAATACCGGATAGGACCTCGGGA TGCATGTTCCGGGGTGGAAAGGTTTTCCGGTGCAGGATGGGCC >gi|117572706|gb|EF028124、1| Rhodococcus sp、Atl25 16S ribosomal RNAgene,partial sequence CGATTAGAGTTTGATCCTGGCTCAGGACGAACGCTGGCGGCGTGCTTAACACATGCAAGTCGAACGATGAAGCCCAGCTTGCTGGGTGGATTAGTGGCGAACGGGTGAGTAACACGTGGGTGA TCTGCCCTGCACTTCGGGATAAGCCTGGGAAACTGGGTCTAATACCGGAT >TS2 TGCAAGTCGAGCGAATGGA TTAAGAGCTTGCTCTTATGAAGTTAGCGGCGGA CGGGTGAGTAACACGTGGGTAACCTGCCCATAAGACTGGGATAACTCCGG GAAACCGGGGCTAATACCGGATAACATTTTGAACTGCATGGTTCGAAATTGAAAGGCGGCTTCGGCTGTCACT >gi|56383044|emb|AJ809498、1|Bacillus cereus partial16S rRNA gene, strainTMW 2、383 GA TGAACGCTGGCGGCGTGCCTAATACATGCAAGTCGAGCGAATGGATTAAGAGCTTGCTCTTATGAAGTTAGCGGCGGACGGGTGAGTAACACGTGGGTAACCTGCCCATAAGACTGGGA TAACTCCGGGAAACCGGGGCTAA TACCGGATAACA TTTTGAACYGCA TGGTTC…………………………、 …………………………、 参考序列选择有几个原则:a,不选非培养(unclutured)微生物为参比;b,所选参考序列要正确,里面无错误碱基;c,在保证同属得前提下,优先选择16S rDNA全长测序或全基因组测序得种;d,每个种属选择一个参考序列,如果自己得序列中同一属得较多,可适当选择两个参考序列。 2、序列比对
MEGA构建系统进化树的步骤(以MEGA7为例)教学文案
M E G A构建系统进化树的步骤(以M E G A7为 例)
MEGA构建系统进化树的步骤(以MEGA7为例) 本文是看中国慕课山东大学生物信息学课程总结出来的 分子进化的研究对象是核酸和蛋白质序列。研究某个基因的进化,是用它的DNA序列,还是翻译后的蛋白质序列呢?序列的选取要遵循以下原则:1)如果DNA序列的两两间的一致度≥70%,选用DNA序列。因为,如果DNA序列都如此相似,它的蛋白质会相似到看不出区别,这对构建系统发生树是不利的。所以这种情况下应该选用DNA序列,而不选蛋白质序列。2)如果DNA 序列的两两间的一致度≤70%,DNA序列和蛋白质序列都可以选用。 1. 将要用于构建系统进化树的所有序列合并到同一个fasta格式文件,注意:所有序列的方向都要保持一致 ( 5’-3’)。 想要做系统发生树先要做多序列比对,然后把多序列比对的结果提交给建树软件进行建树,所以在用MEGA建树时可以输入一个已经比对好的多序列比对,也可以输入一条原始序列,让MEGA先来做多序列比对,再建树(一般我们都是原始序列)。所以我们以后者为例。 2.打开MEGA软件,选择主窗口的”File”→“Open A File”→找到并打开fasta文件,这时会询问以何种方式打开,我们是原始序列,需要先进行多序列比对,所以选择“Align”。如果是比对好的多序列比对可以直接选择“Analyze”。 3.在打开的Alignment Explorer窗口中选择”Alignment”-“Align by ClustalW”进行多序列比对(MEGA提供了ClustalW和Muscle两种多序列比对方法,这
用MEGA作进化树[2]
用MEGA2做进化树的步骤(图示) 1、打开程序 如下图所示: 2、MEGA2只能打开meg格式的文件,但是它可以把其他格式的多序列比对文件转换过来,我们在这里用aln格式(Clustal的输出文件)转换meg文件。点File:Convert to MEGA Format...打开转换文件对话框 如下图所示:
3、选择文件和转换文件对话框,选择aln文件,点OK 如下图所示: 4、转换好的meg文件,点存盘保存meg文件,meg文件会和aln文件保存在同一个目录 如下图所示: 5、关闭转换窗口,回到主窗口,现在点面板上的“Click me to activate a data file”打开刚才的meg 文件 如下图所示:
6、选择meg文件,点“打开” 如下图所示: 7、程序会自动识别序列的类型,如果识别错误,请手工选择数据类型。然后点OK就行了如下图所示:
8、数据输入之后的样子,窗口下面有序列文件名和类型 如下图所示: 9、现在终于可以开始做Bootstrap验证和进化树了,MEGA的主要功能就是做Bootstrap验证的进化树分析,Bootstrap验证是对进化树进行统计验证的一种方法,可以作为进化树可靠性的一个度量。各种算法虽然不同,但是操作方法基本一致,我们在此以UPGMA方法为例进行演示。点下图所示的菜单项。 如下图所示:
10、...会弹出如下的对话框,在此你可以选择计算参数。 如下图所示: 11、Distance Options标签页中的Models可以下拉,其中有若干个计算距离的方法可以选择,在此默认泊松校验(Poisson Correction)作为计算距离的方法。 如下图所示:
用MEGA构建进化树
如何用MEGA构建进化树 MEGA3.1是一个关于序列分析以及比较统计的工具包,其中包括有距离建树法和MP 建树法;可自动或手动进行序列比对,推断进化树,估算分子进化率,进行进化假设测验,还能联机的Web数据库检索。下载后可直接使用,主要包括几个方面的功能软件:i)DNA 和蛋白质序列数据的分析软件。ii)序列数据转变成距离数据后,对距离数据分析的软件。iii)对基因频率和连续的元素分析的软件。iv)把序列的每个碱基/氨基酸独立看待(碱基/氨基酸只有0和1的状态)时,对序列进行分析的软件。v)绘制和修改进化树的软件,进行网上blast搜索。 用MEGA构建进化树有以下步骤: 1. 16S rDNA测序和参考序列选取 从环境中分离到单克隆,去重复后扩增16S rDNA序列并测序,然后与数据库https://www.360docs.net/doc/3e10796817.html,/blast/Blast.cgi比对,找到相似度最高的几个序列,确定一下你分离的细菌大约属于哪个科哪个属,如果相似度达到百分之百那基本可以确定你分离得到的就是Blast到的那个,然后找一到两个同科的,再找一到两个同目的,再找一到两个同纲的细菌,把序列全部下下来,以FSATA形式整合在TXT文档中,如 >TS1 GCAGTCGAACGATGAAGCCCAGCTTGCTGGGTGGA TTAGTGGCGAACGGGTGAGTAA CACGTGGGTGATCTGCCCTGCACTTCGGGATAAGCCTGGGAAACTGGGTCTAATACCG GA TAGGACCTCGGGA TGCA TGTTCCGGGGTGGAAAGGTTTTCCGGTGCAGGATGGGCC >gi|117572706|gb|EF028124.1| Rhodococcus sp. Atl25 16S ribosomal RNA gene, partial sequence CGATTAGAGTTTGA TCCTGGCTCAGGACGAACGCTGGCGGCGTGCTTAACACATGCAA GTCGAACGATGAAGCCCAGCTTGCTGGGTGGA TTAGTGGCGAACGGGTGAGTAACAC GTGGGTGATCTGCCCTGCACTTCGGGATAAGCCTGGGAAACTGGGTCTAA TACCGGA T >TS2 TGCAAGTCGAGCGAATGGA TTAAGAGCTTGCTCTTA TGAAGTTAGCGGCGGACGGGTG AGTAACACGTGGGTAACCTGCCCA TAAGACTGGGATAACTCCGGGAAACCGGGGCTAA TACCGGATAACA TTTTGAACTGCATGGTTCGAAA TTGAAAGGCGGCTTCGGCTGTCACT >gi|56383044|emb|AJ809498.1| Bacillus cereus partial 16S rRNA gene, strain TMW 2.383 GA TGAACGCTGGCGGCGTGCCTAA TACATGCAAGTCGAGCGAA TGGATTAAGAGCTTG CTCTTA TGAAGTTAGCGGCGGACGGGTGAGTAACACGTGGGTAACCTGCCCATAAGAC TGGGATAACTCCGGGAAACCGGGGCTAATACCGGATAACATTTTGAACYGCATGGTTC …………………………. …………………………. 参考序列选择有几个原则:a,不选非培养(unclutured)微生物为参比;b,所选参考序列要正确,里面无错误碱基;c,在保证同属的前提下,优先选择16S rDNA全长测序或全基因组测序的种;d,每个种属选择一个参考序列,如果自己的序列中同一属的较多,可适当选择两个参考序列。
怎样使用MEGA建立进化树
怎样使用MEGAt 立进化树 如何使用MEGA4.0#立进化树 1、首先是双击软件打开如下图所示 |M| ijaKMr 3 valj 141 Mrhr ArgrwricQt iVvta “qplii :护 忏冲 i 二客H - I 号筍需.廿星"L IF M ■ H 、- | II ■ DKi -Mjrsrze: H r? r-r r ^c>az^ LCS 2、现在是处于DNA 序列,而我们要做蛋白质的进化树的话,就如下操作
M4. Aligmr>&nl Explof頁 H L lQnmt*Ft ji Edit m e祁 3、接下来我们要进行序列的输入,点击左边那个红箭头,贝U出现下面的窗口
刚M4: Alfgnment Explorer 匚;日屯EJrt S?ar di Aflgmnenl Wfrb $e<)□ d | D ◎日 「蹇輻酋1 41象 Protein S^quer匚弊 1 |主曲色"匕色丄 4、然后右击sequenee 1,修改名字,如改成DPV Frotejn Sequence? 5、然后从Word里复制蛋白质序列,然后在下面的位置粘贴 G 辱Copf PTCtfiT X CU, 書 f sterna 6则可出现如下图的序列了 □ Q CW1C 3 iRWfl Wq^ri[ V ^i>n irequ^Ki 幷册枷? 1話皿讥曲佰i" —喇?ct Mgeirc 惟 ■ sy
7、然后点击窗口上的保存图标保存 8、重复从3开始,直到你的序列输入完 9、序列输入元后进行最后的保存,方法如下 垂邑trit 5|讨之斗和"1 of op?r * dow 亠 P TOUMT 1 假如你要对比你所测序列E的序列与其他物质的亲缘关系,步骤如下:一,首先要先把你获得E的序列去NCBI网站进行比对,步骤如下: 1.登录NCBI网站https://https://www.360docs.net/doc/3e10796817.html,/ 2.找到右侧的BLAST,点进去; 3.找到页面下方的这个图标,点Nucleotide BLAST 4.将测得的序列全部粘贴到页面上的这个框里: 5.找到页面最下方的Algorithm parameters,在最下面的BLAST旁边勾选 “Show XXXX”后点击BLAST 6.然后就会弹出另一个页面,你就得耐心等待了,因为它在比对,比对好后就 会出现这样一个界面: 7.然后往下拉,就看到好多序列的结果,可以选择所有的序列下载,也可以选 择你想要的序列来下载(All/None可全选或都不选),选好后点击“GenBank”。 8.把所有的序列都勾选后,点右上角的“send” 9.出现这个框格,File-FASTA按框格里选择好点Create File就可以批量下载内含你所选的序列的“fasta”格式的文件; 11改好后打开,把自己的序列按“>名称+序列”的格式紧接在已下好的序列后面,添加好后再把后缀改回“fasta”,便可进行下一步 8. 3 12.双击fasta文件,由MEGA6.0打开,如图 13.单击W图标中的“Align DNA”,会提醒你选择序列,单击确定即可,如下图 14.比对后的序列如下图。 15.然后我们需要把“*”号之外的序列全部删除,只留下"*"标注的序列,保存,保存后得到的是“mas”格式文件 MEGA构建系统进化树的步骤 1. 将要用于构建系统进化树的所有序列合并到同一个fasta格式文件,注意:所有序列的方向都要保持一致( 5’-3’)。如图: 2. 打开MEGA软件,选择"Alignment" - "Alignment Explorer/CLUSTAL",在对话框中选择Retrieve sequences from a file, 然后点OK,找到准备好的序列文件并打开,如图: 。 3. 在打开的窗口中选择”Alignment”-“Align by ClustalX” 进行对齐,对齐过程需要一段时间,对齐完成后,最好将序列两端切齐,选择两端不齐的部分,单击右键,选择delete即可,如图: 。 4. 关闭当前窗口,关闭的时候会提示两次否保存,第一次无所谓,保存不保存都可以,第二次一定要保存,保存的文件格式是.meg。根据提示输入Title,然后会出现一个对话框询问是否是Protein-coding nucleotide sequence data, 根据情况选择Yes或No。最后出现一个对话框询问是否打开,选择Yes,如图: 。 5. 回到MEGA主窗口,在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” -“Neighbor-joining”,打开一个窗口,里面有很多参数可以设置,如何设置这些参数请参考详细的MEGA说明书,不会设置就暂且使用默认值, 不要修改,点击下面的Compute按钮,系统进化树就画出来了,如图: 在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“Minimun-evolution”,如图: 在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“Maximun-parsimony”,如图: 在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“UPGMA”,使用mega6做进化树
Mega的使用以及进化树的绘制
