基因组组装新技术研讨-全国巡讲最终版(1)

诺禾致源 support@https://www.360docs.net/doc/5e701519.html,
https://www.360docs.net/doc/5e701519.html, The World’s Leading Genomics Research Solutions Provider
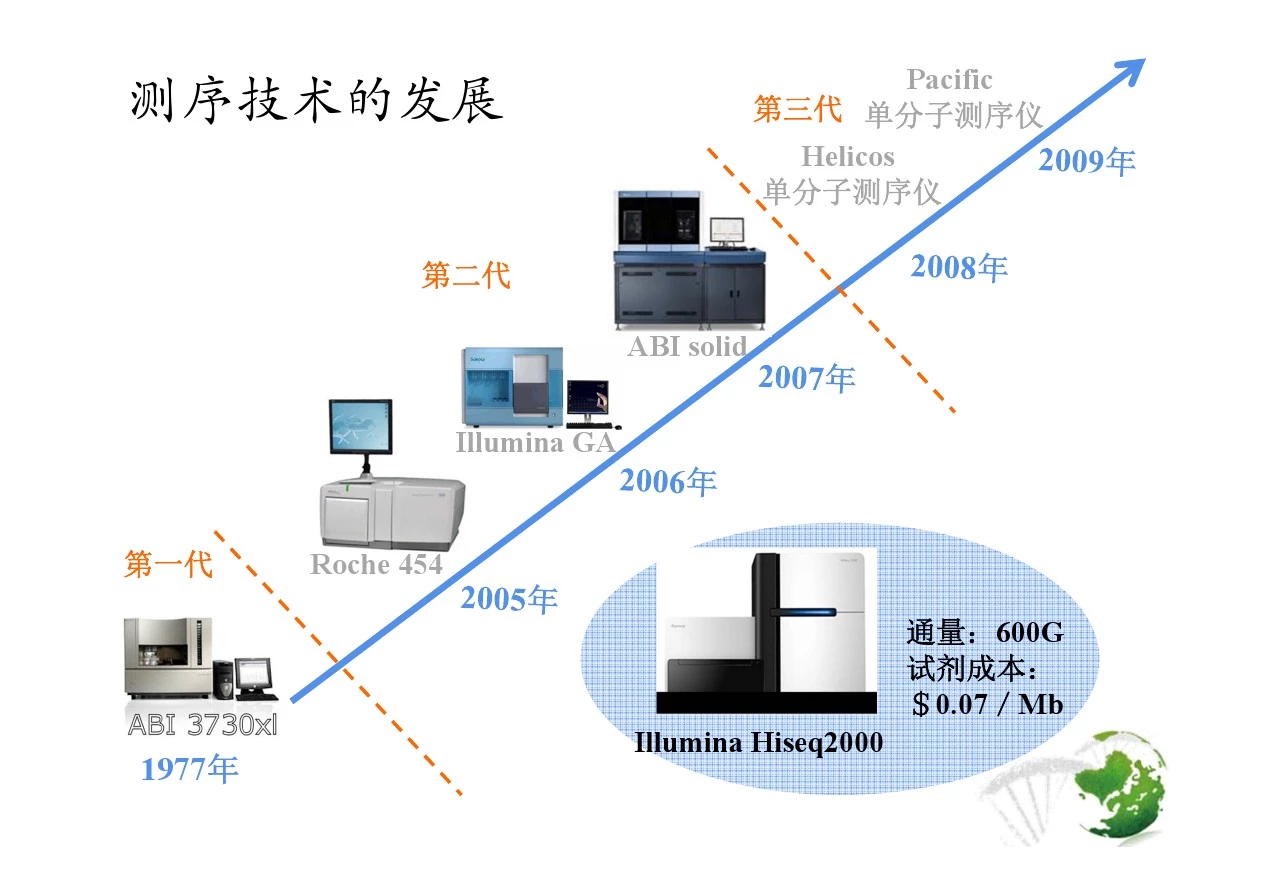
测序技术的发展
第二代 ABI solid Illumina GA
Pacific 第三代 单分子测序仪 Helicos 单分子测序仪
第一代
Roche 454 通量:600G 试剂成本: $0.07/Mb
1977年
Illumina Hiseq2000
DNA测序技术的发展和其最新进展
DNA测序技术的发展及其最新进展 摘要:自从诺贝尔奖得主桑格于1977年发明了第一代DN测序技术以来,DNA测序技术已经作为重要的实验技术广泛的应用于现代生物学研究当中。经过了几十年的发展,DNA测序技术日臻成熟,并且以单分子测序为特点的第三代测序技术也已经诞生。本文主要就每一代测序技术原理和特点及其最新进展做简要介绍。 关键词:DNA测序技术;第三代DNA测序技术;最新进展 The Development and New Progress of DNA Sequencing Technology Abstract: Since Nobel Prize Winner Sanger have founded the first generation of DNA Sequence technology in 1977, DNA sequencing technology has been widely used in modern biological researches as an important experimental. Over decades of year’s development, DNA sequence technology mature gradually and the third generation sequencing technologies characterized by single-molecule sequencing have also emerged. The mechanisms and features of each generation of sequencing technology and their latest progress will be discussed here. Key Words: DNA Sequence technology ; third generation DNA sequencing ;latest development 1.引言 DNA测序技术是分子生物学研究中最常用的技术,它的出现极大地推动了生物学的发展。自从1953年Watson和Crick发现DNA双螺旋结构后[1],人类就开始了对DNA序列的探索,在世界各地掀起了DNA测序技术的热潮。1977年Maxam和Gilbert报道了通过化学降解测定DNA序列的方法[2]。同一时期,Sanger发明了双脱氧链终止法[3]。20世纪90年代初出现的荧光自动测序技术将DNA测序带入自动化测序的时代。这些技术统称为第一代DNA测序技术。最近几年发展起来的第二代DNA测序技术则使得DNA测序进入了高通量、低成本的时代。目前,基于单分子读取技术的第三代测序技术已经出现,该技术测定DNA序列更快,并有望进一步降低测序成本,推进相关领域生物学研究。本文主要介绍DNA测序技术的发展历史及不同发展阶段各种主要测序技术的特点,并针对目前新一代DNA测序技术及目前国际DNA测序最新进展做简要综述。
新一代测序技术的发展及应用前景
2010年第10期杨晓玲等:新一代测序技术的发展及应用前景 等交叉学科的迅猛发展。 1.1第二代测序——高通量低成本齐头并进以高通量低成本为主要特征的第二代测序,不再需要大肠杆菌进行体内扩增,而是直接通过聚合酶或者连接酶进行体外合成测序¨】。根据其原理又可分为两类:聚合酶合成测序和连接酶合成测序。1.1.1聚合酶合成测序法Roche公司推出的454技术开辟了高通量测序的先河。该技术通量可达Sangcr测序的几百倍,而成本却只有几十分之一,因此一经推出,便受到了国际上基因组学专家的广泛关注。454采用焦磷酸合成测序法HJ,避免了传统测序进行荧光标记以及跑胶等繁琐步骤,同时利用乳胶系统对DNA分子进行扩增,实现了大规模并行测序。截止到2010年4月,已有700多篇文献是采用了454测序技术(http://454.com/publications.and—resources/publications.asp),对该技术是一个极大的肯定。 Illumina公司推出的Solexa遗传分析仪是合成技术的进一步发展与延伸。该技术借助高密度的DNA单分子阵列,使得测序成本和效率均有了较大改善。同时Solexa公司提出的可逆终止子”1也是该技术获得认可的原因之一。与454相比。Solexa拥有更高的通量,更低的成本。虽然片段长度较短仍是主要的技术瓶颈,但是对于已有基因组的物种来说,Solexa理所当然成为第二代测序技术的首选。2008年以来,利用该技术开展的研究大幅度上升,报道文献达400多篇(http://www.illumina.com/systems/genome—analyzer_iix.ilmn)o 1.1.2连接酶合成测序法2007年ABI公司在Church小组拍1研究成果的基础上推出了SOLID测序仪。该技术的创新之处在于双碱基编码…的应用,即每个碱基被阅读两次,因此大大减少了测序带来的错误率,同时可以方便的区分SNP和测序错误。在测序过程中,仪器自动加入4种荧光标记的寡核苷酸探针,探针与引物发生连接反应,通过激发末端的荧光标记识别结合上的碱基类型。目前SOLID3.0测序通量可达20G,而测序片段仅有35—50bp,这使得该技术与Solexa相比,应用范围还不够广泛。ABI公司正加快研发进度,争取在片段长度方面做出重大突破。 DanaherMotion公司推出Polonator¨1测序仪同样也是基于Church小组的研究成果,但是该设备的成本要低很多,同时用户在使用时可以根据自己的研究目的设置不同的测序条件。而CompleteGe—nomics公司推出的DNA纳米阵列与组合探针锚定连接测序法"1则具有更高的容错能力,试剂的消耗也进一步减少,目前已顺利完成3个个体基因组的测序工作。 1.2第三代测序——单分子长片段有望实现第二代测序技术虽然在各方面都有了较大的突破,但是仍然建立在PCR扩增的基础上。为了避免PCR扩增带来的偏差,科学家目前正在研制对DNA单个分子直接测序的第三代测序仪。最具代表性的包括Heliscope单分子测序仪,单分子实时合成测序法,纳米孔测序技术等。 Helicos技术仍然是基于合成测序原理¨…,它采用了一种新的荧光类似物和灵敏的监测系统,能够直接记录到单个碱基的荧光,从而克服了其他方法须同时测数千个相同基因片段以增加信号亮度的缺陷。PacificBioscienees公司研发的单分子实时合成测序法充分利用了DNA聚合酶的特性,可以形象的描述为通过显微镜实时观测DNA聚合酶,并记录DNA合成的整个过程。纳米孔测序技术[11’121则是利用不同碱基在通过纳米小孔时引起的静电感应稍有不同,或者不同碱基通过小孔的能力各有差异,来加以区分不同的碱基信号。 2应用与实践 Kahvejian在2008年的一篇综述中提到¨“:“如果你可以随心所欲地测序,你会开展哪些研究?”。人类基因组计划的完成和近年来高通量测序的兴起,使越来越多的科研工作者认识到,我们对于生物界的认识才刚刚起步。基因图谱的绘制并不意味着所有遗传密码的破解,癌症基因组的开展也没有解决所有的医学难题。DNA变异的模式和进化机制,基因调控网络的结构和相互作用方式,复杂性状及疾病的分子遗传基础等,仍是困扰生物学家和医学家的难题,而高通量测序的广泛应用,也许可以让我们知道的更多。 2.1DNA水平的应用 2.1.1全基因组测序新一代测序技术极大地推
高通量测序基础知识
高通量测序基础知识简介 陆桂 什么是高通量测序? 高通量测序技术(High-throughput sequencing,HTS)是对传统Sanger测序(称为一代测序技术)革命性的改变,一次对几十万到几百万条核酸分子进行序列测定, 因此在有些文献中称其为下一代测序技术(next generation sequencing,NGS )足见其划时代的改变, 同时高通量测序使得对一个物种的转录组和基因组进行细致全貌的分析成为可能, 所以又被称为深度测序(Deep sequencing)。 什么是Sanger法测序(一代测序) Sanger法测序利用一种DNA聚合酶来延伸结合在待定序列模板上的引物。直到掺入一种链终止核苷酸为止。每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。由于ddNTP缺乏延伸所需要的3-OH基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止。终止点由反应中相应的双脱氧而定。每一种dNTPs和ddNTPs的相对浓度可以调整,使反应得到一组长几百至几千碱基的链终止产物。它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,凝胶处理后可用X-光胶片放射自显影或非同位素标记进行检测。 什么是基因组重测序(Genome Re-sequencing) 全基因组重测序是对基因组序列已知的个体进行基因组测序,并在个体或群体水平上进行差异性分析的方法。随着基因组测序成本的不断降低,人类疾病的致病突变研究由外显子区域扩大到全基因组范围。通过构建不同长度的插入片段文库和短序列、双末端测序相结合的策略进行高通量测序,实现在全基因组水平上检测疾病关联的常见、低频、甚至是罕见的突变位点,以及结构变异等,具有重大的科研和产业价值。 什么是de novo测序 de novo测序也称为从头测序:其不需要任何现有的序列资料就可以对某个物种进行测序,利用生物信息学分析手段对序列进行拼接,组装,从而获得该物种的基因组图谱。获得一个物种的全基因组序列是加快对此物种了解的重要捷径。随着新一代测序技术的飞速发展,基因组测序所需的成本和时间较传统技术都大大降低,大规模基因组测序渐入佳境,基因组学研究也迎来新的发展契机和革命性突破。利用新一代高通量、高效率测序技术以及强大的生物信息分析能力,可以高效、低成本地测定并分析所有生物的基因组序列。 什么是外显子测序(whole exon sequencing) 外显子组测序是指利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法。外显子测序相对于基因组重测序成本较低,对研究已知基因的SNP、Indel等具有较大的优势,但无法研究基因组结构变异如染色体断裂重组等。
基因测序技术的优缺点及应用
基因测序技术的优缺点及应用 随着人类基因组计划的完成,人类对自身遗传信息的了解和掌握有了前所未有的进步。与此同时,分子水平的基因检测技术平台不断发展和完善,使得基因检测技术得到了迅猛发展,基因检测效率不断提高。从最初第一代以 Sanger 测序为代表的直接检测技术和以连锁分析为代表的间接测序技术,到 2005 年,以Illumina 公司的 Solexa技术和 ABI 公司的 SOLiD 技术为标志的新一代测序 (next-generation sequencing,NGS) 的相继出现,测序效率明显提升,时间明显缩短,费用明显降低,基因检测手段有了革命性的变化。其技术正向着大规模、工业化的方向发展,极大地提高了基因检测的检出率,并扩展了疾病在基因水平的研究范围。2009 年 3 月,约翰霍普金斯大学的研究人员在《Science》杂志上发表了通过 NGS外显子测序技术,发现了一个新的遗传性胰腺癌的致病基因PALB2,标志着 NGS 测序技术成功应用于致病基因的鉴定研究。同年,《Nature》发表了采用 NGS 技术发现罕见弗里曼谢尔登综合征MYH3 致病基因突变和《Nat Genet》发表了遗传疾病米勒综合征致病基因。此后,通过 NGS 技术,与遗传相关的致病基因不断被发现,NGS 技术已成为里程碑式的进步。2010 年,《Science》杂志将这一技术评选为当年“十大科学进展”。 近两年,基因检测成为临床诊断和科学研究的热点,得到了突飞猛进和日新月异的发展,越来越多的临床和科研成果不断涌现出来。同时,基因检测已经从单一的遗传疾病专业范畴扩展到复杂疾病和个体化应用更加广阔的领域,其临床检测范围包括高危疾病的新生儿筛查、遗传疾病的诊断和基因携带的检测以及基因药物检测用于指导个体化用药剂量、选择和药物反应等诸多方面的研究。目前,基因检测在临床诊断和医学研究的应用正越来越受到医生的普遍重视和引起研究人员的极大的兴趣。 本文介绍了几种 DNA 水平基因检测常见的方法,比较其优缺点和在临床诊断和科学研究中的应用,对指导研究生和临床医生课外学习,推进临床科研工作和提升科研教学水平有着指导意义。 1、第一代测序 1.1 Sanger 测序采用的是直接测序法。1977年,Frederick Sanger 等发明了双脱氧链末端终止法,这一技术随后成为最为常用的基因测序技术。2001 年,Allan Maxam 和 Walter Gibert 发明了 Sanger 测序法,并在此后的 10 年里成为基因检测的金标准。其基本原理即双脱氧核苷三磷酸(dideoxyribonucleoside triphosphate,ddNTP) 缺乏PCR 延伸所需的 3'-OH,因此每当 DNA 链加入分子 ddNTP,延伸便终止。每一次 DNA 测序是由 4个独立的反应组成,将模板、引物和 4 种含有不同的放射性同位素标记的核苷酸的ddNTP 分别与DNA 聚合酶混合形成长短不一的片段,大量起始点相同、终止点不同的 DNA 片段存在于反应体系中,具有单个碱基差别的 DNA 序列可以被聚丙烯酰胺变性凝胶电泳分离出来,得到放射性同位素自显影条带。依据电泳条带读取DNA 双链的碱基序列。 人类基因组的测序正是基于该技术完成的。Sanger 测序这种直接测序方法具有高度的准确性和简单、快捷等特点。目前,依然对于一些临床上小样本遗传疾病基因的鉴定具有很高的实用价值。例如,临床上采用 Sanger 直接测序 FGFR 2 基因证实单基因 Apert 综合征和直接测序 TCOF1 基因可以检出多达 90% 的
基因组测序的数学模型
基因组组装 摘要 基因组测序是生物信息学的核心,有着极其重要的应用价值。新的测序技术大量涌现,产生的reads长度更短,数量更多,覆盖率更大,能直接读取的碱基对序列长度远小于基因组长度。所以测序之前DNA分子要经过复制若干份、随机打断成短片段。要获取整个DNA片段,需要把这些片段利用重合部分信息组织连接。如何在保证组装序列的连续性、完整性和准确性的同时设计耗时短、内存小的组装算法是本题的关键。 本文建立改进后OLC算法模型。该模型首先使用了特定的编码规定,通过C++程序对庞大的数据先后进行十进制和二进制的处理,不改变数据准确性的前提下尽可能减小内存和缩短计算机操作时间,并引入解决碱基识别错误问题的一般思路消除初始reads中的碱基错误。然后通过深度优先算法,设定适当的阈值,找出具有重叠关系的碱基片段并形成一有向赋权图,其中点是碱基片段,边代表具有重叠关系,权值代表片段重叠的多少,将问题转化为图论中寻找最大赋权通路的问题,从而对OLC算法进行改进,采用图论的方法更直观和更具操作性的解决DNA的拼接问题,从而对OLC算法进行改进。最后再根据OLC算法对Hamilton 路径进行拼接,生成共有序列,通过多序列比对等方法,获得最终的基因组序列。 关键词:基因组测序 OLC算法深度优先算法Hamilton路径
一问题的重述 1.1 问题背景 快速和准确地获取生物体的遗传信息对于生命科学研究具有重要的意义。对每个生物体来说,基因组包含了整个生物体的遗传信息,这些信息通常由组成基因组的DNA或RNA分子中碱基对的排列顺序所决定。获得目标生物基因组的序列信息,进而比较全面地揭示基因组的复杂性和多样性,成为生命科学领域的重要研究内容。 1.2 问题提出 确定基因组碱基对序列的过程称为测序。目前能直接读取的碱基对序列长度远小于基因组序列长度,因此需要利用一定的方法将测序得到的短片段序列组装成更长的序列。通常的做法是,将基因组复制若干份,无规律地分断成短片段后进行测序,然后寻找测得的不同短片段序列之间的重合部分,并利用这些信息进行组装。例如,若有两个短片段序列分别为 ATACCTT GCTAGCGT GCTAGCGT AGGTCTGA 则有可能基因组序列中包含有ATACCTT GCTAGCGT AGGTCTGA这一段。 由于技术的限制和实际情况的复杂性,最终组装得到的序列与真实基因组序列之间仍可能存在差异,甚至只能得到若干条无法进一步连接起来的序列。对组装效果的评价主要依据组装序列的连续性、完整性和准确性。连续性要求组装得到的(多条)序列长度尽可能长;完整性要求组装序列的总长度占基因组序列长度的比例尽可能大;准确性要求组装序列与真实序列尽可能符合。 利用现有的测序技术,可按一定的测序策略获得长度约为50–100个碱基对的序列,称为读长(reads)。基因组复制份数约为50–100。基因组组装软件可根据得到的所有读长组装成基因组,这些软件的核心是某个组装算法。一个好的算法应具备组装效果好、时间短、内存小等特点。新一代测序技术在高通量、低成本的同时也带来了错误率略有增加、读长较短等缺点,现有算法的性能还有较大的改善空间。具体解决问题如下: (1)建立数学模型,设计算法并编制程序,将读长序列组装成基因组。你的算法和程序应能较好地解决测序中可能出现的个别碱基对识别错误、基因组中存在重复片段等复杂情况。 (2)现有一个全长约为120,000个碱基对的细菌人工染色体,采用Hiseq2000测序仪进行测序,测序策略以及数据格式的简要说明见附录一和附录二,测得的读长数据见附录三,测序深度约为70×,即基因组每个位置平均被测到约70次。试利
新一代DNA测序技术总览
作者:尹银亮、陈会平、毛良伟译来源:生物谷 原文刊登于《分析化学》综述Analytical Chemistry 原文标题:Landscape of Next-Generation Sequencing Technologies 索引信息:https://www.360docs.net/doc/5e701519.html,/10.1021/ac2010857 | Anal. Chem. 2011, 83, 4327–4341 原文作者:Thomas P. Niedringhaus, Denitsa Milanova, Matthew B. Kerby, Michael P. Snyder,and Annelise E. Barro 译者资料: 尹银亮,香港华大基因研发中心有限公司email:stevenyinbio@https://www.360docs.net/doc/5e701519.html, 陈会平,毛良伟,武汉华大基因科技有限公司 【内容】 第二代测序 第二代测序成本 第三代测序技术 单分子测序法 边连接边测序法 边合成边测序法 纳米孔测序技术 蛋白质纳米孔测序法 固态纳米孔测序法 长距离阅读DNA的扩展方法 总结性评论 DNA测序正处在技术上天翻地覆剧变的阵痛之中,其突出特点是,测序通量(测序数据量)的大幅增长,原始数据中每个碱基的测序成本急剧下跌,并伴随着以巨资购买仪器以引进新技术的需求。以前看似高不可攀的奢侈性研究活动(如个人基因组测序,宏基因组学研究,以及对大量重要物种的测序),在短短几年之间,正以急速的步伐而变得越来越切实可行了。本篇综述将集中讨论在第三,第四代测序方法背后的故事:它们所面临的挑战;各种方法的局限性;以及它们带给我们的充满诱惑的前景。 第一代DNA测序技术用的是1975年由桑格(Sanger)和考尔森(Coulson)开创的链终止法或者是1976-1977年由马克西姆(Maxam)和吉尔伯特(Gilbert)发明的化学法(链降解). 在1977年,桑格测定了第一个基因组序列,是噬菌 体X174的,全长5375个碱基。其测序方法和历史过程以前已做过详细回顾。 后来的四色荧光桑格测序法(每一种荧光代表四种碱基中的一种)被用在自动毛细管电泳测序系统中,此系统由应用生物系统有限公司(Applied Biosystems Inc.)推上市场,后来该公司被整合入生命技术公司(Life Technologies)和贝克曼.考尔特公司(Beckman Coulter inc.)(见表1)。发表于2001年的第一个人类基因组
基因组组装 数学建模
基因组组装 摘要 基因组组装是生物信息学的核心,有着极其重要的应用价值。本文针对提高基因组组装问题的不同途径和规模,利用了图论中的De Bruijn图法和欧拉路径问题的思想建立模型,并对传统De Bruijn图模型中存在的一些问题(如overlap 部分判定速度较慢、内存占用大等)建立了相应模型进行改进,利用所建模型对附录中给出的reads进行了组装,并对原文件中错误和低质量的reads进行了筛选,提高了原始数据的质量,对问题进行了拓展。 首先,在模型的建立方面,我们利用了图论中de Bruijn图法和欧拉路径问题的思想并结合实际,建立了基因组序列组装模型,基于de Bruijn图法的模型不仅避免了使用OLC方法组装第二代基因测序技术所产生的高通量、短序列、高覆盖的基因组易产生错误、运行较慢的弊端,并且还可以减少冗余数据量,提高了内存效率。 其次,在模型的优化改进方面,我们通过建立基于De Bruijn sequence的碱基序列替换改进模型和k值选择模型对传统De Bruijn 图模型进行了改进,很好的解决了原有模型存在的overlap比对速度慢、不同k取值导致资源占用不同等问题,提高了基因组组装过程中的时间效率和容错率。 最后,在对于原始reads数据的处理方面,我们利用了Hash算法的思想,对每条k-mer建立Hash值,并建立了基于Phred法的reads记录评分筛选模型,对于低质量和错误的reads记录进行了筛选去除,提高了原始reads数据的质量,使最终得到的contig更加准确。 关键词:De Bruijn图欧拉路径Phred质量评分Hash算法
快速和准确地获取生物体的遗传信息对于生命科学研究具有重要的意义,对每个生物体来说,基因组包含了整个生物体的遗传信息,这些信息通常由组成基因组的DNA或RNA分子中碱基对的排列顺序所决定。获得目标生物基因组的序列信息,进而比较全面地揭示基因组的复杂性和多样性,成为生命科学领域的重要研究内容。 确定基因组碱基对序列的过程称为测序(sequencing)。利用现有的测序技术,按一定的测序策略获得长度约为50–100个碱基对的序列,称为读长(reads)。基因组复制份数约为50–100。基因组组装软件可根据得到的所有读长组装成基因组,这些软件的核心是某个组装算法。常用的组装算法主要基于OLC (Overlap/Layout/Consensus)方法、贪婪图方法、de Bruijn图方法等。一个好的算法应具备组装效果好、时间短、内存小等特点。新一代测序技术在高通量、低成本的同时也带来了错误率略有增加、读长较短等缺点,现有算法的性能还有较大的改善空间。 1.1问题一的重述 问题一要求建立数学模型,设计算法并编制程序,将读长序列组装成基因组。对算法和程序的要求如下: (1)能较好地解决测序中出现的个别碱基对识别错误 (2)能较好地解决基因组中存在重复片段的情况 1.2问题二的重述 问题二要求将一个全长约为120,000个碱基对的细菌人工染色体(BAC),采用Hiseq2000测序仪进行测序,测序深度(sequencing depth)约为70×,即基因组每个位置平均被测到约70次。利用解决问题一建立的算法和程序进行组装,并使之具有良好的组装效果。
基因组组装
数学建模暑假培训 论文题目:基因组组装 姓名1:李建平学号:201220370107 专业:物理学姓名1:肖震南学号:201220370115 专业:物理学姓名1:肖丽霞学号:201220300325 专业:应用化学
摘要 快速和准确地获取生物体的遗传信息对于生命科学研究具有重要的意义。伴随着人类基因组计划的实施而突飞猛进。从第一代到现在普遍应用的第二代,以及近年来正在兴起的第三代,测序技术正向着高通量、低成本的方向发展。尽管如此,目前能直接读取的碱基对序列长度远小于基因组序列长度,因此需要利用一定的方法将测序得到的短片段序列组装成更长的序列。 随着测序技术的发展,测序过程中获得DNA片段越来越短,基于Euler路径拼接算法在处理这种短片段拼接时更具优势,在Euler路径算法中,构建de Bruijn图。该算法能够快速地处理海量测序数据,而且能得到质量较高的重叠群。 问题一:首先,构造de Bruijn图,通过Euler路径,确定基因组组装的结果。对于个别碱基对识别错误、基因组中存在重复片段等复杂情况,通过设定一个固定的长度阈值,直接去除和设计一个Tour Bus算法来解决这些问题。 问题二:问题二是把上面的模型具体化,对问题一的模型进行检验。由于问题二的数据庞大,所以先对其进行筛选。筛选出读长为88bp,然后建立Euler路径。 关键词:基因组组装de Bruijn图Euler路径
一.问题的重述 随着测序技术的不断发展,新一代测序技术产生的在高通量、低成本的同时也使得错误率略有增加、读长较短等缺点。本题要求利用数学模型,设计算法要求解决如下几个复杂问题: (1)测序过程中可能出现的个别碱基对识别错误; (2)基因组中存在重复片段; (3)能够处理海量的序列比对; 二.模型的假设 根据题设与模型的要求,作出如下假设: 1)假设所给read的质量值较大的数量足够多; 2)假设所给read的重复片段的数量少; 3)假设所给能够配对read的碱基重复数量大于模型所给的值; 三.符号说明
新一代高通量测序技术SOLiD简介
新一代高通量测序技术SOLiD简介 目前市场上有四种高通量测序仪,分别是Solexa,454 (GS-FLX),SOLiD和Polonator。根据测序原理,它们可以被分为两大类:使用合成法测序(Sequencing by Synthesis)的Solexa和454,及使用连接法测序(Sequencing by Ligation)的Polonator和SOLiD。这些高通量测序仪的共同点是不需要大肠杆菌系统进行DNA模板扩增,且测序所得序列较短:其中的454序列最长,为200~300个碱基,其余三种序列都只有几十个碱基。测序原理及序列长度的差异决定了各种高通量测序仪具有不同的应用领域。这就要求我们在熟悉各种高通量测序仪内在技术特点的基础上进行选择。 基因组所引进的SOLiD (Sequencing by Oligonucleotide Ligation and Detection)是ABI(Applied Biosystems)公司生产的高通量测序仪。目前这台SOLiD运行稳定,SOLiD实验及数据分析小组也可以为大家提供专业的技术服务。所以接下来的关键是如何把SOLiD测序仪应用到符合其技术特点的科研项目中。本短文将简单介绍SOLiD测序流程,双碱基编码原理及数据分析原理,以帮助大家了解SOLiD测序仪的技术特点和应用范围。 1.SOLiD关键技术及其原理 SOLiD使用连接法测序获得基于“双碱基编码原理”的SOLiD颜色编码序列,随后的数据分析比较原始颜色序列与转换成颜色编码的reference序列,把SOLiD颜色序列定位到reference上,同时校正测序错误,并可结合原始颜色序列的质量信息发现潜在SNP位点。 1.1. SOLiD文库构建 使用SOLiD测序时,可根据实际需要,制备片段文库(fragment library)或末端配对文库(mate-paired library)。简单地说,制备片段文库就是在短DNA片段(60~110 bp)两端加上SOLiD 接头(P1、P2 adapter)。而制备末端配对文库,先通过DNA环化、Ecop15I酶切等步骤截取长DNA片段(600bp到10kb)两末端各25 bp进行连接,然后在该连接产物两端加上SOLiD接头。两种文库的最终产物都是两端分别带有P1、P2 adapter的DNA双链,插入片段及测序接头总长为120~180 bp。 1.2:油包水PCR 我们知道,文库制备得到大量末端带P1、P2 adapter但内部插入序列不同的DNA双链模板。和普通PCR一样,油包水PCR也是在水溶液进行反应,该水相含PCR所需试剂,DNA模板及可分别与P1、P2 adapter结合的P1、P2 PCR引物。但与普通PCR不同的是,P1引物固定在P1磁珠球形表面(SOLiD将这种表面固定着大量P1引物的磁珠称为P1磁珠)。PCR反应过程中磁珠表面的P1引物可以和变性模板的P1 adapter负链结合,引导模板合成,这样一来,P1引物引导合成的DNA链也就被固定到P1磁珠表面了。 油包水PCR最大的特点是可以形成数目庞大的独立反应空间以进行DNA扩增。其关键技术是“注水到油”,基本过程是在PCR反应前,将包含PCR所有反应成分的水溶液注入到高速旋转的矿物油表面,水溶液瞬间形成无数个被矿物油包裹的小水滴。这些小水滴就构成了独立的PCR 反应空间。理想状态下,每个小水滴只含一个DNA模板和一个P1磁珠,由于水相中的P2引物和磁珠表面的P1引物所介导的PCR反应,这个DNA模板的拷贝数量呈指数级增加,PCR反应结束后,P1磁珠表面就固定有拷贝数目巨大的同来源DNA模板扩增产物。A BI公司提供的SOLiD 实验手册已经把小水滴体积及水相中DNA模板和磁珠的个数比等重要参数进行了技术优化和流程固定,尽可能提高“优质小水滴”(水滴中只含一个DNA模板一个P1磁珠)的数量,为后续SOLiD 测序提供只含有一种DNA模板扩增产物的高质量P1磁珠。
基因组序列的差异分析
基因组序列的差异分析 ----mVISTA的在线使用说明 当然,除了在线版的,我们还可以在网站上填写信息申请离线的软件。但我试用了一下,需要先自己比对,然后要按照一定的格式来制作文件,当然你还必须得安装java才能运行软件;总之,我感觉没有在线版的方便。 1 将数据放入服务器中 在首页,你将被要求确定你想要分析的基因组序列的数量。输入这个数字之后,点击“提交”,将带你到主提交页面。 mVISTA服务器最多可以同时处理100条序列。 1.1主提交页面必填的内容 E-mail 地址 通过E-mail,我们可以提示你的在线处理已经得到结果。
序列 你可以用2种方式来上传你的序列: 1.使用“Browse”按钮从你的电脑上,上传纯文本的Fasta格式文件。如果是一个作为参 考的生物体的DNA序列必须作为一个contig提交(可以进行一定的定向排列将多个片段合并为一个contig),而其他非参考序列可以在一个或多个contig中提交(draft)。 Fasta格式的示例序列(您可以在NCBI站点上找到关于该格式的更多细节): >mouse ATCACGCTCTTTGTACACTCCGCCATCTCTCTCT … !!!注意:序列里面我们只接受字母CAGTN和X。请确保提交序列是作为一种纯文本格式,而不是Word或HTML文件格式。 如果您以FASTA格式提交序列,我们建议您为它取一个有意义的名称(比如直接是你的物种名之类的),因为这些名称将出现在我们生成的图形中。如果您使用的是一个draft草图序列,那么结果中每个contigs的命名都将按照您在“>”符号后指示的命名进行。 2.您可以给出它的GenBank登录号,系统将自动从GenBank数据库里进行检索序列。 在这两种情况下,序列的总大小都不应超过10M,而且任何一条序列都不应超过2M。 1.2主提交页面选填的内容 这些选项允许您自定义您的VISTA分析。您可以使用独立获得的基因注释,选择合适的Repeat Masker选项,给分析的序列指定名称,并改变序列保存分析的参数。如果您没有填写这些选填选项,我们将使用它们的默认值。 比对程序 根据您分析的具体内容(参见“about”-链接中的详细信息),您可以选择以下比对程序之一:1、AVID----全局两两比对。如果您选择使用这个程序,其中一个序列应该被完成比对,其他 所有序列可以完成或以草图draft格式完成。对于集合中所有已完成的序列,AVID生成所有相对所有成对的比对结果,可以使用任何序列作为基础(参考)来显示。如果某些序列是草图格式,AVID将生成它们与最终序列的比对,这将被用作基础(参考)。这是该服务器上唯一可以处理草图序列的比对程序。 (小知识:草图序列与完整序列DNA sequence, draft: Sequence of a DNA with less accuracy than a finished sequence. In a draft sequence, some segments are missing or are in the wrong order or are oriented incorrectly. A draft sequence is as opposed to a finished DNA sequence.)2、LAGAN----完成完整序列的全局两两比对和多重比对。如果某些序列是草图格式,您的查 询将被重定向到AVID以获得两两比对。多重比对将由VISTA可视化,它将计算并显示序列的保守区,以您指示的任何序列作为参考。这是该服务器上唯一能够产生真正的多重
高通量测序:第二代测序技术详细介绍
在过去几年里,新一代DNA 测序技术平台在那些大型测序实验室中迅猛发展,各种新技术犹如雨后春笋般涌现。之所以将它们称之为新一代测序技术(next-generation sequencing),是相对于传统Sanger 测序而言的。Sanger 测序法一直以来因可靠、准确,可以产生长的读长而被广泛应用,但是它的致命缺陷是相当慢。十三年,一个人类基因组,这显然不是理想的速度,我们需要更高通量的测序平台。此时,新一代测序技术应运而生,它们利用大量并行处理的能力读取多个短DNA 片段,然后拼接成一幅完整的图画。 Sanger 测序大家都比较了解,是先将基因组DNA 片断化,然后克隆到质粒载体上,再转化大肠杆菌。对于每个测序反应,挑出单克隆,并纯化质粒DNA。每个循环测序反应产生以ddNTP 终止的,荧光标记的产物梯度,在测序仪的96或384 毛细管中进行高分辨率的电泳分离。当不同分子量的荧光标记片断通过检测器时,四通道发射光谱就构成了测序轨迹。 在新一代测序技术中,片断化的基因组DNA 两侧连上接头,随后运用不同的步骤来产生几百万个空间固定的PCR 克隆阵列(polony)。每个克隆由单个文库片段的多个拷贝组成。之后进行引物杂交和酶延伸反应。由于所有的克隆都是系在同一平面上,这些反应就能够大规模平行进行。同样地,每个延伸所掺入的荧光标记的成像检测也能同时进行,来获取测序数据。酶拷问和成像的持续反复构成了相邻的测序阅读片段。
Solexa高通量测序原理
--采用大规模并行合成测序法(SBS,Sequencing-By-Synthesis)和可逆性末端终结技术(ReversibleTerminatorChemistry) --可减少因二级结构造成的一段区域的缺失。 --具有高精确度、高通量、高灵敏度和低成本等突出优势 --可以同时完成传统基因组学研究(测序和注释)以及功能基因组学(基因表达及调控,基因功能,蛋白/核酸相互作用)研究 ----将接头连接到片段上,经PCR扩增后制成Library。 ----随后在含有接头(单链引物)的芯片(flowcell)上将已加入接头的DNA片段变成单链后通过与单链引物互补配对绑定在芯片上,另一端和附近的另外一个引物互补也被固定,形成“桥” ----经30伦扩增反应,形成单克隆DNA簇 ----边合成边测序(Sequencing By Synthesis)的原理,加入改造过的DNA 聚合酶和带有4 种荧光标记的dNTP。这些dNTP是“可逆终止子”,其3’羟 基末端带有可化学切割的基团,使得每个循环只能掺入单个碱基。此时,用激光扫描反应板表面,读取每条模板序列第一轮反应所聚合上去的核苷酸种类。之后,将这些基团化学切割,恢复3'端粘性,继续聚合第二个核苷酸。如此继续下去,直到每条模板序列都完全被聚合为双链。这样,统计每轮收集到的荧光信号结果,就可以得知每个模板DNA 片段的序列。目前的配对末端读长可达到2×50 bp,更长的读长也能实现,但错误率会增高。读长会受到多个引起信号衰减的因素所影响,如荧光标记的不完全切割。 Roche 454 测序技术 “一个片段= 一个磁珠= 一条读长(One fragment =One bead = One read)”1)样品输入并片段化:GS FLX 系统支持各种不同来源的样品,包括基因组DNA、PCR 产物、BAC、cDNA、小分子RNA 等等。大的样品例如基因组DNA 或者BAC 等被打断成300-800 bp 的片段;对于小分子的非编码RNA 或者PCR 扩增产物,这一步则不需要。短的PCR 产物则可以直接跳到步骤3)。 2)文库制备:借助一系列标准的分子生物学技术,将A 和B 接头(3’和5’端具有特异性)连接到DNA 片段上。接头也将用于后续的纯化,扩增和测序步
基因组组装算法研究(已审核)
基因组组装算法研究 摘要 基因组测序是生物信息学的核心,有着极其重要的应用价值。近些年来,新的测序技术大量涌现,与传统的Sanger方法相比,这些方法产生的read(由测序仪直接测得的DNA 片段)长度更短,数量更多,覆盖率更大。然而,传统的拼接算法并不适用于利用短read 进行拼接,新的拼接算法在拼接效果上仍有待提高。 本文首先介绍了传统的基因组拼接所用的贪婪算法和overlap-layout-consensus 算法,这两种算法仅适用用于第一代测序技术所得的reads,并不适用于第二代基因测序。对于第二代测序技术所得的reads,可以建立de bruijn 图算法的数学模型,然后编写程序,组装基因片段。利用第二代测序技术可以在一次实验中获得高通量短 read,然而第二代测序技术并不完美,由于在测序前要通过 PCR 手段对待测片段进行扩增,因此增加了测序的错误率。因此,本文利用HiTEC纠错算法对de bruijn 图算法进行优化。 另外,本文还利用了基于概率模型的基因组从头测序算法克服了原有拼接算法过度依赖碱基片段之间重叠信息的缺陷,创造性地将 DNA 拼接过程抽象为二阶离散马尔可夫过程,与此同时,每一条碱基片段被抽象为系统中的一个状态。 关键词:贪婪算法,OLC算法,de bruijn 图算法,HiTEC纠错算法
一、问题重述 遗传信息是生物遗传与进化的主要研究依据。能否快速和准确地获取生物体的遗传信息对于生命科学研是否有重大发现具有重要意义。对每个生物体来说,基因组包含了整个生物体的遗传信息,这些信息通常由组成基因组的DNA或RNA分子中碱基对的排列顺序所决定。获得目标生物基因组的序列信息,进而比较全面地揭示基因组的复杂性和多样性,成为生命科学领域的重要研究内容。 确定基因组碱基对序列的过程称为测序。测序技术始于20世纪70年代,伴随着人类基因组计划的实施而突飞猛进。从第一代到现在普遍应用的第二代,以及近年来正在兴起的第三代,测序技术正向着高通量、低成本的方向发展。现有的测序技术中,可按一定的测序策略获得长度约为50–100个碱基对的序列,称为读长(reads)。基因组复制份数约为50–100。基因组组装软件可根据得到的所有读长组装成基因组,这些软件的核心是某个组装算法。常用的组装算法主要基于OLC(Overlap/Layout/Consensus)方法、贪婪图方法、de Bruijn图方法等尽管如此,目前能直接读取的碱基对序列长度远小于基因组序列长度,因此需要利用一定的方法将测序得到的短片段序列组装成更长的序列。 一个好的算法应具备组装效果好、时间短、内存小等特点。新一代测序技术在高通量、低成本的同时也带来了错误率略有增加、读长较短等缺点,现有算法的性能还有较大的改善空间。 本题要求我们尝试建立模型,由程序计算得到基因组的长须组装。算法与程序要求能有效地解决在测序过程中出现的碱基对识别错误,或则基因中出现重复片段的情况。 将所建立的模型检查运行后,本题要求我们进一步对其进行探究。针对一个全长约为120,000个碱基对的细菌人工染色体(BAC),采用Hiseq2000测序仪进行测序,结合附录中的测序策略、数据格式以及读长数据,在测序长度约为70×的情况下,对上述所建立的模型与算法程序进行组装验算。 二、问题分析 本题是基于新一代测序技术的基因组装算法问题,要求设计算法针对性的解决新一代测序技术带来的一些弊端。 2.1 read长度较短,数量较多——de bruijn图
三代基因组测序技术简介及其原理整理.
三代基因组测序技术简介及其原理整理 第一代测序技术 第一代DNA测序技术用的是1975年由桑格(Sanger)和考尔森(Coulson)开创的链终止法以及1976-1977年由马克西姆(Maxam)和吉尔伯特(Gilbert)发明的化学法(链降解)。 1977年,桑格测定了第一个基因组序列——噬菌体X174,全长5375个碱基。自此,人类获得了窥探生命遗传差异本质的能力,并以此为开端步入基因组学时代。研究人员在Sanger法的多年实践之中不断对其进行改进。在2001年,完成的首个人类基因组图谱就是以改进了的Sanger法为其测序基础。 Sanger法原理: 1)在模板指导下,DNA聚合酶不断将dNTP(N=A/G/T/ C)加到引物的3’- OH末端,合成出新的互补链。在4个DNA合成反应体系中分别加入一定比例带有放射性同位素标记的ddNTP,在互补链在DNA聚合酶作用下延伸时,一旦连接上ddNTP,由于双脱氧核糖的2’和3’都不含羟基,故不能同后续的dNTP形成磷酸二酯键而终止反应,随即形成一系列不同长度的、以同样引物为起始、以同一碱基终止的短片段混合物。 2)双脱氧核苷酸在每个DNA分子中掺入的位置不同,采用聚丙烯酰胺凝胶电泳区分长度差一个核苷酸的单链DNA,从而读取DNA核苷酸序列。 化学裂解法原理: 与Sanger法类似,将DNA模板分成4个反应。在每个反应中,先在模板5’端进行放射性标记,再加入能特异性在其中一种碱基处切开DNA的化学试剂。反应进行时,平均一个DNA分子只在随机位点产生一次裂解。接着,通过凝胶电泳和放射自显影后可以根据电泳带的位置确定待测分子的DNA序列。 第二代测序技术 第一代测序技术的主要特点是测序读长可达1000bp,准确性高达99.999%,但其测序成本高,通量低等方面的缺点,严重影响了其真正大规模的应用。因而第一代测序技术并不是最理想的测序方法。经过不
三代测序技术的比较
一代、二代、三代测序技术 张祥瑞 2013/04/22 11:43 第一代测序技术-Sanger链终止法 一代测序技术是20世纪70年代中期由Fred San ger及其同事首先发明。其基本原理是,聚丙烯酰胺凝胶电泳能够把长度只差一个核苷酸的单链DNA分子区分开来。一代测序实验的起始材料是均一的单链DNA分子。第一步是短寡聚核苷酸 在每个分子的相同位置上退火,然后该寡聚核苷酸就充当引物来合成与模板互补的新的DNA链。用双脱氧核苷酸作为链终止试剂(双脱氧核苷酸在脱氧核糖上 没有聚合酶延伸链所需要的3-OH基团,所以可被用作链终止试剂)通过聚合酶的引物延伸产生一系列大小不同的分子后再进行分离的方法。测序引物与单链DNA莫板分子结合后,DNA R合酶用dNTP延伸引物。延伸反应分四组进行,每一 组分别用四种ddNTP(双脱氧核苷酸)中的一种来进行终止,再用PAG吩析四组样品。从得到的PAGE交上可以读出我们需要的序列。 第二代测序技术-大规模平行测序 大规模平行测序平台(massively parallel DNA sequencing platform )的出现不仅令DNA测序费用降到了以前的百分之一,还让基因组测序这项以前专属于大型测序中心的“特权”能够被众多研究人员分享。新一代DNA测序技术有助于 人们以更低廉的价格,更全面、更深入地分析基因组、转录组及蛋白质之间交互作用组的各项数据。市面上出现了很多新一代测序仪产品,例如美国Roche Applied Scienee 公司的454基因组测序仪、美国Illumina 公司和英国Solexa techno logy 公司合作开发的Illumi na 测序仪、美国Applied Biosystems 公司的SOLiD测序仪。Illumina/Solexa Genome Analyzer 测序的基本原理是边合成边测序。在Sanger 等测序方法的基础上,通过技术创新,用不同颜色的荧光标记四种不同的dNTP当DNA聚合酶合成互补链时,每添加一种dNTP就会释放出不同的荧光,根据捕捉的荧光信号并经过特定的计算机软件处理,从而获得待 测DNA勺序列信息。以Illumina测序仪说明二代测序的一般流程,(1)文库制备,将DNA用雾化或超声波随机片段化成几百碱基或更短的小片段。用聚合酶和外切核酸酶把DNA片段切成平末端,紧接着磷酸化并增加一个核苷酸黏性末端。然后将川umina测序接头与片段连接。(2)簇的创建,将模板分子加入芯片用于产生克隆簇和测序循环。芯片有8个纵向泳道的硅基片。每个泳道内芯片表面有无数的被固定的单链接头。上述步骤得到的带接头的DNA片段变性成 单链后与测序通道上的接头引物结合形成桥状结构,以供后续的预扩增使用。通 过不断循环获得上百万条成簇分布的双链待测片段。(3)测序,分三步:DNA 聚合酶结合荧光可逆终止子,荧光标记簇成像,在下一个循环开始前将结合的核苷酸剪切并分解。(4)数据分析 第三代测序技术-高通量、单分子测序
