芯片数据预处理方法
基因芯片数据预处理过程

基因芯片数据预处理过程
基因芯片数据预处理是指对原始基因芯片数据进行处理、清洗和标准化的过程。
下面是基因芯片数据预处理的主要步骤:
1. 数据导入和存储:将基因芯片数据从原始格式导入到计算机中,并确定存储格式,如矩阵形式。
2. 数据清洗:去除无效数据、缺失数据、异常值和重复数据,以确保数据的质量和一致性。
3. 数据标准化:由于基因芯片数据通常具有不同的量级和分布,需要对数据进行标准化,以便在后续的分析中比较和综合不同样本或基因的表达数据。
常用的标准化方法有Z-score标准化
和最大最小值归一化等。
4. 数据变换:对数据进行变换,以满足统计分析的假设前提。
常见的变换方法包括对数变换、幂变换和Box-Cox变换等。
5. 数据分割:将数据按照实验组和对照组分割,以便在差异分析中进行比较。
6. 批次效应校正:由于实验过程中可能存在批次效应,即同一批次下的样本可能具有相似的表达模式,因此需要对数据进行批次效应校正,以消除批次效应对差异分析的影响。
7. 基因筛选:基因芯片数据通常包含大量的基因,为了减少多重比较问题和提高模型的可解释性,需要对基因进行筛选,选
择具有显著差异表达的基因进行后续分析。
8. 数据集成和整合:将不同芯片平台或实验中得到的数据进行整合,以增加样本量和数据的可靠性。
以上是基因芯片数据预处理的一般步骤,根据具体的研究目的和数据特点,可能还会有其他特定的处理方法。
基因芯片图像的处理和分析方法研究

第35卷,增刊、,r 01.35Suppl em ent 红外与激光工程I nf r ar ed aIl d Las e r En gi ne er i n g 撕年l O 月oct .2006基因芯片图像的处理和分析方法研究张瑜(西南技术物理研究所,四川成都610041)摘要:对基因芯片图像进行预处理和分析。
分别采用伪中值滤波和网格定位对图像进行预处理和分析。
经过预处理后,图像的噪声得到了有效的消除。
经过不同的网格定位方法后,图像得到了全面的分析。
用网格定位的方法对经过预处理后的图像进行分析,为获得杂交后基因芯片探针二维荧光信息的关键特征奠定了基础。
为了提高运算效率和准确性,采用了中值滤波的改进法——伪中值滤波法对图像进行预处理。
同时使用了自适应阈值分割,对图像进行自动网格定位。
关键词:伪中值滤波;网格定位;自适应阈值分割中圈分类号l 1N 911.73文献标识码l A 文章编号:1007.2276(2006)增D .0219.04R es ear ch about t he m e t hod of pr oc ess i ng and anal ysi ng t he i m agesobt ai ne d w i t h ge ne -c hi p s c a n ne rZ H A N G Y u(S 伽恤w es t h 伽地of Tech 血al Phy s i cs ,aI 蛐gd I I 610041.aI ㈣A bs tr 躯t :11l e i m age s obt ai ned w i m gene-c 11i p s ca l l ner a r e pre —pr oces s ed aI l d anal yzed .U s i ng ps eudo m e di 柚fi l 锄ng and 班ddi ng ,m e i I I l ages ar e pre —pr oc es s ed aI l d aI l al yzed .A 讹r pre —pr ocess i ng ,血e i m age s ni oses a 陀ef !F &t i Vel y el i I ni n at ed .A n d t he i m ages ar e r oundl y anal y zed by 鲥ddi ng di 仃ere nt l y .T t l e ke y char act er of h ybr i di z ed gene —chi p ’2一D nuo r es cen ce i I l f om at i on is ob 妇ed by gr i d di ng 吐l e pre —pr oces s ed i I l l a ges .To i I n pr o V e Ⅱl e oper at i on e ff i c i e nc y and V er aci t y'ps eud o m edi aI l f i l t er i I 峪i s adoppt ed .A t t he s 锄e t i m e Ⅱl e i m ages a r e gr i d ed aut om at i ca ny by ail 印tiV e ‰shold se gI nent a t i on .K ey w or ds :P s eudo m edi 锄f i l t er i ng ;G r !i dding ;A dapdV e 吐鹏shol d se gm ent ;a t i onO引言基因是指导细胞或生物体生命活动的信息单位,它调控着细胞的活动和人的生老病死,基因探测被认为是当代生命科学的核心技术之一。
bioconductor系列教程之一分析基因芯片上
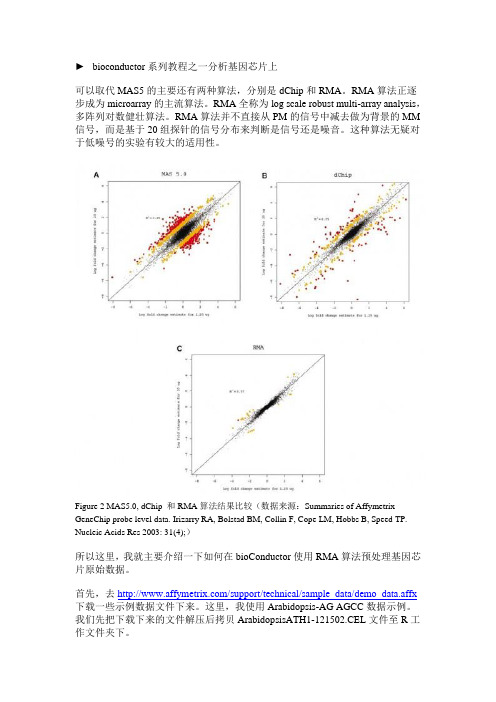
►bioconductor系列教程之一分析基因芯片上可以取代MAS5的主要还有两种算法,分别是dChip和RMA。
RMA算法正逐步成为microarray的主流算法。
RMA全称为log scale robust multi-array analysis,多阵列对数健壮算法。
RMA算法并不直接从PM的信号中减去做为背景的MM 信号,而是基于20组探针的信号分布来判断是信号还是噪音。
这种算法无疑对于低噪号的实验有较大的适用性。
Figure 2 MAS5.0, dChip 和RMA算法结果比较(数据来源:Summaries of Affymetrix GeneChip probe level data. Irizarry RA, Bolstad BM, Collin F, Cope LM, Hobbs B, Speed TP. Nucleic Acids Res 2003: 31(4);)所以这里,我就主要介绍一下如何在bioConductor使用RMA算法预处理基因芯片原始数据。
首先,去/support/technical/sample_data/demo_data.affx 下载一些示例数据文件下来。
这里,我使用Arabidopsis-AG AGCC数据示例。
我们先把下载下来的文件解压后拷贝ArabidopsisATH1-121502.CEL文件至R工作文件夹下。
首先是一个快速上手教程:1 2 3 4 5 6 7 8 910111213141516171819202122232425262728293031323334353637 > library(affy)##加载库文件Loading required package: BiobaseWelcome to BioconductorVignettes contain introductory material. To view, type'openVignette()'. To cite Bioconductor, see'citation("Biobase")'and for packages 'citation(pkgname)'.> Data <- ReadAffy()##读取工作目录下的CEL文件> eset <- rma(Data)##用RMA算法预处理数据,这时它会自动下载CDF文件,所以需要联网。
蛋白芯片分析流程

#流程大放送#蛋白质组芯片分析
Agonyr
蛋白质芯片是一种高通量的蛋白功能分析技术,可用于蛋白质表达谱分析,研究蛋白质与蛋白质的相互作用,甚至DNA-蛋白质、RNA-蛋白质的相互作用,筛选药物作用的蛋白靶点等。
应用蛋白质组芯片可以进行基因表达的筛选、抗原抗体检测、蛋白筛选、生化反应检测、药物筛选以及疾病诊断等,相比于质谱的昂贵报价以及数据量来说,蛋白质组芯片的优势更加明显。
常规分析流程:
1.芯片预处理,样本peak双向聚类
2.过滤分析,PCA分析,发现离群样本,PCA分析是一种降维技术,可以将多维(即蛋白peak数)的芯片数据投射到低维空间中。
相似的样本所在的点将彼此靠近,可以通过PCA 分析找到那些“离群”的样本。
3.分组差异peak的筛选,根据实验样本的分组情况进行差异峰的筛选,并进行聚类分析,确定差异峰(蛋白)及不同样本之间的互作关系。
一般认为在聚类图上距离越近的样本或差异峰之间的关系越密切。
4.疾病预测模型的构建,利用决策树,神经网络,SVM等机器学习方法来建立了分组诊断的模型,目的在于利用实验数据来筛选出一批靶标peak,并以此构建模型,以进行早期诊断、疾病预测和预后分析。
5.peak注释。
功能分析,从基因水平上研究差异peak的功能信息,从不能的处理实验对比发现疾病的分子机制。
chip实验

Chip实验存在的问题和挑战
• Chip实验技术仍面临成本高、实验操作复杂、数据分析难度大
等问题,需要进一步改进和优化
• 通过改进芯片设计和实验技术,可以降低实验成本和误差,提
高实验结果的可靠性
• 通过引入新的数据分析和生物信息学方法,可以提高数据分析
的准确性和效率,挖掘更多生物信息
对未来Chip实验的
信度和生物学意义
表达谱,常用的芯片类型有抗体芯片、
多肽芯片等
Chip实验的优缺点
Chip实验的缺点主要有成本高、实验操作复杂、数据分析难度大
• 芯片制作和实验操作需要较高的技术要求,成本较高
• 实验过程中容易产生误差,需要严格的实验质量控制
• 数据量庞大,需要专业的生物信息学知识和统计分析方法进行分析
特异性结合
Chip实验通常采用荧光标记或放射性
标记方法
• 探针是一段与目标分子互补的DNA
• 荧光标记法是通过荧光染料标记目标
或RNA序列
分子,然后通过荧光扫描仪检测信号
• 通过探针与目标分子的结合,实现对
• 放射性标记法是通过放射性同位素标
目标分子的检测
记目标分子,然后通过放射性探测器检
测信号
Chip实验的技术手段
• 网络图是一种用于展示基因或蛋白质之间相互关系的图像,可
以帮助理解生物过程中的相互作用
06
Chip实验技术的发展趋
势
Chip实验技术的创
新
• Chip实验技术的创新主要体现在芯片设计、实验技术、数据分
析方法等方面
• 芯片设计方面,可以通过优化探针排列、提高探针密度等方法,
提高芯片的检测灵敏度和特异性
1990年代末期,蛋白质组学芯片技术逐渐兴起
生物芯片技术原理

生物芯片技术原理生物芯片技术是一种在微型芯片上集成了生物学实验室所需基本组件的技术,它允许在单个芯片上进行高通量、高灵敏度和高可重复性的生物分子检测。
生物芯片技术在基因组学、蛋白质组学等领域具有广泛的应用前景。
生物芯片技术可分为两类:基于DNA和RNA的芯片和基于蛋白质的芯片。
本文将主要介绍基于DNA和RNA的芯片。
DNA芯片技术主要用于基因表达的研究。
其基本原理是在芯片表面上固定一系列已知基因序列的DNA探针,通过杂交实验检测样品中的核酸是否与探针杂交,从而实现对基因表达水平的分析。
生物芯片技术的主要流程包括样品处理、芯片制备、试验操作和数据分析。
一、样品处理:样品处理是整个实验中最为关键的一步。
主要包括RNA/DNA提取、放大、标记、杂交等。
样品的选择和质量的好坏决定了分析结果的准确性和可重复性。
二、芯片制备:芯片制备的主要步骤包括芯片表面处理、探针的合成和连接、芯片包覆等。
芯片表面的化学修饰能够改变探针的亲和性和特异性,从而优化芯片的检测性能。
三、试验操作:试验操作包括芯片杂交、成像和数据获取等。
芯片样品通过加热和振动使样品中的RNA/DNA与芯片上的探针结合,随后将样品从芯片上洗掉并用成像仪或扫描仪获得芯片上的图像数据。
四、数据分析:数据分析是生物芯片技术中最为繁琐和复杂的一个环节。
数据分析主要有三个方面:首先是图像预处理,包括背景校准、排除异常值等;其次是数据提取,包括简单或复杂的数据处理和统计分析;最后是结果呈现,通常通过聚类、差异表达分析等手段对结果进行可视化展示。
生物芯片技术具有样品需求量小、实验周期短、重现性强等优点。
它在医学、农业、环境保护等领域有着广泛的应用,如基因突变、疾病诊断、药物筛选、农作物育种、环境污染检测等领域。
近年来,生物芯片技术已经得到了广泛的应用和发展。
在医学方面,生物芯片技术被广泛应用于疾病的早期诊断、疗效评估和药物筛选等方面。
生物芯片技术也能从基因水平为疾病的发生与发展提供关键信息,对于个体化医疗有着巨大的潜力。
甲基化芯片标准流程

甲基化芯片标准流程全文共四篇示例,供读者参考第一篇示例:甲基化芯片是一种用于测量DNA甲基化水平的工具,通过芯片上的探针与被测样本中的DNA相互作用,可以快速而准确地获得DNA 甲基化信息。
甲基化芯片标准流程是指在进行甲基化芯片实验时需要遵循的一系列标准步骤,以确保实验的可靠性和重复性。
本文将介绍甲基化芯片标准流程,并详细解释每个步骤的操作方法和注意事项。
甲基化芯片标准流程主要包括样本准备、DNA提取、DNA甲基化反应、芯片杂交、芯片扫描和数据分析等步骤。
下面将逐步介绍这些步骤的具体操作流程。
第一步:样本准备在进行甲基化芯片实验之前,首先需要准备样本。
样本可以是从组织、血液、细胞等来源提取的DNA。
在提取样本之前,需要注意样本的保存和处理条件,避免DNA降解或受污染。
还需根据实验设计确定所需样本量,确保实验的顺利进行。
第二步:DNA提取DNA提取是甲基化芯片实验的关键步骤,它直接影响后续实验结果的准确性和可靠性。
DNA提取方法有很多种,常用的包括酚-氯仿提取法、离心柱法等。
在进行DNA提取时,需要注意避免污染和降解,确保提取的DNA质量和浓度符合实验要求。
第三步:DNA甲基化反应DNA甲基化反应是将DNA中的甲基化位点与甲基化标记物结合的过程。
在进行DNA甲基化反应时,需要选择适当的甲基化反应试剂和条件,确保反应的有效性和特异性。
还需对反应体系进行控制,避免非特异性反应的发生。
第四步:芯片杂交芯片杂交是将经甲基化的DNA样本与甲基化芯片上的探针相互作用的过程。
在进行芯片杂交时,需要注意控制温度、时间和杂交液的成分,以确保探针和样本之间的特异性结合。
还需避免芯片的污染和损坏,确保实验的顺利进行。
第五步:芯片扫描芯片扫描是将杂交后的芯片放入扫描仪中进行扫描,获取甲基化信号的过程。
在进行芯片扫描时,需要注意设置扫描仪的参数和检查芯片的扫描质量,确保获取准确的甲基化数据。
还需避免芯片的移位和损坏,以保证数据的可靠性和重复性。
LncRNA芯片分析自己总结

•lncRNA芯片分析lncRNA芯片分析修改时间2010/6/16 13:57:12 点击3210次1. 归一化lncRNA芯片采用的归一化的方法为quantile normalization。
2. 差异LncRNA的筛选lncRNA芯片中既有lncRNA的探针又有mRNA的探针,分别做差异基因的筛选,筛选方法同表达谱的筛选方法是一致的,参见表达谱的差异基因筛选。
3. 差异lncRNA的重注释lncRNA芯片注释不完善,因此需要将筛选出来的lncRNA进行重注释。
将差异lncRNA在基因组上位置上下游延伸,以寻找lncRNA附近的有功能的基因。
差异lncRNA重注释示例4. 差异lncRNA靶基因的预测lncRNA可能通过调控相应的mRNA发挥功能,因此有必要预测lncRNA的靶基因。
我们提取差异lncRNA和mRNA的序列,首先用blast进行初筛,之后用RNAplex进行进一步筛选,以预测lncRNA可能调控的mRNA。
差异lncRNA靶基因预测结果示例5. 差异lncRNA与靶基因共表达网络预测出lncRNA的靶基因后,并可进一步在mRNA的数据中探寻该mRNA是否发生表达量的变化。
由此构建差异lncRNA与靶基因相互作用网络图。
差异lncRNA与靶基因相互作用网络图。
方框代表lncRNA,圆形代表mRNA。
连线表示可能的调控关系。
节点面积越大,表示调控的mRNA越多,预示该lncRNA在调控网络中所起的作用可能越大。
6. 差异lncRNA与差异mRNA的共表达分析SBC Human lncRNA芯片能同时检测出差异表达的lncRNA和mRNA。
我们将差异lncRNA和差异mRNA在一组样品中进行共表达分析,可以发现与某个lncRNA具有相同表达模式的mRNA。
要求:每组数据3个或3个以上生物学重复实验组:对照组:lncRNA与mRNA共表达分析作用图,圆形带圈代表lncRNA,圆形代表mRNA。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
2.3 提取表达值
由于芯片数据的小样本和大变量的特点,导致数据分布呈偏态、标准差大。 对数转换能使上调、下调的基因连续分布在0的周围,更加符合正态分布,同时 对数转换使荧光信号强度的标准差减少,利于进一步的数据分析。
cDNA芯片:对双通道数据使用Cy5(红)和Cys3(绿)两种荧光标记分别标记 case和control样本的cDNA序列。扫描仪采用两种波长对基因芯片的图像进行扫 描,根据每个点的光密度值计算相对应的绝对表达量(intensity);然后图像分 析软件通过芯片的背景噪音以及杂交点的光密度分析,对每个点的intensity校 准,利用Cy5/Cy3的值获取case与control组不同基因的表达值ratio((R/G ratio);一般选择以2为底的对数转化数据,比如R/G=1,则 log2R/G=0,即认 为表达量没有发生变化,当R/G=2 或者,R/G=0.5,则log值为1 或–1,这是可 以认为表达量都发生两倍的变化。
信号检测与分析
基因芯片的实验流程(双通道)
单通道/双通道基因芯片实例
基因芯片数据分析:对从基因芯片高密度杂交点阵图中提取 的杂交点荧光信号进行定量分析,通过有效数据筛选和相关基因 表达谱聚类,发现基因的表达谱和功能之间的联系。
杂交完成后,要对基因芯片进行“读片”,即应用激光共聚焦荧光扫 描显微镜,对基因芯片表面的每个位点进行检测。
对数据的删除,通常是删去所在的列向量或行向量。一个比较常用的做法是,事 先定义个阈值M。若行(列)向量中的缺失数据量达到阈值M,则删去该向量。若未 达到M,有两种方法处理,一是以0或者用基因表达谱中的平均值或中值代替,另一 个是分析基因表达谱的模式,从中得到相邻数据点之间的关系,据此利用相邻数据点 估算得到缺失值(类似于插值)。填补缺失值( k临近法):利用与待补缺基因距离 最近的k个临近基因的表达值来预测待填补基因的表达值。根据邻居基因在样本中的 加权平均估计缺失值。
列向量mj=(m1j,m2j,…,mGj)表示在第j个条件下各基因的表达水平 (即一张芯片的数据);
元素mij表示第基因i在第j个条件下(绝对)基因表达数据。m可以 是R(红色,Cy5,代表样品组)。也可以是G(绿色,Cy3,代表对照 组)。
2.2 数据清洗(data cleaning)
经过背景校正后的芯片数据中可能会产生负值,还有一些单个异常大(或小)的 峰(谷)信号(随机噪声)。对于负值和噪声信号,通常的处理方法就是将其去除, 常见数据经验型舍弃方法有:标准值或奇异值舍弃法;变异系数法;前景值<200; 前景值-平均数/前景值-中位数<80%等等。然而,数据的缺失对后续的统计分析(尤 其是层式聚类和主成分分析)有致命的影响。Affy公司的芯片分析系统会直接将负值 修正为一个固定值。
在样品中呈高表达的基因其杂交点呈红色,相反,在对照组中高表达的 基因其杂交点呈绿色,在两组中表达水平相当的显黄色, 这些信号就代表了样品中基因的转录表达情况。
数据预处理分析流程:算法 (以cDNA芯片为例)
探针水平数据获得(计算机扫描图像)
数据预处理:背景处理、数据清洗、提取表达值、标准化、汇总
获取基因表达数据:判断差异基因表达
聚类和分析
1 探针水平数据(probe-level data)的获得
提取生物样品的mRNA并反转录成cDNA,同时用荧光素或同位素标记。在液 相中与基因芯片上的探针杂交,经洗膜后用图像扫描仪捕获芯片上的荧光或同位 素信号,由此获得的图像就是基因芯片的原始数据(raw data),也叫探针水平 数据。
获取探针水平的数据是芯片数据处理的第一步,然后需要对其进行预处理( pre-processing),以获得基因表达数据(gene expression data)。基因表达数据 是芯片数据处理的基础。
基因芯片探针水平数据处理的R软件包有affy, affyPLM, affycomp, gcrma等。
以下的数据处理都是对log2R/G的形式进行分析。
2.4 归一化
经过背景处理和数据清洗处理后的修正值反映了基因表达的水平。然而在芯片试验中, 各个芯片的绝对光密度值是不一样的,在比较各个试验结果之前必需将其归一化 (normalization,也称作标准化)。
2 预处理 2.1 背景(background)处理
背景处理即过滤芯片杂交信号中属于非特异性的背景噪音部分。一 般以图像处理软件对芯片划格后,每个杂交点周围区域各像素吸光度的 平均值作为背景,但此法存在芯片不同区域背景扣减不均匀的缺点。也 可利用芯片最低信号强度的点(代表非特异性的样本与探针结合值)或 综合整个芯片非杂交点背景所得的平均吸光值做为背景。
探针 荧光值
基因 表达值
计算机“读片”机理
将样品中的DNA/RNA标上荧光标记,则可 以定量检验基因的表达水平。
cDNA芯片、载有较长片段的寡核苷酸芯片采用双色荧光系统:目前常用 Cy3一dUTP(绿色)标记对照组mRNA,Cy5一dUTP(红色)标记样品组 mRNA
用不同波长的荧光扫描芯片,将扫描所得每一点荧光信号值自动输入计 算机并进行信息处理,给出每个点在不同波长下的荧光强度值及其比值,同 时计算机还给出直观的显色图。
基因芯片数据预处理
基因芯片(gene chip),又称DNA微阵列(microarray),是 由大量DNA或寡核苷酸探针密集排列所形成的探针阵列,其工作的基 本原理是通过碱基互补配对检测生物信息。
4个技 术环节
分类
基因芯片制备 样品制备mRNA提取等
杂交反应
实验要求:单通道—— 一张芯片检验一种状态 ; 双通道——差异表达基 因的筛选 储存的生物信息:寡核 苷酸芯片(常为单通 道)、cDNA芯片(常为 双通道)
背景处理之后,我们可以将芯片数据放入一个矩阵中:
m11
M
=
m21
M mG1
m12 L m22 L M mG2 L
m1N
m2 N
M
mGN
其中,各字母的意义如下:
N:条件数; G:基因数目(一般情况下,G>>N); 行向量mi=(mi1,mi2,…,miN)表示基因i在N个条件下的表达水平(这里 指绝对表达水平,亦即荧光强度值);
