文本挖掘在生物医学领域中的应用及其系统工具
计算机在医学中的应用

05
人工智能在医学领域的前沿探 索
Chapter
深度学习在医学影像识别中的应用
卷积神经网络(CNN)在医学影像识别中的应用
通过训练大量的医学影像数据,CNN能够自动学习和提取图像特征,实现病灶的自动检 测和定位。
生成对抗网络(GAN)在医学影像合成中的应用
GAN能够生成与真实医学影像相似的合成图像,为医学研究和教学提供丰富的数据资源 。
光学成像技术
如荧光成像、光学相干断层扫描等,应用于微观 层面的生物医学研究,揭示细胞和组织的结构和 功能。
分子成像技术
如PET、SPECT等,能够在分子水平上观察生物 体内的生理和病理过程,为精准医疗提供有力支 持。
生物信息学在基因组学等领域的应用
基因组学数据分析
利用计算机技术对大规模基因组数据进行处理、分析和挖掘,揭 示基因与疾病之间的关联。
分析医院业务流程,优化信息管理系统的功能和流程设计。
多系统集成
实现医院内部不同系统间的集成,提高信息共享和协同工作效率 。
决策支持功能增强
利用数据挖掘和分析技术,为医院管理层提供决策支持。
区域卫生信息平台构建
信息共享机制建立
01Байду номын сангаас
制定区域卫生信息共享标准和机制,实现不同医疗机构间的信
息互通。
平台可扩展性考虑
计算机在医学中的应用
汇报人:XX 2024-01-24
目录
• 计算机辅助诊断与治疗 • 医学信息系统建设与管理 • 生物医学工程中的计算机技术 • 药物研发过程中的计算机辅助技术 • 人工智能在医学领域的前沿探索
01
计算机辅助诊断与治疗
Chapter
医学影像处理技术
系统生物学和p4医学模式_理论说明

系统生物学和p4医学模式理论说明1. 引言1.1 概述系统生物学和P4医学模式作为现代生物医学研究领域中的重要理论模型,受到了广泛关注和应用。
系统生物学是研究生物体内大量组分相互作用及其整体行为的科学,涉及多个学科领域,如生物信息学、计算机科学和数学等。
P4医学模式则是以预防、个性化、精准和参与为核心原则,旨在构建更加全面、高效和个性化的医疗健康体系。
1.2 文章结构本文将首先对系统生物学进行定义和原理的介绍,包括其研究对象、基本概念以及相关技术与方法。
接着将探讨系统生物学在不同应用领域中的具体应用情况,并结合实际案例进行说明。
随后,将对P4医学模式进行界定,并概述其特点及发展。
进一步,将介绍P4医学模式在相关研究领域中的最新进展,并探讨其在临床实践中可能产生的深远影响和前景。
最后一部分,则是对系统生物学与P4医学模式的关系进行理论阐述,包括系统生物学在P4医学模式中的应用以及P4医学模式对系统生物学发展的影响。
1.3 目的本文旨在深入探讨系统生物学和P4医学模式这两个重要理论模型之间的关系并阐明它们在现代生物医学研究和临床实践中的重要性和应用价值。
通过对其定义、原理、应用领域、技术与方法等方面进行详细说明,并结合实际案例,将为读者提供全面而清晰的认识,帮助其更好地理解和运用这些理论模型。
最终旨在促进系统生物学和P4医学模式在医疗健康领域中的进一步发展和应用,为构建更加精准、个性化的未来医疗体系作出贡献。
2. 系统生物学2.1 定义和原理系统生物学是一门综合性的研究领域,它将生物学、数学、物理学和计算机科学等多个学科融合在一起,以整体的角度研究生物系统。
其基本原理是将生物体视为一个整体系统,通过分析组成该系统的各个部分之间的相互作用和关系,从而揭示生物系统的结构和功能。
2.2 应用领域系统生物学在诸多领域中都有广泛的应用。
例如,在基因调控网络研究中,通过利用大量实验数据进行建模和仿真分析,可以揭示基因间复杂的互作关系,并探索调控网络对于特定生理过程的影响。
基于关键词共现的可视化技术的精神病专科医院文献主题分析

由图 1 见 , 可 与关 键词 “ 郁症 ” 现 次数 较 多 抑 共 的药 物关 键 词 主 要 有 “ 西 汀 ” “ 酞 普 兰 ” 说 明 氟 、西 , 该 院较 关 注 这 两 种 药 物 在 治 疗 抑 郁 症 中 的 应 用 。 与“ 抑郁 症 ” 现 次 数 较 多 的 诊 断 检 查 方 面 的 关 键 共
河南省精神病院论文关键词共现关系见 图2 。
图 2 河南省精神病 院论文关键词共现网络图
图 2显示 , 河南省精神病 院对儿童和青少年的
精神 疾病 有一 定 研 究 。 图 2还 显 示 , 抑郁 症 共 现 与
・
4 6
中华医学图书情报杂志 2 1 9月 第 2 0 1年 0卷第 9期
n lg o o y;bi i me r bl o ty;me a o p t ntlh s ia l
浙 江 省 杭 州 市 医学 情 报 中 心 应 杭 州 市 第 七 人
l 材料 与方 法
1 1 数 据 来源及 处理 方 法 .
民医院的要求 , 对全 国精神疾病治疗领域实力较强
的2 7家精 神病 专科 医院 20 — 0 0年在 国 内外 发 06 2 1 表 的科研 论 文 进 行 了统 计 分 析 。单 从 发 文 量 上 来 看 , 京 脑科 医 院 、 南 省 精 神 病 院 、 南 河 山东 省安 康 医 院、 中南 大 学 湘 雅 二 院 精 神 卫 生 研 究 所 、 海 市 精 上 神卫 生 中心 这 5家 医疗 机 构 的发 文 数量 最 多 , 与 且 其他 2 2家机 构发 文量 相 差 较 大 。然 而 传 统 的文 献 计 量方 法 只能对 文 献 的数 量 特 征 进 行分 析 , 能深 不 入 文献 的 主题 内容 。而 关 键 词 能 够 反 映 文 献 涉 及 的领域 和 内容 … 。因 此 我 们 采 用 关 键 词 共 现 的 方 法 , 过 可视 化技术 对 发 文 量 居 前 的 5家 医 院 的论 通 文 内容进 行揭 示 , 以发 现其 近 5年的研 究热 点 。
基于整合的TCGA数据库探索基因组学与临床数据关系

基于整合的TCGA数据库探索基因组学与临床数据关系一、本文概述随着生物信息学和临床研究的不断深入,基因组学与临床数据之间的关联日益成为生物医学领域的研究热点。
本文旨在通过整合和分析公开的The Cancer Genome Atlas(TCGA)数据库,探索基因组学与临床数据之间的关系。
我们将系统介绍如何利用TCGA数据库的资源,运用生物信息学方法,挖掘基因组学数据中的潜在信息,并与临床数据进行整合分析,以期揭示癌症发生、发展过程中的关键基因和分子机制,为癌症的诊断、治疗和预后评估提供新的思路和方法。
本文将首先介绍TCGA数据库的概况和数据特点,阐述选择TCGA 数据库作为研究基础的原因。
随后,我们将详细介绍基因组学数据的处理方法,包括数据清洗、基因表达分析、基因变异检测等,并阐述如何将这些方法与临床数据进行有效整合。
在结果展示部分,我们将通过图表和统计分析,展示基因组学与临床数据之间的关联,并解释这些关联在癌症研究中的意义。
我们将讨论本文的局限性,并对未来的研究方向进行展望。
通过本文的研究,我们期望能够为深入理解癌症的基因组学特征和临床表型提供新的视角和工具,为癌症的精准医疗提供科学支持。
我们也希望本文的研究方法和结果能够为其他领域的生物医学研究提供借鉴和参考。
二、TCGA数据库概述The Cancer Genome Atlas (TCGA) 是一个由美国国家癌症研究所(NCI)和国家人类基因组研究所(NHGRI)共同发起的项目,旨在通过应用高通量的基因组测序技术,对多种类型的人类癌症进行深入的基因组学研究。
自2006年启动以来,TCGA已经产生了海量的多维度数据,包括基因组、转录组、表观组、蛋白质组以及临床数据等,涵盖了超过33种不同类型的癌症,总计数千个患者的样本。
TCGA数据库不仅提供了丰富的原始测序数据,还通过严格的数据处理和分析流程,生成了大量的二级和三级数据,如基因变异注释、基因表达量统计、生存分析等。
常用的生物信息学软件的介绍和文献依据
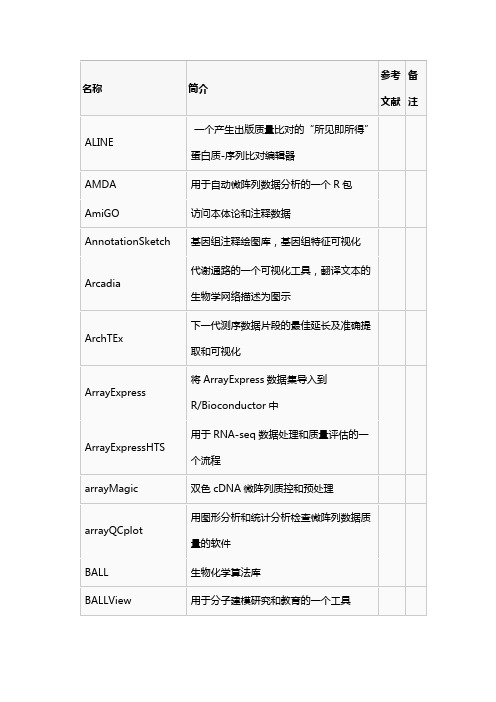
BioWarehouse
一个生物信息学数据仓库整合工具包
birgHPC
为生物信息学和分子动力学创建即时计算集群,自启动linux发行版
Biskit
python编写的一个结构生物信息学软件平台(库)
BisoGenet
一个新的基因网络构建、可视化和分析工具,cytoscape插件
一个促进高通量测序分析的基于云计算的框架
ESBTL
用于生物大分子结构和几何分析的高效PDB剖析器和数据结构
Expander
一个整合的基因表达数据分析软件平台,支持微阵列数据
分析的所有阶段
ExpressionPlot
一个分析RNA-Seq和微阵列基因表达数据的基于网络的框架
EZ-Viz
用标签和按钮简化PyMOL中分子查看
ChIPpeakAnno
一个注释ChIP-seq和ChIP-chip数据(峰)的Bioconductor包
ChIPseqR
核小体定位和组蛋白修饰ChIP-seq实验分析
Chipster
用于微阵列和其他高通量数据的用户友好的分析软件
CisGenome
一个分析ChIP-chip和ChIP-Seq的整合软件系统
病毒的传播和重组事件
J-Express
使用Java来探索基因表达数据
Jalview
Java多重序列比对编辑器
Java Treeview
微阵列数据可视化,树状图查看
JBrowse
下一代基因组浏览器,通过平滑地动态移动,缩放,导航基因组注释
jClust
一个聚类和可视化工具箱
JColorGrid
生物学测量值可视化,绘制热图,颜色网格等
HotDataSpider,一个生物医学文献热点数据自动化抽取工具

计 算 机 与数 字工 程
C mp tr& Dii lEn ie r g o ue gt gn ei a n
Vo. 8 No 1 13 . 1
6 9
21 0 0年第 1 期 1
HoDaa pd r 一 个 生 医 学 文 献 热 点 数 据 t tS ie , 物 自动 化 抽 取 工 具
i u l e o i r s n p b i r p st i .Th s k n fs p lme t r t r l l b a d Ho Da a n t i p p r c o e i id o u p e n a yma e i l en me t t .I h s a e ,wed v l p d Ho Da a p - a wi e eo e t t S i
生 了大量有 意 义 的数 据 。在 学 术 期 刊 以文 献 形 式
站 的附 加数 据正 在 呈 爆 炸性 的速 度增 长 。以 E m— b jh t :/ o ( t / www. aue cm/mb j) 志 为例 , p n tr. o e o/ 杂
2 0 仅 有 5 文 献 提供 了不 到 1 M 的 附加数 0 0年 0篇 8
( iaUK Chn - HUS RRe o n t gn eig a d Ge o c on b rt re T- sCr pGe ei En ie rn n n misJ itLa o ao y ,W u a 4 0 7 ) c h n 3 04
( h n h i n e o on o ma i n Te h oo y ’ h n h i 2 0 3 ) S a g a Ce t rf rBii f r t c n l g a ,S a g a 0 2 5 o
生物医学基因测序数据的分析方法

生物医学基因测序数据的分析方法生物医学领域中的基因测序技术,可以对个体基因序列进行扫描,识别出存在的个人基因突变或者DNA多态性等情况。
但是,基因测序数据的处理是一个非常复杂的难题,因为数据量庞大,而且各种测序技术和仪器的校准也存在一定的误差。
因此,在生物医学领域,如何分析基因测序数据,是一个关键的问题。
基因测序数据分析方法主要分为以下几个方面:1. 数据预处理针对基因测序原始数据,首先需要进行质量控制。
包括去除低质量的序列,读长校验,去除3’和5’端低质量数据等。
另外,需要将测序结果转化成bam格式,便于后续进一步分析。
2. 变异检测基因测序数据中,会出现多种类型的变异。
如单核苷酸多态性(SNP),结构变异(SV)等。
针对这些变异,需要对测序数据进行多阶段的处理,包括对序列比对、变异检测和筛选等步骤。
在变异检测的过程中,一般使用SNP、INDEL、SV等不同类型的变异检测工具,进行变异的检测和过滤,这些工具会根据不同类型的变异执行不同的检测流程,并提供一些基因注释信息等。
3. 数据注释注释是将生物实体(如基因、蛋白质等)与相关知识库中的信息进行对应和链接。
基因测序数据中的变异类型有很多,常规的注释包括突变的位置、基因功能和疾病相关信息等。
现在,随着大数据、深度学习等技术的不断发展,生物医学数据分析技术也在快速进化。
比如,深度学习文本挖掘技术,可以将海量的文献资料进行深入挖掘,挖掘到有用的医学研究信息,从而更好地指导医学研究,并提高基因测序数据的准确性。
4. 数据可视化数据可视化是将复杂的数据信息进行展示和呈现,以便更好地理解生物医学数据。
目前常用的可视化工具包括R、Python、IGV等,可以将变异信息、基因表达等结果图形化展示。
同时,基于网络和交互式的可视化技术,可以实现更精细的可视化效果。
综上所述,生物医学领域中基因测序数据的分析方法,需要从多个角度入手,例如数据预处理、变异检测、数据注释和数据可视化等。
semeds原理

semeds原理一、什么是semeds?semeds是一种语义推理引擎,全称为Semantic MEDline database。
它是通过在文献数据库中搜索、解析和分析生物医学文献来构建关联性网络的。
semeds的目标是帮助研究人员从大量的文献中提取有用的信息并进行语义推理,以便更好地理解和发现关于疾病、基因、药物等领域的知识。
二、semeds原理semeds的工作原理可以分为以下几个步骤:1. 文献收集首先,semeds通过在主要的生物医学文献数据库(如PubMed)中进行关键词搜索来收集相关的文献。
这些文献涵盖了各种生物医学领域的研究,包括疾病、基因、药物等。
2. 文献解析接下来,semeds对收集到的文献进行解析。
它使用自然语言处理技术和文本挖掘算法来提取文献中的关键信息,如标题、摘要、作者、关键词等。
3. 语义标注在文献解析的基础上,semeds对提取到的关键信息进行语义标注。
它使用一些已有的词汇表和本体文件来将文献中的词语与特定的概念进行关联。
这些概念可以是疾病、基因、药物等。
4. 关联网络构建通过语义标注的结果,semeds构建了一张关联网络。
在这个网络中,每个节点表示一个概念,如疾病或基因,而连接不同节点的边表示它们之间的关系。
这个关联网络可以帮助研究人员识别不同概念之间的相互作用和关联。
5. 语义推理最后,semeds利用构建的关联网络进行语义推理。
它可以根据已有的知识和关联性,推断出新的关联或提供相关的推荐。
这有助于研究人员发现新的研究方向、分析相关概念之间的相互影响等。
三、semeds的应用semeds在生物医学领域有着广泛的应用。
以下是semeds在不同领域的应用实例:1. 疾病研究semeds可以帮助研究人员系统地分析和理解不同疾病之间的关联。
通过分析疾病相关的文献,semeds可以识别不同疾病之间的共同基因、药物治疗等信息,从而有助于研究人员发现新的治疗方法或确定疾病之间的遗传关系。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
・56- 中华医学图书情报杂志2010年4月第19卷第4期 Chin J Med Libr Inf Sei,Vo1.19 No.4 Apr.2010 ・生物医药信息研究 文本挖掘在生物医学领域中的应用及其系统工具 吕婷 ,姜友好 [摘要]系统介绍了生物医学文本挖掘的具体流程和文本挖掘技术在生物医学领域中的应用情况,并着重从自然语言处 理和本体、命名实体识别、关系抽取、文本分类与聚类、共现分析、系统工具及评价、可视化等方面分别做了阐述。 [关键词】生物医学文本挖掘;自然语言处理;命名实体识别;关系抽取;共现分析 [中图分类号]R318;G254.0 文献标识码:A [文章编号]1671-3982(2010)04-0056-09
Application of text mining in biomedical field and its system tools LU Ting ,Jiang You—hao。 (1.Medical Library of Chinese PLA,Beijing 100039,China; 2.Department of Medical Information,Zhongnan University,Changsha 410013,Hunan Province,China)
[Abstract]the specific processes of text mining in biomedicine and the application of text mining technology in biomedical field were introduced in detail with stress laid on the natural language processing,ontology,named entity recognition,relationship extraction,text classification and clustering,CO—occurrence analysis,system tools and their evaluation,and visualization. [Key Words]text mining in biomedicine;natural language processing;named entity recognition;relation- ship extraction;CO—occurrence analysis
1文本挖掘概述 1.1概念 数据挖掘(Data mining),又称数据库知识发现 (Knowledge discovery in database),是指从结构化信 息中提取人们感兴趣的知识。这些知识是隐含的、事 先未知的、潜在的有用信息。文本挖掘(Text mining) 是数据挖掘的一个方向,它所挖掘的对象是非结构 化或半结构化,即从数以百万计的文本数据中寻找 潜在规律和趋势。文本挖掘在商业、传媒、教育、政 府、银行及生物技术、医疗卫生等行业领域都发挥 着不可忽视的作用 。搜索引擎、自动邮件回复、 垃圾邮件过滤、客户关系管理、自动简历评审等都 是典型的文本挖掘技术。 1.2流程及模型 文本挖掘的基本思想是利用文本切分技术抽
[作者单位】1.解放军医学图书馆,北京100039;2.中南大学医学 信息系,湖南长沙410013 [作者简介]吕婷(1985一),女,陕西宝鸡人,本科,发表论文5篇。
取文本特征,将文本数据转化为计算机能识别的结 构化数据,然后利用聚类、分类等数据挖掘技术形 成结构化文本,并根据该结构发现新的概念及获取 相应的关系。构成模型如图1所示。
数据源h叫源文本卜_T_叫结构化数据r_T叫知识或模型卜_T—叫知识 旦旦 唪 l
巨 ! i固 l共 竺析I l 盒 图1文本挖掘基本模型 1.3技术 文本挖掘涉及多个学科领域,如数据库、信息 检索、信息提取、机器学习、自然语言处理、计算语 言学、统计数据分析、图论等。文本挖掘按照挖掘 对象分为两类。一是单文本的数据挖掘,主要涉及 的挖掘技术有文本摘要、信息提取(包括名字提取、 短语提取和关系提取等)。二是文本集的数据挖 掘,主要技术有文本分类、文本聚类、个性化文本过 中华医学图书情报杂志2010年4月第19卷第4期 Chin J Med Libr Inf Sci。Vo1.19 No.4 Apr.2010 ・57・ 滤、文档作者归属、因素分析等。 以“预处理”过程为例,需要对文本数据做以下 预处理:消除噪声和冗余数据,推算缺失数据,数据 缩减,对元数据进行标记,词性标记,短语边界辨 认,对特征项量化处理等口J,最后形成计算机可处 理的结构化信息。 2生物医学文本挖掘 几个世纪以来,虽然科学信息都呈指数级增 长,但现代医学文献数量之多仍让人印象深刻 J。 遗憾的是,人们对信息处理及分析的速度远远落后 于信息本身的增长,从而产生了信息过载的问 题_4]。生物信息文本挖掘就是通过计算机,帮助人 们从爆炸式增长的生物医学自然语言文本数据中 发现知识、抽取关系,减轻研究人员信息超载的负 担。总的来说,生物医学文本挖掘可以从文献中抽 取出特定的事实信息(主要是生物实体如基因、蛋 白质、药物、疾病之间的关系),对整个生物知识网 络的建立、生物体关系的预测、新药的研制等均具 有重要的意义 。 2.1 自然语言处理与本体 2.1.1 自然语言的模糊性 计算语言学的研究使人们更关注对语言的理 解分析。自然语言的模糊性使找到句子含义变得 复杂,常常会出现不同的理解,如词汇歧义、句法歧 义、语义歧义等。词汇歧义也称词类歧义和类别歧 义,主要是因为一个单词可能有不止一种词性。自 然语言的文法通常是模棱两可的,这就出现了句法 歧义,如“AFB 1 binds preferentially to DNA with an alternating G-C sequence compared to DNA with a se- quence of contiguous Gs or Cs”与“GMPPCP binds to tubulin with a low affinity relative to GTP or GDP”。 第一句中的“with”引出的介词短语修饰前面的 “DNA”,第二句中的介词短语则是修饰“bind”前的 “GMPPCP”,而并非“tubulin”。因为一个句子通常 可能有多棵剖析树(Parse Tree),只有依靠语意及前 后文意思,才能在其中选择一棵最适合的树。语义 歧义涉及句子意思解释的问题,单词有不同含义时 就会出现,如figure在“figure indicate”中指的是数 字,而在“a good figure”中则指的是身材 。所以, 要真正理解人类语言,需要有广泛的知识并要结合 语境,而不是仅了解语言本身。 2.1.2自然语言处理的应用 自然语言处理(Natural language processing, NLP)是人工智能(Artificial Intelligence,AI)和语言 学领域的分支学科,主要用于中文自动分词(Chi. nese word segmentation)、词性标注(Part—of—speech tagging)、句法分析(Parsing)、自然语言生成(Natural language generation)、文本分类(Text categorization)、 信息检索(Information retrieva1)、信息抽取(Informa. tion extraction)、问答系统(Question answering)、机器 翻译(Machine translation)、自动摘要(Automatic summarization)等。自然语言处理也可用于临床决 策支持。如Joshua Denny等 调查发现,利用NLP 和正则表达式查询心脏病专家对心电图的解释,可 以更有效地识别QTc延长和其他心电图异常报告。 2.1.3 基于自然语言处理技术的文本挖掘系统 基于自然语言处理技术的文本挖掘系统有 MetaMap,IndexFinder,MedScanI ,GeneWays E9-10],
PASTA… 等。MedLEE系统提取UMLS概念的查全
率和查准率已分别达到83%和89%l12]。P.Karina Tulipano等 将BioMedLEE系统应用于分子成像 领域,使用自然语言处理技术,通过结构化自由文 本,找到相关的图像说明和文献,以协助自动标引 和组织图像。如果没有一种方法能组织这些图像, 很难完成图像的比较研究,成像技术解决方案如基 于内容的图像检索将受到限制。结果此次 BioMedLEE的查全率和查准率达到了0.74和0. 70。袁毅等 称其开发的基因相关文献挖掘网络 平台是我国唯一基于自然语言处理的文本挖掘系 统,能够通过文献获取、语法处理、语义处理、信息 整合及可视化等步骤实现基因功能、基因与疾病关 系、生物分子相互作用网络知识发现,辅助形成生 物科学研究创新假设,准确率达86%。 2.1.4本体 在生物医学领域中,本体(Ontology)已经广泛 用于领域专业知识的结构化组织。本体是对概念 体系的明确的、形式化、可共享的规范说明。大量 面向医学的本体被集成在一体化医学语言系统 (UMLS) 。 2.1.4.1 UMLS
一体化医学语言系统(UMLS)是对生物医学科 学领域内许多受控词表的一部纲目式汇编,收录了 100多部受控词表和分类系统,如ICD-9一CM,ICD- 10,MeSH,SNOMED CT,LOINC,世界卫生组织药物
