宏基因组实验计划
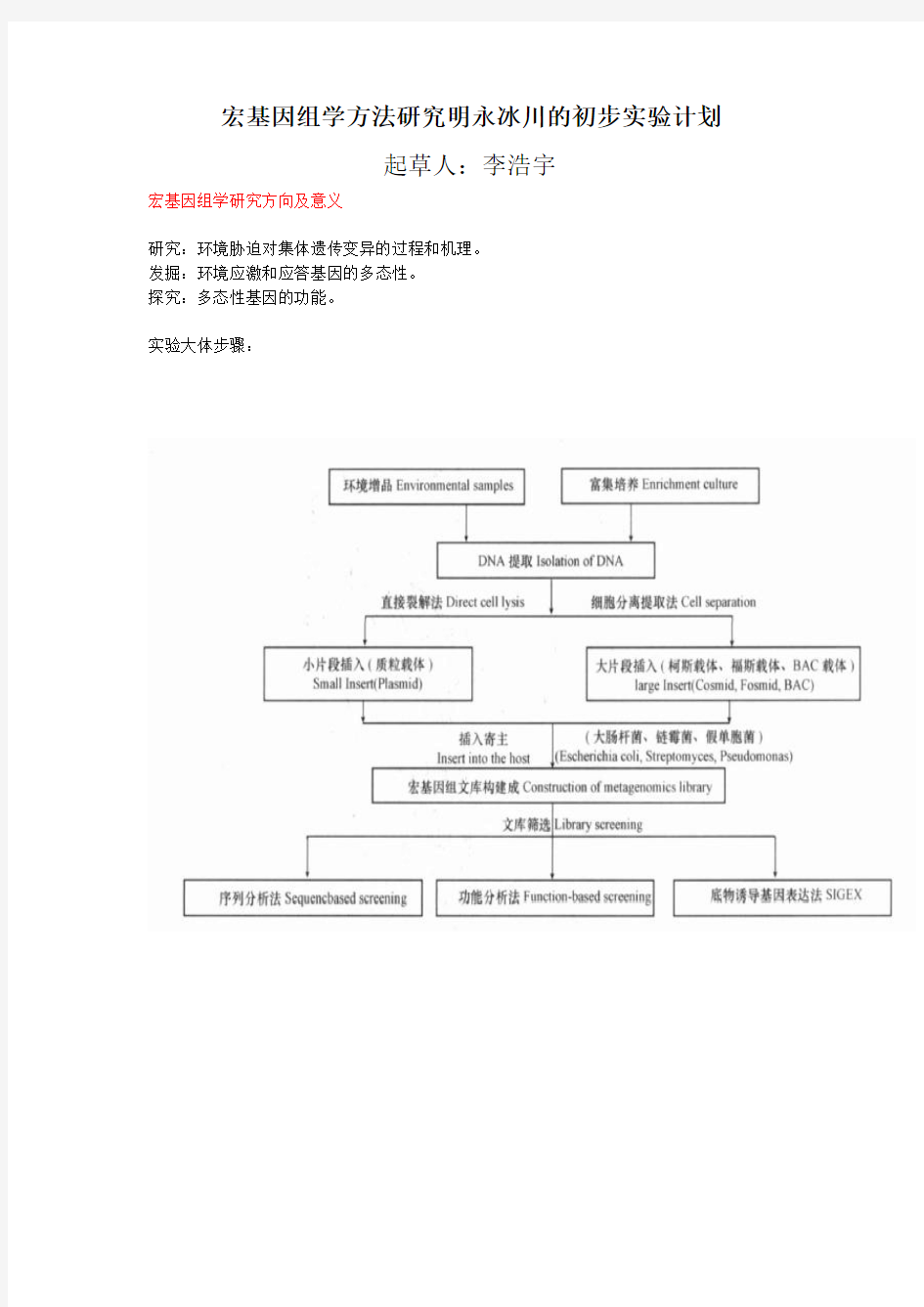

宏基因组学方法研究明永冰川的初步实验计划
起草人:李浩宇
宏基因组学研究方向及意义
研究:环境胁迫对集体遗传变异的过程和机理。
发掘:环境应激和应答基因的多态性。
探究:多态性基因的功能。
实验大体步骤:
I.环境样品的采集:
1)避免人源污染,采样的器具都要高温灭菌,采样过程中要戴手套。
2)样品的迅速处理保存。有条件最好用液氮冷冻运输,没有条件可以将装有样品的容器至于干冰泡沫箱中。带回实验室之后要尽快处理,特别是固体样品,迅速用预冷缓冲液(多为PBS)重悬,离心收集沉淀。分装成小份,冻存于液氮或者‐80 ℃冰箱中,避免反复冻融。
3)如果后续用间接法提取总DNA 的话,此处需要收集菌体,采取的策略是稀释之后的差速离心,一般适用于水样。
II.DNA提取方法的选择:
总DNA 的提取按照处理样品方式的不同划分为直接提取法和间接提取法。
直接提取法就是直接对样品进行裂解,释放其中的DNA。
间接提取法则是先分离样品中的微生物,后对微生物进行裂解。两种方法的适用范围不同,各自都有优缺点。
直接提取法的优缺点:直接提取法多用在提取固体样品(如土壤、污泥、湖泊沉积物等) 总DNA 的试验中。其最大的优点在于得率高,在某些环境样品总DNA 提取实例中,比间接提取法高数倍至数百倍。造成这种情况的最主要原因一是环境样品中存在丰富的胞外DNA;二是许多微生物与基质形成复杂的结构,间接提取法不易对其进行分离。在样品较珍贵时多采用此法。其缺点同样明显,所得DNA 的纯度远不及间接提取法所得,基质中含的腐殖质一并被保留下来,使得所得DNA 呈现黄褐色甚至黑色。
可以考虑MoBio 和Epicentre的DNA提取的商业试剂盒(主要用于提取土壤样品)。
间接提取法的优缺点:间接提取法则既可以用来提取固相样品总DNA 也可以用来提取液体样品总DNA。在提取液体样品(如海水、河流等样品)总DNA 时,需要用抽滤的方式浓缩。与直接提取法刚好相反,其最大的优点在于提取DNA 纯度高,既使在提取固体样品微生物总DNA 时,也可以用缓冲液对收集的菌体进行反复冲洗以去除表面所带杂质。其缺点是DNA 得率低。
土壤DNA 直接提取法:
(1) 从超低温冰箱中取出保存样品(5 g);
(2) 于室温融化;
(3) 加入13.5 ml CTAB 抽提缓冲液和50 μl 蛋白酶K 贮存液;
(4) 离心管于37oC 225 rpm 水平振荡30 min;
(5) 加入1.5 mL20%SDS;
(6) 65oC 水浴2 h,每隔15 min 轻轻颠倒数次;
(7) 室温6,000g 离心10 min,收集上清液,转移至一个新的50 ml 离心管中;
(8) 沉淀中再加入4.5 ml 提取液和0.5 ml 20%SDS,轻轻涡旋至混匀;
(9) 浸入液氮中冷冻3 min 后,于沸水中煮沸3 min;
(10) 室温6,000g 离心10 min,收集上清液,与上次上清液合并;
(11) 重复(8)、(9)、(10)一次;
(12) 将三次提取所获上清液与等体积的苯酚:氯仿(1:1 v/v) 混合,摇匀后于4 ℃12,000g 离心10 min;
(13) 将上层水相转移至一新的50 ml 离心管中,加入等体积的氯仿:异戊醇(24:1),混匀后于4 ℃12,000g 离心10 min;
(14) 再次转移上清至一新的离心管中,加入0.6 倍体积的异丙醇于室温沉淀1 h;
(15) 室温12,000g 离心20 min;
(16) 收集沉淀,用预冷的70%乙醇洗涤沉淀;
(17) 向离心管中加入1 ml TE,于4 ℃放置过夜,分装保存于‐20 ℃。
实验过程中的注意事项及一些技巧:
(1) 整个提取过程吹吸动作要轻柔,避免剧烈震荡(试剂盒提取除外)。
(2) 用到的枪头在灭菌之前最好将头部剪掉,避免机械剪切。
(3)酚氯仿抽提时,中间相一定不要吸到,宁可多丢弃一些。
(4) 异丙醇沉淀时不要放在低温,0.7 倍体积室温沉淀足够了。
(5) DNA 溶解时可以放在4℃静置过夜,次日离心取上清去除多余杂质。
(6) 水平震荡可以用左右摇晃的震荡器。
水样DNA 间接提取法:
(1)抽滤的方式浓缩采集到的水样。
(2)采取差速离心取得水样中的菌体。
(3)取得微生物后进行裂解提取。
此方法还在查证中,具体前期对样品的处理方法还在整理中。
例如:用多少的水样进行浓缩,差速离心的转速选择。
会不会有DNA的丢失等等。
Epicentre推出了宏基因组DNA分离试剂盒(Metagenomic DNA Isolation Kit for Water),能从水样中分离已随机剪切的,可直接用于fosmid克隆的DNA片段。
此试剂盒能从水样中的培养或未培养微生物(包括革兰氏阳性菌)中提取40kb大小的DNA。提取的DNA可立即进行末端修复,并在乙醇沉淀洗涤后克隆到Epicentre的PCC1FOS 载体上。
优势:
1.无需化学剪切,琼脂糖块或大小选择。
2.无需纯化柱或酚/氯仿提取。
3.产量更高,从低丰度的微生物中回收DNA的概率更大。
4.Fosmid载体与DNA的克隆只需要90分钟。
下图为方法与流程:
粗提DNA 的纯化
采用直接提取法提取的总DNA 或多或少都含有腐殖质的存在。它的存在对后续的分子生物学操作会有影响,时常会导致PCR 扩增不出来,酶切困难,连接转化率低。需要进行纯化。纯化的方法包括:胶回收,柱纯化,电泳/电洗脱,梯度离心。
a 胶回收
胶回收最大的优点在于快捷,可以在很短的时间内完成。纯化后的DNA 往往能够满足PCR 要求。但其缺点在于,膜截留式的纯化方式难免会对DNA 分子造成机械损伤,使得DNA 弥散,完整度下降。如果后续的实验室分析16S r DNA,则这种纯化方式不失为一种好方法。
b 柱纯化
柱纯化相对于胶回收来说更为快速,十几分钟内可完成。与胶回收相比,其纯化效果要差一些。但通常情况下,纯化之后的DNA 也能够满足PCR 需求。这种方法也会造成DNA 分子的机械损伤,使得DNA 弥散,完整度下降。
c 电泳/电洗脱
电泳/电洗脱法相对于前两种有很多优点:其一,其价格低;其二,整个过程都很温和,能够保证DNA 的完整性;其三,纯化后的DNA 纯度较之前两种要好一些。但是其亦有些许不便:其一,过程耗时长,包括透析袋的准备,电洗脱,洗脱液的浓缩等;其二,DNA 损失量较前两种大,透析袋上会残留DNA。如果样品不是很珍贵,并且后续要进行Metagenomic 研究,这种方法不失为一种好方法。如果有脉冲场凝胶电泳系统,用此方法可以得到高纯大片段DNA,可以满足后续的metagenomic C/Fosmid 文库构建。
d 蔗糖/氯化铯密度梯度离心
如果实验室有超速离心机(100,000g 以上),那么这种方法无疑也是一种好方法。与电泳电洗脱法相同,这种方法在连续介质中对DNA 及杂质进行分离,可以很好的纯化DNA。纯化得到的DNA 经过低熔点凝胶电泳切胶回收,可以得到高纯度大片段的DNA,可以满足后续的metagenomic C/Fosmid 文库构建。
纯化质量的检测
DNA 纯化完毕之后往往需要对纯化效果做一个初步的判断,主要包括两方面:其一,完整度判断;其二,纯度判断。
a 完整度判断
完整度判断同前所述,千万要记得的是加纯化之前的DNA 样品做对照。结果可以告诉我们纯化得率及完整度。同样,最好能跑脉冲场凝胶电泳。
b 纯度判断
纯度判断通常可以采用两种方法:分光光度计法和PCR 扩增法。分光光度计可以测定双链DNA 在260nm 处的吸收值及杂质在其他波长的吸收值。通过不同波长吸收值的比,可以对DNA 纯度做出判定。通常这种方法由于灵敏性较高,所以在存在杂质时误差较大。
III. 所提DNA 完整性检测
DNA 的完整性主要采用普通琼脂糖凝胶电泳进行指示。最好采用0.8% 琼脂糖凝胶电泳,低电压长时间跑。至少要用lambda DNA/Hind III 做marker,如果在23 kb 处有一条完整或略拖尾的带,则证明DNA 完整性尚好。如有条件,可以采用脉冲场凝胶电泳对提取DNA 的完整度进行分析。用脉冲场凝胶电泳分析时,除了上面提及的那个maker,至少还应该有完整lambda DNA (约48 kb)充当另一个maker。
以上为送测序前样品的采集,DNA的提取,验证等前期处理与方法的大致步骤与方法。
Illumina所进行的测序是由Genome Analyzer测序仪所完成的:
以上为Genome Analyzer技术的基本原理IV.在DNA提取,检验完成后送BGI进行后续测序。
宏基因组学的研究进展
宏基因组学的研究状况及其发展 摘要:宏基因组学是近年来发展起来的一门新兴学科,主要技术包括从环境样品中提取微生物混合基因组DNA、利用可培养的宿主菌建立宏基因组文库及筛 选目的基因。该技术可以克服传统培养技术的不足,是研究未培养微生物、寻找新功能基因和开发获得新资源的重要新途径。目前宏基因组学已广泛应用于各个领域,并在医药、农业、能源开发、环境修复、生物技术、生物防御等方面有了较深入的研究。 关键词:宏基因组学、宏基因组、基因组文库构建、文库筛选、未培养微生物、研究进展 随着微生物学的发展,微生物基因组全序列测定计划正在全球被快速地推行,但现有技术条件下,自然界存在的可培养微生物不到总数的1%,阻碍了该计划 的发展,使得绝大多数的微生物资源不能被开发和利用。21世纪初,随着测序能力的提高和基因组学的发展,科学家提出了一种研究不可培养微生物基因组的新思路——直接对含有各种不可培养的微生物的群体进行基因组序列的测定。这类研究称为Metagenomics,前缀“Meta”源于希腊语。意思是“超越”。科学家选择它来表示这种基因组研究超越了传统意义上分析单一物种的基因组学,将研究对象定为由种类众多的微生物组成的整个菌落。国内的研究者也据此将该术语翻译为“宏基因组学”。 1 宏基因组的概念 宏基因组 (也称微生物环境基因组、宏基因组学、元基因组学、生态基因组学) 是由Handelsman等1998年提出的新名词, 其定义为“the genomes of the total microbiota found in nature”,即生境中全部微小生物遗传物质的总和。它包含了可培养的和未可培养的微生物的基因, 目前主要指环境样品中的细菌 和真菌的基因组总和。而所谓宏基因组学就是一种以环境样品中的微生物群体基因组为研究对象, 以功能基因筛选和测序分析为研究手段, 以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系为研究目的的新的微生物研究方法。一般包括从环境样品中提取基因组 DNA, 克隆DNA到合适 的载体,导入宿主菌体,筛选目的转化子等工作。宏基因组文库既包含了可培养的又包含了不能培养的微生物基因,避开了微生物分离培养的问题,极大地扩展了微生物资源的利用空间,增加了获得新的生物活性物质的机会,为新的医药产业和发现新的生物技术提供丰富的基因文库,并利于环境微生物有机群体的分布和功能的研究。 2 宏基因组学的研究过程 2.1 宏基因组文库的构建 宏基因组文库的构建沿用了分子克隆的基本原理和技术方法,并根据具体环境样品的特点和建库目的采用了一些特殊的步骤和策略。一般包括样品总DNA的 提取、与载体连接和克隆到宿主中。 2.1.1样品总DNA的提取 宏基因组文库构建的关键之一是获得高质量的目的样品的总DNA。目的样品 的采集是第一步,除了需严格遵循取样规则外,取样中应尽量避免对样品的干扰,缩短保存和运输的时间,使样品能更好地代表自然状态下的微生物原貌。 根据提取样品总DNA前是否分离细胞,提取方法可以分为原位裂解法和异位 裂解法。原位裂解法主要是通过去污剂处理(如SDS)、酶解法(如蛋白酶K)、机械
宏基因组实验计划
宏基因组实验计划
————————————————————————————————作者: ————————————————————————————————日期:
宏基因组学方法研究明永冰川的初步实验计划 起草人:李浩宇 宏基因组学研究方向及意义 研究:环境胁迫对集体遗传变异的过程和机理。 发掘:环境应激和应答基因的多态性。 探究:多态性基因的功能。 实验大体步骤:
I.环境样品的采集: 1)避免人源污染,采样的器具都要高温灭菌,采样过程中要戴手套。 2) 样品的迅速处理保存。有条件最好用液氮冷冻运输,没有条件可以将装有样品的容器至于干冰泡沫箱中。带回实验室之后要尽快处理,特别是固体样品,迅速用预冷缓冲液(多为PBS)重悬,离心收集沉淀。分装成小份,冻存于液氮或者‐80 ℃冰箱中,避免反复冻融。 3)如果后续用间接法提取总DNA 的话,此处需要收集菌体,采取的策略是稀释之后的差速离心,一般适用于水样。 II.DNA提取方法的选择: 总DNA的提取按照处理样品方式的不同划分为直接提取法和间接提取法。 直接提取法就是直接对样品进行裂解,释放其中的DNA。 间接提取法则是先分离样品中的微生物,后对微生物进行裂解。两种方法的适用范围不同,各自都有优缺点。 直接提取法的优缺点:直接提取法多用在提取固体样品(如土壤、污泥、湖泊沉积物等)总DNA 的试验中。其最大的优点在于得率高,在某些环境样品总DNA 提取实例中,比间接提取法高数倍至数百倍。造成这种情况的最主要原因一是环境样品中存在丰富的胞外DNA;二是许多微生物与基质形成复杂的结构,间接提取法不易对其进行分离。在样品较珍贵时多采用此法。其缺点同样明显,所得DNA的纯度远不及间接提取法所得,基质中含的腐殖质一并被保留下来,使得所得DNA呈现黄褐色甚至黑色。 可以考虑MoBio和Epicentre的DNA提取的商业试剂盒(主要用于提取土壤样品)。 间接提取法的优缺点:间接提取法则既可以用来提取固相样品总DNA也可以用来提取液体样品总DNA。在提取液体样品(如海水、河流等样品)总DNA 时,需要用抽滤的方式浓缩。与直接提取法刚好相反,其最大的优点在于提取DNA 纯度高,既使在提取固体样品微生物总DNA 时,也可以用缓冲液对收集的菌体进行反复冲洗以去除表面所带杂质。其缺点是DNA 得率低。? 土壤DNA 直接提取法:
宏基因组学概述
宏基因组学概述
————————————————————————————————作者: ————————————————————————————————日期: ?
宏基因组学概述 王莹,马伊鸣 (北京交通大学土木建筑工程学院环境1402班) 摘要:随着分子生物学技术的快速发展及其在微生物生态学和环境微生物学研究中的广泛应用,促进了以环境中未培养微生物为研究对象的新兴学科——微生物环境基因组学(又叫宏基因组学、元基因组学,英文名Metagenomics)的产生和快速发展。宏基因组学通过直接从环境样品中提取全部微生物的DNA,构建宏基因组文库,利用基因组学的研究策略研究环境样品所包含的全部微生物的遗传组成及其群落功能.在短短几年内,宏基因组学研究已渗透到各个领域,包括海洋、土壤、热液口、热泉、人体口腔及胃肠道等,并在医药、替代能源、环境修复、生物技术,农业、生物防御及伦理学等各方面显示了重要的价值。本文对宏基因组学的主要研究方法、热点内容及发展趋势进行了综述 关键词:宏基因组宏基因组学环境基因组学基因文库的构建 Macro summary of Metagenomics WangYing,Ma Yi-Ming (BeijingJiaotongUniversity, Institute of civil engineering,)Key words:Metagenome; Metagenomics;The environmental genomics 宏基因组学(Metagenomics)又叫微生物环境基因组学、元基因组学。它通过直接从环境样品中提取全部微生物的DNA,构建宏基因组文库,利用基因组学的研究策略研究环境样品所包含的全部微生物的遗传组成及其群落功能。它是在微生物基因组学的基础上发展起来的一种研究微生物多样性、开发新的生理活性物质(或获得新基因)的新理念和新方法。其主要含义是:对特定环境中全部微生物的总DNA(也称宏基因组,metagenomic)进行克隆,并通过构建宏基因组文库和筛选等手段获得新的生理活性物质;或者根据rDNA数据库设计引物,通过系统学分析获得该环境中微生物的遗传多样性和分子生态学信息。 1.起源 宏基因组学这一概念最早是在1998年由威斯康辛大学植物病理学部门的Jo Handelsman等提出的,是源于将来自环境中基因集可以在某种程度上当成一个单个基因组研究分析的想法,而宏的英文是"meta-",具有更高层组织结构和动态变化的含义。后来伯克利分校的研究人员Kevin Chen和LiorPachter将宏基因组定义为"应用现代基因组学的技术直接研究自然状态下的微生物的有机群落,而不需要在实验室中分离单一的菌株"的科学。 2 研究对象 宏基因组学(Metagenomics)是将环境中全部微生物的遗传信息看作一个整体自上而下地研究微生物与自然环境或生物体之间的关系。宏基因组学不仅克服了微生物难以培养的困难, 而且还可以结合生物信息学的方法, 揭示微生物之间、微生物与环境之间相互作用的规律, 大大拓展了微生物学的研究思路与方法, 为从群落结构水平上全面认识微生物的生态特征和功能开辟了新的途径。目前, 微生物宏基因组学已经成为微生物研究的热点和前沿, 广泛应用于气候变化、水处理工程系统、极端环境、人体肠道、石油污染、生物冶金等领域, 取得了一系列引人瞩目的重要成果。 3 研究方法
宏基因组测序技术检测方法
宏基因组测序技术检测标准 简介: 宏基因组测序介绍 宏基因组学是以环境样品中的微生物群体基因组为研究对象,通过现代基因组技术手段包括功能基因的筛选和测序分析,对环境中微生物多样性、种群结构、进化关系、功能活性、相互协作关系以及环境之间的关系进行研究的新的微生物研究方法。随着高通量测序技术的发展,为宏基因组学研究提供了新的理想研究方法。高通量测序的方法无需分离环境中各种微生物,也无需构建克隆文库就可以直接对环境中所有微生物进行测序。可以真实客观的反映环境中微生物的多样性、种群结构、进化关系等。目前又可以分为针对16s DNA/18sDNA/ITS测序和针对宏基因组全序列的测序研究。下面就是对这两者的具体介绍。 一、16s DNA/18s DNA/ITS测序 16sDNA是最常用的微生物物种分子鉴定的标签,,通过对样品中16sDNA测序可以鉴定其中微生物物种的丰度和分布情况。目前,普遍使用Roche 454平台来对环境样品进行16s DNA测序。因为16s DNA序列比较相似,读长短的话,难以进行有效的比对,而454平台的平均读长在400bp左右,可以很好的避免此类问题。 二、宏基因组全测序 在这种测序方式中,我们可以假定一个环境中的所有微生物就是一个整体,然后对其中所有的微生物进行测序。这样我们就可以研究样品中的功能基因以及其在环境中所起的作用而不用关心其来自哪个微生物。可以发现新的基因,可以进行基因的预测,甚至有可能得到某个细菌基因组的全序列。此外,该项测序不单可以针对DNA水平,也可以针对全RNA进行基因表达水平的研究。 样品处理:
宏基因组样品收集主要有口腔,下呼吸道痰液,下呼吸道灌洗液,皮肤和粪便。样品采集遵照样品采集规范(人)所规定的操作来进行。尽量留足备份样品。核酸提取: 宏基因组核酸提取主要有两种方法:膜过滤法和直接裂解提取。对于液体样品如痰液,灌洗液两种方法都适用,对于固体样品如粪便宜采用直接裂解的方法。核酸提取后用NanoDrop ND-1000测定,260/280 = , 260/230 = ,电泳检测DNA 应是完整的一条带。 测序Sequencing 1)16S/18S测序: Sanger测序: 用于低通量的16S/18S DNA测序,提取宏基因组后,首先通过PCR将16S/18S 序列扩增出来,再将其连接到克隆载体上,导入感受态细胞,涂平板做蓝白斑筛选,选出阳性克隆提质粒,对质粒进行测序反应,测序反应后纯化后用ABI 3130或ABI 3730进行毛细管电泳测序。 由于其测序准确率比较高,而通量非常低,现通常用做二代测序结果的验证。454 Platform: 454平台主要包括两种测序系统:454 GS FLX+ System和454 GS Junior System。454 GS FLX+ System测序读长可以达到600-1000bp,通量450-700M,GS Junior System测序读长在400bp左右,通量在35M。
宏基因组学的一般研究策略
宏基因组学的一般研究策略 摘要: 宏基因组学是目前微生物基因工程的一个重要方向与热点。它把微生物的总群体特性与基因组学实验手段结合了起来,包括从环境样品中提取总DNA、再用可培养的宿主微生物建立文库及筛选目的克隆和基因。该法是研究不可培养微生物、寻找新的基因和开发新活性产物的重要新途径。它避开了微生物分离、纯化和培养的步骤,大大扩展了微生物资源的利用范围。本文旨在介绍宏基因组学的一般研究方法并结合我们的实验情况,对这一崭新领域中的最新研究策略进行了简要综述。 关键词: 宏基因组学, 不可培养微生物, 文库构建, 文库筛选,研究策略 Strategies for accessing metagenomics for desired applications Abstract: Metagenomics is a new field of microbial genetic engineering. It has the characteristics of microbial ecology and the methodology of genomics. Metagenomics includes genomic DNA isolation, library construction and screening strategies, and can be used in the discovery of new gene and biocatalysts and in the study of uncultured microorganism. Metagenomics can overcome the advantages of isolation and cultivation procedures in traditional microbial method, and thus greatly broaden the space of microbial resource utilization. In this paper, we mainly reviewed the metagenomic methodology, together with the latest advances and novel strategy in this research field. Keywords:Metagenomics; Uncultured microorganism;Library construction;Library screening Research strategies 大自然中蕴藏着无数具有重要价值的微生物及其活性产物,也是新基因及生物学资源的重要源泉,对其进行研究成为微生物学和分子生物学研究的一个重要方向。然而人们现在能够培养与利用的不到环境中总微生物的1%[1]。宏基因组学(metagenomics)是直接从环境样品中提取全部微生物的总DNA, 避开了分离、纯化和培养微生物的过程来构建宏基因组文库,用基因组学的研究策略来研究环境样品中的总微生物的组成及其在群落中的功能等。现在,宏基因组学技术方法已在微生物多样性,微生物细胞间的相互作用,新基因和新型生物催化剂的开发,新的抗生素的开发及环境生态等方面得到了广泛应用[2]。本文旨在介绍宏基因组学的一般实验方法并结合我们的研究情况,对这一崭新领域中的最新研究策略进行了简要综述。深化了我们对这一学科的认识,促进了该学科的进步。 1 宏基因组学研究策略 1.1宏基因组学概要 宏基因组学是Handelsman等于1998年提出的[3], 可见是一门很新的学科,其随着基因组实验手段,生物信息学和测序技术等的日新月异也迅猛发展了起来,这个新学科是以环境样品的总微生物基因组为实验对象,通过测序分析、文库评价、产活性物质及其基因的克隆的获取和基因功能的鉴别,对微生物种群组成与生物量、生态学关系、生物化学关系与环境关系以及功能活性进行研究[4]。其主要过程包括样品和基因的富集和提取; 宏基因组文库的构建; 目的基因的筛选; 目的基因活性产物的表达(图1)。 1.2 微生物及其基因的富集 在文库筛选过程中由于目的基因比例较小, 对环境中微生物的富集不但可提高基因总量,有利于基因的提取,还可增加目的基因的比例,如Kouker 等用橄榄油富集产脂肪酶的微生物收到了很好的效果[5 ],橄榄油不仅可作为底物,还可诱导脂肪酶的合成。目前富集技术主要分为细胞水平和基因水平。其中细胞水平主要是用选择培养基来富集某些微生物, 常
宏基因组测序技术检测方法模板
宏基因组测序技术 检测方法
宏基因组测序技术检测标准 简介: 宏基因组测序介绍 宏基因组学是以环境样品中的微生物群体基因组为研究对象,经过现代基因组技术手段包括功能基因的筛选和测序分析,对环境中微生物多样性、种群结构、进化关系、功能活性、相互协作关系以及环境之间的关系进行研究的新的微生物研究方法。随着高通量测序技术的发展,为宏基因组学研究提供了新的理想研究方法。高通量测序的方法无需分离环境中各种微生物,也无需构建克隆文库就能够直接对环境中所有微生物进行测序。能够真实客观的反映环境中微生物的多样性、种群结构、进化关系等。当前又能够分为针对16s DNA/18sDNA/ITS测序和针对宏基因组全序列的测序研究。下面就是对这两者的具体介绍。 一、16s DNA/18s DNA/ITS测序 16sDNA是最常见的微生物物种分子鉴定的标签,,经过对样品中16sDNA测序能够鉴定其中微生物物种的丰度和分布情况。当前,普遍使用Roche 454平台来对环境样品进行16s DNA测序。因为16s DNA序列比较相似,读长短的话,难以进行有效的比对,而454平台的平均读长在400bp左右,能够很好的避免此类问题。 二、宏基因组全测序
在这种测序方式中,我们能够假定一个环境中的所有微生物就是一个整体,然后对其中所有的微生物进行测序。这样我们就能够研究样品中的功能基因以及其在环境中所起的作用而不用关心其来自哪个微生物。能够发现新的基因,能够进行基因的预测,甚至有可能得到某个细菌基因组的全序列。另外,该项测序不单能够针对DNA水平,也能够针对全RNA进行基因表示水平的研究。 样品处理: 宏基因组样品收集主要有口腔,下呼吸道痰液,下呼吸道灌洗液,皮肤和粪便。样品采集遵照样品采集规范(人)所规定的操作来进行。尽量留足备份样品。 核酸提取: 宏基因组核酸提取主要有两种方法:膜过滤法和直接裂解提取。对于液体样品如痰液,灌洗液两种方法都适用,对于固体样品如粪便宜采用直接裂解的方法。核酸提取后用NanoDrop ND-1000测定,260/280 = 1.8-2.0, 260/230 = 1.8-2.0,电泳检测DNA应是完整的一条带。 测序Sequencing 1)16S/18S测序: Sanger测序: 用于低通量的16S/18S DNA测序,提取宏基因组后,首先经过PCR将16S/18S序列扩增出来,再将其连接到克隆载体上,导
宏基因组学研究方法及应用概述
宏基因组学研究方法及应用概述彭昌文 (山东省济宁学院生物学系 273155) 颜 梅 (山东省曲阜师范大学生命科学学院 273165) 摘 要 本文简要介绍了宏基因组的概念,概述了其原理及应用。 关键词 宏基因组 宏基因组学 环境基因组学 基因文库的构建 迄今,人们对微生物世界的认识基本都来源于对占细菌总种数不到1%的微生物的单个种群的孤立研究结果。然而微生物是通过其群落而非单一种群来执行在自然界物质与能量循环中的作用的,对微生物群落作为整体的功能认识远远落后于对其个体的认识。这种状况不利于全面认识微生物在自然界所扮演的重要角色。为了获得完整的环境微生物基因表达产物,早在1978年许多学者就提出了直接从环境中提取微生物DNA的思路,1998年,AR I A D phar maceutical公司的科学家Handels man等首次提出宏基因组的概念[1]。宏基因组(the genomes of the total m icrobi ota found in nature)是指生境中全部微生物基因的总和[2]。它包含了可培养的和未培养的微生物的基因总和,微生物主要包括环境样品中的细菌和真菌。而宏基因组学就是一种以环境样品中的微生物群体基因组为研究对象,以功能基因筛选和测序分析为研究手段,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系等为研究目的的新的微生物研究方法,也称为微生物环境基因组学、元基因组学或生态基因组学。它主要研究从环境样品获得的基因组中所包含的微生物的遗传组成及其群落功能,为充分认识和开发利用非培养微生物,并从完整的群落水平上认识微生物的活动、最大限度地挖掘微生物资源,提供了可能,已成为国际生命科学技术研究的热点和前沿。 1 宏基因组学的研究方法 宏基因组学的研究过程一般包括从环境样品中提取基因组DNA,克隆DNA到合适的载体,导入宿主菌体,筛选目的转化子等工作,可分为三个步骤。 1.1 宏基因组的提取 在宏基因组筛选过程中,目的基因是整个核苷酸链中的一部分,因此样品前期的富集能够提高筛选命中率。DNA的提取是宏基因文库构建的关键步骤。提取步骤通常需要满足两个条件:既要尽可能提取样品所有微生物的基因,又要保持片段的完整和纯度。目前所开发的DNA提取方法有两种:细胞提取法和直接裂解法。直接裂解法包括物理法(冻融法、超声法、玻璃球珠击打法、液氮碾磨法)、化学法(常用化学试剂有表面活性剂、盐类、有机溶剂等)及酶裂解法。另外,依据提取样品总DNA前是否分离细胞,可以分为原位裂解法和异位裂解法。原位裂解法可以直接破碎样品中的微生物细胞而使DNA 得以释放,由于无需对样品微生物进行复苏,且黏附颗粒上的微生物细胞亦能被裂解,所得DNA能更好地代表样品微生物的多样性。此法操作容易、成本低,DNA 提取率高,但由于机械剪切作用较强,所提取的DNA 片段小(1~50kb),通常适用于构建小片段插入文库(以质粒和λ噬菌体为载体)的DNA提取。异位裂解法则先采用物理方法将微生物从样品中分离出来,然后采用较温和的方法抽提DNA。此法条件温和,可获得大片段DNA(20~500kb),纯度高,但操作繁琐、成本高、得率低,通常适用于构建大片段插入文库(以柯斯质粒或者细菌人工染色体为载体)的DNA提取。1.2 宏基因组文库的构建 宏基因组文库的构建需适宜的克隆载体。通常用于DNA克隆的载体主要包括质粒、黏粒和细菌人工染色体等。质粒一般用于克隆小于10kb的DNA片段,适用于单基因的克隆与表达。黏粒的插入片段可达40kb左右,细菌人工染色体插入片段可达350kb,可用来制备由多基因簇调控的微生物活性物质的完整代谢途径的相关片段文库。1.3 目的基因的筛选 目的基因的筛选方法包括序列分析和功能分析两种。序列分析适用于小片段DNA文库的基因筛选;而功能分析通常适用于大片段DNA文库的筛选。序列分析筛选不依赖于重组基因在外源宿主中的表达,因为所使用的寡聚核苷酸引物是直接通过DNA序列中的保守区域设计的,反映了氨基酸序列的保守性,可获得未知序列的目的基因。该方法对DNA量的要求不高,筛选到新活性物质的可能性较大。序列分析的另一个手段是对宏基因组克隆测序,无论是全部或随机测序都是发现新基因的有效手段。 对于功能分析而言,首先需获得目的克隆,然后通过序列和生化分析对其进行表征。此法能快速鉴定出全新且有开发价值的活性物质,可用于医药、工农业等行业。由于此法检出率较低,工作量较大,且受检测手段的限制,所以常要借助于高通量筛选。 2 宏基因组学的应用 2.1 在生态学方面的应用 当今微生物生态学研究的主要目的之一是将微生物与其所在环境中的代谢过程相联系。应用16s r DNA作为系统发育锚去鉴定属于某种微生物的克隆,然后对基因进行测序,从而获得
宏基因组实验计划
宏基因组学方法研究明永冰川的初步实验计划 起草人:李浩宇 宏基因组学研究方向及意义 研究:环境胁迫对集体遗传变异的过程和机理。 发掘:环境应激和应答基因的多态性。 探究:多态性基因的功能。 实验大体步骤:
I.环境样品的采集: 1)避免人源污染,采样的器具都要高温灭菌,采样过程中要戴手套。 2)样品的迅速处理保存。有条件最好用液氮冷冻运输,没有条件可以将装有样品的容器至于干冰泡沫箱中。带回实验室之后要尽快处理,特别是固体样品,迅速用预冷缓冲液(多为PBS)重悬,离心收集沉淀。分装成小份,冻存于液氮或者‐80 ℃冰箱中,避免反复冻融。 3)如果后续用间接法提取总DNA 的话,此处需要收集菌体,采取的策略是稀释之后的差速离心,一般适用于水样。 II.DNA提取方法的选择: 总DNA 的提取按照处理样品方式的不同划分为直接提取法和间接提取法。 直接提取法就是直接对样品进行裂解,释放其中的DNA。 间接提取法则是先分离样品中的微生物,后对微生物进行裂解。两种方法的适用范围不同,各自都有优缺点。 直接提取法的优缺点:直接提取法多用在提取固体样品(如土壤、污泥、湖泊沉积物等) 总DNA 的试验中。其最大的优点在于得率高,在某些环境样品总DNA 提取实例中,比间接提取法高数倍至数百倍。造成这种情况的最主要原因一是环境样品中存在丰富的胞外DNA;二是许多微生物与基质形成复杂的结构,间接提取法不易对其进行分离。在样品较珍贵时多采用此法。其缺点同样明显,所得DNA 的纯度远不及间接提取法所得,基质中含的腐殖质一并被保留下来,使得所得DNA 呈现黄褐色甚至黑色。 可以考虑MoBio 和Epicentre的DNA提取的商业试剂盒(主要用于提取土壤样品)。 间接提取法的优缺点:间接提取法则既可以用来提取固相样品总DNA 也可以用来提取液体样品总DNA。在提取液体样品(如海水、河流等样品)总DNA 时,需要用抽滤的方式浓缩。与直接提取法刚好相反,其最大的优点在于提取DNA 纯度高,既使在提取固体样品微生物总DNA 时,也可以用缓冲液对收集的菌体进行反复冲洗以去除表面所带杂质。其缺点是DNA 得率低。
宏基因组学概述
宏基因组学概述 王莹,马伊鸣 (北京交通大学土木建筑工程学院环境1402班) 摘要:随着分子生物学技术的快速发展及其在微生物生态学和环境微生物学研究中的广泛应用,促进了以环境中未培养微生物为研究对象的新兴学科——微生物环境基因组学(又叫宏基因组学、元基因组学,英文名Metagenomics)的产生和快速发展。宏基因组学通过直接从环境样品中提取全部微生物的DNA,构建宏基因组文库,利用基因组学的研究策略研究环境样品所包含的全部微生物的遗传组成及其群落功能.在短短几年内,宏基因组学研究已渗透到各个领域,包括海洋、土壤、热液口、热泉、人体口腔及胃肠道等,并在医药、替代能源、环境修复、生物技术,农业、生物防御及伦理学等各方面显示了重要的价值。本文对宏基因组学的主要研究方法、热点内容及发展趋势进行了综述 关键词:宏基因组宏基因组学环境基因组学基因文库的构建 Macro summary of Metagenomics Wang Ying, Ma Yi-Ming (BeijingJiaotongUniversity, Institute of civil engineering,) Key words: Metagenome; Metagenomics; The environmental genomics 宏基因组学(Metagenomics)又叫微生物环境基因组学、元基因组学。它通过直接从环境样品中提取全部微生物的DNA,构建宏基因组文库,利用基因组学的研究策略研究环境样品所包含的全部微生物的遗传组成及其群落功能。它是在微生物基因组学的基础上发展起来的一种研究微生物多样性、开发新的生理活性物质(或获得新基因)的新理念和新方法。其主要含义是:对特定环境中全部微生物的总DNA (也称宏基因组,metagenomic)进行克隆,并通过构建宏基因组文库和筛选等手段获得新的生理活性物质;或者根据rDNA数据库设计引物,通过系统学分析获得该环境中微生物的遗传多样性和分子生态学信息。 1.起源 宏基因组学这一概念最早是在1998年由威斯康辛大学植物病理学部门的Jo Handelsman等提出的,是源于将来自环境中基因集可以在某种程度上当成一个单个基因组研究分析的想法,而宏的英文是"met a-",具有更高层组织结构和动态变化的含义。后来伯克利分校的研究人员Kevin Chen和Lior Pachter 将宏基因组定义为"应用现代基因组学的技术直接研究自然状态下的微生物的有机群落,而不需要在实验室中分离单一的菌株"的科学。 2 研究对象 宏基因组学(Metagenomics)是将环境中全部微生物的遗传信息看作一个整体自上而下地研究微生 物与自然环境或生物体之间的关系。宏基因组学不仅克服了微生物难以培养的困难, 而且还可以结合生物信息学的方法, 揭示微生物之间、微生物与环境之间相互作用的规律, 大大拓展了微生物学的研究思路与方法, 为从群落结构水平上全面认识微生物的生态特征和功能开辟了新的途径。目前, 微生物宏基因组学已经成为微生物研究的热点和前沿, 广泛应用于气候变化、水处理工程系统、极端环境、人体肠道、石油污染、生物冶金等领域, 取得了一系列引人瞩目的重要成果。 3 研究方法 宏基因组学的研究过程一般包括样品和基因(组)的富集;提取特定环境中的基因组 DNA;构建宏基因组 DNA 文库;筛选目的基因;目的基因活性产物表达(图 1)五个步骤。
宏基因组测序讲解
宏基因组测序讲解
宏基因组测序 目的 研究藻类物种的分类,研究与特定环境与相关的代谢通路,以及通过不同样品的比较研究微生物内部,微生物与环境,与宿主的关系。技术简介 宏基因组( Metagenome)(也称微生物环境基因组Microbial Environmental Genome, 或元基因组) 。是由 Handelsman 等 1998 年提出的新名词,其定义为"the genomes of the total microbiota found in nature" , 即生境中全部微小生物遗传物质的总和。它包含了可培养的和未可培养的微生物的基因,目前主要指环境样品中的细菌和真菌的基因组总和。而所谓宏基因组学 (或元基因组学, metagenomics) 就是一种以环境样品中的微生物群体基因组为研究对象,以功能基因筛选和/或测序分析为研究手段,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系为研究目的的新的微生物研究方法。一般包括从环境样品中提取基因组 DNA, 进行高通量测序分析,或克隆DNA到合适的载体,导入宿主菌体,筛选目的转化子等工作。 宏基因组( Metagenome)(也称微生物环境基因组Microbial Environmental Genome, 或元基因组) 。是由 Handelsman 等 1998 年提出的新名词,其定义为"the genomes of the total microbiota found in nature" , 即生境中全部微小生物遗传物质的总和。它包含了可培养的和未可培养的微生物的基因,目前主要指环境样品中的细菌和真菌的基因组总和。而所谓宏基因组学 (或元基因组学, metagenomics) 就是一种以环境样品中的微生物群体基因组为研究对象,以功能基因筛选和/或测序分析为研究手段,以微生物多样
(完整word版)宏基因组测序讲解
宏基因组测序 目的 研究藻类物种的分类,研究与特定环境与相关的代谢通路,以及通过不同样品的比较研究微生物内部,微生物与环境,与宿主的关系。技术简介 宏基因组( Metagenome)(也称微生物环境基因组Microbial Environmental Genome, 或元基因组) 。是由 Handelsman 等 1998 年提出的新名词,其定义为"the genomes of the total microbiota found in nature" , 即生境中全部微小生物遗传物质的总和。它包含了可培养的和未可培养的微生物的基因,目前主要指环境样品中的细菌和真菌的基因组总和。而所谓宏基因组学 (或元基因组学, metagenomics) 就是一种以环境样品中的微生物群体基因组为研究对象,以功能基因筛选和/或测序分析为研究手段,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系为研究目的的新的微生物研究方法。一般包括从环境样品中提取基因组 DNA, 进行高通量测序分析,或克隆DNA到合适的载体,导入宿主菌体,筛选目的转化子等工作。 宏基因组( Metagenome)(也称微生物环境基因组Microbial Environmental Genome, 或元基因组) 。是由 Handelsman 等 1998 年提出的新名词,其定义为"the genomes of the total microbiota found in nature" , 即生境中全部微小生物遗传物质的总和。它包含了可培养的和未可培养的微生物的基因,目前主要指环境样品中的细菌和真菌的基因组总和。而所谓宏基因组学 (或元基因组学, metagenomics) 就是一种以环境样品中的微生物群体基因组为研究对象,以功能基因筛选和/或测序分析为研究手段,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系为研究
宏基因组测序技术检测方法
宏基因组测序技术检测方法
宏基因组测序技术检测标准 简介: 宏基因组测序介绍 宏基因组学是以环境样品中的微生物群体基因组为研究对象,通过现代基因组技术手段包括功能基因的筛选和测序分析,对环境中微生物多样性、种群结构、进化关系、功能活性、相互协作关系以及环境之间的关系进行研究的新的微生物研究方法。随着高通量测序技术的发展,为宏基因组学研究提供了新的理想研究方法。高通量测序的方法无需分离环境中各种微生物,也无需构建克隆文库就可以直接对环境中所有微生物进行测序。可以真实客观的反映环境中微生物的多样性、种群结构、进化关系等。目前又可以分为针对16s DNA/18sDNA/ITS测序和针对宏基因组全序列的测序研究。下面就是对这两者的具体介绍。 一、16s DNA/18s DNA/ITS测序 16sDNA是最常用的微生物物种分子鉴定的标签,,通过对样品中16sDNA 测序可以鉴定其中微生物物种的丰度和分布情况。目前,普遍使用Roche 454平台来对环境样品进行16s DNA测序。因为16s DNA序列比较相似,读长短的话,难以进行有效的比对,而454平台的平均读长在400bp左右,可以很好的避免此类问题。 二、宏基因组全测序 在这种测序方式中,我们可以假定一个环境中的所有微生物就是一个整体,然后对其中所有的微生物进行测序。这样我们就可以研究样品中的功能基因以及其在环境中所起的作用而不用关心其来自哪个微生物。可以发现新的基因,可以进行基因的预测,甚至有可能得到某个细菌基因组的全序列。此外,该项测序不单可以针对DNA水平,也可以针对全RNA进行基因表达水平的研究。 样品处理: 宏基因组样品收集主要有口腔,下呼吸道痰液,下呼吸道灌洗液,皮肤和粪便。样品采集遵照样品采集规范(人)所规定的操作来进行。尽量留足备份样品。
宏基因组及其应用
宏基因组及其应用 学习笔记 吕涛15010906 一、宏基因组及宏基因组学 1.概念 宏基因组( Metagenome)(也称微生物环境基因组Microbial Environmental Genome, 或元基因组)是由Handelsman 等1998 年提出的新名词,其定义为 “the genomes of the total microbiota found in nature” , 即环境中全部微小生物遗 传物质的总和。它包含了可培养的和未可培养的微生物的基因,目前主要指环境 样品中的细菌和真菌的基因组总和。 2.宏基因组学 宏基因组( Metagenome)(也称微生物环境基因组Microbial Environmental Genome, 或元基因组)是由Handelsman 等1998 年提出的新名词,其定义 为“the genomes of the total microbiota found in nature” , 即环境中全部微小生 物遗传物质的总和。它包含了可培养的和未可培养的微生物的基因,目前主要指 环境样品中的细菌和真菌的基因组总和。 3.发展历程 环境基因组学——微生物基因组学——宏基因组学——人类基因组学 人类基因组学: 把人体内所有微生物菌群基因组的总和称为“人体宏基因组”(human metagenome)。人类宏基因组学(human metagenomics)研究人体宏基因组结构和 功能、相互之间关系、作用规律和与疾病关系的学科。它不仅要把总体基因组序 列信息都测定出来,而且还要研究与人体发育和健康有关的基因功能。人类宏基 因组计划目标是:把人体内共生菌群的基因组序列信息都测定出来,而且要研究 与人体发育和健康有关的基因功能。 4.研究步骤
宏基因组学的研究
因组学研究进展及其应用 摘要: 本文先简要介绍了当前生物化学的一些研究热点,再针对因组学展开论述,介绍了因组学的产生背景和概念,当前的研究进展及应用。 因组学尝试通过免培方法获得微生物的纯培养,主要技术包括DNA的提取、文库的构建和目标基因克隆的筛选,可用于开发新型酶、发现新基因、筛选医药等方面。 关键字:因组学;因组学基本策略;文库构建与筛选;因组学研究进展及其应用引言: 微生物是地球上种类最多、数量最大、分布最广的生物群。仅原核生物(细菌和古细菌)即构成地球生物总量的的25~50 %[1]。自然条件下,包括病毒在的微生物,通过群落广泛参与C、N、O 和S等重要元素的循环转化,在人体的食物消化、毒素降解及机体免疫反应,环境污染物降解等方面发挥着重要作用[2]。人们对于微生物的研究主要是建立在纯培养基础上,后来人们发现通过纯培养方法估计的环境微生物多样性只占总量的0.1%~1%[3],多达99%以上的微生物是不可培养的, 其中蕴含着巨大的应用潜能——其代产物中可能有众多具有应用开发价值的化合物[4]。为了研究不能培养的微生物,一个全新的理念——因组学应运而生,该技术不需预先培养就能开发这些微生物基因组,目前已广泛应用于微生物活性物质的开发与利用、环境微生物种群分布及动态变化分析等方面的研究[5]。 因组学的提出为解决上述问题提供了一个可行途径。因组学以生境中全部
DNA作为研究对象,通过克隆、异源表达来筛选有用基因及其产物。由于突破了传统研究领域无法涵盖不可培养微生物的瓶颈,因组学概念及研究方法一经提出,就被广泛接受。尽管在方法上还存在一定缺陷,但并不妨碍不同领域学者利用该方法来研究各种生境中微生物生态以及筛选功能基因的热情,有关因组学研究的文章逐年增多[4]。 1.因组学的概念 因组( metagenome) 的概念是指从生境样本中取得全部微生物的基因组, 而不是采用传统的培养微生物的基因组。因组的样本既包括可培养的微生物,也包括更大量的传统方法无法研究的不可培养微生物[6]。而所谓因组学 (也称元基因组学Metagenomics 、微生物环境基因组学Microbial Environmental Genomics、生态基因组学Ecogenomics ) 就是一种以环境样品中的微生物群体基因组为研究对象,以功能基因筛选和测序分析为研究手段,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系为研究目的的新的微生物研究方法,一般包括克隆、构建文库和功能分析筛选等工作[7]。 2.因组学的基本策略及方法 2.1因组学的基本策略 因组学的研究还处于初期发展阶段,但其研究的基本过程和基本策略已基本清楚。在此要强调的是,因组学研究有着明确的指导思想,它是在反向生物学原则指导下,基于特定生态环境基础上,依据整体、系统、动态变化和相互作用的观点,运用特殊的技术路线和方法,对研究围中所有基因组展开研究的学科。 因组学是一种整体性的研究策略,它建立在微生物基因组学的迅速发展和聚
宏基因组测序
宏基因组测序 环境中超过99%的微生物是不可培养的,很多致力于研究微生物多样性的努力由于培养方法的限制而受到制约,为了克服由培养技术所带来的困难和限制,多种以DNA为基础的分子生物学的方法已经被开发。 目前16s rDNA测序可以提供大量关于环境微生物的群落及种类信息,但是在种群中不同微生物的作用以及其携带的基因组信息基本不能体现出来。相比之下,宏基因组是一种新的,可用于快速分析微生物复杂基因组的方法,它提取环境中的全基因组DNA,构建DNA文库并进行高通量测序。对数据进行分析,不仅能够获得环境中微生物的组成及丰度信息,还可以通过相关功能及代谢通路注释,获得这些微生物全面的微生物基因组信息,以及在环境中可能的功能。 技术参数 样品准备测序策略推荐数据周期 3ug DNA 300bp DNA文库 HiSeq PE150测序 一般测序数据量:5Gb clean data 大测序数据量:10Gb clean data 40个工作日 建库方法技术流程
技术特点 (1)无需分离培养,直接提取样本DNA测序; (2)群落多样性、种群结构、进化关系、功能组成、相互协作关系等多种分析; (3)高效、高通量,一次性获取样本中所有微生物组成等信息。 部分结果展示 进化树分析OTU维恩图 抗生素类型统计图 案例解析 排泄物微生物宏基因组可作为结直肠癌标志物 为了评估利用排泄物诊断结直肠癌的可行性,作者对来自于中国的74个结直肠癌患者和54个健康人的粪便样本进行宏基因组测序,发现除了已经证实的与结直肠癌相关的具核梭杆菌(Fusobacterium nucleatum)和消化链球菌(Peptostreptococcus stomatis)之外,微小微单胞菌(Parvimonas micra)和口臭致病菌(Solobacterium moorei)也与结直肠癌具有显著相关性。作者随后选择了20个微生物基因标志物,通过q-PCR发现,来自于具核梭杆菌的丁酰coA脱氢酶和来自于微小微单胞菌的RNA聚合酶亚基β在患者的粪便微生物的基因组中高度表达;利用这两个基因可以准确区分患有结直肠癌的患者和健康人群。这项研究为通过排泄物中微生物的宏基因组标志物对结直肠癌进行无创早期诊断奠定了坚实的基础。
宏基因组学的研究
宏基因组学的研究
宏基因组学研究进展及其应用 摘要: 本文先简要介绍了当前生物化学的一些研究热点,再针对宏基因组学展开论述,介绍了宏基因组学的产生背景和概念,当前的研究进展及应用。 宏基因组学尝试通过免培方法获得微生物的纯培养,主要技术包括DNA的提取、文库的构建和目标基因克隆的筛选,可用于开发新型酶、发现新基因、筛选医药等方面。 关键字:宏基因组学;宏基因组学基本策略;文库构建与筛选;宏基因组学研究进展及其应用 引言: 微生物是地球上种类最多、数量最大、分布最广的生物群。仅原核生物(细菌和古细菌)即构成地球生物总量的的25~50 %[1]。自然条件下,包括病毒在内的微生物,通过群落广泛参与C、N、O 和S等重要元素的循环转化,在人体的食物消化、毒素降解及机体免疫反应,环境污染物降解等方面发挥着重要作用[2]。人们对于微生物的研究主要是建立在纯培养基础上,后来人们发现通过纯培养方法估计的环境微生物多样性只占总量的0.1%~1%[3],多达99%以上的微生物是不可培养的, 其中蕴含着巨大的应用潜能——其代谢产物中可能有众多具有应用开发价值的化合物[4]。为了研究不能培养的微生物,一个全新的理念——宏基因组学应运而生,该技术不需预先培养就能开发这些微生物基因组,目前已广泛应用于微生物活性物质的开发与利用、环境微生物种群分布及动态变化分析等方面的研究[5]。 宏基因组学的提出为解决上述问题提供了一个可行途径。宏基因组学以生境中全部DNA作为研究对象,通过克隆、异源表达来筛选有用基因及其产物。由于突破了传统研究领域无法涵盖不可培养微生物的瓶颈,宏基因组学概念及研究方法一经提出,就被广泛接受。尽管在方法上还存在一定缺陷,但并不妨碍不同领域学者利用该方法来研究各种生境中微生物生态以及筛选功能基因的热情,有关宏基因组学研究的文章逐年增多[4]。 1.宏基因组学的概念 宏基因组( metagenome) 的概念是指从生境样本中取得全部微生物的基因组, 而不是采用传统的培养微生物的基因组。宏基因组的样本既包括可培养的微生物,也包括更大量的传统方法无法研究的不可培养微生物[6]。而所谓宏基因组学(也称元基因组学Metagenomics 、微生物环境基因组学Microbial Environmental Genomics、生态基因组学Ecogenomics ) 就是一种以环境样品中的微生物群体基因组为研究对象,以功能基因筛选和测序分析为研究手段,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系为研究目的的新的微生物研究方法,一般包括克隆、构建文库和功能分析筛选等工作[7]。 2.宏基因组学的基本策略及方法 2.1宏基因组学的基本策略 宏基因组学的研究还处于初期发展阶段,但其研究的基本过程和基本策略已基本清楚。在此要强调的是,宏基因组学研究有着明确的指导思想,它是在反向生物学原则指导下,基于特定生态环境基础上,依据整体、系统、动态变化
