已测出的全基因组序列
一、问答题(本大题共4小题,每小题25 分,共
100分)
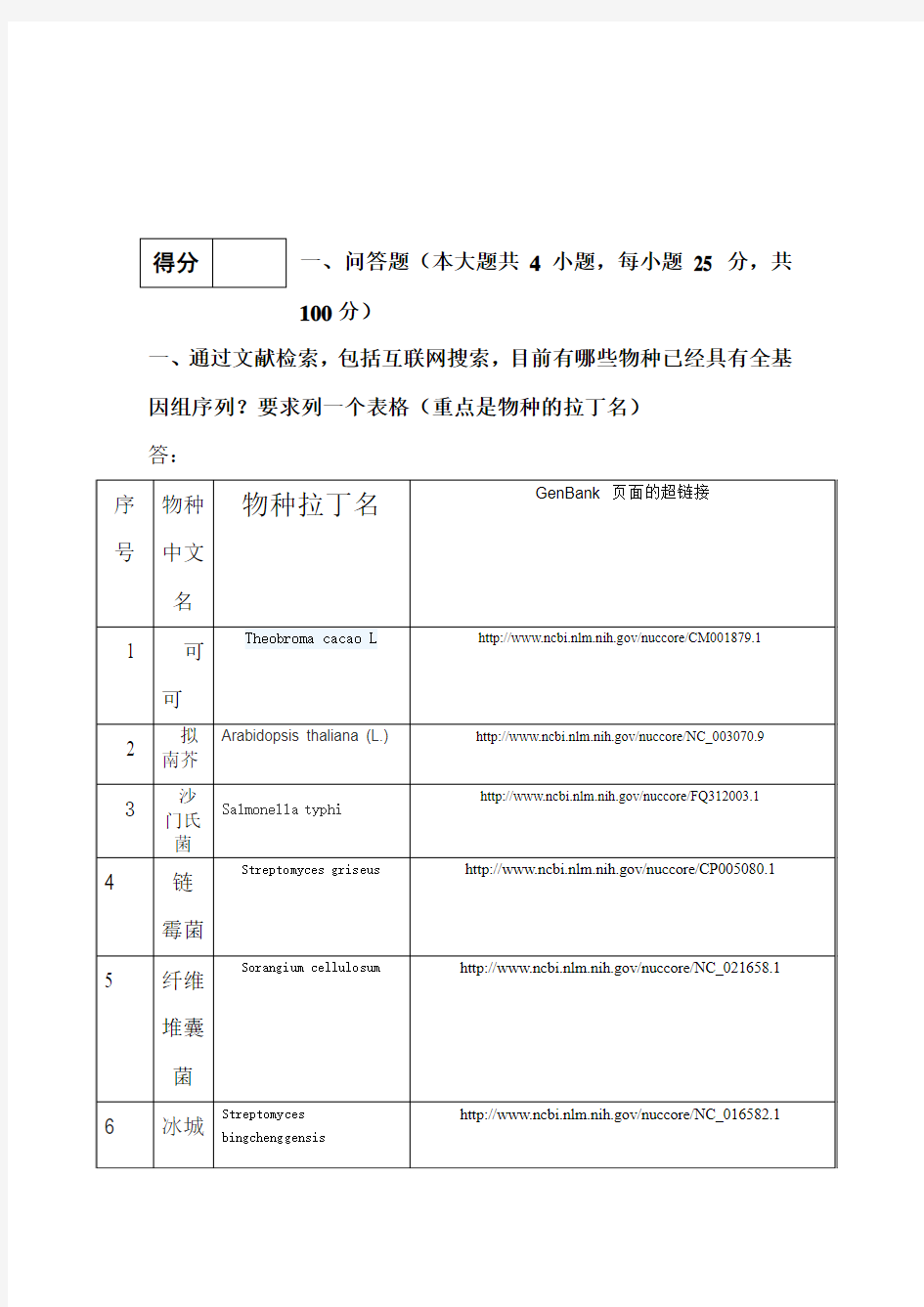
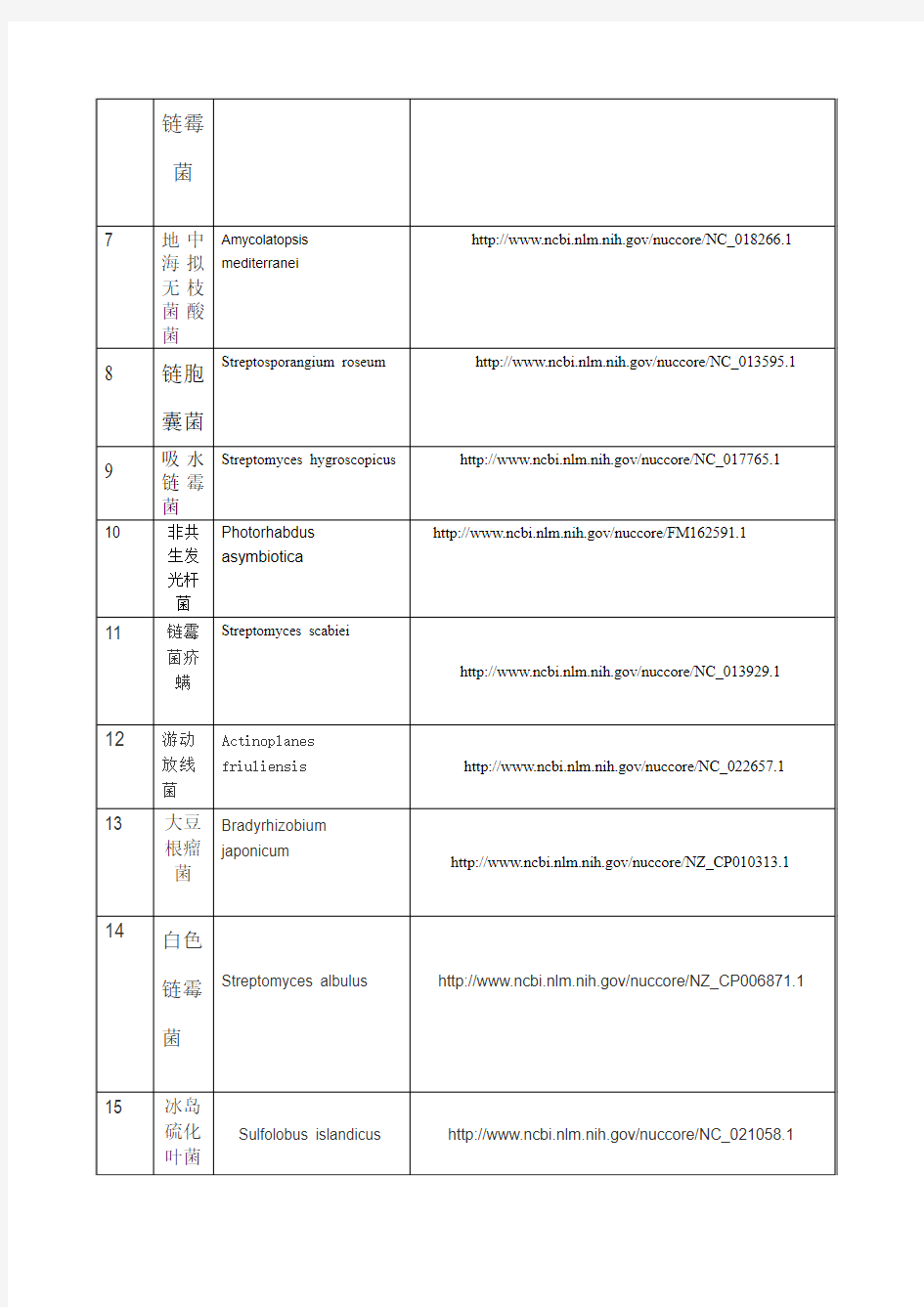
一、通过文献检索,包括互联网搜索,目前有哪些物种已经具有全基因组序列?要求列一个表格(重点是物种的拉丁名)
答:
全基因组测序在遗传病检测中的临床应用专家共识(完整版)
全基因组测序在遗传病检测中的临床应用专家共识(完整版) 遗传病是指由于基因或基因组的结构或功能改变所导致的疾病。下一代测序(next-generation sequencing,NGS)是遗传病检测领域的一项革新性技术。近年来靶向测序和全外显子组测序(whole exome sequencing,WES)得到广泛认可,逐渐成为辅助医生进行遗传病诊断的重要工具[1]。这些检测手段尽管有效,仍然存在一些技术限制,特别是在检测结构变异(structural variations,SV)等方面。全基因组测序(whole genome sequencing,WGS)有望进一步提升临床遗传检测的效能[2]。WGS对受检者基因组中的全部DNA序列进行检测,较WES所覆盖的区域更广,不仅覆盖了几乎全部基因的外显子序列,也覆盖了内含子序列和基因间序列。现在认为WGS可有效避免在对相关基因组区域进行靶向富集时产生的技术偏差,不仅可以检出单核苷酸变异(single nucleotide variations,SNV),还可以对SV进行分析,并常规性地对线粒体基因组(mitochondrial genome DNA,mtDNA)变异进行分析[2,3]。同时其操作步骤相对简化,能更加快速地获得更完整的基因组信息。因此,WGS 应用于临床遗传诊断有望提高诊断率,缩短诊断流程,节省时间及降低诊疗费用[4]。 由于WGS产生的数据涉及受检者的几乎全部遗传信息,其应用于临床遗传病检测需遵循医学伦理中的自愿、患者受益、不伤害和公平原则。
为了实现其应有的临床意义,并妥善处理检测可能带来的复杂遗传咨询问题,本共识列出了WGS作为遗传病诊断检测手段的关键特征,并在检测申请、检测及分析流程、报告及遗传咨询等方面给出建议,但其实施流程及效能验证的具体步骤不在本共识的涵盖范围。本共识适用于以NGS技术为主的高覆盖度WGS(通常>40X)在遗传病临床诊断性检测中的应用,主要针对符合孟德尔遗传规律的基因或基因组疾病。对于WGS在预测性检测、筛查等场景中的应用,不在本共识的讨论范畴。 一、送检目的及临床适应证 1.送检目的: 本共识认为WGS可用于以下送检目的,(1)临床诊断不明但怀疑为遗传病的患者,通过WGS寻求相关的分子诊断和鉴别诊断;(2)临床诊断明确的遗传病患者,为进一步指导治疗或生育寻求分子水平的确诊。 2.临床适应证: WGS适用于怀疑遗传病是患者全部或部分症状原因的所有情况。(1)推荐WGS,①高度怀疑患者有遗传病的可能(如临床症状、体征和其他
自动控制原理复习题(选择和填空)
A.比较元件 B.给定元件 C. 反馈元件 D.放大元件 第一章 自动控制的一般概念 1. 如果被调量随着给定量的变化而变化,这种控制系统叫( ) A. 恒值调节系统 B.随动系统 C. 连续控制系统 D.数字控制系 统 2. 主要用于产生输入信号的元件称为( ) 3. 与开环控制系统相比较,闭环控制系统通常对( )进行直接或间接地测量,通过反馈 环节去影响控制信号。 A.输出量 B. 输入量 C. 扰动量 D . "r 亘. 设定量 4. 直接对控制对象进行操作的元件称为( ) A.给定兀件 B. 放大兀件 C. 比较兀件 D . 执行兀件 5. 对于代表两个或两个以上输入信号进行 ( )的元件又称比较器。 A.微分 B. 相乘 C. 加减 D. 相除 6. 开环控制系统的的特征是没有( ) A.执行环节 B. 给定环节 C. 反馈环节 D . . 放大环节 7. 主要用来产生偏差的兀件称为( ) A.比较兀件 B. 给疋兀件 C. 反馈兀件 D . 放大兀件 8. 某系统的传递函数是 G s _ 1 2s +1 s e , 则该可看成由( ) 环节串联而成。 A.比例.延时 B. 惯性.导前 C. 惯性.延时 D. 惯性.比例 10 . 在信号流图中,在支路上标明的是( ) A.输入 B. 引出点 C. 比较点 D. 传递函数 11. 采用负反馈形式连接后,贝U () A. 一定能使闭环系统稳定; B. 系统动态性能一定会提高; C. 一定能使干扰引起的误差逐渐减小,最后完全消除; D. 需要调整系统的结构参数,才能改善系统性能。
全基因组选择及其在奶牛育种中的应用
发表于《中国奶牛》,2011 全基因组选择育种技术及在奶牛育种中应用进展 范翌鹏1孙东晓1* 张勤1张胜利1张沅1刘林2 (1.中国农业大学动物科技学院,北京,100193; 2.北京奶牛中心. 北京. 100085) 摘要:全基因组选择是指基于基因组育种值(GEBV)的选择方法,指通过检测覆盖全基因组的分子标记,利用基因组水平的遗传信息对个体进行遗传评估,以期获得更高的育种值估计准确度。由于可显著缩短世代间隔,全基因组选择作为一种育种新技术在奶牛育种中具有广阔的应用前景,目前已经成为各国的研究热点。不同国家的试验结果表明,在奶牛育种工作,基于GEBV 的遗传评估可靠性在20-67%之间,如果代替常规后裔测定体系,可节省92%的育种成本。本文综述了全基因组选择的基本原理及其在各国奶牛育种中的应用现状和所面临的问题。 关键词:全基因组选择,奶牛育种 Genome-Wide Selection and its Application in Dairy Cattle FAN YiPeng, SUN Dongxiao, ZHANG Qin, ZHANG Shangli, ZHANG Yuan, LIU Lin (College of Animal Science Technology, China Agricultural University, Beijing, 100193) Abstract: Genomic selection refers to selection decisions based on genomic breeding values (GEBV). The GEBV are calculated as the sum of the effects of dense genetic markers, or haplotypes of these markers, across the entire genome, thereby potentially capturing all the quantitative trait loci (QTL) that contribute to variation in a trait. Genomic selection has become a focus of study in many countries as the new breeding method. Reliabilities of GEBV for young bulls without progeny test results in the reference population were between 20 and 67%. By avoiding progeny testing, bull breeding companies could save up to 92% of their costs [1]. In this paper, we first review the progress of genomic selection, including the principle, methods, accuracy and advantages of genomic selection. We then review the application of genomic selection in dairy cattle. Key words: Genomic Selection, Dairy Breeding 全基因组选择(Genomic Selection,GS),即全基因组范围的标记辅助选择(Marker Assisted Selection, MAS),指通过检测覆盖全基因组的分子标记,利用基因组水平的遗传信息对个体进行遗传评估,以期获得更高的育种值估计准确度。研究已表明,标记辅助选择可提高奶牛育种遗传进展[2][3],但是在目前奶牛育种工作中却无法大规模推广应用标记辅助选择。因为奶牛的生产性状和健康性状均受大量基因座位共同影响,通过有限数量的已知标记无法大幅度加快遗传进展;其次,通过精细定位策略鉴定主效基因需花费大量人力物力和时间;而且利用标记信息估计育种值的计算方法也很复杂。全基因组选择基于基因组育种值(Genomic Estimated Breeding Value, GEBV)进行选择,其实施包括两个步骤:首先在参考群体中使用基因型数据和表型数据估计每个染色体片段的效应;然后在候选群体中使用个体基因型数据估计基因组育种值(genomic breeding value,GEBV)[4],模拟研究证明,仅仅通过标记预测育种值的准确性可以达到0.85(指真实育种值与估计育种值之间的相关,而可靠性则指其平方)。如果在犊牛刚出生时即可达到如此高的准确性,对奶牛育种工作则具有深远意义。模拟研究表明:对于一头刚出生的公犊牛而言,如果其GEBV的估计准确性可以达到经过后
全基因组关联分析的原理和方法
全基因组关联分析(Genome-wide association study;GWAS)是应用基因组中 数以百万计的单核苷酸多态性(single nucleotide ploymorphism ,SNP)为分子 遗传标记,进行全基因组水平上的对照分析或相关性分析,通过比较发现影响复杂性状的基因变异的一种新策略。 随着基因组学研究以及基因芯片技术的发展,人们已通过GWAS方法发现并鉴定了大量与复杂性状相关联的遗传变异。近年来,这种方法在农业动物重要经济性状主效基因的筛查和鉴定中得到了应用。 全基因组关联方法首先在人类医学领域的研究中得到了极大的重视和应用,尤其是其在复杂疾病研究领域中的应用,使许多重要的复杂疾病的研究取得了突破性进展,因而,全基因组关联分析研究方法的设计原理得到重视。 人类的疾病分为单基因疾病和复杂性疾病。单基因疾病是指由于单个基因的突变导致的疾病,通过家系连锁分析的定位克隆方法,人们已发现了囊性纤维化、亨廷顿病等大量单基因疾病的致病基因,这些单基因的突变改变了相应的编码蛋白氨基酸序列或者产量,从而产生了符合孟德尔遗传方式的疾病表型。复杂性疾病是指由于遗传和环境因素的共同作用引起的疾病。目前已经鉴定出的与人类复杂性疾病相关联的SNP位点有439 个。全基因组关联分析技术的重大革新及其应用,极大地推动了基因组医学的发展。(2005年, Science 杂志首次报道了年龄相关性视网膜黄斑变性GWAS结果,在医学界和遗传学界引起了极大的轰动, 此后一系列GWAS陆续展开。2006 年, 波士顿大学医学院联合哈佛大学等多个研究机构报道了基于佛明翰心脏研究样本关于肥胖的GWAS结果(Herbert 等. 2006);2007 年, Saxena 等多个研究组联合报道了与2 型糖尿病( T2D ) 关联的多个位点, Samani 等则发表了冠心病GWAS结果( Samani 等. 2007); 2008 年, Barrett 等通过GWAS发现了30 个与克罗恩病( Crohns ' disrease) 相关的易感位点; 2009 年, W e is s 等通过GWAS发现了与具有高度遗传性的神经发育疾病——自闭症关联的染色体区域。我国学者则通过对12 000 多名汉族系统性红斑狼疮患者以及健康对照者的GWAS发现了5 个红斑狼疮易感基因, 并确定了4 个新的易感位点( Han 等. 2009) 。截至2009 年10 月, 已经陆续报道了关于人类身高、体重、 血压等主要性状, 以及视网膜黄斑、乳腺癌、前列腺癌、白血病、冠心病、肥胖症、糖尿病、精神分 裂症、风湿性关节炎等几十种威胁人类健康的常见疾病的GWAS结果, 累计发表了近万篇 论文, 确定了一系列疾病发病的致病基因、相关基因、易感区域和SNP变异。) 标记基因的选择: 1)Hap Map是展示人类常见遗传变异的一个图谱, 第1 阶段完成后提供了 4 个人类种族[ Yoruban ,Northern and Western European , and Asian ( Chinese and Japanese) ] 共269 个个体基因组, 超过100 万个SNP( 约1
自动控制原理选择题
自动控制原理选择题(48学时) 1.开环控制方式是按 进行控制的,反馈控制方式是按 进行控制的。 (A )偏差;给定量 (B )给定量;偏差 (C )给定量;扰动 (D )扰动;给定量 ( ) 2.自动控制系统的 是系统正常工作的先决条件。 (A )稳定性 (B )动态特性 (C )稳态特性 (D )精确度 ( ) 3.系统的微分方程为 222 )()(5)(dt t r d t t r t c ++=,则系统属于 。 (A )离散系统 (B )线性定常系统 (C )线性时变系统 (D )非线性系统 ( ) 4.系统的微分方程为)()(8)(6)(3)(2233t r t c dt t dc dt t c d dt t c d =+++,则系统属于 。 (A )离散系统 (B )线性定常系统 (C )线性时变系统 (D )非线性系统 ( ) 5.系统的微分方程为()()()()3dc t dr t t c t r t dt dt +=+,则系统属于 。 (A )离散系统 (B )线性定常系统 (C )线性时变系统 (D )非线性系统 ( ) 6.系统的微分方程为()()cos 5c t r t t ω=+,则系统属于 。 (A )离散系统 (B )线性定常系统 (C )线性时变系统 (D )非线性系统 ( ) 7.系统的微分方程为 ττd r dt t dr t r t c t ?∞-++=)(5)(6 )(3)(,则系统属于 。 (A )离散系统 (B )线性定常系统 (C )线性时变系统 (D )非线性系统 ( ) 8.系统的微分方程为 )()(2t r t c =,则系统属于 。 (A )离散系统 (B )线性定常系统 (C )线性时变系统 (D )非线性系统 ( ) 9. 设某系统的传递函数为:,1 2186)()()(2+++==s s s s R s C s G 则单位阶跃响应的模态有: (A )t t e e 2,-- (B )t t te e --,
自动控制原理期末考试题
《 自动控制原理B 》 试题A 卷答案 一、单项选择题(本大题共5小题,每小题2分,共10分) 1.若某负反馈控制系统的开环传递函数为 5 (1) s s +,则该系统的闭环特征方程为 ( D )。 A .(1)0s s += B. (1)50s s ++= C.(1)10s s ++= D.与是否为单位反馈系统有关 2.梅逊公式主要用来( C )。 A.判断稳定性 B.计算输入误差 C.求系统的传递函数 D.求系统的根轨迹 3.关于传递函数,错误的说法是 ( B )。 A.传递函数只适用于线性定常系统; B.传递函数不仅取决于系统的结构参数,给定输入和扰动对传递函数也有影响; C.传递函数一般是为复变量s 的真分式; D.闭环传递函数的极点决定了系统的稳定性。 4.一阶系统的阶跃响应( C )。 A .当时间常数较大时有超调 B .有超调 C .无超调 D .当时间常数较小时有超调 5. 如果输入信号为单位斜坡函数时,系统的稳态误差为无穷大,则此系统为( A ) A . 0型系统 B. I 型系统 C. II 型系统 D. III 型系统 二、填空题(本大题共7小题,每空1分,共10分) 1.一个自动控制系统的性能要求可以概括为三个方面:___稳定性、快速性、__准确性___。 2.对控制系统建模而言,同一个控制系统可以用不同的 数学模型 来描述。 3. 控制系统的基本控制方式为 开环控制 和 闭环控制 。 4. 某负反馈控制系统前向通路的传递函数为()G s ,反馈通路的传递函数为()H s ,则系统 的开环传递函数为()()G s H s ,系统的闭环传递函数为 () 1()() G s G s H s + 。 5 开环传递函数为2(2)(1) ()()(4)(22) K s s G s H s s s s s ++= +++,其根轨迹的起点为0,4,1j --±。 6. 当欠阻尼二阶系统的阻尼比减小时,在单位阶跃输入信号作用下,最大超调量将 增大 。 7.串联方框图的等效传递函数等于各串联传递函数之 积 。 三、简答题(本题10分) 图1为水温控制系统示意图。冷水在热交换器中由通入的蒸汽加热,从而得到一定温度的热水。冷水流量变化用流量计测量。试绘制系统方框图,并说明为了保持热水温度为期望值,系统是如何工作的?系统的被控对象和控制装置各是什么?
全基因组选择育种策略及在水产动物育种中的应用前景(精)
中国水产科学 2011年7月, 18(4: 936?943 Journal of Fishery Sciences of China 综述 收稿日期: 2011?03?14; 修订日期: 2011?04?10. 基金项目: 国家自然基金资助项目(30730071; 30972245; 农业科技成果转化资金项目(2010GB24910700. 作者简介: 于洋(1987?, 硕士研究生. E-mail: yuy8866@https://www.360docs.net/doc/8614317048.html, 通信作者: 张晓军, 副研究员. E-mail: xjzhang@https://www.360docs.net/doc/8614317048.html, DOI: 10.3724/SP.J.1118.2011.00935 全基因组选择育种策略及在水产动物育种中的应用前景 于洋1,2 , 张晓军1 , 李富花1 , 相建海1 1. 中国科学院海洋研究所实验海洋生物学重点实验室, 山东青岛266071; 2. 中国科学院研究生院, 北京 100049 摘要: 全基因组选择的概念自2001年由Meuwissen 等提出后便引起了动物育种工作者的广泛关注。目前, 澳大利亚、新西兰、荷兰、美国的研究小组已经应用该方法进行了优质种牛的选择育种, 并取得了很好的效果。此外在鸡和猪的选择育种中也有该方法的应用, 但在水产动物选育中尚未见该方法使用的报道。本文对“全基因组选择育种”的概念和提出背景进行了归纳, 对全基因组选择育种的优势进行了阐述, 并详细介绍了其具体的策略, 总结了目前全基因组育种所广泛采用的方法以及取得的成果, 旨在为该方法在水产动物育种方面的应用研究提供科学参考。 关键词: 全基因组选择; 水产动物育种; SNP; QTL; 全基因组育种值估计 中图分类号: S96 文献标志码: A 文章编号: 1005?8737?(201104?0935?08 人类对于动物的选择育种由来已久, 最初所进行的只是简单的人工驯化。随着遗传学研究的发展, 尤其是“数量遗传学理论”的提出, 动物育种技术进入快速发展时
全基因组重测序大数据分析报告
全基因组重测序数据分析 1. 简介(Introduction) 通过高通量测序识别发现de novo的somatic和germ line 突变,结构变异-SNV,包括重排突变(deletioin, duplication 以及copy number variation)以及SNP的座位;针对重排突变和SNP的功能性进行综合分析;我们将分析基因功能(包括miRNA),重组率(Recombination)情况,杂合性缺失(LOH)以及进化选择与mutation之间的关系;以及这些关系将怎样使得在disease(cancer)genome中的mutation产生对应的易感机制和功能。我们将在基因组学以及比较基因组学,群体遗传学综合层面上深入探索疾病基因组和癌症基因组。 实验设计与样本 (1)Case-Control 对照组设计; (2)家庭成员组设计:父母-子女组(4人、3人组或多人); 初级数据分析 1.数据量产出:总碱基数量、Total Mapping Reads、Uniquely Mapping Reads统计,测序深度分析。 2.一致性序列组装:与参考基因组序列(Reference genome sequence)的比对分析,利用贝叶斯统计模型检测出每个碱基位点的最大可能性基因型,并组装出该个体基因组的一致序列。 3.SNP检测及在基因组中的分布:提取全基因组中所有多态性位点,结合质量值、测序深度、重复性等因素作进一步的过滤筛选,最终得到可信度高的SNP数据集。并根据参考基因组信息对检测到的变异进行注释。 4.InDel检测及在基因组的分布: 在进行mapping的过程中,进行容gap的比对并检测可信的short InDel。在检测过程中,gap的长度为1~5个碱基。对于每个InDel的检测,至少需要3个Paired-End序列的支持。 5.Structure Variation检测及在基因组中的分布: 能够检测到的结构变异类型主要有:
如何查找基因的序列(全)
如何查找基因序列?(转载) (2010-08-01 11:47:41) 如何查找基因序列? ——在Genbank中寻找目的基因的实例 ——献给受类似问题困扰的广大酷友,以及给我动力和信心发表原创帖的基因酷的朋友们。 酷友感言:网络的世界很精彩,网络的查询很无奈。为了我们的科学研究事业,为了我们能够顺利毕业,我们的广大酷友们在网络的海洋里遨游…遨游…咋就找不到彼岸呢?今天要设计这个基因的PCR引物,明天又要查那个基因的信息,那么大一张网,唉想起来就郁闷……鉴此,我们推出了利用Genbank查找基因序列的帖子,希望对大家有所帮助,并请大家多多指教!当然,如果您已经是此中高手,那就权当我是班门弄斧了,呵呵。 1. 根据文献 搞reasearch肯定要读文献的,如果你曾经在文献中看到过你感兴趣的基因,而且文中还提到了该基因在Genbank中的ID号,那就好办了,直接打开https://www.360docs.net/doc/8614317048.html,,在Search后的下拉框中选择Nucleotide,把Genbank ID号输入GO前面的文本框中,点“GO”,就可以找到他了。 举例说明,例如:在2003年JBC的文章(Conditional Knock-out of
Integrin-linked Kinase Demonstrates an Essential Role in Protein Kinase B/Akt Activation)中出现了“calreticulin (GenBank accession number gi 16151096)”,那么把“16151096”输入GO前面的文本框中,点“GO”,就可以找到该基因了(当然包括基因序列等相关信息)。 在出现了检索结果界面(下图)后,直接点击红箭头所指的 AY047586就可以看到基因的相关信息了...(呵呵,是不是有点太......easy 了) 这里需要指出一下,在显示基因的页面右侧有一个Link,点击后出现一个小菜单,里面是与该基因相关的链接,很有用的,值得一个一个地去看看,这里我就不多说了。点击 AY047586后出现的界面如下:如果你只想获得序列(例如去设计PCR引物的时候),那就可以选择FASTA,这样就得到了FASTA格式的序列文件,没有其他数字和格式的干扰。 (缩略图,点击图片链接看原图)这就是FASTA格式的序列: (缩略图,点击图片链接看原图)2. 根据已经获得的基因的相关信息进行查找(待续......) 鼓励一下吧,累坏了正如路漫漫所说,如果只是知道基因的名字,怎么查序列呢?还是举例说明,比如我想做的基因名称是人的VEGF基因,那么怎么在Genbank中找到它呢?还是一步一步来...打开https://www.360docs.net/doc/8614317048.html,/ 在search后面的下拉框中选择Gene,然后在中间的文本框中输入基
自动控制原理选择填空
自动控制原理选择填空
1、反馈控制又称偏差控制,其控制作用是通过 给定值 与反馈量的差值进行的。 2、复合控制有两种基本形式:即按 输入 的前馈复合控制和按 扰动 的前馈复合控制。 3、两个传递函数分别为G 1(s)与G 2(s)的环节,以并联方式连接,其等效传递函数为()G s ,则G(s)为 G1(s)+G2(s) (用G 1(s)与G 2(s) 表示)。 4、典型二阶系统极点分布如图1所示, 则无阻尼自然频率=n ω 2 , 阻尼比=ξ 20.7072 = , 该系统的特征方程为 2220s s ++= , 该系统的单位阶跃响应曲线为 衰减振荡 。 5、若某系统的单位脉冲响应为0.20.5()105t t g t e e --=+, 则该系统的传递函数G(s)为 1050.20.5s s s s +++ 。 6、根轨迹起始于 开环极点 ,终止于 开环零点 。 7、设某最小相位系统的相频特性为101()()90()tg tg T ?ωτωω--=--,则该系 统的开环传递函数为 (1) (1)K s s Ts τ++ 。 8、PI 控制器的输入-输出关系的时域表达式是 1()[()()]p u t K e t e t dt T =+? , 其相应的传递函数为 1[1]p K Ts + ,由于积分环节的引入,可以改善系统的 稳态性能 性能。 二、选择题(每题 2 分,共20分)
1、采用负反馈形式连接后,则 ( D ) A 、一定能使闭环系统稳定; B 、系统动态性能一定会提高; C 、一定能使干扰引起的误差逐渐减小,最后完全消除; D 、需要调整系统的结构参数,才能改善系统性能。 2、下列哪种措施对提高系统的稳定性没有效果 ( A )。 A 、增加开环极点; B 、在积分环节外加单位负反馈; C 、增加开环零点; D 、引入串联超前校正装置。 3、系统特征方程为 0632)(23=+++=s s s s D ,则系统 ( C ) A 、稳定; B 、单位阶跃响应曲线为单调指数上升; C 、临界稳定; D 、右半平面闭环极点数2=Z 。 4、系统在2)(t t r =作用下的稳态误差∞=ss e ,说明 ( A ) A 、 型别2 王前飞: (1)为什么要研究表观遗传学? 答: 表观遗传学主要通过DNA 的甲基化、组蛋白修饰、染色质重塑和非编码RNA 调控等方式控制基因表达。表观遗传学是近几年兴起的而且发展迅速的一个研究遗传的分支学科,其研究和应用不仅对基因表达、调控、遗传有重要作用,而且在肿瘤、免疫等许多疾病的发生和防治以及干细胞定向分化研究、基因芯片中亦具有十分重要的意义。表观遗传学补充了“中心法则”忽略的两个问题,即哪些因素决定了基因的正常转录和翻译以及核酸并不是存储遗传信息的唯一载体;在分子水平上,表观遗传学解释了DNA序列所不能解释的诸多奇怪的现象。如: 同一等位基因可因亲源性别不同而产生不同的基因印记疾病,疾病严重程度也可因亲源性别而异。表观遗传学信息还可直接与药物、饮食、生活习惯和环境因素等联系起来,营养状态能够通过改变表观遗传以导致癌症发生,尤其是维生素和必需氨基酸。 此外,表观遗传学信息的改变,对包括人体在内的哺乳动物基因组有广泛而重要的效应,如转录抑制、基因组印记、细胞凋亡、染色体灭活等。DNA 甲基化模式的改变,尤其是某些抑癌基因局部甲基化水平的异常增加,在肿瘤的发生和发展过程中起到了不容忽视的作用。研究发现,肿瘤细胞DNA 存在广泛的低甲基化和局部区域的高甲基化共存现象,以及总的甲基化能力增高,这3个特征各以不同的机制共同参与甲基化在肿瘤发生、发展中的作用。如胃癌、结肠癌、乳腺癌、肺癌、胰腺癌等众多恶性肿瘤都不同程度地存在一个或多个肿瘤抑制基因CpG 岛甲基化。而表观遗传学改变在本质上的可逆性,又为肿瘤的防治提供了新的策略。所以,随着表观遗传学研究的深入,肯定会对人类生长发育、肿瘤发生以及遗传病的发病机制及其防治做出新的贡献,也必将在其他领域中展示其不可估量的作用和广阔的前景。 (2)表观遗传学涉及到哪些方面? 答: 表观遗传学的研究内容主要包括:DNA甲基化、组蛋白的末端修饰和变异体、DNAaseⅠ高敏感位点、非编码RNA、转录因子及其辅助因子、顺式调控元件和基因组印记等。 (3)什么因素会影响基因表达水平? 答: 基因选择性转录表达的调控( DNA甲基化,基因印记,组蛋白共价修饰,染色质重塑) 基因转录后的调控(基因组中非编码RNA,微小RNA(miRNA),反义RNA、内含子、核糖开关等) 1.转录水平的调控:包括DNA转录成RNA时的是否转录及转录频率的调控,DNA 的序列决定了DNA的空间构型,DNA的空间构型决定了转录因子是否可以顺利的结合到DNA的调控序列上,比如结合到TATA等序列上。 2.翻译水平的调控:翻译水平的调控又可以分成翻译前的调控和翻译后的调控。 a、翻译前的调控主要是RNA编辑修饰。 b、翻译后调控主要是蛋白的修饰,蛋白修饰后可以成为有功能的蛋白或者有隐藏功能的蛋白。 在真核和原核细胞中,从基因表达到蛋白质合成,其间有许多地方受到调控,这 全基因组关联分析(GWAS)解决方案 ※ 概述 全基因组关联研究(Genome-wide association study,GWAS)是用来检测全基因组范围的遗传变异与 可观测的性状之间的遗传关联的一种策略。2005年,Science杂志报道了第一篇GWAS研究——年龄相关性黄 斑变性,之后陆续出现了有关冠心病、肥胖、2型糖尿病、甘油三酯、精神分裂症等的研究报道。截至2010年 底,单是在人类上就有1212篇GWAS文章被发表,涉及210个性状。GWAS主要基于共变法的思想,该方法是 人类进行科学思维和实践的最重要工具之一;统计学研究也表明,GWAS很长时期内都将处于蓬勃发展期(如 下图所示)。 基因型数据和表型数据的获得,随着诸多新技术的发展变得日益海量、廉价、快捷、准确和全面:如 Affymetrix和Illumina公司的SNP基因分型芯片已经可以达到2M的标记密度;便携式电子器械将产生海量的表型 数据;新一代测序技术的迅猛发展,将催生更高通量、更多类别的基因型,以及不同类别的高通量表型。基于 此,我们推出GWAS的完整解决方案,协助您一起探索生物奥秘。 ※ 实验技术流程 ※ 基于芯片的GWAS Affymetrix公司针对人类全基因组SNP检测推出多个版本检测芯片,2007年5月份,Affymetrix公司发布了 人全基因组SNP 6.0芯片,包含90多万个用于单核苷酸多态性(SNP)检测探针和更多数量的用于拷贝数变化(CNV)检测的非多态性探针。因此这种芯片可检测超过180万个位点基因组序列变异,即可用于全基因组 SNP分析,又可用于CNV分析,真正实现了一种芯片两种用途,方便研究者挖掘基因组序列变异信息。 Illumina激光共聚焦微珠芯片平台为全世界的科研用户提供了最为先进的SNP(单核苷酸多态性)研究平 台。Illumina的SNP芯片有两类,一类是基于infinium技术的全基因组SNP检测芯片(Infinium? Whole Genome Genotyping),适用于全基因组SNP分型研究及基因拷贝数变化研究,一张芯片检测几十万标签SNP位点,提 供大规模疾病基因扫描(Hap660,1M)。另一类是基于GoldenGate?特定SNP位点检测芯片,根据研究需要挑选SNP位点制作成芯片(48-1536位点),是复杂疾病基因定位的最佳工具。 罗氏NimbleGen根据人类基因组序列信息设计的2.1M超高密度CGH芯片,可以在1.1Kb分辨率下完成全基 因组检测,可有效检测人基因组中低至约5kb大小的拷贝数变异。 自动控制原理1 一、单项选择题(每小题1分,共20分) 1. 系统和输入已知,求输出并对动态特性进行研究,称为( ) A.系统综合 B.系统辨识 C.系统分析 D.系统设计 2. 惯性环节和积分环节的频率特性在( )上相等。 A.幅频特性的斜率 B.最小幅值 C.相位变化率 D.穿越频率 3. 通过测量输出量,产生一个与输出信号存在确定函数比例关系值的元件称为( ) A.比较元件 B.给定元件 C.反馈元件 D.放大元件 4. ω从0变化到+∞时,延迟环节频率特性极坐标图为( ) A.圆 B.半圆 C.椭圆 D.双曲线 5. 当忽略电动机的电枢电感后,以电动机的转速为输出变量,电枢电压为输入变量时,电 动机可看作一个( ) } A.比例环节 B.微分环节 C.积分环节 D.惯性环节 6. 若系统的开环传 递函数为2) (5 10+s s ,则它的开环增益为( ) .2 C 7. 二阶系统的传递函数52 5)(2++= s s s G ,则该系统是( ) A.临界阻尼系统 B.欠阻尼系统 C.过阻尼系统 D.零阻尼系统 8. 若保持二阶系统的ζ不变,提高ωn ,则可以( ) A.提高上升时间和峰值时间 B.减少上升时间和峰值时间 C.提高上升时间和调整时间 D.减少上升时间和超调量 9. 一阶微分环节Ts s G +=1)(,当频率T 1=ω时,则相频特性)(ωj G ∠为( ) ° ° ° ° 10.最小相位系统的开环增益越大,其( ) > A.振荡次数越多 B.稳定裕量越大 C.相位变化越小 D.稳态误差越小 11.设系统的特征方程为()0516178234=++++=s s s s s D ,则此系统 ( ) A.稳定 B.临界稳定 C.不稳定 D.稳定性不确定。 12.某单位反馈系统的开环传递函数为:()) 5)(1(++=s s s k s G ,当k =( )时,闭环系统临界稳定。 .20 C 13.设系统的特征方程为()025103234=++++=s s s s s D ,则此系统中包含正实部特征的个数 有( ) .1 C A f f y m e t r i x全基因组S N P芯片检测 单核苷酸多态性(single nucleotide polymorphism, SNP) 指基因组单个核苷酸的变异,它是最微小的变异单元,是由单个核苷酸对置换、颠换、插入或缺失所形成的变异形式。单核苷酸多态性是基因组上高密度的遗传标志,在人类基因组中已发现的SNP数量超过3000万。作为第三代遗传标记,SNP数量众多、分布密集、易于检测,因而是理想的基因分型目标。SNP分型检测在疾病基因组(如疾病易感性),药物基因组(药效、药物代谢差异和不良反应)和群体进化等研究中具有重大意义。在人研究方面,Affymetrix 公司有分别基于GeneChip和GeneTitan平台的SNP 6.0 芯片和针对中国人群设计的CHB1&2 Array,既可用于全基因组SNP分析,又可用于CNV分析,极大地方便了中国人类疾病GWAS研究。Affymetrix公司针对多个农业物种也开发了多款商品化的基因分型芯片,如鸡、牛、水牛、鲑鱼、水稻、小麦、辣椒、草莓等,为农业育种研究、遗传图谱构建、群体基因组学研究提供研究手段。此外,Affymetrix公司还支持定制芯片,最低起订量为480个样品。 检测原理|?技术优势|?产品列表|?定制芯片|?数据分析| 基于GeneChip平台的人SNP 6.0 芯片实验流程: 基于GeneTitan平台的Axiom基因分型芯片检测流程: 从SNP原理谈SNP分析技术之SNP芯片 日期:2012-05-21 ? ? 来源:网络 标签:?SNP原理?SNP分析?SNP芯片 摘要:?SNP是近年来基因突变的热点研究之一。它是指在单个的核苷酸上发生了变异,有四种不同的变异形式,而实际上只发生转换和颠换这两种。当科学家弄清了SNP的突变原理以后,他们就着手对SNP进行分析,以求找到疾病相对应的突变位点或者是进行个性化药物治疗研究。其中应用到的技术多达上百余种,其中包括有测序技术、质谱分析技术、HRM技术、Taqman技术以及SNP芯片技术。 恩必美生物新一轮2-5折生物试剂大促销! Ibidi细胞灌流培养系统-模拟血管血液流动状态下的细胞培养系统 广州赛诚生物基因表达调控专题 SNP是近年来基因突变的热点研究之一。它是指在单个的核苷酸上发生了变异,有四种不同的变异形式,而实际上只发生转换和颠换这两种。当科学家弄清了SNP的突变原理以后,他们就着手对SNP进行分析,以求找到疾病相对应的突变位点或者是进行个性化药物治疗研究。其中应用到的技术多达上百余种,其中包括有测序技术、质谱分析技术、HRM 技术、Taqman技术以及SNP芯片技术。 1、反馈控制又称偏差控制,其控制作用是通过 给定值 与反馈量的 差值进行的。 2、复合控制有两种基本形式:即按 输入 的前馈复合控制和按 扰动 的前馈复合控制。 3、两个传递函数分别为G 1(s)与G 2(s)的环节,以并联方式连接,其等效传 递函数为()G s ,则G(s)为 G1(s)+G2(s) (用G 1(s)与G 2(s) 表示)。 4、典型二阶系统极点分布如图1所示, 则无阻尼自然频率=n ω 阻尼比=ξ 0.707= , 该系统的特征方程为 2220s s ++= , 该系统的单位阶跃响应曲线为 衰减振荡 。 5、若某系统的单位脉冲响应为0.20.5()105t t g t e e --=+, 则该系统的传递函数G(s)为 1050.20.5s s s s +++ 。 6、根轨迹起始于 开环极点 ,终止于 开环零 点 。 7、设某最小相位系统的相频特性为101()()90()tg tg T ?ωτωω--=--,则该 系统的开环传递函数为 (1) (1)K s s Ts τ++ 。 8、PI 控制器的输入-输出关系的时域表达式是 1()[()()]p u t K e t e t d t T =+? , 其相应的传递函数为 1[1]p K Ts + ,由于积分环节的引入,可以改善系统的 稳态性能 性能。 二、选择题(每题 2 分,共20分) 1、采用负反馈形式连接后,则 ( D ) A 、一定能使闭环系统稳定; B 、系统动态性能一定会提高; C 、一定能使干扰引起的误差逐渐减小,最后完全消除; D 、需要调整系统的结构参数,才能改善系统性能。 2、下列哪种措施对提高系统的稳定性没有效果 ( A )。 A 、增加开环极点; B 、在积分环节外加单位负反 馈; C 、增加开环零点; D 、引入串联超前校正装置。 3、系统特征方程为 0632)(23=+++=s s s s D ,则系统 ( C ) A 、稳定; B 、单位阶跃响应曲线为单调指数上 升; C 、临界稳定; D 、右半平面闭环极点数2=Z 。 4、系统在2)(t t r =作用下的稳态误差∞=ss e ,说明 ( A ) A 、 型别2 全基因组选择在猪育种上的研究进展 自野生动物被驯化以来,科学家一直致力于提高畜禽育种值的研究。近半个世纪来,畜禽育种值估计的方法主要经历了综合选择指数法、同期群体比较法、最佳线性无偏预测法(Best LinearUnbiased Prediction,BLUP)、分子标记辅助选择育种(MAS)以及近几年快速发展的GS 法。同时,随着高密度基因芯片的出现和高通量测序技术的快速发展,单核苷酸多态性(SingleNucleotide Polymorphism,SNP)分型成本快速下降,GS 才逐渐引起畜禽界的关注。特别是Schaeffer发现,在奶牛育种中利用GS比后裔测定可节约成本97%,且遗传进展可提高3~4倍后,全球掀起了一股研究GS的热潮。 全基因组选择(GS) 什么是GS 2001年,Meuwissen等人最先提出GS,实质为全基因组范围的标记辅助选择。其理论基础是应用整个基因组的标记信息和各性状值来估计每个标记或染色体片段的效应值,然后将效应值加和即得到基因组育种值(GenomicEstimated Breeding Value,GEBV)。GS在某种程度上是MAS的延伸,弥补了在MAS 中标记数量只能解释一部分遗传方差以及数量性状位点(QuantitativeTrait Locus,QTL) 定位困难的缺点。其中心任务是提高GEBV值的准确性,并尽可能准确地估计每个标记的效应。而估计标记效应的方法在实际运用中以BLUP法为主;Bayes法虽其准确性高于BLUP,但因其计算复杂,需在超级计算机上运行而限制其应用。不过随着快速算法的开发和计算机硬件的改进,Bayes法的运算效率有望提高。 为什么选用GS GS的优势 与MAS相比,GS的优势主要表现在: 1)能对所有的遗传和变异效应做出准确的估计。而MAS 只能对部分遗传变异进行检测,且容易高估其遗传效应。 2)缩短世代间隔、提高畜禽年遗传进展、降低生产成本等,这在需要后裔测定的家畜中尤为明显。如GS给奶牛育种带来了巨大经济效益。 3)早期选择准确率高。 4)对于较难实施选择的性状具有重大影响。如低遗传力性状、难以测定的性状等。 5)GS在提高种群的遗传进展前提下,还能降低群体的近交增量。 GS的可靠性 利用全基因组重测序技术对某一物种个体或群体的基因组进行测序及差异分析,可获得SNP、InDel、SV、CNV、PAV、转座子等大量的遗传多态性信息,建立遗传多态性数据库,为后续揭示进化关系、功能基因挖掘等奠定基础。基于全基因组重测序技术进行变异检测,可在全基因组范围内精确检测多种变异,与传统的分子标记和芯片相比,具有周期短、密度高、检测全面、性价比高等技术优势。诺禾致源使用的检测软件及参数为国际高影响因子文章通用标准,对获 得的变异进行全方位评估以确保变异准确、验证率高。技术参数SNP检测、注释及统计 InDel检测、注释及统计 SV检测、注释及统计 CNV检测、注释及统计 转座子检测、注释及统计 样品要求 文库类型测序策略分析内容项目周期 变异检测(基于全基因组重测序)SNP检测周期为30天;全部变异检测周期为40天,个性化分析时间需根据项目实际情况进行评估 HiSeq PE150SNP、InDel(≥10X),SV(≥20X),CNV(≥30X),转座子(≥20X) DNA文库 DNA样品总量: ≥3 μg 参考文献 [1] Cheng Z, Lin J, Lin T, et al . Genome-wide analysis of radiation-induced mutations in rice (Oryza sativa L. ssp. indica) [J]. Molecular BioSystems, 2014, 10(4): 795-805. [2] Jansen S, Aigner B, Pausch H, et al . Assessment of the genomic variation in a cattle population by re-sequencing of key animals at low to medium coverage [J]. BMC genomics, 2013, 14(1): 446.案例解析 [案例一] 水稻辐射诱导突变体的全基因组变异分析[1] 辐射是进行水稻种质资源创新的一种有效手段。然而,通过辐射诱发突 变的分子机制尚不清楚。该研究对由9311 经γ-射线诱变而来,富含对人 体有益成分的突变体Red-1进行全基因组测序(20X),并与参考基因组 比对,全面揭示辐射诱导水稻突变体的序列情况。结果显示,9.19%的 基因组序列发生改变,突变位于14,493个基因中,其中点突变是主要的 变异类型。与结合功能、催化活性、代谢过程的相关的基因更易被γ-射 线诱变。该研究首次将全基因组测序技术应用于辐射诱变机理研究,为 诱变育种研究提供了新的思路。 [案例二] 牛种群关键个体中低深度测序评估基因组多态性[2] 该研究对43个牛关键个体(代表Fleckvieh种群68%多态性)进行二代 测序,测序深度为4.17-24.98X,平均深度7.46X。与参考基因组比对, 检测出1700万个多态性位点,91,733个变异在18,444个基因编码区 中,且46%为非同义突变。非同义突变中,575个变异可能与转录提前 终止相关,3个变异出现在OMIA数据库中并与特定表型相关。结果表 明,通过对能代表种群主要多态性的关键个体进行低、中深度测序,能 够高效、准确地获得该物种大量多态性信息。 图1 Red-1水稻基因组的变异特征 图2 变异注释基因组学(复习)
全基因组关联分析(GWAS)解决方案
自动控制原理选择题库
Affymetrix全基因组SNP芯片检测
自动控制原理选择填空
全基因组选择在猪育种上的研究进展
全基因组的变异检测
