动植物全基因组测序

2013.07
2013.102014.09
2014.11
2015.04
图1 异源多倍体棉花基因组共线性分析与非对称进化分析
图2 MYB基因家族表达模式分析
, Jiang W, et al. Sequencing of allotetraploid cotton (Gossypium hirsutum L. acc. TM-1) provides a resource for fiber improvement [J]. Nature Biotechnology, 2015, 33(5): 531-537.
图3 金丝猴植食性机制的分析图4 金丝猴有效群体大小分析
参考文献
Zhou X, Wang B, Pan Q, Li R, Li M. Whole-genome sequencing of the nub-nosed monkey provides insights into folivory and evolutionary history [J]. Nature Genetics, 2014, 46(12):1303-1310.
图5 藏猪及其它猪种的群体遗传结构分析
参考文献
Li M, Tian S, Jin L, et al. Genomic analyses identify distinct patterns of selection in domesticated pigs and Tibetan wild boars [J]. Nature genetics, 2013, 45(12): 1431-1438.
图6 进化分析结果图7 脂肪酸能量代谢途径
蓝色表示正选择基因;红色表示特异性基因
参考文献
Qu Y, Zhao H, Han N, et al. Ground tit genome reveals avian adaptation to living at high altitudes in the Tibetan plateau [J]. Nature communications, 2013, 4.
图1 7株野生大豆共有和特有基因集图2 野生大豆开花时间调控基因SNP和InDel变异
参考文献
Li Y, Zhou G, Ma J, Jiang W, Li R#, et al. De novo assembly of soybean wild relatives for pan-genome analysis of diversity and agronomic traits [J]. Nature biotechnology, 2014.32(10):1045-1052.
图3 部分novel sequence在世界人群中的分布
参考文献
Li R, Li Y, Zheng H, et al. Building the sequence map of the human pan-genome [J]. Nature biotechnology, 2010, 28(1): 57-63.
诺禾致源高分文章集锦-植物基因组
陆地棉基因组测序揭示四倍体棉进化与纤维发育机制Sequencing of allotetraploid cotton (Gossypium hirsutum L. acc. TM-1) provides a resource for fiber improvement 研究对象:陆地棉遗传标准系TM-1 期刊:Nature Biotechnology 影响因子:41.514 合作单位:南京农业大学 发表时间:2015年4月 摘 要 Upland cotton is a model for polyploid crop domestication and transgenic improvement. Here we sequenced the allotetraploid Gossypium hirsutum L. acc. TM-1 genome by integrating whole-genome shotgun reads, bacterial artificial chromosome (BAC)-end sequences and genotype-by-sequencing genetic maps. We assembled and annotated 32,032 A-subgenome genes and 34,402 D-subgenome genes. Structural rearrangements, gene loss, disrupted genes and sequence divergence were more common in the A subgenome than in the D subgenome, suggesting asymmetric evolution. However, no genome-wide expression dominance was found between the subgenomes. Genomic signatures of selection and domestication are associated with positively selected genes (PSGs) for fiber improvement in the A subgenome and for stress tolerance in the D subgenome. This draft genome sequence provides a resource for engineering superior cotton lines.关键词 陆地棉;de novo;四倍体 研究背景 陆地棉(Gossypium hirsutum L.)隶属锦葵目(Malvales),锦葵科(Malvaceae),棉属(Gossypium),因最早在美洲大陆种植而得名,是世界上最重要的棉花栽培品种,占全球棉花种植面积的90%以上。尽管陆地棉在棉花产业中占据核心地位,但由于其为异源四倍体,相关的全基因组测序工作一直难以开展。来自南京农业大学、北京诺禾致源、美国德克斯大学的国际团队,利用最新测序技术,成功构建了高质量的陆地棉全基因组图谱,为进一步改良棉花的农艺性状提供了基础,同时也为多倍体植物的形成和演化机制提供了新的启示。
植物数量性状全基因组选择研究进展
4期吴永升等:植物数量性状全基因组选择研究进展1511 全基因组选择的概念和原理 全基因组选择(Genome-wideselection,GWS),又称基因组选择(Genomicselection,GS),由Meu—wissen于2001年首先提出∞J。主要是通过全基因组中大量的分子标记和参照群体(trainingpopula—tion)的表型数据建立BLUP模型估计出每一标记的育种值,然后仅利用同样的分子标记估计出后代个体育种值并进行选择[7】。 全基因组选择理论主要利用连锁不平衡信息,即假设标记与其相邻的QTL处于连锁不平衡状态,因而由相同标记估计的不同群体的染色体片段效应是相同的,这就要求标记密度足够高以使所有的QTL与标记处于连锁不平衡(LD)状态哺J。而目前随着拟南芥、水稻、玉米等植物基因组序列图谱及SNP图谱的完成或即将完成,提供了大量的SNP标记用于基因组研究。而随着SNP芯片等大规模高通量SNP检测技术的发展和成本的降低,使得全基因组选择应用成为可能。 2全基因组选择的基本方法及案例说明 2.1全基因组选择的基本方法 全基因组选择在实施过程中应该包括以下几个基本步骤:在需要实行选择的参照群体中获取参照群体的基因型数据和表现型数据;然后,通过BLUP程序估计出每个标记位点的标记效应值,从而获得育种值;最后,在接下来每一轮的选择中,不再需要表型数据,根据每一轮次群体基因型信息估计育种值,直接选择群体的优良单株【9j。 全基因组选择的核心过程就是用从参照群体中每一个体的表现型数据和基因型数据建立的数学模型来估算接下来的育种群体中仅有基因型数据的个体的GEBV值。由既有表现型数据又有基因型数据的每一个体组成的群体被成为参照群体。参照群体用来估计数学模型的参数,这个参数接着用来计算仅有基因型数据的育种个体GEBV值,然后根据计算的GEBV值对育种群体进行选择并提升到下一轮次的选择中。因此,通过模型来预测个体的育种值,可以不进行表型鉴定就直接对育种群体的个体进行选择(Meuvissen,2001)。为了使估算的GEBV值尽可能地准确,参照群体必须具有代表性,尽可能地代表接下来在育种过程中用全基因组选择方法来进行选择的分离群体。 2.2全基因组选择方法案例 如图l所示,在这个例子中,笔者的目标是把外来种质中的优良性状基因(包括产量、矮杆、抗逆等)导入本地优良的自交系,从而实现种质的改良 图1在玉米中利用全基因组选择方法导入外源种质 Fig.1Genomewideselectionto introgr%exotictraitsintoadaptedmaize
植物功能基因组学及其研究技术_崔兴国
第9卷 第1期2007年3月 衡水学院学报 J o u r n a l o f H e n g s h u i U n i v e r s i t y V o l.9,N o.1 Ma r.2007植物功能基因组学及其研究技术 崔兴国 (衡水学院 生命科学系,河北 衡水053000) 摘 要:植物基因组的研究已经由以全基因组测序为目标的结构基因组学转向以基因功能鉴定为目标的功能基因组学研究.植物功能基因组学研究是利用结构基因组学积累的数据,从中得到有价值的信息,阐述D N A序列的功能,从而对所有基因如何行使其职能并控制各种生命现象的问题作出回答.近年来植物功能基因组学的研究技术主要包括表达序列标签、基因表达的系列分析、D N A微阵列和反向遗传学等.对植物功能基因组学的研究将有利于我们对基因功能的理解和对植物形状的定性改造和利用. 关键词:植物;功能基因组学;研究技术 中图分类号:Q3-3 文献标识码:A 文章编号:1673-2065(2007)01-0023-04 基因是细胞的遗传物质,决定细胞的生物学形状,细胞的生物学功能最终是由大量的基因表达完成的.随着人类基因组“工作框架图”的完成,生命科学研究的重点已经从结构基因组学转移到了功能基因组学的研究,特别是模式植物拟南芥(A r a b i d o p-s i s t h a l i a n a)和水稻(O r y z a s a t i v a)基因组测序的完成,公共数据库中已经积累了大量基因序列信息,获得了许多与植物发育相关的功能基因,在此基础上应用实验分析方法并结合统计和计算机分析来研究基因的表达、调控与功能,并相应诞生和发展了一批新的研究技术,为功能基因组学的研究提供了必要而有效的技术支撑.功能基因组学研究的最终目标是解析所有基因的功能,即从基因水平上大规模批量鉴定基因的功能,进而全面研究控制植物生长发育及响应环境变化的遗传机制,在基因组序列与细胞学行为之间起到桥梁作用,共同承担起从整体水平上解析生命现象的重任. 1 植物功能基因组学研究 植物的生长和发育是一个有机体或有机体的一部分形态建成和功能按一定次序而进行的一系列生化代谢反应的总合,反应在分子水平上,它要求相应的遗传代谢途径必须按照特定的时空次序严格进行以保证正常发育.植物功能基因组研究就是要利用植物全基因组序列的信息,通过发展和应用系统基因组水平的实验方法来研究和鉴别基因组序列的作用;研究基因组的结构、组织与植物功能在细胞、有机体和进化上的关系以及基因与基因间的调控关系;从表达时间、表达部位和表达水平3个方面对目的基因在植物中的精细调控进行系统研究.当前植物功能基因组学研究主要集中于一年生的拟南芥与水稻两个物种上,这主要是由于它们的遗传背景清楚,基因组较小,基因结构简单而且易于进行分子生物学操作.拟南芥研究组“2010计划”的宏伟目标是充分利用拟南芥基因组计划获得的序列信息并结合功能基因组研究技术来获知其25000个基因的全部功能,例如开花的诱导过程是植物生活周期中最奇妙的过程,目前从拟南芥中鉴定了提早开花和延迟开花的多种突变体,显示植物开花受多个遗传基因的控制,如延迟开花的两个突变体是由等位基因 C O(C O N S T A N S)和L D(C O L D L U M I N I D E P E N- D E N S)突变引起,这两个基因均已被克隆,并使其在转基因植物的叶片中进行表达,将C O基因转移到拟南芥中,高效表达C O蛋白的转基因植株即使处于短日照条件下也会开花,这说明C O基因具有激活开花基因的作用.对模式植物功能基因组的研究将有助于整个植物基因组学的研究. 目前的功能基因组研究主要包括以下几个方面:(1)c D N A全长克隆与测序;(2)获得D N A芯片 ①收稿日期:2006-10-12 作者简介:崔兴国(1963-),女,河北冀州市人,衡水学院生命科学系副教授.
已完成基因组测序的生物(植物部分)分析解析
水稻、玉米、大豆、甘蓝、白菜、高粱、黄瓜、西瓜、马铃薯、番茄、拟南芥、杨树、麻风树、苹果、桃、葡萄、花生 拟南芥籼稻粳稻葡萄番木瓜高粱黄瓜玉米栽培大豆苹果蓖麻野草莓马铃薯白菜野生番茄番茄梨甜瓜香蕉亚麻大麦普通小麦西瓜甜橙陆地棉梅毛竹桃芝麻杨树麻风树卷柏狗尾草属花生甘蓝 物种基因组大小和开放阅读框文献 Sesamum indicum L. Sesame 芝麻(2n = 26)293.7 Mb, 10,656 orfs 1 Oryza brachyantha短药野生稻261 Mb, 32,038 orfs 2 Chondrus crispus Red seaweed爱尔兰海藻105 Mb, 9,606 orfs 3 Pyropia yezoensis susabi-nori海苔43 Mb, 10,327 orfs 4 Prunus persica Peach 桃226.6 of 265 Mb 27,852 orfs 5 Aegilops tauschii 山羊草(DD)4.23 Gb (97% of the 4.36), 43,150 orfs 6 Triticum urartu 乌拉尔图小麦(AA)4.66 Gb (94.3 % of 4.94 Gb, 34,879 orfs 7 moso bamboo (Phyllostachys heterocycla) 毛竹2.05 Gb (95%) 31,987 orfs 8 Cicer arietinum Chickpea鹰嘴豆~738-Mb,28,269 orfs 9 520 Mb (70% of 740 Mb), 27,571 orfs 10 Prunus mume 梅280 Mb, 31,390 orfs 11 Gossypium hirsutum L.陆地棉2.425 Gb 12 Gossypium hirsutum L. 雷蒙德氏棉761.8?Mb 13 Citrus sinensis甜橙87.3% of ~367 Mb, 29,445 orfs 14 甜橙367 Mb 15 Citrullus lanatus watermelon 西瓜353.5 of ~425 Mb (83.2%) 23,440 orfs 16 Betula nana dwarf birch,矮桦450 Mb 17
全基因组重测序数据分析
全基因组重测序数据分析 1. 简介(Introduction) 通过高通量测序识别发现de novo的somatic和germ line 突变,结构变异-SNV,包括重排 突变(deletioin, duplication 以及copy number variation)以及SNP的座位;针对重排突变和SNP的功能性进行综合分析;我们将分析基因功能(包括miRNA),重组率(Recombination)情况,杂合性缺失(LOH)以及进化选择与mutation之间的关系;以及这些关系将怎样使 得在disease(cancer)genome中的mutation产生对应的易感机制和功能。我们将在基因组 学以及比较基因组学,群体遗传学综合层面上深入探索疾病基因组和癌症基因组。 实验设计与样本 (1)Case-Control 对照组设计; (2)家庭成员组设计:父母-子女组(4人、3人组或多人); 初级数据分析 1.数据量产出:总碱基数量、Total Mapping Reads、Uniquely Mapping Reads统计,测序深度分析。 2.一致性序列组装:与参考基因组序列(Reference genome sequence)的比对分析,利用贝叶斯统计模型检测出每个碱基位点的最大可能性基因型,并组装出该个体基因组的一致序列。3.SNP检测及在基因组中的分布:提取全基因组中所有多态性位点,结合质量值、测序深度、重复性等因素作进一步的过滤筛选,最终得到可信度高的SNP数据集。并根据参考基 因组信息对检测到的变异进行注释。 4.InDel检测及在基因组的分布: 在进行mapping的过程中,进行容gap的比对并检测可信的short InDel。在检测过程中,gap的长度为1~5个碱基。对于每个InDel的检测,至少需 要3个Paired-End序列的支持。 5.Structure Variation检测及在基因组中的分布: 能够检测到的结构变异类型主要有:插入、缺失、复制、倒位、易位等。根据测序个体序列与参考基因组序列比对分析结果,检测全基因组水平的结构变异并对检测到的变异进行注释。
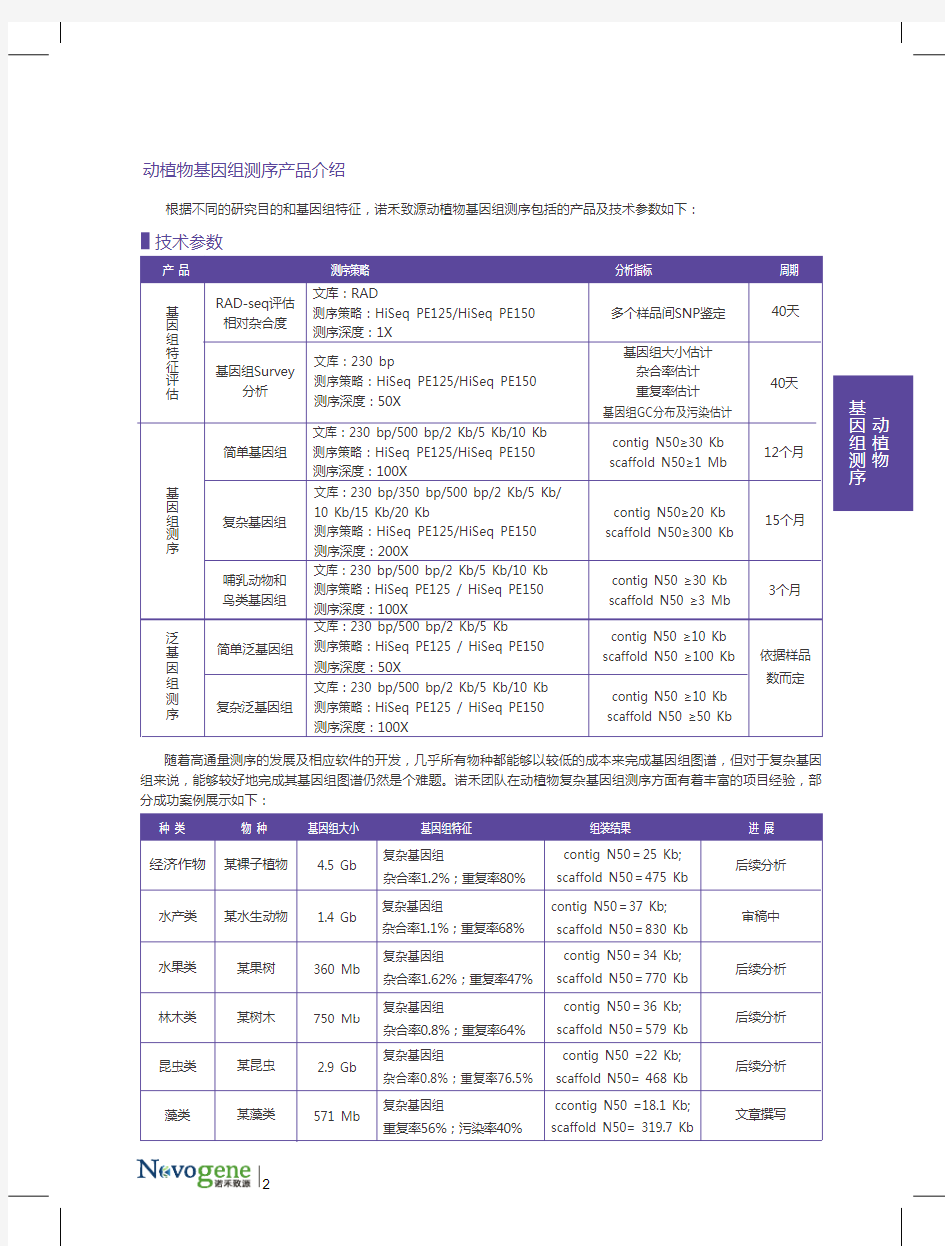
植物基因组测序
千年基因将应邀参加第十六届全国植物基因组学大会 第十六届全国植物基因组学大会将于2015年8月19日-22日在陕西杨凌召开,千年基因应邀参加此次会议,并将在会场学术交流区设立展台。届时千年基因的技术团队会向大家展示我们最全面的测序平台、一站式的基因组学解决方案以及近年来在植物基因组学领域取得的科研成果,欢迎广大科研人员莅临指导交流! 在测序平台方面,千年基因目前拥有国内最全面的测序平台,能够为科研人员提供一站式解决方案。以PacBio RS II三代平台为例,千年基因自去年提供PacBio RS II测序以来,通过项目经验的积累及严格的质量控制,目前各项数据指标已达国内最高水平。数据产出已稳步升级至1.4Gb/ SMRT cell,读长最长可达42 Kb,reads N50高达18Kb,远超PacBio官方提供的数据标准!在植物基因组de novo测序的研究中,千年基因提供的超长读长测序可更好地跨越基因组高重复序列、转座子区域以及大的拷贝数变异区域和结构变异区,从而实现对高杂合及高重复基因组的完美组装。在植物转录组测序的研究中,千年基因提供的超长读长测序无需拼接即可获得全长转录组序列信息,同时可获得全面的可变剪切、融合基因以及Isoform信息。另外,千年基因提供的HiSeq 4000及HiSeq 2000/2500测序可解决研究人员在植物基因组重测序、转录组测序、小RNA测序等方面的科研需求。 在项目经验方面,千年基因与来自全球的科研人员合作开展了大量植物基因组项目,相关成果已发表于Nature、Nature Genetics、Science等杂志。例如,油棕榈基因组项目在Nature 杂志同时发表两篇文章,辣椒基因组项目的成果发表于Nature Genetics,玉米基因组项目的成果发表于Science。在国外合作方面,千年基因与美国爱荷华州立大学Patrick Schnable教授领导的国际玉米基因组团队合作开展的上万份玉米样本重测序项目也正在进行中;千年基因与国际半干旱热带作物研究所建立长期战略合作关系,正在开展上千份木豆、鹰嘴豆及高粱样本的群体遗传学研究;同时千年基因与华盛顿大学的Evan Eugene Eichler院士及佐治亚大学的Jeffrey Lynn Bennetzen院士也有大量基因组项目合作。在国内合作方面,千年基因与广东省农科院、山东省农科院共同启动的花生基因组项目已全部完成de novo测序及数据挖掘,同时与中国科学院、北京大学、中国农业大学、中国科学技术大学、上海交通大学、
全基因组从头测序(de novo测序)
全基因组从头测序(de novo测序) https://www.360docs.net/doc/aa3488370.html,/view/351686f19e3143323968936a.html 从头测序即de novo 测序,不需要任何参考序列资料即可对某个物种进行测序,用生物信息学分析方法进行拼接、组装,从而获得该物种的基因组序列图谱。利用全基因组从头测序技术,可以获得动物、植物、细菌、真菌的全基因组序列,从而推进该物种的研究。一个物种基因组序列图谱的完成,意味着这个物种学科和产业的新开端!这也将带动这个物种下游一系列研究的开展。全基因组序列图谱完成后,可以构建该物种的基因组数据库,为该物种的后基因组学研究搭建一个高效的平台;为后续的基因挖掘、功能验证提供DNA序列信息。华大科技利用新一代高通量测序技术,可以高效、低成本地完成所有物种的基因组序列图谱。包括研究内容、案例、技术流程、技术参数等,摘自深圳华大科技网站 https://www.360docs.net/doc/aa3488370.html,/service-solutions/ngs/genomics/de-novo-sequencing/ 技术优势: 高通量测序:效率高,成本低;高深度测序:准确率高;全球领先的基因组组装软件:采用华大基因研究院自主研发的SOAPdenovo软件;经验丰富:华大科技已经成功完成上百个物种的全基因组从头测序。 研究内容: 基因组组装■K-mer分析以及基因组大小估计;■基因组杂合模拟(出现杂合时使用); ■初步组装;■GC-Depth分布分析;■测序深 度分析。基因组注释■Repeat注释; ■基因预测;■基因功能注释;■ ncRNA 注释。动植物进化分析■基因家族鉴定(动物TreeFam;植物OrthoMCL);■物种系统发育树构建; ■物种分歧时间估算(需要标定时间信息);■基因组共线性分析; ■全基因组复制分析(动物WGAC;植物WGD)。微生物高级分析 ■基因组圈图;■共线性分析;■基因家族分析; ■CRISPR预测;■基因岛预测(毒力岛); ■前噬菌体预测;■分泌蛋白预测。 熊猫基因组图谱Nature. 2010.463:311-317. 案例描述 大熊猫有21对染色体,基因组大小2.4 Gb,重复序列含量36%,基因2万多个。熊猫基因组图谱是世界上第一个完全采用新一代测序技术完成的基因组图谱,样品取自北京奥运会吉祥物大熊猫“晶晶”。部分研究成果测序分析结果表明,大熊猫不喜欢吃肉主要是因为T1R1基因失活,无法感觉到肉的鲜味。大熊猫基因组仍然具备很高的杂合率,从而推断具有较高的遗传多态性,不会濒于灭绝。研究人员全面掌握了大熊猫的基因资源,对其在分子水平上的保护具有重要意义。 黄瓜基因组图谱黄三文, 李瑞强, 王俊等. Nature Genetics. 2009. 案例描述国际黄瓜基因组计划是由中国农业科学院蔬菜花卉研究所于2007年初发起并组织,并由深圳华大基因研究院承担基因组测序和组装等技术工作。部分研究成果黄瓜基因组是世界上第一个蔬菜作物的基因组图谱。该项目首次将传
DNA测序标准实验流程(V1.3版)
DNA测序标准实验流程(V1.2版)1.对DNA的要求 纯度:OD 260 / OD 280 = 1.6 ~ 2.0, PCR产物用量:每反应15 -20ng(片段大于3KB可加两倍DNA)。 质粒DNA用量:每反应20 -25ng(插入片段大于3KB质粒要加两倍DNA)。 1300载体本身序列就比较长,我们建议每反应加50-80ng。 每个小组一次配100份BD MIX(BD 0.4ul,5*buffer 1.8ul,water 2.8ul)长期保存,每个反应体系加5ul 2.P CR产物的测序PCR反应(测序PCR反应中只要加一个引物就可以,需要加热盖) 标准反应体系: 10ul体系 试剂用量 纯化的P CR产物(15-20 ng / μL) 1 μL (片段大于3KB可加两倍DNA) 引物(2 pmol / μL) 1 μL BigDye (2.5 x) 0.4 μL BigDye Seq Buffer (5 x) 1.8μL 灭菌去离子水 5.8μL 96 °C 1 min → (96 °C 10 sec → 50 °C 5 sec → 60 °C 2 min) x 25个循环→ 4 °C保温 质粒DNA的测序PCR反应 标准反应体系: 10ul体系 试剂用量 质粒DNA (20-25 ng / μL) 1 μL (插入片段大于3KB质粒要加两倍DNA) 引物(2 pmol / μL) 1 μL BigDye (2.5 x) 0.4 μL BigDye Seq Buffer (5 x) 1.8 μL 灭菌去离子水 5.8 μL 96 °C 1 min → (96 °C 10 sec → 50 °C 5 sec → 60 °C 2 min) x 25个循环→ 4 °C保温 注意:BigDye (2.5 x)是一种含有DNA聚合酶和荧光物质的混合物,非常昂贵,平时都放在-20度保存。加之前拿出来放在冰上融化,用完马上放回-20冰箱。BigDye (2.5 x)和BigDye Seq Buffer (5 x)可以混合后一起加到反应体系,有多的话可以放在-20冰箱,下次还能使用。 BIGDYE尽量避光,一般用铝珀纸遮盖。P CR样品处理过程中如在室温放置和酒精挥发阶段都尽量用铝珀纸遮盖或者放入抽屉,有利于样品的稳定性。 3.测序产物纯化 单个0.2 mL离心管离心方法: 1. 每孔加入1μL 7.5M NH3Ac,26μL 100%酒精,盖好,震荡4次。(酒精和NH3Ac先混合好,而且要比样品数多预算几个) 2. 台式离心机12000 x g 4°C离心20 min,马上用枪吸尽上清液。(DNA很微量,基本看不到,所以枪头不要碰到DNA沉积处) 3. 每孔加入100μL 75% 酒精,12000 x g 4°C离心10 min,马上用枪吸尽上清液。(如果不是马上操作,DNA沉淀很可能 浮起,被吸走,所以如果没有及时吸去上清的话,要重新离心5MINS。) 4. 让酒精在室温避光(抽屉)挥发干净(至少20mins),加入10 μL Hi-Di Formamide溶解DNA。 5. 在PCR仪上变性:95 °C 4 min,4 °C 4 min。上机测序。 96孔板整板离心方法: 1. 每孔加入1μL 7.5M NH3Ac,26μL 100%酒精,盖好,震荡4次。(酒精和NH3Ac先混合好,而且要比样品数多预算几个) 2. 板式离心机4000 x rpm 4°C离心30min;马上倒置96孔板,弃上清,倒置在洗水纸上,离心500rpm,1mins。 3. 加100μL 75% 酒精,4000 rpm 4°C离心20 min;马上倒置96孔板,弃上清,离心500rpm,1mins。 4.让酒精在室温避光(抽屉)挥发干净(至少15mins),加入10 μL Hi-Di For mamide溶解DNA。 5. 在PCR仪上变性:95 °C 4 min,4 °C 4 min。上机测序。 4. 部分相关试剂 酒精:100%酒精使用国产分析纯;75%酒精用去离子水配制。 BigDye (2.5 x) -20度保存 BigDye Seq Buffer (5 x) 4度保存 7.5M NH3Ac 4度保存 Hi-Di For mamide -20度保存 黄方亮 2009.10.27日整理
植物功能基因组学概述
植物功能基因组学概述 XXX* (XXXXX) 摘要:植物功能基因组学是从整体水平研究基因的功能及表达规律的科学。对植物功能基因组学的研究将助于我们对基因功能的理解和对植物性状的定性改造和利用。本文简要介绍了植物功能基因组学的概念、研究内容和研究方法。 关键词:植物;功能基因组学;ESTs;SAGE Summarize of Plant Functional Genomics XXX (XXXXX) Abstract:Plant functional genomics studies provide a novel approach to the identification of genome-wide gene expression. It is currently being widely focused on the gene expression by transcript profiling and takes us rapidly forward in our understanding of plant biological traits. In this review, comprehensive of concepts, research contents and methodologies regarding plant functional genomics and transcript profiling are described. Key words: Plant; functional genomics; ESTs; SAGE 1 植物功能基因组学 基因组学(Genomics)是20世纪最后10年研究最活跃的领域之一。基因组学是指对所有基因的结构和功能进行分析的一门学科, 1986年由美国科学家Thomas Roderick提出, 兴起于20世纪90年代[1]。基因组学研究分为结构基因组学( structural genomics) 和功能基因组学( functional genomics)。结构基因组学代表基因组分析的早期阶段, 以建立生物体高分辨率遗传、物理和转录图谱为主, 以研究基因序列为目标。功能基因组学(Functional genomics)的研究又被称为后基因组学(Post genomics)研究,它是利用结构基因组学提供的信息和产物,通过在基因组或系统水平上全面分析基因的功能,使得生物学研究从对单一基因或蛋白质的研究转向对多个基因或蛋白质同时进行系统研究。 植物功能基因组学是植物后基因时代研究的核心内容,它强调发展和应用整体的(基因 组水平或系统水平)实验方法分析基因组序列信息、阐明基因功能,其特点是采用高通量的实验方法结合大规模的数据统计计算方法进行研究。基本策略是从研究单一基因或蛋白质上升到从系统角度研究所有基因或蛋白质。在植物功能基因组学的研究中,拟南芥和水稻是两种最常用的模式植物。目前, 功能基因组学在水稻、拟南芥等模式植物中取得了较快进展, 主要原因在于这两种植物已完成全基因组测序工作[2], 获得了结构基因组数据, 且遗传背景清楚, 易于开展分子生物学研究, 已率先步入后基因组时代。 2 植物功能基因组学研究内容 2、1基因组多样性研究[1] *联系人Tel:XXXXX;E-mail:XXXXX
已基因组测序物种
已完成植物基因组测序情况(更新至2014年11月) 中文名拉丁名发表时间刊物科、属基因组大小拟南芥Arabidopsis thaliana 2000.12 Nature 十字花科、鼠耳芥属125M 水稻Oryza sativa. ssp. indica 2002.04 Science 禾本科、稻属466M 水稻Oryza sativa. ssp. japonica 2002.04 Science 禾本科、稻属466M 杨树Populus trichocarpa 2006.09 Science 杨柳科、杨属480M 葡萄Vitis vinifera 2007.09 Nature 葡萄科、葡萄属490M 衣藻Chlamydomonas reinhardtii 2007.01 Science 衣藻科、衣藻属130 M 小立碗藓Physcomitrella pattens 2008.01 Science 葫芦藓科、小立碗藓属480M 番木瓜Carica papaya 2008.04 Nature 番木瓜科、番木瓜属370M 百脉根Lotus japonicus 2008.05 DNA Res. 豆科472 Mb 三角褐指藻Phaeodactylum tricornutum 2008.11 Nature 褐指藻属27.4M 高粱Sorghum bicolor 2009.01 Nature 禾本科、高粱属730M 玉米Zea mays ssp. mays 2009.11 Science 禾本科、玉米属2300M 黄瓜Cucumis sativus 2009.11 Nature Genetics 葫芦科、黄瓜属350M 大豆Glycine max 2010.01 Nature 豆科、大豆属1100M 二穗短柄草Brachypodium distachyon 2010.02 Nature 禾本科、短柄草属260M 褐藻Ectocarpus 2010.06 Nature 水云属196M 团藻Volvox carteri 2010.07 Science 团藻属138M 蓖麻Ricinus communis 2010.08 Nature Biotechnology 大戟科、蓖麻属350M 小球藻Chlorella variabilis 2010.09 Plant Cell 小球藻科46M 苹果Malus × domestica 2010.09 Nature Genetics 蔷薇科、苹果属742M 森林草莓Fragaria vesca 2010.12 Nature Genetics 蔷薇科、草莓属240M 可可树Theobroma cacao 2010.12 Nature Genetics 梧桐科、可可属430-Mb 野生大豆Glycine soja 2010.12 PNAS 豆科、大豆属915.4 Mb 褐潮藻类Aureococcus anophagefferens 2011.02 PNAS 57M 麻风树Jatropha curcas 2010.12 DNA Res. 大戟科、麻风树属410M 卷柏Selaginella moellendorffii 2011.05 Science 卷柏属212M 枣椰树Phoenix dactylifera 2011.05 Nature biotechnology 棕榈科685M 琴叶拟南 芥 Arabidopsis lyrata 2011.05 Nature Genetics 十字花科、鼠耳芥属206.7 Mb 马铃薯Solanum tuberosum 2011.07 Nature 茄目、茄科、茄属844M 条叶蓝芥Thellugiella parvula 2011.08 Nature Genetics 盐芥属140M
紫花苜蓿基因组测序及分析
紫花苜蓿基因组测序及分析 近年来,随着基因组测序技术的不断发展越来越多的植物基因组序列得到完善。通过基因组测序,可以系统地解析植物基因功能。 紫花苜蓿具有优良的农艺性状,是全世界种植范围最广的牧草作物,具有“牧草之王”的美称。在我国,紫花苜蓿生长主要集中在北方地区,这些地区气候寒冷,容易发生低温冻害,造成苜蓿减产,大大降低苜蓿的生产效益。 肇东苜蓿作为紫花苜蓿的地方品种之一,具有良好的抗寒特性。因此,本研究以肇东苜蓿为主要研究材料,进行基因组测序、组装及注释,获得基因组草图;对其它四种紫花苜蓿品种:阿尔冈金、WL168、WL525和WL440进行基因组重测序, 挖掘抗寒相关的SNP。 同时,对紫花苜蓿AP2/ERF转录因子进行挖掘分析,构建紫花苜蓿AP2/ERF 基因调控网络,系统的解析紫花苜蓿AP2/ERF转录因子在抗寒胁迫中的调控机制。主要研究成果如下:1.紫花苜蓿全基因组组装及注释分析通过高通量测序,评估 紫花苜蓿基因组大小为1107M左右,杂合率为1.77%,通过组装获得紫花苜蓿基因组草图。 进一步注释分析,挖掘119194个蛋白质编码基因,它们主要的功能集中在信号转导机制、蛋白质翻译后修饰、防御机制以及转录调控等生物过程。2.紫花苜蓿抗寒性状相关遗传变异的挖掘通过四个紫花苜蓿品种进行基因组重测序,发掘紫花苜蓿品种间的遗传变异(SNP),获得8909604个遗传变异;结合5个紫花苜蓿品种的抗寒性数据,挖掘82838个SNP与紫花苜蓿抗寒性状形成相关。 这些SNP分布在14372个基因上,它们主要涉及转录调控、还原代谢和信号转导等过程。进一步富集分析发现,AP2/ERF、bHLH、MADS、NAC、MYB以及WRKY
农作物重要品种全基因组de novo测序
首页 科技服务 医学检测 科学与技术 市场与支持 加入我们 关于我们 提供领先的基因组学解决方案Providing Advanced Genomic Solutions 参考文献 [ 1 ] Li Y, Zhou G, Jiang W, et al. De novo assembly of soybean wild relatives for pan-genome analysis of diversity and agronomic traits[J]. Nature biotechnology, 2014, 32(10): 1045-1052. [ 2 ] Da Silva C, Zamperin G, et al. The high polyphenol content of grapevine cultivar tannat berries is conferred primarily by genes that are not shared with the reference genome. Plant Cell, 2013, 25(12):4777-88. [ 3 ] Qi XP, Li M, et al. Identification of a novel salt tolerance gene in wild soybean by whole-genome sequencing. Nature Communica- tions, 2014(5). 挖掘特异基因 解析特有性状 重要品种 全基因组 de novo 所研究品种 小片段文库大片段文库 HiSeq测序(>100X) 基因组组装 注释 全基因组序列比对 转录组遗传图谱等辅助验证 重要农艺性状解析 基因家族聚类分析 所研究品种基因组序列 已发表品种 基因组序列 已发表品种基因集合 所研究品种基因集合 变异检测 小的插入 缺失 SNP 倒位 易位大的插入 缺失 基因挖掘 新基因鉴定拷贝数扩增基因基因丢失正选择基因鉴定 物种 品种 发表杂志(年份) 物种 品种发表杂志(年份) 大豆 棉花 番茄 土豆 水稻 葡萄猪栽培大豆7种野生大豆 野生耐盐大豆雷德氏棉 亚洲棉 陆地棉栽培番茄 抗病番茄栽培土豆 耐寒土豆 Nature (2010) Nature Biotechnology (2014) Nature Communications (2014) Nature (2012)Nature Genetics (2014)Nature Biotechnology (2015)Nature (2012) Nature Genetics (2014) Nature (2011) Plant Cell (2015) 栽培水稻(粳稻)栽培水稻(籼稻) 短花药野生稻 非洲栽培稻五种野生稻三种栽培稻葡萄 丹娜葡萄杜洛克猪藏猪 Science (2002)Science (2002) 野生大豆泛基因组 阅读原文>> 诺禾致源的项目经验 诺禾致源在动植物全基因组测序领域一直处于领先地位, 以第一通讯作者发表基因组文章5篇(影响因子累计152.474),其中2篇为杂志封面文章。 近年来,诺禾通过自主研发软件与技术革新,成功地将项目周期压缩至14个月内,费用降低一半以上。 特有基因检测 对7株代表性野生大豆品种进行全基因组de novo 测序及比较基因组分析,发现每个大豆品种中有1,000~3,000个品种特有的基因。 高变区变异检测 在传统测序方法中,将研究物种短reads 比对参考基因组无法检测到变异位点;在全基因组de novo 方法中,将组装后的超长序列比对参考基因组可准确识别高变区域内的所有变异位点。性状解析方案设计 通过对重要品种高深度(>100X)测序,并进行基因组组装注释: 找到传统测序无法鉴定的高度变异位点,找到更多更准确的SNP位点;找到参考基因组中 所不存在的基因——品种特有基因。 2. 耐盐大豆耐盐基因的发现 2014年,研究人员对一株耐盐大豆开展了全基因组de novo 测序,并与栽培大豆基因组进行全基因组比对,通过一条跨过长达388 Kb 的重要功能区的scaffold,发现了巨大的结构变异,从而成功鉴定出耐盐基因。该基因在栽培大豆中被插入了一个长达3.4 Kb 的反转座子,影响了阳离子转运体功能,从而使栽培大豆失去了耐盐能力。 传统的测序手段,采用的是短reads 比对,因而对这类大的结构变异检测精度差、灵敏度低、甚至难以实现检测,而全基因组de novo 测序则能很好的克服该问题。阅读原文>> 案例分享 1. 丹娜葡萄全基因组测序揭示高丹宁含量性状的分子机制 丹娜葡萄被认为是丹宁含量最高的葡萄品种之一,由于富含丹宁等抗氧化分子,被认为有延缓衰老的作用。通过对其基因组测序,研究人员发现与丹宁合成有关的关键酶,几乎都能找到新的基因。很显然,只依赖已有的参考基因组,完全无法了解丹娜葡萄高丹宁含量这一性状的遗传基础,而全基因组de novo 测序则完美回答了该问题。阅读原文>> 重要品种基因组的价值 一些物种,虽然已有参考基因组,但仍然无法找到性状关联基因。 一方面,参考基因组与研究物种差异太大; 另一方面,性状相关基因处于基因组快速进化区域,变异极大,传统测序手段难以鉴定。 目前,de novo测序在有参品种重要性状探究方面的应用愈发广泛, 相关研究结果常见于国际顶级杂志上。
人类全基因组测序
1 技术优势 全基因组测序(Whole Genome Sequencing,WGS)是利用高通量测序平台对人类不同个体或群体进行全基因组测序,并在个体或群体水平上进行生物信息分析。可全面挖掘DNA 水平的遗传变异,为筛选疾病的致病及易感基因,研究发病及遗传机制提供重要信息。 全基因组测序 平台优势 HiSeq X 测序平台 读长:PE150 通量:1.8T/run 测序周期:3 天 专为人全基因组测序准备、测序周期短、通量高
生物信息分析 技术路线 技术参数 样品要求 样本类型:DNA 样品 样本总量:≥1.0 μg DNA (提取自新鲜及冻存样本) ≥1.5 μg DNA (提取自FFPE 样本)样品浓度:≥ 20 ng/μl 测序平台及策略HiSeq X PE150 测序深度 肿瘤:癌组织(50X),癌旁组织/血液样本(30X)遗传病:30~50 X 项目周期37天
3 案例解析 该研究选取3个家系中6个患者和1个正常个体,首先使用基因芯片寻找纯合突变位点,然后对其中无亲缘关系的2例患者采用全基因组测序研究,在2例患者非编码区域均发现相同的变异,10号染色体PTF1A 末端发生一个点突变(chr10:23508437 A>G),且变异在患病人群和细胞试验中均得到了验证。研究解释了生长发育启动子隐性变异是罕见孟德尔遗传病的常见致病原因,同时说明许多疾病的致病突变也可能位于非编码区。 图1 检出的变异信息 智力障碍是影响新生儿心智发育的一类疾病。这项研究选取50个经过基因芯片和全外显子测序未确诊致病因子的trio 家系,全基因组测序检出84个de novo SNVs 和8个de novo CNVs,及一些结构变异(如VPS13B、STAG1、IQSEC2-TENM3),检出率为42%。揭示编码区的de novo SNVs 和de novo CNVs 是导致智力障碍的主要因素,全基因组测序可以作为可靠的遗传性检测应用工具。 案例一 单基因病研究——全基因组测序鉴定PTF1A末端增强子常染色体隐性突变导致胰腺 发育不全[1] 案例二 复杂疾病研究——全基因组测序解析智力障碍的主要致病因素[2] 图2 PTF1A 的家系图谱
基因组重测序分析流程-代码文件
差异位点分析流程步骤分解 数据准备: mkdir 1.QC cd 1.QC ln -s /root/mdna-data/reseq/1.QC/*.fastq . Ls cd .. mkdir 2.mapping cd 2.mapping ln -s /root/mdna-data/reseq/2.mapping/ref.fasta . 步骤1:参考基因建索引 cd 2.mapping ##bwa建索引: bwa index ref.fasta Expected Result:得到一系列BWA 进行alignment 需要的文件。 ##samtools建索引: samtools faidx ref.fasta Expected Result:生成refgene.fasta.fai。每行都是fasta 文件中每条contig 的record,每条record 由contig name, size, location, basesPerLine 和bytesPerLine 组成。 ##生成字典: java -jar /root/mdna_software/picard-tools-1.102/CreateSequenceDictionary.jar R=ref.fasta O=ref.dict Expected Result:生成refgene.dict。描述fasta 文件内容,类似SAM header 格式。 步骤2:bwa比对 ##用bwa作比对: nohup bwa aln -e 3 -i 10 -t 1 -R 100 -q 20 ref.fasta ../1.QC/test_trim1.fastq -f 1.sai & nohup bwa aln -e 3 -i 10 -t 1 -R 100 -q 20 ref.fasta ../1.QC/test_trim2.fastq -f 2.sai & nohup bwa aln -e 3 -i 10 -t 1 -R 100 -q 20 ref.fasta ../1.QC/test_trim_unpaired.fastq -f s.sai & jobs
