测序常见问题分析实例

测序常见问题分析实例
峰型整齐,在某一点前后突然变乱
信号迅速衰减
信号极弱或无信号
整条序列信号杂乱
峰型整齐,在某一点前后突然变乱:
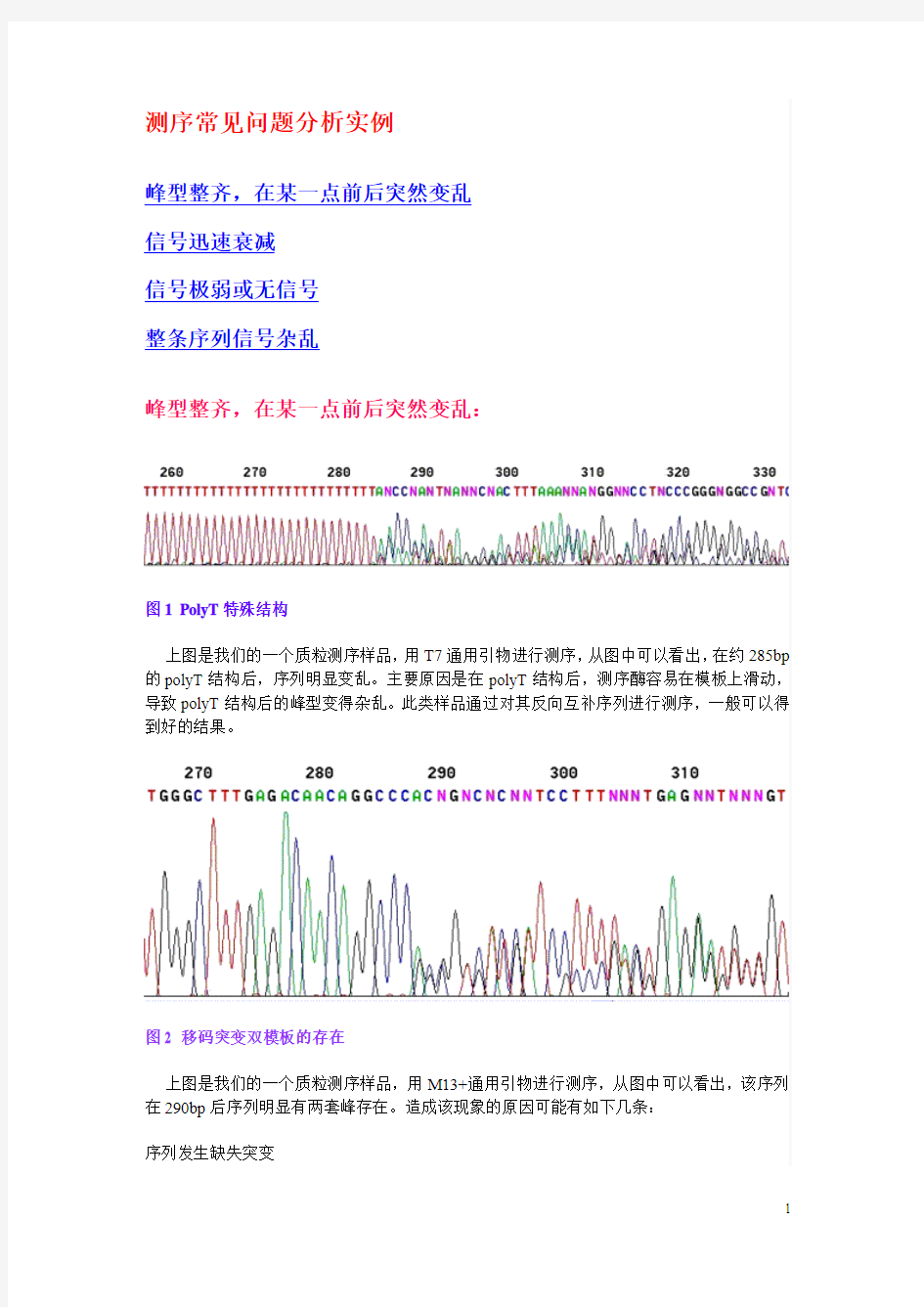
图1 PolyT特殊结构
上图是我们的一个质粒测序样品,用T7通用引物进行测序,从图中可以看出,在约285bp 的polyT结构后,序列明显变乱。主要原因是在polyT结构后,测序酶容易在模板上滑动,导致polyT结构后的峰型变得杂乱。此类样品通过对其反向互补序列进行测序,一般可以得到好的结果。
图2 移码突变双模板的存在
上图是我们的一个质粒测序样品,用M13+通用引物进行测序,从图中可以看出,该序列在290bp后序列明显有两套峰存在。造成该现象的原因可能有如下几条:
序列发生缺失突变
插入外援片段的载体和未插入外援片段的载体同时存在
PCR产物用T载体进行克隆时,PCR片段可以以两个方向克隆进T载体
所挑克隆不纯
两个大小相近的PCR产物同时存在,无法纯化分开
解决的办法:
对于质粒模板,重新挑选克隆,或从另一段进行测序
对于PCR模板,用另一端引物进行测序,或克隆后进行测序
图3 等位基因双模板的存在
上图是针对一个质粒进行的测序结果,从图中可以明显看出,在序列的80bp到120bp之间有两套峰存在,但是没有发生移码突变。该情况与图2所举的例子有所不同,该情况下从反向进行测序仍然不可能得到好的测序结果。该种情况下只能采取克隆的方法将两套模板分开,分别进行测序。
信号迅速衰减返回
图4 CTT重复结构
如上图,在大约260碱基后出现了一个严重的CTT重复结构,导致信号迅速衰减,很难得到跨过该区后的信息。该种情况下,只能从另一端进行测序,一般来说,AAG重复结构不会太影响测序。也可以对片段进行亚克隆,使每个片段大小不大于200bp,然后再进行测序。不过,该方法要麻烦很多。
图5 GGT重复结构严重影响测序
上图下半部分是一个质粒模板用M13+通用引物进行测序,大约在130bp开始,出现GGT 重复结构,测序信号迅速减弱至消失。上图的上半部分是用M13-通用引物从反向进行测序,在反向序列上该重复是GGT的反向互补序列,即ACC,大约在500bp处,测序很轻松地就读过了ACC重复序列区。GGT重复结构严重影响测序的原因是在测序试剂盒中,为了得到均匀的测序峰型图,G和T碱基均进行了替代,分别替代成I和U,因此与模板的结合能力减弱,测序酶反应到此后就比较难延伸下去,导致该区域后信号迅速减弱。通过优化多种条件,可以对结果有一点改善,但仍然不能得到可用的结果。对该类型的模板,对反向互补链进行测序,可以很轻松地跨过该区域。
图6 GC特殊结构区
上图是一个质粒模板用M13+通用引物进行测序的结果,序列中存在一个GC特殊结构区,在该区域后,信号迅速减弱。上图的下半部分是对测序反应进行优化后的测序结果,在GC 特殊结构后,测序信号得到一定程度的改善,但是离一般的测序结果还是相差甚远。针对该类型的模板,一般应从反向进行测序,然后在该特殊结构区附近将两个方向的测序结果拼接起来,得到完整的序列。
图7 模板特殊结构
上图是一个pGEM-T载体用M13+引物进行测序,可以看出,序列在载体后迅速衰减,造成此现象的原因一直不明,我们试用了多种方法试图解决该测序问题,但几乎毫无效果。该种情况很有可能是在全序列中有大的特殊结构,造成严重的二级结构结构,使测序酶无法
读过此区域。正常情况下pGEM-T载体测序结果非常理想。返回
信号极弱或无信号
造成该现象主要有两个原因:
模板质量极差;
引物与模板序列不匹配。
图8 pGEM质粒用T3通用引物进行测序,测序结果无可用信息,因为pGEM载体上无T3通用引物结合位点。上图中两处峰前面的一个主要是未去除的测序反应单体造成的,靠右边的一处峰主要是引物的非特异性反应造成的。
在我们每天的测序样品中,有相当一部分测序失败的样品是由于缺少引物结合序列造成的,主要有下面几种原因可能造成该种结果:
客户提供了错误的载体信息或引物信息,
用客户自己在克隆片段上的引物对质粒进行测序,但是该质粒为空载体,不包含插入片段。载体由于突变,丢失了原来的引物结合位点。
图9 一个PCR产物的测序结果,整个序列信号极低,几乎无可用信息。造成该现象主要是在测序反应中,模板的量太低,所得信号太弱。
重新提供足够量的模板一般可以得到较好的测序结果。返回
整条序列信号杂乱
模板本身的问题:
图10 一个污染的PCR产物测序结果,可以看出,整条序列都有极高的背景。
有以下几个原因可能造成测序模板污染:
PCR产物有杂带
质粒类型的模板克隆不纯
对于PCR类型的模板,我们现在都采取切胶的方法进行纯化。该纯化方法一般情况下可以将PCR产物的杂带及多余的引物去掉。但是,当扩增的片段中包含有大小非常近似的片段时,切胶纯化的方法也无济于事了。该情况下只能采取将待测的PCR产物进行克隆测序了。
质粒类型的测序模板一般有两种原因可以造成模板污染。一为所挑选的克隆不纯,包含两种或两种以上的克隆,如不包含插入片段的克隆和包含插入片段的克隆的混合体,用T载体进行PCR产物克隆时,正向插入的克隆和反向插入的克隆的混合体等。通常重新挑选独立的克隆可以解决该问题。
第二种情况是一些产量很低的质粒,测序时需要加入较多体积的模板,因此相应包含的杂质就要多了,这些增加的杂质对测序将产生极大的影响。通常我们要求质粒的产量要达到每毫升菌液能够提取到至少200 ng的质粒,低于此产量的质粒用于测序成功率将大大下降。对于低产量的质粒,一般让客户自己采取大量提取的方法,拿到足够用于测序的量的质粒,最少1ug。
图11PCR产物的测序结果,该结果信号很强,峰型整齐,但是在该测序结果中有多个位置有重叠峰,出现N值。
造成该情况的主要原因很可能是该PCR产物中有突变体的存在。在每个突变位点上有一个重叠的峰,由于仪器无法正确识别该处的碱基,就只能以N值代替。在有的测序结果中,整条序列信号很好,在个别位置有N值,一定要确认该处N值的具体情况,如果却是两个
重叠峰存在,重新测序也解决不了问题。返回
DNA测序常见问题及分析
DNA测序过程可能遇到的问题及分析 对于一些生物测序公司(如Invitrogen等),我们的菌液或质粒经过PCR和酶切鉴定都没问题,但几天后的测序结果却无法另人满意。 为什么呢? PCR产物直接进行测序,在PCR产物长度以后将无反应信号,机器将产生许多N值。这是由于Taq酶能够在PCR反应的末端非特异性地加上一个A碱基,我们所用的T载体克隆PCR产物就是应用该原理,通常PCR产物结束的位点,PCR产物测序一般末端的一个碱基为A(绿峰),也就是双脱氧核甘酸ddNTP终止反应的位置之前的A,A后的信号会迅速减弱。 N值情况一般是由于有未去除的染料单体造成的干扰峰。该干扰峰和正常序列峰重叠在一起,有时机器377以下的测序仪无法正确判断出为何碱基。有时,在序列的起始端的小片段容易丢失,导致起始区信号过低,机器有时也无法正确判读。在序列的3’端易产生N值。一个测序反应一般可以读出900bp以上的碱基(ABI3730可以达到1200bp),但是,只有一般600bp以前的碱基是可靠的,理想条件下,多至700bp的碱基都是可以用的。一般在650bp以后的序列,由于测序毛细管胶的分辩率问题,会有许多碱基分不开,就会产生N值。测序模板本身含杂合序列,该情况主要发生在PCR产物直接测序,由于PCR产物本身有突变或含等位基因,会造成在某些位置上有重叠峰,产生N值。这种情况很容易判断,那就是整个序列信号都非常好,只有在个别位置有明显的重叠峰,视杂合度不同N值也不同。 测序列是从引物3’末端后第一个碱基开始的,所以就看不到引物序列。有两种方法可以得到引物序列。1.对于较短的PCR产物 (<600bp),可以用另一端的引物进行测序,从另一端测序可以一直测通,可以在序列的末端得到该引物的反向互补序列。对于较长的序列,一个测序反应测不通,就只能将PCR产物片段克隆到载体中,用载体上的通用引物(T7/SP6)进行测序。载体上的通用引物与所插入序列间
高通量测序基础知识
高通量测序基础知识简介 陆桂 什么是高通量测序? 高通量测序技术(High-throughput sequencing,HTS)是对传统Sanger测序(称为一代测序技术)革命性的改变,一次对几十万到几百万条核酸分子进行序列测定, 因此在有些文献中称其为下一代测序技术(next generation sequencing,NGS )足见其划时代的改变, 同时高通量测序使得对一个物种的转录组和基因组进行细致全貌的分析成为可能, 所以又被称为深度测序(Deep sequencing)。 什么是Sanger法测序(一代测序) Sanger法测序利用一种DNA聚合酶来延伸结合在待定序列模板上的引物。直到掺入一种链终止核苷酸为止。每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。由于ddNTP缺乏延伸所需要的3-OH基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止。终止点由反应中相应的双脱氧而定。每一种dNTPs和ddNTPs的相对浓度可以调整,使反应得到一组长几百至几千碱基的链终止产物。它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,凝胶处理后可用X-光胶片放射自显影或非同位素标记进行检测。 什么是基因组重测序(Genome Re-sequencing) 全基因组重测序是对基因组序列已知的个体进行基因组测序,并在个体或群体水平上进行差异性分析的方法。随着基因组测序成本的不断降低,人类疾病的致病突变研究由外显子区域扩大到全基因组范围。通过构建不同长度的插入片段文库和短序列、双末端测序相结合的策略进行高通量测序,实现在全基因组水平上检测疾病关联的常见、低频、甚至是罕见的突变位点,以及结构变异等,具有重大的科研和产业价值。 什么是de novo测序 de novo测序也称为从头测序:其不需要任何现有的序列资料就可以对某个物种进行测序,利用生物信息学分析手段对序列进行拼接,组装,从而获得该物种的基因组图谱。获得一个物种的全基因组序列是加快对此物种了解的重要捷径。随着新一代测序技术的飞速发展,基因组测序所需的成本和时间较传统技术都大大降低,大规模基因组测序渐入佳境,基因组学研究也迎来新的发展契机和革命性突破。利用新一代高通量、高效率测序技术以及强大的生物信息分析能力,可以高效、低成本地测定并分析所有生物的基因组序列。 什么是外显子测序(whole exon sequencing) 外显子组测序是指利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法。外显子测序相对于基因组重测序成本较低,对研究已知基因的SNP、Indel等具有较大的优势,但无法研究基因组结构变异如染色体断裂重组等。
DNA测序结果分析
学习 通常一份测序结果图由红、黑、绿和蓝色测序峰组成,代表不同的碱基序列。测序图的两端(本图原图的后半段被剪切掉了)大约50个碱基的测序图部分通常杂质的干扰较大,无法判读,这是正常现象。这也提醒我们在做引物设计时,要避免将所研究的位点离PCR序列的两端太近(通常要大于50个碱基距离),以免测序后难以分析比对。 我的课题是研究基因多态性的,因此下面要介绍的内容也主要以判读测序图中的等位基因突变位点为主。 实际上,要在一份测序图中找到真正确实的等位基因多态位点并不是一件容易的事情。由于临床专业的研究生,这些东西是没人带的,只好自己研究。开始时大概的知道等位基因位点在假如在测序图上出现像套叠的两个峰,就是杂合子位点。实际比对了数千份序列后才知道,情况并非那么简单,下面测序图中标出的两
个套峰均不是杂合子位点,如图并说明如下: 说明:第一组套峰,两峰的轴线并不在同一位置,左侧的T峰是干扰峰;第二组套峰,虽两峰轴线位置相同,但两峰的位置太靠近了,不是杂合子峰,蓝色的C峰是干扰峰通常的杂合子峰由一高一略低的两个轴线相同的峰组成,此处的序列被机器误判为“C”,实际的序列应为“A”,通常一个高大碱基峰的前面1~2个位点很容易产生一个相同碱基的干扰峰,峰的高度大约是高大碱基峰的1/2,离得越近受干扰越大。一个摸索出来的规律是:主峰通常在干扰峰的右侧,干扰峰并不一定比主峰低。最关键的一点是一定要拿疑似为杂合子峰的测序图位点与测序结果的文本序列和基因库中的比对结果相比较;一个位点的多个样本相比较;你得出的该位点的突变率与权威文献或数据库中的突变率相比较。通常,对于一个疑似突变位点来说,即使是国际上权威组织大样本的测序结果中都没有报道的话,那么单纯通过测序结果就判定它是突变点,是并不严谨的,因一份PCR产物中各个碱基的实际含量并不相同,很难避免不产生误差的。对于一个未知
转录组高通量测序
转录组高通量测序 2010-11-22 09:48 (第二代高通量测序技术-454) 转录组即特定细胞在某一功能状态下所能转录出来的所有RNA的总和,是研究细胞表型和功能的一个重要手段。与基因组不同的是,转录组的定义中包含了时间和空间的限定。同一细胞在不同的生长时期及生长环境下,其基因表达情况是不完全相同的。罗氏GS-FLX-Titanium第二代高通量测序仪平均读长超过 400bp,在测序读长上遥遥领先于其它第二代高通量测序仪,使其成为转录组学研究的首选测序平台,已被广泛应用于基础研究、临床诊断和药物研发等领域。 一、罗氏454测序技术在环境微生物生态多样性研究中的突出优势体现在:(1)测序序列长,便于聚类拼接,可以对转录本进行从头组装(de novo assembly)。 (2)测序通量高,可以检测到低丰度转录本信息。 (3)可以对无基因组参考序列的新物种进行转录组测序,发现新的转录本和亚型。 (4)实验操作简单、结果稳定,可重复性强。无需进行克隆的文库构建,双链cDNA连接454接头后可以直接进行测序,实验周期短。 (5)测序数据便于进行生物信息分析,可以进行基因差异表达分析、鉴定基因的可变剪切以及预测新基因。 二、美吉公司在环境微生物生态多样性研究中的突出优势体现在: (1)拥有自主实验室和高通量测序平台,可以根据客户要求灵活安排实验,实验周期短,取样方便,质量可靠。 (2)技术人员经验丰富,可以稳定地进行总RNA的提取和双链cDNA的合成,可以根据顾客要求第一时间提供实验方案。 (3)有专业的生物信息团队和大型计算机,可以为客户提供个性化的生物信息分析服务。 (4)开放式实验室,参与式服务。客户不但可以参与整个实验过程,而且可以参与生物信息分析,提供最为增值的售后服务。 三、服务流程 (1)客户提供样本背景信息、实验目的和实验预期。 (2)美吉公司设计实验方案,提供测序深度建议和生物信息分析建议。 (3)客户认可实验方案,双方签订项目合作协议。 (4)项目开始运作,美吉公司指定专人和客户保持无障碍沟通。 (5)项目结束,美吉公司提供标准结题报告。 (6)客户可以和美吉公司签订长期合作协议,享受折扣和VIP服务。 四、送样要求 (1)动物、植物、微生物组织: > 请提供足量的新鲜样品,样品量≥5g;植物材料应避免过老的组织,尽量用柔嫩部位。 > 新鲜程度要求:采样后将样品立即液氮速冻-80℃保存(保存期不超过1个月),干冰运输,运输时间不超过72h。 > 样本保存期间切忌反复冻融。
高通量测序生物信息学分析(内部极品资料,初学者必看)
基因组测序基础知识 ㈠De Novo测序也叫从头测序,是首次对一个物种的基因组进行测序,用生物信息学的分析方法对测序所得序列进行组装,从而获得该物种的基因组序列图谱。 目前国际上通用的基因组De Novo测序方法有三种: 1. 用Illumina Solexa GA IIx 测序仪直接测序; 2. 用Roche GS FLX Titanium直接完成全基因组测序; 3. 用ABI 3730 或Roche GS FLX Titanium测序,搭建骨架,再用Illumina Solexa GA IIx 进行深度测序,完成基因组拼接。 采用De Novo测序有助于研究者了解未知物种的个体全基因组序列、鉴定新基因组中全部的结构和功能元件,并且将这些信息在基因组水平上进行集成和展示、可以预测新的功能基因及进行比较基因组学研究,为后续的相关研究奠定基础。 实验流程: 公司服务内容 1.基本服务:DNA样品检测;测序文库构建;高通量测序;数据基本分析(Base calling,去接头, 去污染);序列组装达到精细图标准 2.定制服务:基因组注释及功能注释;比较基因组及分子进化分析,数据库搭建;基因组信息展 示平台搭建 1.基因组De Novo测序对DNA样品有什么要求?
(1) 对于细菌真菌,样品来源一定要单一菌落无污染,否则会严重影响测序结果的质量。基因组完整无降解(23 kb以上), OD值在1.8~2.0 之间;样品浓度大于30 ng/μl;每次样品制备需要10 μg样品,如果需要多次制备样品,则需要样品总量=制备样品次数*10 μg。 (2) 对于植物,样品来源要求是黑暗无菌条件下培养的黄化苗或组培样品,最好为纯合或单倍体。基因组完整无降解(23 kb以上),OD值在1.8~2.0 之间;样品浓度大于30 ng/μl;样品总量不小于500 μg,详细要求参见项目合同附件。 (3) 对于动物,样品来源应选用肌肉,血等脂肪含量少的部位,同一个体取样,最好为纯合。基因组完整无降解(23 kb以上),OD值在1.8~2.0 之间;样品浓度大于30 ng/μl;样品总量不小于500 μg,详细要求参见项目合同附件。 (4) 基因组De Novo组装完毕后需要构建BAC或Fosmid文库进行测序验证,用于BAC 或Fosmid文库构建的样品需要保证跟De Novo测序样本同一来源。 2. De Novo有几种测序方式 目前3种测序技术 Roche 454,Solexa和ABI SOLID均有单端测序和双端测序两种方式。在基因组De Novo测序过程中,Roche 454的单端测序读长可以达到400 bp,经常用于基因组骨架的组装,而Solexa和ABI SOLID双端测序可以用于组装scaffolds和填补gap。下面以solexa 为例,对单端测序(Single-read)和双端测序(Paired-end和Mate-pair)进行介绍。Single-read、Paired-end和Mate-pair主要区别在测序文库的构建方法上。 单端测序(Single-read)首先将DNA样本进行片段化处理形成200-500bp的片段,引物序列连接到DNA片段的一端,然后末端加上接头,将片段固定在flow cell上生成DNA簇,上机测序单端读取序列(图1)。 Paired-end方法是指在构建待测DNA文库时在两端的接头上都加上测序引物结合位点,在第一轮测序完成后,去除第一轮测序的模板链,用对读测序模块(Paired-End Module)引导互补链在原位置再生和扩增,以达到第二轮测序所用的模板量,进行第二轮互补链的合成测序(图2)。 图1 Single-read文库构建方法图2 Paired-end文库构建方法
一代测序常见问题及解决策略
测序常见问题及解决策略 一、PCR常见问题 1.假阴性,不出现扩增条带 PCR出现假阴性结果,可从以下几个方面来寻找原因: 1)模板:①模板中有杂蛋白;②模板中有Taq酶抑制剂;③在提取制备模板时丢失过多;④模板核酸变性不彻底。 2)酶:酶失活或反应时忘了加酶。 3)Mg2+浓度:Mg2+浓度过高可降低PCR扩增的特异性,浓度过低则影响PCR 扩增产量甚至使PCR扩增失败而不出扩增条带。 4)反应条件:变性对PCR扩增来说相当重要,如变性温度低,变性时间短,极有可能出现假阴性;退火温度过低,可致非特异性扩增而降低特异性扩增效率退火温度过高影响引物与模板的结合而降低PCR扩增效率。 5)靶序列变异:靶序列发生突变或缺失,影响引物与模板特异性结合,或因靶序列某段缺失使引物与模板失去互补序列,其PCR扩增是不会成功的。 2.假阳性 假阳性:出现的PCR扩增条带与目的靶序列条带一致,有时其条带更整齐,亮度更高。常见原因有: 1)引物设计不合适:选择的扩增序列与非目的扩增序列有同源性,因而在进行PCR扩增时,扩增出的PCR产物为非目的性的序列。靶序列太短或引 物太短,容易出现假阳性。需重新设计引物。 2)靶序列或扩增产物的交叉污染:这种污染有两种原因:一是整个基因组或大片段的交叉污染,导致假阳性。这种假阳性可用以下方法解决:操作时应小心轻柔,防止将靶序列吸入加样枪内或溅出离心管外。二是空气中的 小片段核酸污染,这些小片段比靶序列短,但有一定的同源性。可互相拼接,与引物互补后,可扩增出PCR产物,而导致假阳性的产生,可用巢式PCR方法来减轻或消除。 3.出现非特异性扩增带 PCR扩增后出现的条带与预计的大小不一致,或大或小,或者同时出现特异性扩增带与非特异性扩增带。非特异性条带的出现,其原因:一是引物
高通量测序RNA-seq数据的常规分析
案例一 虽然RNA-seq早已被大家所熟知,特别是在高通量测序越来越便宜的今天,但是RNA-seq数据的分析仍令多数小菜抓狂。多个软件的使用,参数设置,参考基因组准备,输出结果的解读等等,都让很多初次接触测序数据或者非生物信息专业的人头疼不已。 哈哈,不用怕,有云生信,这都不是事儿!今天我就向大家简单介绍一下如何用云生信做RNA-seq数据的常规分析。不过在此之前,我要稍稍啰嗦一下RNA-seq的常规分析流程,请不要拍砖头。图1是RNA-seq数据从产生到分析的常规分析流程:根据实验设计,提取细胞RNA,并将RNA提交给测序公司,就可以坐等测序数据了。测序公司会根据客户提供的RNA进行建库,上机测序。拿到测序数据后,就到了我们大显身手的时候了。首先,我们要对测序结果做个简单的质量评估,剔除低质量的数据。然后,根据基因组数据(这里我们讲的是基因组数据已知的物种,基因组未知的有套独立的流程,这里不讲),将测序数据组装。根据组装结果,计算基因或转录本的表达量。最后,同芯片数据一样,我们可以根据表达量数据做很多分析,如差异表达分析,网络分析(包括蛋白互作网络,共表达网络等),也可以结合临床数据做分析(如预后,亚型分类、关联,药效等)。 图1. RNA-seq常规分析流程
叨叨完毕,进入正题。 进入尔云后,打开“测序数据处理”模块,我们会看到图2的结果。在这一模块,我们可以完成RNA-seq数据分析的前两步:1、数据质控和过滤低质量数据;2、基因组组装,计算基因表达量。对于上面两部,尔云又根据是双端测序还是单端测序,分了两块。以edgeR 为例,输出的DEGs.txt就是根据我们设定的参数得到的差异表达基因的列表,有geneSymbol, logCPM, PVlue信息。 图2. 测序数据处理模块 质控结束后,尔云会给出全部的质控结果。图3是以demo数据为例的双端测序的质控结果,好多好多呀,可以下了慢慢看。建议主要关注一下xxx_qc_TABLE,该表格是对质控前后的数据统计,反应了测序的好坏。Clean_xxx.fq是质控后的干净的fastq数据,是第2步组装的输入文件。 图3.质控结果 组装完成后,会返回一个expression.txt的表达矩阵文件,该文件是下一步差异表达分析的输入分析。 得到表达矩阵后,我们就可以进入到第3步差异表达数据分析。进入尔云的“差异分析”模块(如下图所示),它针对芯片和测序两种检测技术提供了不同的分析方案。对于RNA-seq
大基因组大数据与生物信息学英文及翻译
Big Genomic Data in Bioinformatics Cloud Abstract The achievement of Human Genome project has led to the proliferation of genomic sequencing data. This along with the next generation sequencing has helped to reduce the cost of sequencing, which has further increased the demand of analysis of this large genomic data. This data set and its processing has aided medical researches. Thus, we require expertise to deal with biological big data. The concept of cloud computing and big data technologies such as the Apache Hadoop project, are hereby needed to store, handle and analyse this data. Because, these technologies provide distributed and parallelized data processing and are efficient to analyse even petabyte (PB) scale data sets. However, there are some demerits too which may include need of larger time to transfer data and lesser network bandwidth, majorly. 人类基因组计划的实现导致基因组测序数据的增殖。这与下一代测序一起有助于降低测序的成本,这进一步增加了对这种大基因组数据的分析的需求。该数据集及其处理有助于医学研究。 因此,我们需要专门知识来处理生物大数据。因此,需要云计算和大数据技术(例如Apache Hadoop项目)的概念来存储,处理和分析这些数据。因为,这些技术提供分布式和并行化的数据处理,并且能够有效地分析甚至PB级的数据集。然而,也有一些缺点,可能包括需要更大的时间来传输数据和更小的网络带宽,主要。 Introduction The introduction of next generation sequencing has given unrivalled levels of sequence data. So, the modern biology is incurring challenges in the field of data management and analysis. A single human's DNA comprises around 3 billion base pairs (bp) representing approximately 100 gigabytes (GB) of data. Bioinformatics is encountering difficulty in storage and analysis of such data. Moore's Law infers that computers double in speed and half in size every 18 months. And reports say that the biological data will accumulate at even faster pace [1]. Sequencing a human genome has decreased in cost from $1 million in 2007 to $1 thousand in 2012. With this falling cost of sequencing and after the completion of the Human Genome project in 2003, inundate of biological sequence data was generated. Sequencing and cataloguing genetic information has increased many folds (as can be observed from the GenBank database of NCBI). Various medical research institutes like the National Cancer Institute are continuously targeting on sequencing of a million genomes for the understanding of biological pathways and genomic variations to predict the cause of the disease. Given, the whole genome of a tumour and a matching normal tissue sample consumes 0.1 T B of compressed data, then one million genomes will require 0.1 million TB, i.e. 103 PB (petabyte) [2]. The explosion of Biology's data (the scale of the data exceeds a single machine) has made it more expensive to store, process and analyse compared to its generation. This has stimulated the use of cloud to avoid large capital infrastructure and maintenance costs. In fact, it needs deviation from the common structured data (row-column organisation) to a semi-structured or unstructured data. And there is a need to develop applications that execute in parallel on distributed data sets. With the effective use of big data in the healthcare sector, a
20个测序常见的问题
20个测序常见的问题 1.为什么需要新鲜的菌液? 首先,新鲜的菌液易于培养,可以获得更多的DNA,同时最大限度地保证菌种的纯度。2.如何提供菌液? 如果您提供新鲜菌液,用封口膜封口以免泄漏;也可以将培养好的4~5ml菌液沉淀下来,倒去上清以方便邮寄。同时邮寄时最好用盒子以免邮寄过程中压破。 3.如何制作穿刺菌? 用灭菌过1.5ml或2ml离心管加入LB琼脂(7g/L)斜面凝固,用接种针挑取分散良好的单菌落穿过琼脂直达管底,不完全盖紧管盖适当温度培养过夜,然后盖紧盖子加封口膜,室温或4度保存。 4.PCR产物直接测序有什么要求? (1)扩增产物必须特异性扩增,条带单一。如果扩增产物中存在非特异性扩增产物,一般难以得到好的测序结果; (2)必须进行胶回收纯化; (3)DNA纯度在1.6—2.0之间,浓度50ng/ul以上。 5.为什么PCR产物直接测序必须进行Agarose胶纯化? 如果不进行胶纯化而直接用试剂盒回收,经常会导致测序出现双峰甚至乱峰,这主要是非特异性扩增产物或者原来的PCR引物去除不干净所导致。大多所谓的PCR“纯化试剂盒”实际上只是回收产物而不能起到纯化的作用的。对于非特异性扩增产物肯定无法去除,而且通常他们不能够完全去除所有的PCR引物,这会造成残留的引物在测序反应过程中参与反应而导致乱峰。 6.如何进行PCR产物纯化? PCR产物首先必须用Agarose胶电泳,将特异扩增的条带切割下,然后纯化。使用凝胶回收试剂盒回收,产物用ddH2O溶解。 7.PCR产物直接测序的好处? (1) PCR产物直接测序可以反映模板的真实情况; (2) 省去克隆的实验费用和时间; (3) PCR产物测序正确的片段进行下一步克隆实验使结果更有保障; (4) 混合模板进行PCR的产物直接测序可以发现其中的点突变。 8.对用于测序的质粒DNA的要求有哪些? 对测序模板DNA的一般要求:(1)DNA纯度要求高,1.6—2.0之间,不能有混合模板,也不能含有RNA,染色体DNA,蛋白质等;(2)溶于ddH2O中,溶液不能含杂质,如盐类,或EDTA等螯合剂,将干扰测序反应正常进行。 9.如何鉴定质粒DNA浓度和纯度? 我们使用水平琼脂糖凝胶电泳,并在胶中加入0.5ug/ml的EB(电泳缓冲液中不必加E,加一个已知浓度的标准样品。电泳结束以后在紫外灯下比较亮度,判断浓度和纯度。此方法可以更直接、准确地判断样品中是否含有染色体DNA、RNA等,也可以鉴别抽提的质粒DNA 的不同构型。 质粒DNA的3种构型是指在抽提质粒DNA过程中,由于各种原因的影响,使得超螺旋的共价闭合环状结构的质粒(SC)的一条链断裂,变成开环状(OC)分子,如果两条链发生断裂,就变成为线状(L)分子。这3种分子有不同的迁移率,通常,超螺旋型(SC)迁移速度最快,其次为线状(L)分子,最慢为开环状(OC)分子。使用紫外分光光度计检测,或者用溴乙锭-标准浓度DNA比较法只能检测抽提到的产物中的浓度,甚至由于抽提的质粒DNA中含有RNA、蛋白质、染色体DNA等因素的干扰,浓度检测的数值也是没有多少意义的。
高通量测序的生物信息学分析
附件三生物信息学分析 一、基础生物信息学分析 1.有效测序序列结果统计 有效测序序列:所有含样品barcode(标签序列)的测序序列。 统计该部分序列的长度分布情况。 注:合同中约定测序序列条数以有效测序序列为准。 图形示例为: 2.优质序列统计 优质序列:有效测序序列中含有特异性扩增引物、不含模糊碱基、长度大于可供分析标准的序列。 统计该部分序列的长度分布情况。 图形示例为:
3.各样本序列数目统计: 统计各个样本所含有效测序序列和优质序列数目。 结果示例为: 4.OTU生成: 根据序列的相似性,将序列归为多个OTU(操作分类单元),以便后续分析。 5.稀释曲线(rarefaction 分析) 根据第4条中获得的OTU数据,做出每个样品的Rarefaction曲线。本合同默认生成OTU相似水平为0.03的rarefaction曲线。 rarefaction曲线结果示例:
6.指数分析 计算各个样品的相关分析指数,包括: ?丰度指数:ace\chao ?多样性指数:shannon\simpson ?本合同默认生成OTU相似水平为0.03的上述指数值。 多样性指数分析结果示例: 注:默认分析以上所列指数,如有特殊需要请说明。 7.Shannon-Wiener曲线 利用各样品的测序量在不同测序深度时的微生物多样性指数构建曲线,反映各样本在不同测序数量时的微生物多样性。当曲线趋向平坦时,说明测序数据量足够大,可以反映样品中绝大多数的微生物信息。绘制默认水平为:0.03。 例图:
8.Rank_Abuance 曲线 根据各样品的OTU丰度大小排序作丰度分布曲线图。结果文件默认为PDF格式(其它格式请注明)。 例图: 9.Specaccum物种累积曲线(大于10个样品) 物种累积曲线( species accumulation curves) 用于描述随着抽样量的加大物种增加的状况,是理解调查样地物种组成和预测物种丰富度的有效工具,在生物多样性和群落调查中,被广泛用于抽样量充分性的判断以及物种丰富度( species richness) 的估计。因此,通过物种累积曲线不仅可以判断抽样量是否充分,在抽样量充分的前提下,运用物种累积曲线还可以对物种丰富度进行预测。
高通量测序:环境微生物群落多样性分析
(5)高通量测序:环境微生物群落多样性分析 微生物群落多样性的基本概念 环境中微生物的群落结构及多样性和微生物的功能及代谢机理是微生物生态学的研究 热点。长期以来,由于受到技术限制,对微生物群落结构和多样性的认识还不全面, 对微生物功能及代谢机理方面了解的也很少。但随着高通量测序、基因芯片等新技术 的不断更新,微生物分子生态学的研究方法和研究途径也在不断变化。第二代高通量 测序技术(尤其 是Roche 454高通量测序技术)的成熟和普及,使我们能够对环境微生物进行深度测序,灵 敏地探测出环境微生物群落结构随外界环境的改变而发生的极其微弱的变化,对于我 们研究微生物与环境的关系、环境治理和微生物资源的利用以及人类医疗健康有着重 要的理论和现实意义。 在国内,微生物多样性的研究涉及农业、土壤、林业、海洋、矿井、人体医学等诸多领域。以在医疗领域的应用为例,通 过比较正常和疾病状态下或疾病不同进程中人体微生物群落的结构和功能变化,可以 对正常人群与某些疾病患者体内的微生物群体多样性进行比较分析,研究获得人体微 生物群
落变化同疾病之间的关系;通过深度测序还可以快速地发现和检测常见病原及新发传 染病病原微生物。研究方法进展 环境微生物多样性的研究方法很多,从国内外目前采用的方法来看大致上包括以下四 类:传统的微生物平板纯培养方法、微平板分析方法、磷脂脂肪酸法以及分子生物学 方法等等。 近几年,随着分子生物学的发展,尤其是高通量测序技术的研发及应用,为微生物分 子生态学的研究策略注入了新的力量。 目前用于研究微生物多样性的分子生物学技术主要包 括:DGGE/TGGE/TTGE 、 T-RFLP 、SSCP、FISH 、印记杂交、定量 PCR、基因芯片等。 DGGE 等分子指纹图谱技术,在其实验结果中往往只含有数十条条带,只能反映出样品中少数 优势菌的信息;另一方面,由于分辨率的误差,部分电泳条带中可能包含不只一种 16S rDNA 序列,因此要获悉电泳图谱中具体的菌种信息,还需 对每一条带构建克隆文库,并筛选克隆进行测序,此实验操 作相对繁琐;此外,采用这种方法无法对样品中的微生物做 到绝对定量。生物芯片是通过固定在芯片上的探针来获得微
CHIP SEQ分析常见问题集锦
ChIP-Seq分析常见问题集锦 染色质免疫共沉淀测序(ChIP-Seq)是指对染色质免疫共沉淀(ChIP)获得的DNA片段进行大规模测序,并能把所研究蛋白的DNA结合位点精确定位到基因组上。 Roche GS FLX Titanium、Illumina Solexa GA IIx和AB SOLID4这3种测序技术均可以用于ChIP-seq,其中采用Illumina Solexa GA IIx进行ChIP-Seq已有较多文献报道。 ChIP-Seq技术高质量、高通量、低成本的数据产出,为表观遗传组学研究奠定了技术基础。研究者可以在以下几方面展开研究:(1)判断DNA链的某一特定位置会出现何种组蛋白修饰;(2)检测RNA polymerase II及其它反式因子在基因组上结合位点的精确定位;(3)研究组蛋白共价修饰与基因表达的关系;(4)CTCF转录因子研究。 ChIP-Seq有什么样品要求? 答:(1)请提供浓度≥10ng/ul、总量≥200ng、OD260/280为1.8~2.2的DNA样品;若单次ChIP后DNA量不够,建议将2~3次ChIP的DNA合并在一起。 (2)请提供DNA打断时检测胶图,要求打断后DNA电泳主带在200-500bp范围内;请对于ChIP 获得DNA设计引物进行QPCR验证和定量,能够提供检测位点的检测报告。附阳性和阴性对照。(3)样品请置于1.5ml管中,管上注明样品名称、浓度以及制备时间,管口使用Parafilm 封口。在运输前将所有样品管固定于50ml带盖离心管中,再将50ml管放在封口袋中。 ChIP-Seq相比ChIP-chip有哪些优势? 答:第一,ChIP-Seq能实现真正的全基因组分析。目前所能获得的芯片上固定的探针只能代表全基因组部分序列,所获得的杂交信息具有偏向性;第二,对于结合位点分析,ChIP-Seq 通过寻找“峰”,结合分辨率可精确到10~30bp,而芯片上探针由于长度所限,无法精确定位,即使目前最高水平的商业芯片都无法提供可与ChIP-Seq媲美的分辨率;第三是所需样本数量。ChIP-chip需要多达4~5μg的起始样本,在杂交之前需要进行LM-PCR,但可能导致背景增高,竞争性扩增等导致假阳性。而ChIP-Seq仅需要纳克级起始材料,如SOLiD起始材料可低至20ng。两者技术特点如下: 研究方法CHIP-on-chip CHIP-Seq 分辨率30~100bp1bp 覆盖范围受芯片容量限制,只能选择性地扫 描特定区域,无法覆盖全基因组只要测定的序列(Reads)能够定位到基因组上,就能获得全部基因组信息 缺陷探针和非特异性区域杂交测序数据会有一些GC含量偏向 性价比只能研究在基因组上广泛存在的目 的位点(Broading bingding)可以扫描全基因组;可以研究在基因组上存在的稀有目的位点(Sharp bingding) 需要的DNA 量 高低(10~50bp)动态量程弱信号会被遗弃;强信号会饱和没有局限 选择数据产 出量 不可以可以
高通量测序 名词解释
高通量测序基础知识汇总 一代测序技术:即传统的Sanger测序法,Sanger法是根据核苷酸在待定序列模板上的引物点开始,随机在某一个特定的碱基处终止,并且在每个碱基后面进行荧光标记,产生以A、T、C、G结束的四组不同长度的一系列核苷酸,每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。由于ddNTP缺乏延伸所需要的3-OH 基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止,使反应得到一组长几百至几千碱基的链终止产物。它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,通过检测得到DNA碱基序列。 二代测序技术:next generation sequencing(NGS)又称为高通量测序技术,与传统测序相比,二代测序技术可以一次对几十万到几百万条核酸分子同时进行序列测定,从而使得对一个物种的转录组和基因组进行细致全貌的分析成为可能,所以又被称为深度测序(Deep sequencing)。NGS主要的平台有Roche(454 & 454+),Illumina(HiSeq 2000/2500、GA IIx、MiSeq),ABI SOLiD等。 基因:Gene,是遗传的物质基础,是DNA或RNA分子上具有遗传信息的特定核苷酸序列。基因通过复制把遗传信息传递给下一代,使后代出现与亲代相似的性状。 DNA:Deoxyribonucleic acid,脱氧核糖核酸,一个脱氧核苷酸分子由三部分组成:含氮碱基、脱氧核糖、磷酸。脱氧核糖核酸通过3',5'-磷酸二酯键按一定的顺序彼此相连构成长链,即DNA链,DNA链上特定的核苷酸序列包含有生物的遗传信息,是绝大部分生物遗传信息的载体。
基因测序(PCR常见问题)
基因测序(PCR常见问题)生物专业很实用 PCR常见问题 PCR常见问题分析及对策(无扩增产物、非特异性扩增、拖尾、假阳性) 问题1:无扩增产物 现象:正对照有条带,而样品则无 原因: 1.模板:含有抑制物,含量低 2.Buffer对样品不合适 3.引物设计不当或者发生降解 4.反应条件:退火温度太高,延伸时间太短 对策: 1.纯化模板或者使用试剂盒提取模板DNA或加大模板的用量 2.更换Buffer或调整浓度 3.重新设计引物(避免链间二聚体和链内二级结构)或者换一管新引物 4.降低退火温度、延长延伸时间 问题2:非特异性扩增 现象:条带与预计的大小不一致或者非 特异性扩增带
原因: 1.引物特异性差 2.模板或引物浓度过高 3.酶量过多 4.Mg2+浓度偏高 5.退火温度偏低 6.循环次数过多 对策: 1.重新设计引物或者使用巢式PCR 2.适当降低模板或引物浓度 3.适当减少酶量 4.降低镁离子浓度 5.适当提高退火温度或使用二阶段温度法 6.减少循环次数 问题3:拖尾 现象:产物在凝胶上呈Smear状态。 原因: 1.模板不纯 2.Buffer不合适 3.退火温度偏低 4.酶量过多 5.dNTP、Mg 2+浓度偏高 6.循环次数过多 对策: 1.纯化模板 2.更换Buffer 3.适当提高退火温度 4.适量用酶 5.适当降低dNTP和镁离子的浓度 6.减少循环次数 问题4:假阳性 现象:空白对照出现目的扩增产物 原因: 靶序列或扩增产物 的交*污染 对策: 1.操作时应小心轻柔,防止将靶序列吸入加样枪内或溅出离心管外; 2.除酶及不能耐高温的物质外,所有试剂或器材均应高压消毒。所用离心管及加样枪头等均应一次性使用。 3.各种试剂最好先进行分装,然后低温贮存 PCR引物设计的黄金法则(转自tiangen)
高通量测序及分析
高通量测序与功能分析 微生物群落测序是指对微生物群体进行高通量测序,通过分析测序序列的构成分析特定环境中微生物群体的构成情况或基因的组成以及功能。借助不同环境下微生物群落的构成差异分析我们可以分析微生物与环境因素或宿主之间的关系,寻找标志性菌群或特定功能的基因。对微生物群落进行测序包括两类,一类是通过16s rDNA,18s rDNA,ITS区域进行扩增测序分析微生物的群体构成和多样性;还有一类是宏基因组测序,是不经过分离培养微生物,而对所有微生物DNA进行测序,从而分析微生物群落构成,基因构成,挖掘有应用价值的基因资源。 以16s rDNA扩增进行测序分析主要用于微生物群落多样性和构成的分析,目前的生物信息学分析也可以基于16s rDNA的测序对微生物群落的基因构成和代谢途径进行预测分析,大大拓展了我们对于环境微生物的微生态认知。 目前我们根据16s的测序数据可以将微生物群落分类到种(species)(一般只能对部分菌进行种的鉴定),甚至对亚种级别进行分析, 几个概念: 16S rDNA(或16S rRNA):16S rRNA基因是编码原核生物核糖体小亚基的基因,长度约为1542bp,其分子大小适中,突变率小,是细菌系统分类学研究中最常用和最有用的标志。16S rRNA基因序列包括9个可变区和10个保守区,保守区序列反映了物种间的亲缘关系,而可变区序列则能体现物种间的差异。16S rRNA基因测序以细菌16S rRNA基因测序为主,核心是研究样品中的物种分类、物种丰度以及系统进化。 OTU:operational taxonomic units (OTUs)在微生物的免培养分析中经常用到,通过提取样品的总基因组DNA,利用16S rRNA或ITS的通用引物进行PCR 扩增,通过测序以后就可以分析样品中的微生物多样性,那怎么区分这些不同的序列呢,这个时候就需要引入operational taxonomic units,一般情况下,如
全基因组重测序大数据分析报告
全基因组重测序数据分析 1. 简介(Introduction) 通过高通量测序识别发现de novo的somatic和germ line 突变,结构变异-SNV,包括重排突变(deletioin, duplication 以及copy number variation)以及SNP的座位;针对重排突变和SNP的功能性进行综合分析;我们将分析基因功能(包括miRNA),重组率(Recombination)情况,杂合性缺失(LOH)以及进化选择与mutation之间的关系;以及这些关系将怎样使得在disease(cancer)genome中的mutation产生对应的易感机制和功能。我们将在基因组学以及比较基因组学,群体遗传学综合层面上深入探索疾病基因组和癌症基因组。 实验设计与样本 (1)Case-Control 对照组设计; (2)家庭成员组设计:父母-子女组(4人、3人组或多人); 初级数据分析 1.数据量产出:总碱基数量、Total Mapping Reads、Uniquely Mapping Reads统计,测序深度分析。 2.一致性序列组装:与参考基因组序列(Reference genome sequence)的比对分析,利用贝叶斯统计模型检测出每个碱基位点的最大可能性基因型,并组装出该个体基因组的一致序列。 3.SNP检测及在基因组中的分布:提取全基因组中所有多态性位点,结合质量值、测序深度、重复性等因素作进一步的过滤筛选,最终得到可信度高的SNP数据集。并根据参考基因组信息对检测到的变异进行注释。 4.InDel检测及在基因组的分布: 在进行mapping的过程中,进行容gap的比对并检测可信的short InDel。在检测过程中,gap的长度为1~5个碱基。对于每个InDel的检测,至少需要3个Paired-End序列的支持。 5.Structure Variation检测及在基因组中的分布: 能够检测到的结构变异类型主要有:
