Taq DNA Polymerase
DNA聚合酶

240发布评论
用户|网站地图2在手机打开
下载
2下载券
用手机扫此二维码:×
以下结果由提供Βιβλιοθήκη ×翻译百科词条:×
百科分享到:QQ空间新浪微博人人网微信
评价文档:
领取
下载券1
新版反馈
/52下载券
下载
加入VIP,免券下载本文
下载文档到电脑,查找使用更方便
2下载券857人已下载
下载
还剩2页未读,继续阅读
1234567890ABCDEFGHIJKLMNabcdefghijklmn!@#$%^&&*()_+.一三五七九贰肆陆扒拾,。青玉案元夕东风夜放花千树更吹落星如雨宝马雕车香满路凤箫声动玉壶光转一夜鱼龙Astar vPrimer3(在线服务)3、模板的制备PCR的模板可以是DNA,也可以是RNA。模板的取材主要依据PCR的扩增对象,可以是病原体标本如病毒、细菌、真菌等。也可以是病理生理标本如细胞、血液、羊水细胞等。法医学标本有血斑、精斑、毛发等。标本处理的基本要求是除去杂质,并部分纯化标本中的核酸。多数样品需要经过SDS和蛋白酶K处理。难以破碎的细菌,可用溶菌酶加EDTA处理。所得到的粗制DNA,经酚、氯仿抽提纯化,再用乙醇沉淀后用作PCR反应模板。4、PCR反应条件的控制①PCR反应的缓冲液提供合适的酸碱度与某些离子②镁离子浓度总量应比dNTPs的浓度高,常用1.5mmol/L③底物浓度dNTP以等摩尔浓度配制,20~200umol/L④TaqDNA聚合酶2.5U(100ul)⑤引物浓度一般为0.1~0.5umol/L⑥反应温度和循环次数变性温度和时间95℃,30s退火温度和时间低于引物Tm值5℃左右,一般在45~55℃延伸温度和时间72℃,1min/kb(10kb内)Tm值=4(G+C)+2(A+T)循环次数:一般为25~30次。循环数决定PCR扩增的产量。模板初始浓度低,可增加循环数以便达到有效的扩增量。但循环数并不是可以无限增加的。一般循环数为30个左右,循环数超过30个以后,DNA聚合酶活性逐渐达到饱和,产物的量不再随循环数的增加而增加,出现了所谓的“平台期”。编辑本段[PCR步骤]标准的PCR过程分为三步:1.DNA变性(90℃-96℃):双链DNA模板在热作用下,氢键断裂,形成单链DNA 2.退火(25℃-65℃):系统温度降低,引物与DNA模板结合,形成局部双链。3.延伸(70℃-75℃):在Taq酶(在72℃左右,活性最佳)的作用下,以dNTP为原料,从引物的5′端→3′端延伸,合成与模板互补的DNA链。每一循环经过变性、退火和延伸,DNA含量即增加一倍。
taqman荧光探针的原理

taqman荧光探针的原理TaqMan荧光探针是一种常用于实时荧光定量PCR(Polymerase Chain Reaction,聚合酶链式反应)的探针。
其原理基于PCR扩增过程中的特定核酸序列的扩增和荧光信号的检测。
TaqMan荧光探针由三个部分组成:1. 引物(primers):引物是设计用于扩增待测核酸序列的短小DNA 片段,通常有两个引物,一个用于扩增待测序列的起始位点,另一个用于扩增终止位点。
2. 探针(probe):探针是一个含有荧光染料和一个对荧光染料发出信号具有抑制作用的化学修饰的短小DNA片段。
探针的序列与待测核酸序列的中间部分完全匹配。
3. Taq DNA聚合酶(Taq DNA polymerase):Taq DNA聚合酶是一种热稳定的DNA聚合酶,能够在PCR反应中扩增DNA序列。
TaqMan荧光探针的工作原理如下:1. 引物与Taq DNA聚合酶一起作用,将待测核酸序列进行扩增。
引物会识别并结合到待测序列的起始位点和终止位点,Taq DNA聚合酶会沿着待测序列进行DNA合成,生成大量的扩增产物。
2. 在PCR反应中,TaqMan探针与待测序列中间部分的完全匹配,探针的5'端和3'端分别连接着两种不同的荧光染料(通常是荧光发射染料和荧光供体染料)。
3. 在PCR反应过程中,当Taq DNA聚合酶在扩增过程中到达探针的位置时,它会剪切探针,将发射染料和供体染料分离,导致荧光信号的释放。
4. 荧光信号可以通过实时荧光PCR仪器进行实时检测和记录。
荧光信号的强度与PCR反应中扩增产物的数量成正比,从而可以定量测量待测核酸的起始量。
TaqMan荧光探针的原理可实现高度特异性和灵敏度的实时定量PCR 分析,广泛应用于基因表达分析、病原体检测、基因突变分析等领域。
希望以上解释对您有所帮助。
如有任何进一步的问题,请随时提问。
pcr试剂盒原理

pcr试剂盒原理PCR试剂盒原理PCR(Polymerase Chain Reaction,聚合酶链反应)试剂盒是一种用于分子生物学实验的试剂盒,它包含了进行PCR反应所需的各种试剂。
PCR试剂盒的原理是利用DNA的特性,在体外模拟DNA 复制过程,通过不断的循环反应,扩增目标DNA片段,从而获得足够数量的DNA用于后续实验分析。
PCR试剂盒的主要组成包括模板DNA、引物、Taq DNA聚合酶、dNTPs(脱氧核苷酸三磷酸盐)、缓冲液和辅助试剂等。
PCR试剂盒中的模板DNA是待扩增的目标DNA片段,可以是从细胞中提取出的全基因组DNA或特定基因的DNA。
模板DNA是PCR反应的起始物质,决定了PCR扩增的目标。
PCR试剂盒中的引物是一对短的单链DNA分子,它们的序列与目标DNA片段的两端序列互补。
引物在PCR反应中的作用是指导Taq DNA聚合酶在目标DNA上合成新的DNA链。
引物的选择非常重要,合适的引物序列可以确保扩增出特异性强、准确的目标DNA片段。
然后,PCR试剂盒中的Taq DNA聚合酶是一种特殊的热稳定DNA 聚合酶,能够在高温下保持活性。
Taq DNA聚合酶通过在引物的引导下,在PCR反应的不同温度环境中,依次进行DNA链的延伸、分离和合成。
Taq DNA聚合酶的热稳定性使得PCR反应可以在高温下进行,从而实现DNA的扩增。
PCR试剂盒中的dNTPs是PCR反应中的四种脱氧核苷酸三磷酸盐,它们是DNA合成的基本原料。
在PCR反应中,dNTPs被Taq DNA聚合酶利用,与模板DNA进行互补配对,从而合成新的DNA链。
PCR试剂盒中的缓冲液是为了提供适宜的反应环境。
缓冲液中含有适当的盐离子浓度和pH值,可以维持Taq DNA聚合酶的活性和稳定性,促进PCR反应的进行。
PCR试剂盒中还包含一些辅助试剂,如Mg2+离子、BSA(牛血清白蛋白)和阻止试剂等。
Mg2+离子是Taq DNA聚合酶催化反应的必需离子,它参与dNTPs与模板DNA的结合和酶的催化活性。
fast_taq_dna_polymerase
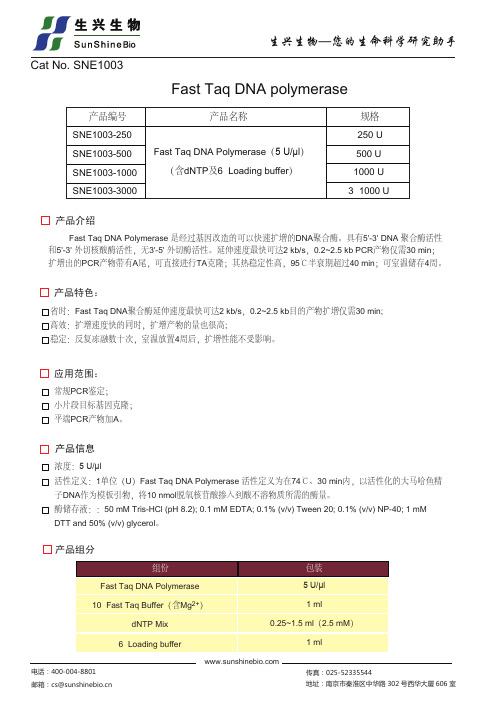
电话:400-004-8801 邮箱:cs@
传真:025-52335544 地址:南京市秦淮区中华路 302 号西华大厦 606 室
以上两反应PCR反应体系:
生兴生物—您的生命科学研究助手
组份 10×PCR反应缓冲液(含Mg2+)
*本品仅供实验室研究使用 感谢并欢迎您使用SunShineBio产品!如您发表的科研文章中提及了SunShineBio产品,请通知我们,我们将为您提 供丰厚的实物礼品,以示祝贺。详情请登陆: /literature_encouragement.html
传真:025-52335544 地址:南京市秦淮区中华路 302 号西华大厦 606 室
生兴生物—您的生命科学研究助手
质量控制
SDS-PAGE检测纯度大于99﹪,经检测无外源核酸酶活性;PCR方法检测无宿主残余DNA,能有效地扩增人基 因组中的单拷贝基因;室温存放4周,无明显活性改变。
dNTP Mix (2.5 mM each) 引物1 (10 μM) 引物2 (10 μM) 模板DNA
Fast Taq DNA聚合酶 ddH2O
体积 5 μl 4 μl 1 μl 1 μl 500 ng 2.5 U 至50 μl
热循环扩增条件:95℃ 2 min;95℃ 1 s,60℃ 1 s,72℃ 1 s;30 cycles;72℃ 10 s。
储存条件
-20℃, 保存1年。
主要技术参数
Fast Taq DNA聚合酶在扩增过程中的延伸速度为2 kb/s;最佳温度为55~65℃;dNTP的工作浓度为100~300 μM, 最佳Mg2+浓度为2~3 mM,最佳pH为8.1~9.1。
PCR推荐反应体系及Mg2+终浓度
taq酶标准

TAQ酶标准一、酶的分类TAQ酶(Thermostable DNA polymerase)是一种热稳定DNA聚合酶,属于DNA聚合酶的一种。
根据酶的来源和性质,可以将酶分为多种不同的类型,包括DNA聚合酶、RNA聚合酶、酯酶、氧化还原酶、蛋白水解酶等等。
每种酶都有其特定的生物学功能和特点,在生物体内发挥着不同的作用。
二、酶的特性TAQ酶具有热稳定性,能够在高温条件下保持活性。
这种特性使得TAQ酶在PCR等高温反应中具有广泛的应用。
此外,TAQ酶还具有高催化效率和高度特异性,能够催化DNA合成等反应,且不易发生误合成。
三、酶的应用TAQ酶在生物学、医学和生物工程领域都有广泛的应用。
在生物学研究中,TAQ酶可以用于基因克隆、DNA序列分析、基因表达等实验中。
在医学领域,TAQ酶可以用于诊断和治疗各种疾病,例如遗传性疾病、癌症、病毒感染等。
在生物工程领域,TAQ酶可以用于基因工程、蛋白质工程等研究中。
四、酶的检测与定量对于TAQ酶的检测和定量,可以使用各种方法,例如活性测定、免疫分析、光谱分析等。
其中,活性测定是常用的方法之一,可以通过测定酶促反应速率来评估TAQ酶的活性。
免疫分析则可以通过检测抗体与TAQ酶的结合情况来定量。
光谱分析可以通过测定光谱变化来定量TAQ酶的浓度。
五、酶的纯化与制备TAQ酶的纯化与制备是保证其质量和应用的关键步骤之一。
通常采用的方法包括离子交换色谱、凝胶色谱、亲和色谱等。
其中,离子交换色谱是常用的方法之一,可以通过交换TAQ酶上的离子来达到纯化的目的。
亲和色谱则可以利用抗体与TAQ酶的特异性结合来纯化。
此外,还可以使用基因工程技术生产重组TAQ酶。
六、酶的结构与功能关系TAQ酶的结构与功能关系是理解其生物学活性的关键。
TAQ酶是一种球状蛋白,由多个亚基组成,每个亚基都具有催化活性。
结构决定功能,TAQ酶的结构决定了其在高温条件下的稳定性和催化效率。
此外,TAQ酶的结构也与其特异性有关,例如识别和结合DNA模板的能力。
赛默飞taq酶说明书 Thermo Scientific EP0402 Taq DNA Polymerase

PRODUCT INFORMATIONTaq DNA Polymerase (recombinant)#EP0402 500 ULot: __ Expiry Date: __ Concentration: 5 U/µLStore at -20°C/onebioOrdering InformationTaq DNA Polymerase (recombinant)Component #EP0401 #EP0402 #EP0405 #EP0406 Taq DNA Polymerase,5 U/µL100 U 500 U 5 x 500 U 10 x 500 U 10X Taq Buffer withKCl0.6 mL 2x1.25 mL 10x1.25 mL 20x1.25 mL 10X Taq Buffer with(NH4)2SO40.6 mL 2x1.25 mL 10x1.25 mL 20x1.25 mL 25 mM MgCl2 0.6 mL 2x1.25 mL 10x1.25 mL 20x1.25 mL Taq DNA Polymerase (recombinant), LCComponent #EP0403 #EP0404Taq DNA Polymerase, 1 U/µL 100 U 500 U10X Taq Buffer with KCl 0.6 mL 2x1.25 mL10X Taq Buffer with (NH4)2SO4 0.6 mL 2x1.25 mL25 mM MgCl2 0.6 mL 2x1.25 mL Rev.12 V DescriptionTaq DNA Polymerase is a highly thermostable DNApolymerase of the thermophilic bacterium Thermusaquaticus. The enzyme catalyzes 5’→3’ synthesis of DNA,has no detectable 3’→5’ exonuclease (proofreading)activity and possesses 5’→3’ exonuclease activity. Inaddition, Taq DNA Polymerase exhibits deoxynucleotidyltransferase activity, which frequently results in the addition ofextra adenines at the 3’-end of PCR products.Recombinant Taq DNA Polymerase is ideal for standardPCR of amplicons 5 kb or shorter.Applications•Routine PCR amplification of DNA fragments up to5 kb (1).•Generation of PCR product for TA cloning.•DNA labeling (2-4).•DNA sequencing (5).SourceE.coli cells with a cloned pol gene from Thermus aquaticusYT1.Definition of Activity UnitOne unit of the enzyme catalyzes the incorporation of10 nmol of deoxyribonucleotides into a polynucleotidefraction (adsorbed on DE-81) in 30 min at 70°C.Enzyme activity is assayed in the following mixture:67 mM Tris-HCl (pH 8.8 at 25°C), 6.7 mM MgCl2,1 mM 2-mercaptoethanol, 50 mM NaCl, 0.1 mg/mL BSA,0.75 mM activated salmon milt DNA, 0.2 mM of each dNTP,0.4 MBq/mL [3H]-dTTP.Storage BufferThe enzyme is supplied in: 20 mM Tris-HCl (pH 8.0),1 mM DTT, 0.1 mM EDTA, 100 mM KCl,0.5% (v/v) Nonidet® P40, 0.5% (v/v) Tween® 20 and50% (v/v) glycerol.10X Taq Buffer with KCl100 mM Tris-HCl (pH 8.8 at 25°C), 500 mM KCl,0.8% (v/v) Nonidet P40.10X Taq Buffer with (NH4)2SO4750 mM Tris-HCl (pH 8.8 at 25°C), 200 mM (NH4)2SO4,0.1% (v/v) Tween 20.Inhibition and Inactivation•Inhibitors: ionic detergents (deoxycholate, sarkosyland SDS) at concentrations higher than 0.06, 0.02 and0.01%, respectively (6).•Inactivated by phenol/chloroform extraction.PROTOCOLTo prepare several parallel reactions and to minimize thepossibility of pipetting errors, prepare a PCR master mix bymixing water, buffer, dNTPs, primers and Taq DNAPolymerase. Prepare sufficient master mix for the numberof reactions plus one extra. Aliquot the master mix intoindividual PCR tubes and then add template DNA.1.Gently vortex and briefly centrifuge all solutions afterthawing.2.Place a thin-walled PCR tube on ice and add the followingcomponents for each 50 µL reaction:10X Taq Buffer 5 µLdNTP Mix, 2 mM each (#R0241) 5 µL (0.2 mM of each)Forward primer 0.1-1.0 µMReverse primer 0.1-1.0 µM25 mM MgCl2* 1-4 mMTemplate DNA 10 pg - 1 µgTaq DNA Polymerase 1.25 UWater, nuclease-free (#R0581) to 50 µLTotal volume 50 µL*Volumes of 25 mM MgCl2, required for specific final MgCl2concentration:Final concentration of MgCl2, mM 1 1.5 2 2.5 3 4Volume of 25 mM MgCl2 to be addedfor 50 µL reaction, µL2 3 4 5 6 83.Gently vortex the samples and spin down.4.If using a thermal cycler that does not use a heated lid,overlay the reaction mixture with 25 µL of mineral oil.5.Perform PCR using recommended thermal cyclingconditions:StepTemperature,°CTimeNumber ofcyclesInitial denaturation 95 1-3 min 1Denaturation 95 30 s25-40Annealing Tm-5 30 sExtension72 1 min/kbFinal Extension 72 5-15 min 1 GUIDELINES FOR PREVENTING CONTAMINATIONOF PCR REACTIONDuring PCR more than 10 million copies of template DNA are generated. Therefore, care must be taken to avoid contamination with other templates and amplicons that may be present in the laboratory environment. General recommendations to lower the risk of contamination are as follows:•Prepare your DNA sample, set up the PCR mixture, perform thermal cycling and analyze PCR products in separate areas.•Set up PCR mixtures in a laminar flow cabinet equipped with an UV lamp.•Wear fresh gloves for DNA purification and reaction set up.•Use reagent containers dedicated for PCR. Use positive displacement pipettes, or use pipette tips with aerosol filters to prepare DNA samples and perform PCR set up. •Use PCR-certified reagents, including high quality water (e.g., Water, nuclease-free, (#R0581)).•Always perform “no template control” (NTC) reactions to check for contamination.GUIDELINES FOR PRIMER DESIGNUse the ThermoScientific REviewer primer design software at /reviewer or follow general recommendations for PCR primer design as outlined below: •PCR primers are generally 15-30 nucleotides long. •Optimal GC content of the primer is 40-60%. Ideally, C and G nucleotides should be distributed uniformly along the primer.•Avoid placing more than three G or C nucleotides at the 3’-end to lower the risk of non-specific priming.•If possible, the primer should terminate with a G or C at the 3’-end.•Avoid self-complementary primer regions, complementarities between the primers and direct primer repeats to prevent hairpin formation and primer dimerization.•Check for possible sites of undesired complementary between primers and template DNA.•When designing degenerate primers, place at least3 conservated nucleotides at the 3’-end.•When introducing restriction enzyme sites into primers, refer to the table “Cleavage efficiency close to the termini of PCR fragments” located on/onebio to determine the number of extra bases required for efficient cleavage. •Differences in melting temperatures (Tm) between the two primers should not exceed 5°C.(continued on reverse page)Estimation of primer melting temperatureFor primers containing less than 25 nucleotides, the approx. melting temperature (Tm) can be calculated using the following equation:Tm= 4 (G + C) + 2 (A + T),where G, C, A, T represent the number of respective nucleotides in the primer.If the primer contains more than 25 nucleotides specialized computer programs e.g., REviewer™ (/reviewer), are recommended to account for interactions of adjacent bases, effect of salt concentration, etc.COMPONENTS OF THE REACTION MIXTURE Template DNAOptimal amounts of template DNA in the 50 µL reaction volume are 0.01-1 ng for both plasmid and phage DNA, and 0.1-1 µg for genomic DNA. Higher amount of template increases the risk of generation of non-specific PCR products. Lower amount of template reduces the accuracy of the amplification.All routine DNA purification methods are suitable for template preparation e.g., Thermo Scientific GeneJET Genomic DNA Purification Kit (#K0721) or GeneJET™ Plasmid Miniprep Kit (#K0502). Trace amounts of certain agents used for DNA purification, such as phenol, EDTA and proteinase K, can inhibit DNA polymerases. Ethanol precipitation and repeated washes of the DNA pellet with 70% ethanol normally removes trace contaminants from DNA samples.MgCl2 concentrationDue to the binding of Mg2+ to dNTPs, primers and DNA templates, Mg2+ concentration needs to be optimized for maximal PCR yield. The recommended concentration range is 1-4 mM. If the Mg2+ concentration is too low, the yield of PCR product could be reduced. On the contrary, non-specific PCR products may appear and the PCR fidelity may be reduced if the Mg2+ concentration is too high.If the DNA samples contain EDTA or other metal chelators, the Mg2+ ion concentration in the PCR mixture should be increased accordingly (1 molecule of EDTA binds one Mg2+). dNTPsThe recommended final concentration of each dNTP is0.2 mM. In certain PCR applications, higher dNTP concentrations may be necessary. Due to the binding ofMg2+ to dNTPs, the MgCl2 concentration needs to be adjusted accordingly. It is essential to have equal concentrations of all four nucleotides (dATP, dCTP, dGTP and dTTP) present in the reaction mixture. To achieve 0.2 mM concentration of each dNTP in the PCRmixture, use the following volumes of dNTP mixes:Volume ofPCR mixturedNTP Mix,2 mM each(#R0241)dNTP Mix,10 mM each(#R0191)dNTP Mix,25 mM each(#R1121)50 µL 5 µL 1 µL 0.4 µL25 µL 2.5 µL 0.5 µL 0.2 µL20 µL 2 µL 0.4 µL 0.16 µLPrimersThe recommended concentration range of the PCR primersis 0.1-1 µM. Excessive primer concentrations increase theprobability of mispriming and generation of non-specific PCRproducts.For degenerate primers higher primer concentrations in therange of 0.3-1 µM are often favorable.CYCLING PARAMETERSInitial DNA denaturationIt is essential to completely denature the template DNA atthe beginning of PCR to ensure efficient utilization of thetemplate during the first amplification cycle. If the GC contentof the template is 50% or less, an initial 1-3 min denaturationat 95°C is sufficient. For GC-rich templates this step shouldbe prolonged up to 10 min. If longer initial denaturation stepis required, or the DNA is denatured at a higher temperature,Taq DNA Polymerase should be added after the initialdenaturation step to avoid a decrease in its activity.DenaturationA DNA denaturation time of 30 seconds per cycle at 95°Cis normally sufficient. For GC-rich DNA templates, this stepcan be prolonged to 3-4 min. DNA denaturation can also beenhanced by the addition of either 10-15% glycerol or10% DMSO, 5% formamide or 1-1.5 M betaine. The meltingtemperature of the primer-template complex decreasessignificantly in the presence of these reagents. Therefore,the annealing temperature has to be adjusted accordingly.In addition, 10% DMSO and 5% formamide inhibit DNApolymerases by 50%. Thus, the amount of the enzymeshould be increased if these additives are used.Primer annealingThe annealing temperature should be 5°C lower than themelting temperature (Tm) of the primers. Annealing for30 seconds is normally sufficient. If non-specific PCRproducts appear, the annealing temperature should beoptimized stepwise in 1-2°C increments. When additiveswhich change the melting temperature of the primer-templatecomplex are used (glycerol, DMSO, formamide and betaine),the annealing temperature must also be adjusted.ExtensionThe optimal extension temperature for Taq DNA Polymeraseis 70-75°C. The recommended extension step is 1 min at72°C for PCR products up to 2 kb. For larger products, theextension time should be prolonged by 1 min/kb.Number of cyclesThe number of cycles may vary depending on the amount oftemplate DNA in the PCR mixture and the expected PCRproduct yield.If less than 10 copies of the template are present in thereaction, about 40 cycles are required. For higher templateamounts, 25-35 cycles are sufficient.Final extensionAfter the last cycle, it is recommended to incubate the PCRmixture at 72°C for additional 5-15 min to fill-in any possibleincomplete reaction products. If the PCR product will becloned into TA vectors (for instance, using Thermo ScientificInsTAclone PCR Cloning Kit (#K1213)), the final extensionstep may be prolonged to 30 min to ensure the highestefficiency of 3’-dA tailing of PCR product. If the PCR productwill be used for cloning using Thermo Scientific CloneJETPCR Cloning Kit (#K1231), the final extension step can beomitted.TroubleshootingFor troubleshooting please visit/onebioReferences1. Innis, M.A., et al., PCR Protocols and Applications: A LaboratoryManual, Academic, New York, 1989.2. Celeda, D., et al., PCR amplification and simultaneous digoxigeninincorporation of long DNA probes for fluorescence in situ hybridization,BioTechniques, 12, 98-102, 1992.3. Finckh, U., et al., Producing single-stranded DNA probes with theTaq DNA polymerase: a high yield protocol, BioTechniques, 10, 35-39,1991.4. Yu, H. et al., Cyanine dye dUTP analogs for enzymatic labeling ofDNA probes, Nucleic Acids Res., 22, 3226-3232, 1994.5. Innis, M.A., et al., DNA sequencing with Thermus aquaticus DNApolymerase and direct sequencing of polymerase chain reaction-amplified DNA, Proc. Natl. Acad. Sci. USA, 85, 9436-9440, 1988.6. Weyant, R.S., et al., Effect of ionic and nonionic detergents on theTaq polymerase, Biotechniques, 9, 309-308, 1990.7. Lundberg, K.S., et al., High-fidelity amplification using a thermostableDNA polymerase isolated from Pyrococcus furiosus, Gene, 108, 1-6,1991.CERTIFICATE OF ANALYSISEndodeoxyribonuclease AssayNo conversion of covalently closed circular DNA to nickedDNA was detected after incubation of 10 U of Taq DNAPolymerase with 1 µg of pUC19 DNA for 4 hours at 37°C.Exodeoxyribonuclease AssayNo degradation of DNA was observed after incubation of1 µg of lambda DNA/HindIII fragments with 10 U Taq DNAPolymerase for 4 hours at 37°C.Ribonuclease AssayNo contaminating RNase activity was detected afterincubation of 10 U of Taq DNA Polymerase with 1 µg of[3H]-RNA for 4 hours at 37°C.Functional AssayTaq DNA Polymerase was tested for amplification of 950 bpsingle copy gene from human genomic DNA and foramplification of cDNA.Quality authorized by: Jurgita ZilinskieneNOTICE TO PURCHASER:Use of this product is covered by US Patent No. 6,127,155. The purchaseof this product includes a limited, non-transferable immunity from suitunder the foregoing patent claims for using only this amount of product forthe purchaser’s own internal research. No right under any other patentclaim, no right to perform any patented method and no right to performcommercial services of any kind, including without limitation reporting theresults of purchaser's activities for a fee or other commercialconsideration, is conveyed expressly, by implication, or by estoppel. Thisproduct is for research use only. Diagnostic uses under Roche patentsrequire a separate license from Roche. Further information on purchasinglicenses may be obtained by contacting the Director of Licensing, AppliedBiosystems, 850 Lincoln Centre Drive, Foster City, California.PRODUCT USE LIMITATIONThis product is developed, designed and sold exclusively for researchpurposes and in vitro use only. The product was not tested for use indiagnostics or for drug development, nor is it suitable for administration tohumans or animals. Please refer to /onebio forMaterial Safety Data Sheet of the product.© 2012 Thermo Fisher Scientific, Inc. All rights reserved. Tween is aregistered trademark of ICI America, Inc. Nonidet is a trademark of Shell.All other trademarks are the property of Thermo Fisher Scientific Inc. and itssubsidiaries.。
热启动酶的原理及应用

热启动酶的原理及应用前言在现代生物科学研究中,热启动酶被广泛应用于DNA特异性放大技术,例如聚合酶链式反应(PCR)。
热启动酶是一种酶类蛋白,能够在高温条件下保持活性,具有特异性结合和扩增DNA的能力。
本文将介绍热启动酶的原理及其在科学研究和生物工程中的应用。
1. 热启动酶的原理热启动酶的原理基于一种天然存在的酶类蛋白,称为热稳定DNA多聚核苷酸激酶(Taq DNA polymerase)。
Taq DNA polymerase是从热温泉中的细菌Thermus aquaticus中分离得到的。
这种细菌栖息在温泉中,能够耐受高温环境。
因此,Taq DNA polymerase具备了在高温条件下保持稳定和具有DNA聚合能力的特点。
热启动酶通过添加特定的抑制剂和改变酶的活性形式,使其在低温下失去或降低DNA聚合酶活性,从而避免在PCR反应体系中的非特异性扩增。
当温度升高时,抑制剂被去除,酶活性被迅速激活,从而实现特异性DNA扩增。
2. 热启动酶的应用热启动酶作为PCR技术中常用的酶类蛋白,其应用非常广泛。
以下列举了一些热启动酶的主要应用领域:•分子生物学研究:热启动酶可用于DNA扩增、基因克隆、DNA测序等领域。
它的高温稳定性保证了PCR反应在高温环境下能够顺利进行,同时能够在DNA复制过程中起到高效率和高特异性的作用。
•医学诊断:PCR技术结合热启动酶的应用在医学诊断领域取得了重大突破。
例如,热启动酶可以检测疾病相关基因的突变,帮助诊断遗传性疾病、癌症等疾病。
•遗传学研究:热启动酶可以用于DNA指纹技术,通过扩增DNA重复序列,快速鉴定个体之间的遗传关系,对犯罪侦破、亲子鉴定等领域非常重要。
•生物工程:热启动酶在基因工程中常被用来扩增外源基因,从而用于遗传转化、重组蛋白表达等目的。
它的高特异性和高效率使得基因工程实验更加准确可靠。
•食品安全检测:热启动酶可以用于食品中病原菌和污染物的检测与鉴定,如大肠杆菌、沙门菌等。
Taq DNA 聚合酶 说明书

Taq DNA Polymerase Taq DNA Polymerase 是耐热的DNA 聚合酶,具有5’-3’ DNA 聚合酶活性和双链DNA 特异的5’-3’外切核酸酶活性,无3’-5’外切酶活性。
使用该产品扩增得到的PCR 产物的3’末端附有一个“A ”碱基,可直接用于TA 克隆。
■产品说明PCR 、TA 克隆。
■应用范围用活性化的大马哈鱼精子DNA 作为模板/引物,在74℃,30分钟内,将10 nmol 脱氧核苷酸摄入为酸不溶物质所需要的酶量定义为1个活性单位(U )。
■活性定义无核酸内切、外切酶活性,也无核酸污染,其酶纯度检测大于99%。
■品质保证产品内容Taq DNA Polymerase (5U/µl )2+10×Taq Reaction Buffer (Mg plus )10 mM dNTP 保存条件:-20 ℃. E coli 重组蛋白。
■来源20mM Tris-Cl (pH 8.0), 100mM KCl, 0.1mM EDTA, 1mM DTT, 0.5%Nonidet P-40, 0.5%Tween-20和50% 甘油。
■储存缓冲液10×Taq Reaction Buffer :100mM Tris-Cl(pH8.3@25℃), 500mM KCl, 15mM MgCl 。
2■反应缓冲液GeneCopoeia Inc.19520 Amaranth DriveGermantown, Maryland 20874USATel: 301-515-6982; 1-866-360-9531Fax: 301-515-6983Web: 产品套装编号: C0101A , 本套装包含以下产品:产品编号C01010A C01011A C10010C包装规格200µl1.25ml ×20.5mlTaq DNA 聚合酶生产经销商:能基因广州复能基因有限公司广州高新技术产业开发区广州科学城掬泉路3号广州国际企业孵化器D 区8楼 (邮编: 510663)技术热线: 020-******** 电子邮箱: support @ 网址:■基本反应条件体系:1、PCR 10× Reaction Buffer Primer1Primer210mM dNTP TemplateTaq DNA Polymerase ddH O22.5µl0.5µl1~100ng(质粒)10~1,000ng(基因组)0.2µl up to 25µl反应物组成体积终浓度l ×0.2~1µM 0.2~1µM0.2mM1U /Reaction条件:2、PCR 94℃94℃Tm -5℃72℃72℃4℃2 min 30 sec 30 sec 1kb/min 7 min hold30 cycles}1. PCR 反应条件应根据模板、目的片段大小、引物结构等具体条件不同,设定最佳反应条件。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Taq DNA Polymerase
简介:
Taq DNA Polymerase 是通过大肠杆菌表达纯化的重组酶,其基因来源于 Thermus aquaticus polymerase ,该蛋白分子量为 94 kDa ,具有 5′→ 3′DNA 聚合酶活性和 5′→ 3′外切核酸酶活性,无 3′→ 5′外切核酸酶活性,扩增得到的 PCR 产物 3′端附有一个“A ”碱基,因此可直接用于 T/A 克隆。
本产品具有延伸速度快、扩增效率高的特点,主要适用于 PCR 法扩增 DNA 片段、DNA 序列测定等实验。
组成:
单位定义:用活性化的大马哈鱼精子 DNA 作为模板 / 引物,在74℃,30 分钟内,将
10nmol 脱氧核苷酸掺入到酸性不溶物质所需的酶量定义为1个活性单位(U ),浓度:5U/ul
质控:经过多次柱纯化,SDS-PAGE 检测其纯度大于 99%;经检测无外源核酸酶活性;
PCR 方法检测无宿主残余DNA ;能有效地扩增人基因组中的单拷贝基因;室温存放一个月,无明显活性改变。
操作步骤(仅供参考):
以下举例为常规 PCR 反应体系和反应条件,实际操作中应根据模板、引物结构和目的片段大小不同进行相应的改进和优化。
(1)PCR 反应体系(50μl)
试剂
加入量 终浓度
10×T aq PCR Buffer μl 1× dNTP Mix ,2.5 mM each μl μM each Forward Primer ,10μM μl μM Reverse Primer ,10μM μl μM Template DNA <μg <1μg/reaction Taq DNA Polymerase(5U/μl) μl
RNase-Free Water
up to μl
编号 名称
NP0101
NP0101
Storage Taq DNA Polymerase(5U/μl) 500U
1000U
-20℃ 10×T aq PCR Buffer 1 ml 2×1ml -20℃
使用说明书
1份
注意:
a 、引物浓度请以终浓度作为设定范围的参考,扩增效率不高的情况下,可提高引物的浓度;发生非特异性反应时,可降低引物浓度。
b 、本产品的10×T aq PCR Buffer 中含镁离子(MgCl2 20mM),无需单独配制。
(2)PCR 反应条件
步骤 温度 时间 预变性 94℃ 2min
变性 94℃ 30s
退火 54-65℃ 30s 25-35 个循环 延伸 72℃ 30s
终延伸 72℃ 2min
注意事项:
1、一般退火温度比扩增引物的熔解温度Tm 低5℃,无法得到理想的扩增效率时,适当降低退火温度;发生非特异性反应时,提高退火温度,由此优化反应条件。
2、延伸时间应根据所扩增片段大小设定,本产品 Taq DNA Polymerase 的扩增效率为 1kb/30s 。
3可根据扩增产物的下游应用设定循环数。
如果循环次数太少,扩增量不足;如果循环次数太多,错配机率会增加,非特异性背景严重;在保证产物得率的前提下应尽量减少循环次数。
4、 避免反复冻融,否则效率会降低;10×T aq PCR Buffer 可以分装成小份使用。
5、为了您的安全和健康,请穿实验服并戴一次性手套操作。
有效期:Taq DNA Polymerase -20℃ 36个月有效;10×T aq PCR Buffer -20℃ 12个
月有效。
相关:
编号 名称
CS0001 ACK 红细胞裂解液(ACK Lysis Buffer) DF0111 中性福尔马林固定液(10%) DM0007 瑞氏-姬姆萨复合染色液 PE0080 Tris-HCl 缓冲液(1mol/L,pH6.8) PW0040
Western blot 一抗稀释液
TC1213
总胆固醇(TC)检测试剂盒(COD-PAP 单试剂比色法
)。
