基本遗传算法的C源程序。doc【精品毕业设计】(完整版)
GA-遗传算法-C#代码【精品毕业设计】(完整版)

using System;using System.IO;using System.Collections;using System.Collections.Generic;using System.Text;using ponentModel;using System.Data;using System.Data.OleDb;namespace ConsoleApplication1{public class Genetic_Algorithm{Random rand=new Random();int MaxTime;//最大运行时间int popsize;//种群数量int ChromosomeLength;//染色体长度double CrossRate;//交叉率double MutateRate;//变异率double[] f;//适应度值int[] selected;//定义selected数组,用于表示需要进行交叉操作的染色体序号double[] wheel;//轮盘int[,] pregeneration;//上一代int[,] nextgeneration;//下一代int[] Best;//定义当前最优解int convergence;//定义当前最优解的已持续代数int[,] timeconstrait;public Genetic_Algorithm(int populationsize, int chromolength)//GA--构造函数,变量初始化{rand = new Random(lisecond);MaxTime = 50;popsize=populationsize;ChromosomeLength = chromolength;CrossRate = 0.8;MutateRate = 0.2;f = new double[2*popsize];selected = new int[popsize];wheel = new double[popsize + 1];pregeneration = new int[popsize, ChromosomeLength];//当前的染色体种群nextgeneration = new int[popsize, ChromosomeLength];//下一代(子代)染色体种群Best = new int[ChromosomeLength];convergence = 1;timeconstrait = new int[20, 2] { { 2, 6 }, { 1, 2 }, { 3, 4 }, { 1, 4 }, { 4, 7 }, { 3, 5 }, { 2, 6 }, { 3, 5 }, { 1, 4 }, { 3, 7 }, { 5, 7 }, { 2, 7 }, { 2, 4 }, { 4, 5 }, { 2, 5 }, { 4, 6 }, { 3, 5 }, { 1, 4 }, { 1, 5 }, { 3, 6 } };}public void RunGA()//运行{int i;CreateFirstPop();//产生初始种群i = 0;bool quit = true;while (quit){for (; i < MaxTime; i++){Console.WriteLine("The {0}th Generation..........", i + 1);CalFitness(ref pregeneration, popsize);//计算适应值PrintResult();//输出每步的结果WheelSelect();//此步确定了selected[i]的值CreateNextGeneration();//产生子代,包括被选择为selected[i]的染色体的交叉,还有变异ProduceNext();}Console.WriteLine("Press 'q' to quit, press Enter to continue.....");if (Console.Read() == 'q'){quit = false;}else{MaxTime += 50;}}}void CreateFirstPop()//产生初始种群{Console.WriteLine("Creating first generation..........\n");int i,j,r;for(i=0;i<popsize;i++){for(j=0;j<ChromosomeLength;j++){r=rand.Next(1,11);pregeneration[i, j] = r;}}}void CreateNextGeneration()//产生下一代种群(经交叉、变异){int i;for (i = 0; i < popsize; i+=2){Crossover(selected[i], selected[i + 1], i, i + 1);//将序号为selected[i]和selected[i + 1]的染色体进行交叉,产生的子代放在pregeneration中i和i+1的位置}Mutation(ref nextgeneration);//变异}void CalFitness(ref int[,] curgeneration,int number)//计算适应度值的函数{for (int i = 0; i < number; i++){double fitness = 0;for (int j = 0; j < ChromosomeLength; j++){fitness += Math.Abs(curgeneration[i, j]-j-1);}f[i] = fitness;}}void FindMax(ref double[] f, out int max)//寻找数组中最大值{int i;max = 0;for (i = 1; i < popsize; i++){if (f[i] > f[max]){max = i;}}}void FindMin(ref double[] f, out int min)//寻找数组中最小值{int i;min = 0;for (i = 1; i < popsize; i++){if (f[i] < f[min]){min = i;}}}void WheelSelect() //轮盘选择popsize个染色体(可重复),并将序号放入selected[]中,作为交叉的染色体{int i,j ,r;double sum;wheel[0] = 0; sum = 0;for (i = 0; i < popsize; i++){sum += f[i];wheel[i + 1] = wheel[i] + f[i];}for (i = 0; i < popsize; i++){r = rand.Next((int)sum);for (j = 0; j < popsize; j++){if (r > wheel[j] && r < wheel[j + 1]){selected[i] = j;break;}}}}void Crossover(int p1, int p2, int c1, int c2)//交叉==>将序号为selected[i]和selected[i + 1](这里形参是p1,p2)的染色体进行交叉,产生的子代放在pregeneration中i和i+1(这里形参是c1,c2)的位置{double dr = rand.NextDouble();if (dr < CrossRate){int[] covering_code = new int[ChromosomeLength];for (int i = 0; i < ChromosomeLength; i++)covering_code[i] = rand.Next(0, 2);for (int i = 0; i < ChromosomeLength; i++){if (covering_code[i] == 0){nextgeneration[c1, i] = pregeneration[p1, i];nextgeneration[c2, i] = pregeneration[p2, i];}else{nextgeneration[c1, i] = pregeneration[p2, i];nextgeneration[c2, i] = pregeneration[p1, i];}}}else{for (int i = 0; i < ChromosomeLength; i++){nextgeneration[c1, i] = pregeneration[p1, i];nextgeneration[c2, i] = pregeneration[p2, i];}}}void Mutation(ref int[,] curgeneration)//变异{int is_not_mutation;double dr;for (int i = 0; i < popsize; i++){dr = rand.NextDouble();if (dr < MutateRate){for (int j = 0; j < ChromosomeLength; j++){is_not_mutation = rand.Next(0, 2);if (is_not_mutation == 1)curgeneration[i, j] = rand.Next(1, 11);}}}}void PrintResult()//计算每次迭代后种群中最优解及其适应度值,平均适应值 {int i,j;int min;double average;average = 0;for (i = 0; i < popsize; i++){average += f[i];}average = (double) average / popsize;Console.Write("Average profit is {0}\n", average);FindMin(ref f, out min);//计算稳定的次数for (j = 0; j < ChromosomeLength; j++){if (pregeneration[min, j] != Best[j]){convergence = 1;goto G2;}}convergence++;G2:for (j = 0; j < ChromosomeLength; j++){Best[j] = pregeneration[min, j];}//打印相关的数据Console.Write("染色体 ");for (j = 0; j < ChromosomeLength; j++){Console.Write(pregeneration[min, j] + ",");}Console.WriteLine("");Console.WriteLine("综合目标 {0} of individual ", f[min]);Console.WriteLine("已经稳定的代数 {0} of individual ",convergence);Console.WriteLine("");}void ProduceNext()//选择==>父代和子代中popsize个最优的解进入下一代{int[,] temgeneration=new int [2*popsize,ChromosomeLength];//定义临时种群,用来将父代和子代放在一起,进行选优//将父代放入临时种群for (int i = 0; i <= popsize - 1; i++){for (int j = 0; j <= ChromosomeLength - 1; j++){temgeneration[i, j] = pregeneration[i, j];}}//将子代放入临时种群for (int i = 0; i <= popsize - 1; i++){for (int j = 0; j <= ChromosomeLength - 1; j++){temgeneration[i + popsize, j] = nextgeneration[i, j];}}CalFitness(ref temgeneration, popsize * 2);//计算临时种群(父代和子代)的各染色体适应值int []tem=new int [ChromosomeLength];//定义临时染色体,用来染色体排序时的交换...//根据临时种群(父代和子代)的各染色体适应值,进行排序for (int i = 0; i < 2*popsize - 1; i++){for (int j = i + 1; j <= 2 * popsize - 1; j++){if (f[i] > f[j]){double tem_f = f[i];f[i] = f[j];f[j] = tem_f;for (int k = 0; k < ChromosomeLength; k++){tem[k] = temgeneration[i, k];temgeneration[i, k] = temgeneration[j, k];temgeneration[j, k] = tem[k];}}}}//取临时种群中前popsize个好的染色体作为下一代种群,并将子代变为父代for (int i = 0; i <= popsize - 1; i++){for (int j = 0; j <= ChromosomeLength - 1; j++){pregeneration[i, j] = temgeneration[i, j];}}}}class Program{static void Main(string[] args){int chromosomelength = 10;int populationsize = 300;int cycle = 5;Console.WriteLine("Press Enter to start running Genetic Algorithm");Console.ReadKey();Genetic_Algorithm GA = new Genetic_Algorithm(populationsize, chromosomelength); GA.RunGA();}}}。
遗传算法解释及代码(一看就懂)【精品毕业设计】(完整版)

遗传算法( GA , Genetic Algorithm ) ,也称进化算法。
遗传算法是受达尔文的进化论的启发,借鉴生物进化过程而提出的一种启发式搜索算法。
因此在介绍遗传算法前有必要简单的介绍生物进化知识。
一.进化论知识作为遗传算法生物背景的介绍,下面内容了解即可:种群(Population):生物的进化以群体的形式进行,这样的一个群体称为种群。
个体:组成种群的单个生物。
基因 ( Gene ) :一个遗传因子。
染色体 ( Chromosome ):包含一组的基因。
生存竞争,适者生存:对环境适应度高的、牛B的个体参与繁殖的机会比较多,后代就会越来越多。
适应度低的个体参与繁殖的机会比较少,后代就会越来越少。
遗传与变异:新个体会遗传父母双方各一部分的基因,同时有一定的概率发生基因变异。
简单说来就是:繁殖过程,会发生基因交叉( Crossover ) ,基因突变( Mutation ) ,适应度( Fitness )低的个体会被逐步淘汰,而适应度高的个体会越来越多。
那么经过N代的自然选择后,保存下来的个体都是适应度很高的,其中很可能包含史上产生的适应度最高的那个个体。
二.遗传算法思想借鉴生物进化论,遗传算法将要解决的问题模拟成一个生物进化的过程,通过复制、交叉、突变等操作产生下一代的解,并逐步淘汰掉适应度函数值低的解,增加适应度函数值高的解。
这样进化N代后就很有可能会进化出适应度函数值很高的个体。
举个例子,使用遗传算法解决“0-1背包问题”的思路:0-1背包的解可以编码为一串0-1字符串(0:不取,1:取);首先,随机产生M个0-1字符串,然后评价这些0-1字符串作为0-1背包问题的解的优劣;然后,随机选择一些字符串通过交叉、突变等操作产生下一代的M个字符串,而且较优的解被选中的概率要比较高。
这样经过G代的进化后就可能会产生出0-1背包问题的一个“近似最优解”。
编码:需要将问题的解编码成字符串的形式才能使用遗传算法。
遗传算法的C语言程序案例
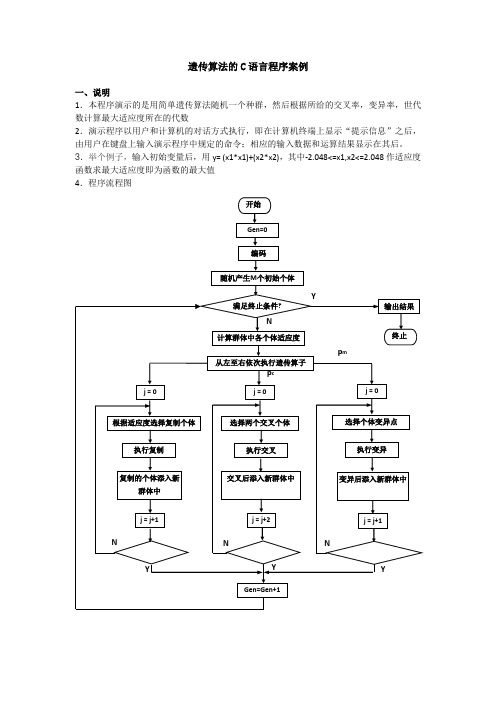
遗传算法的C语言程序案例一、说明1.本程序演示的是用简单遗传算法随机一个种群,然后根据所给的交叉率,变异率,世代数计算最大适应度所在的代数2.演示程序以用户和计算机的对话方式执行,即在计算机终端上显示“提示信息”之后,由用户在键盘上输入演示程序中规定的命令;相应的输入数据和运算结果显示在其后。
3.举个例子,输入初始变量后,用y= (x1*x1)+(x2*x2),其中-2.048<=x1,x2<=2.048作适应度函数求最大适应度即为函数的最大值4.程序流程图5.类型定义int popsize; //种群大小int maxgeneration; //最大世代数double pc; //交叉率double pm; //变异率struct individual{char chrom[chromlength+1];double value;double fitness; //适应度};int generation; //世代数int best_index;int worst_index;struct individual bestindividual; //最佳个体struct individual worstindividual; //最差个体struct individual currentbest;struct individual population[POPSIZE];3.函数声明void generateinitialpopulation();void generatenextpopulation();void evaluatepopulation();long decodechromosome(char *,int,int);void calculateobjectvalue();void calculatefitnessvalue();void findbestandworstindividual();void performevolution();void selectoperator();void crossoveroperator();void mutationoperator();void input();void outputtextreport();6.程序的各函数的简单算法说明如下:(1).void generateinitialpopulation ()和void input ()初始化种群和遗传算法参数。
(完整版)遗传算法简介及代码详解

遗传算法简述及代码详解声明:本文内容整理自网络,认为原作者同意转载,如有冒犯请联系我。
遗传算法基本内容遗传算法为群体优化算法,也就是从多个初始解开始进行优化,每个解称为一个染色体,各染色体之间通过竞争、合作、单独变异,不断进化。
遗传学与遗传算法中的基础术语比较染色体:又可以叫做基因型个体(individuals)群体/种群(population):一定数量的个体组成,及一定数量的染色体组成,群体中个体的数量叫做群体大小。
初始群体:若干染色体的集合,即解的规模,如30,50等,认为是随机选取的数据集合。
适应度(fitness):各个个体对环境的适应程度优化时先要将实际问题转换到遗传空间,就是把实际问题的解用染色体表示,称为编码,反过程为解码/译码,因为优化后要进行评价(此时得到的解是否较之前解优越),所以要返回问题空间,故要进行解码。
SGA采用二进制编码,染色体就是二进制位串,每一位可称为一个基因;如果直接生成二进制初始种群,则不必有编码过程,但要求解码时将染色体解码到问题可行域内。
遗传算法的准备工作:1) 数据转换操作,包括表现型到基因型的转换和基因型到表现型的转换。
前者是把求解空间中的参数转化成遗传空间中的染色体或者个体(encoding),后者是它的逆操作(decoding)2) 确定适应度计算函数,可以将个体值经过该函数转换为该个体的适应度,该适应度的高低要能充分反映该个体对于解得优秀程度。
非常重要的过程。
遗传算法基本过程为:1) 编码,创建初始群体2) 群体中个体适应度计算3) 评估适应度4) 根据适应度选择个体5) 被选择个体进行交叉繁殖6) 在繁殖的过程中引入变异机制7) 繁殖出新的群体,回到第二步实例一:(建议先看实例二)求 []30,0∈x 范围内的()210-=x y 的最小值1) 编码算法选择为"将x 转化为2进制的串",串的长度为5位(串的长度根据解的精度设 定,串长度越长解得精度越高)。
自然计算遗传算法【精品毕业设计】(完整版)

自然计算大作业一.二进制编码在遗传算法中,首先要将数据进行编码,这里采用二进制的方式进行编码。
第一步,我们根据题目的介绍可以得知该函数含有两个变量,以及各自的定义域。
在二进制编码中,我们首先要先计算它的编码长度。
计算公式如下: $${2^{{m_j} - 1}} < ({b_j} - {a_j})*precision \le {2^{{m_j}}} - 1$$其中precision为精度,如小数点后5位,则precision=10^5,mj为编码长度,${x_j} \in [{a_j},{b_j}]$二.二进制解码解码即编码的逆过程:$${x_j} = {a_j} + {\rm{decimal}}(substrin{g_j}) \times \frac{{{b_j} - {a_j}}}{{{2^{{m_j}}} - 1}}$$三.种群初始化编码完成后,开始对种群初始化,为了简便采用随机地方式进行初始化。
初始群体的生成:随机产生N个初始串结构数据,每个串结构数据称为一个个体,N个个体构成了一个群体。
GA以这N个串结构数据作为初始点开始进化。
def rand_init(self):for i in range(self.code_x1_length):self.code_x1 += str(random.randint(0, 1))for i in range(self.code_x2_length):self.code_x2 += str(random.randint(0, 1))四.适应度评估适应度表明个体或解的优劣性。
不同的问题,适应度函数的定义方式也不同。
def decoding(self, code_x1, code_x2):self.x1 = self.bounds[0][0] + int(code_x1, 2) * (self.bounds[0][1] - self.bounds[0][0]) / (2 ** self.code_x1_length - 1)self.x2 = self.bounds[1][0] + int(code_x2, 2) * (self.bounds[1][1] - self.bounds[1][0]) / (2 ** self.code_x2_length - 1)五.选择选择的目的是为了从当前群体中选出优良的个体,使它们有机会作为父代为下一代繁殖子孙。
(完整版)遗传算法c语言代码

srand((unsigned)time(NULL));
for(i=0;i<num;i++)
{
bianyip[i]=(rand()%100);
bianyip[i]/=100;
}
//确定可以变异的染色体
t=0;
for(i=0;i<num;i++)
{
if(bianyip[i]<pm)
printf("\n******************是否想再一次计算(y or n)***********************\n");
fflush(stdin);
scanf("%c",&choice);
}while(choice=='y');
return 0;
}
{
flag=0;
break;
}
}
if(flag)
{
group[i].city[j]=t;
j++;
}
}
}
printf("************初始种群如下****************\n");
for(i=0;i<num;i++)
{
for(j=0;j<cities;j++)
printf("%4d",group[i].city[j]);
{
group[i].p=1-(double)group[i].adapt/(double)biggestsum;
biggestp+=group[i].p;
遗传算法程序实现
遗传算法程序实现#include <stdlib.h>#include <stdio.h>#include <time.h>#include <math.h>#include "GA.h"unsigned seed=1;//产生一个不大于x的随机数double crandom(int x){seed++;srand((unsigned)(seed*time(NULL)));return(rand()%x);}//产生一个0~1的随机数double random(){seed++;srand((unsigned)(seed*time(NULL)));return (double)rand()/(double)RAND_MAX;}//产生(0.1,0.2,...,1)之间的一个随机数double crandom(void){double t;t=random();if((10*t-floor(10*t))>=0.5) return (floor(10*t)+1)/10;elsereturn(floor(10*t)/10);}//初始化种群void Initial_group(double group[M][N_para], double up_limit[N_para],double low_limit[N_para]){ double beta;int i,j;for (i=0;i<M;i++)for (j=0;j<N_para;j++){beta=crandom();group[i][j]=low_limit[j]+beta*(up_limit[j]-low_limit[j]);}}}//加载实时测试数据void Load_data(double realvalue[][4]){FILE *fp;float x;//changedif((fp=fopen("realtime\\Realdata.txt","r"))==NULL){printf("cannot open the file Realvalue.txt\n");exit(0);}for(int i=0;i<N_hit+N_Begin;i++){for(int j=0;j<4;j++)//修改了{fscanf(fp,"%f ",&x);if(i>=N_Begin)realvalue[i-N_Begin][j]=(double)x;//修改了}}fclose(fp);}//计算适应度值void Fit_calculate(double group[][N_para],double realvalue[][4],double fit_degree[],int n){int i;//double Mcar,m1,r1,r2,L,J1,J2,c1,c2,b;double mcar,m1,l1,J1,b,g,f1,eks,eksd,theta1,theta1d,h;double x[4],x_temp[4],k[4][4],lefA[4];double err,A11,A12,A21,A22,B1,B2,B3,B4,C11,C12,C21,C22,D2;g=9.8;h=0.005;for (i=0;i<n;i++){mcar=group[i][0];m1=group[i][1];l1=group[i][2];J1=group[i][3];b=group[i][4];f1=group[i][5];//printf("mcar=%6.3f,m1=%6.3f,l1=%6.3f,J1=%6.3f,b=%6.3,f1=%6.3\n,",mcar,m1, l1,J1,b,f1);err=0;for (int j=0;j<4;j++) //新加的{x[j]=realvalue[0][j];x_temp[j]=realvalue[0][j];}/*for (int j=0;j<4;j++)//修改了{x[j]=Initivalue[j];x_temp[j]=Initivalue[j];}*/for (int q=0;q<N_hit;q++){eks=x[0];eksd=x[1];theta1=x[2];theta1d=x[3];while(fabs(x[2])>(2*Pi)){printf("Error");printf(" q=%d",q);getchar();}A11=mcar+m1;A12=-m1*l1*cos(theta1);A22=m1*l1*l1+J1;A21=A12;C11=b;C12=m1*l1*sin(theta1)*theta1d;C21=0;C22=f1;D2=-m1*g*l1*sin(theta1);B1=eksd;B2=-C11*eksd-C12*theta1d;B3=theta1d;B4=-C22*theta1d-D2;/*%A=[1 0 0 0;% 0 A11 0 A12 ;% 0 0 1 0 ;% 0 A21 0 A22];%lefA = inv(A)*B;%A11x+A12y=B(2)%A21x+A22y=B(4)*/lefA[0]=B1;lefA[1]=(B2*A22-B4*A12)/(A22*A11-A21*A12);lefA[2]=B3;lefA[3]=(B2*A21-B4*A11)/(A12*A21-A22*A11);for (j=0;j<4;j++) //龙格-库塔法{k[j][0]=lefA[0];k[j][1]=lefA[1];k[j][2]=lefA[2];k[j][3]=lefA[3];for (int p=0;p<4;p++){if(j==0) x[p]=x_temp[p]+h/2*k[j][p];if(j==1) x[p]=x_temp[p]+h/2*k[j][p];if(j==2) x[p]=x_temp[p]+h*k[j][p];}}for(j=0;j<4;j++){x[j]=x_temp[j]+(k[0][j]+2*k[1][j]+2*k[2][j]+k[3][j])*h/6;x_temp[j]=x[j];}//getchar();//test uerr=err+pow((x[2]-realvalue[q][2]),2);//err=err+80*pow(x[1],2)+80*pow(x[3],2)+pow(u,2);}//end of for qfit_degree[i]=2+log(2/err)/log(200);///log(2);//9改成200了}}//种群排序void sort(double order_fit[],int index_fit[],int n){int i,j;int t;double temp;for (i=0;i<n;i++)index_fit[i]=i;for (j=1;j<=n-1;j++)for(i=1;i<=n-j;i++){if(order_fit[i]<order_fit[i-1]){temp=order_fit[i];order_fit[i]=order_fit[i-1];order_fit[i-1]=temp;t=index_fit[i];index_fit[i]=index_fit[i-1];index_fit[i-1]=t;}}}//反馈式突变void Feedback_mut(double group[][N_para],double up_limit[],double low_limit[],int index_fit[],double fit_opitimal,int nb){// printf("\nFeedback1 ok\n");double beta;int i,j,best,worse;int n=M;best=index_fit[M-1];for (i=0;i<n*0.9;i++){beta=crandom();worse=index_fit[i];if(nb>50||fit_opitimal<Sigma){if(nb>50) nb=0;for(j=0;j<N_para;j++)group[worse][j]=low_limit[j]+beta*(up_limit[j]-low_limit[j]);}elsefor(j=0;j<N_para;j++){group[worse][j]=(Nu+beta*Vi)*group[best][j];if (group[worse][j]<low_limit[j]||group[worse][j]>up_limit[j])group[worse][j]=group[best][j];}}//printf("\nFeedback 2 ok\n");}//选择操作void Slected(double cross[],double group[][N_para],double fit_degree[],double fit_sum){int j,i=0;double beta,slectvalue[M];beta=crandom();slectvalue[0]=fit_degree[0]/fit_sum;for(i=1;i<M;i++)slectvalue[i]=slectvalue[i-1]+fit_degree[i]/fit_sum;for(i=1;i<M;i++){if(slectvalue[i-1]<beta&&slectvalue[i]>beta)break;}for(j=0;j<N_para;j++)cross[j]=group[i][j];}//计算两个染色体的海明距离int haiming(double crossa[],double crossb[],int n){int num=0;int i;for (i=0;i<n;i++)if(crossa[i]!=crossb[i]) num++;return num;}//交叉操作void Cro_operation(double crossa[],double crossb[],double up_limit[],double low_limit[],int D_hm){int i,j,cro_point,d,zeta=0,sum=0;double cro_temp,beta;double x_temp[N_para];double y_temp[N_para];int rec[6]={0,0,0,0,0,0};if(D_hm==1){for(i=0;i<N_para;i++){if(crossa[i]!=crossb[i])break;}//cro_temp=crossa[i];//crossa[i]=crossb[i];//crossb[i]=cro_tempcro_point=i;}else{//zeta=(int)(crandom(4)+1);for(int ii=0;ii<N_para;ii++){for (int k=0;k<N_para;k++){x_temp[k]=0;y_temp[k]=0;}for(i=0;i<=ii;i++){x_temp[i]=crossa[i];y_temp[i]=crossb[i];}d=haiming(x_temp,y_temp,N_para);if(d>0&&d<D_hm){zeta++;rec[ii]=1;}//break;//>=改成<???}ii=(int)(crandom(zeta)+1);sum=0;for(i=0;sum<ii;i++){sum=sum+rec[i];cro_point=i;;}}//End of elsefor (j=0;j<cro_point;j++){cro_temp=crossa[j];crossa[j]=crossb[j];crossb[j]=cro_temp;}//cro_point--;//改了beta=crandom();cro_temp=(crossa[cro_point]<=crossb[cro_point])?crossa[cro_point]:crossb[cro_point];crossa[cro_point]=cro_temp+beta*fabs(crossa[cro_point]-crossb[cro_point]);beta=crandom();crossb[cro_point]=low_limit[cro_point]+beta*(up_limit[cro_point]-low_limit[cro_poi nt]);}//正交实验产生一个较优染色体void Cro_opiti(double crossa[N_para],double crossb[N_para],double cro_result[N_para],double realvalue[][4])//改了{double matrix[8][6]={//四因素两水平正交试验表1,1,1,1,1,1,1,1,1,2,2,2,1,2,2,1,1,2,1,2,2,2,2,1,2,1,2,1,2,1,2,1,2,2,1,2,2,2,1,1,2,2,2,2,1,2,1,1};int i,j;double fit_temp[8];double kx[N_para]={0,0,0,0,0,0};//changedouble ky[N_para]={0,0,0,0,0,0};//changefor(i=0;i<8;i++)for(j=0;j<N_para;j++){if(matrix[i][j]==1)matrix[i][j]=crossa[j];elsematrix[i][j]=crossb[j];}Fit_calculate(matrix,realvalue,fit_temp,8);for(i=0;i<8;i++)// 改了{for(j=0;j<N_para;j++){if(matrix[i][j]==crossa[j])kx[j]=kx[j]+fit_temp[i];if(matrix[i][j]==crossb[j])ky[j]=ky[j]+fit_temp[i];}}for(j=0;j<N_para;j++){if(kx[j]>ky[j])cro_result[j]=crossa[j];elsecro_result[j]=crossb[j];}}//变异操作void mut_operation(double cro_result[N_para],double up_limit[], double low_limit[]) {int mut_point1,mut_point2;double beta,x1,x2;do{mut_point1=(int)crandom(6);//改了13mut_point2=(int)crandom(6);//改了13}while(mut_point1==mut_point2);x1=(cro_result[mut_point1]-low_limit[mut_point1])/(up_limit[mut_point1]-low_limit [mut_point1]);x2=(cro_result[mut_point2]-low_limit[mut_point2])/(up_limit[mut_point2]-low_limit [mut_point2]);beta=crandom();if(beta<Pm){beta=crandom();cro_result[mut_point1]=(1-beta)*cro_result[mut_point1]+beta*low_limit[mut_point 1]+x2*(up_limit[mut_point1]-cro_result[mut_point1]);cro_result[mut_point2]=(1-beta)*cro_result[mut_point2]+beta*low_limit[mut_point 2]+x1*(up_limit[mut_point2]-cro_result[mut_point2]);}}//产生新一代种群void New_group(double group_temp[][N_para],double group[][N_para],double newfit_degree[],double fit_degree[]){int i,j;int temp;int index[M+Ncross];sort(newfit_degree,index,M+Ncross);for(i=0;i<M;i++){temp=index[Ncross+i];for(j=0;j<N_para;j++){group[i][j]=group_temp[temp][j];}fit_degree[i]=newfit_degree[Ncross+i];}}//种群最优个体,适应度值数据存盘void Data_record(double group[][N_para],double fit_degree[],double fit_population,int index_fit[]){FILE *fp;if((fp=fopen("Result\\Result.txt","a"))==NULL){printf("cannot open the fiel Result.txt\n");exit(0);}int nbest=index_fit[M-1];for(int i=0;i<N_para;i++)fprintf(fp,"%6.5f ",group[nbest][i]);printf("\npara=%6.5f %6.5f %6.5f %6.5f %6.5f %6.5f\n",group[nbest][0],group[n best][1],group[nbest][2],group[nbest][3],group[nbest][4],group[nbest][5]);//家的fprintf(fp,"%6.5f %6.5f\n",fit_degree[M-1],fit_population);fclose(fp);。
遗传算法c源程序
{
for(j=0;j<chromsize;j++)
{
oldpop[i].chrom[j]=0;
if(j==(chromsize-1))
{
stop=lchrom-(j*(8*sizeof(unsigned)));
#include <math.h>
#include <iostream.h>
#include <iomanip.h>
/////////////////////////////////////////////////
//静态变量定义(产生随机数专用)
static int inextp,iff,inext;
{
unsigned mask=1;
unsigned bitpos;
unsigned tp;
double pow(),bitpow;
int j,k,stop;
critter->varible=0.0;
for(k=0;k<chromsize;k++)
{
if(k==(chromsize-1))
};
/////////////////////////////////////////////////////////
struct individual oldpop[20]; //当前代种群
struct individual newpop[20]; //新一代种群
struct bestever bestfit; //最佳个体
遗传算法 c语言代码
以下是一个简单的遗传算法的C语言代码示例:c#include <stdio.h>#include <stdlib.h>#include <time.h>#include <math.h>#define POPULATION_SIZE 100#define GENE_LENGTH 10#define MAX_GENERATIONS 1000#define MUTATION_RATE 0.01#define CROSSOVER_RATE 0.8typedef struct Individual {char genes[GENE_LENGTH];double fitness;} Individual;double calculate_fitness(Individual* individual) {// 计算适应度函数,这里使用简单的二进制字符串中1的个数作为适应度 int count = 0;for (int i = 0; i < GENE_LENGTH; i++) {if (individual->genes[i] == '1') {count++;}}return count;}void initialize_population(Individual* population) {// 初始化种群for (int i = 0; i < POPULATION_SIZE; i++) {for (int j = 0; j < GENE_LENGTH; j++) {population[i].genes[j] = rand() % 2 ? '0' : '1';}population[i].fitness = calculate_fitness(&population[i]); }}void selection(Individual* population, Individual* parents) {// 选择操作,采用轮盘赌算法选择两个父代个体double total_fitness = 0;for (int i = 0; i < POPULATION_SIZE; i++) {total_fitness += population[i].fitness;}double rand1 = rand() / (double)RAND_MAX * total_fitness;double rand2 = rand() / (double)RAND_MAX * total_fitness;double cumulative_fitness = 0;int parent1_index = -1, parent2_index = -1;for (int i = 0; i < POPULATION_SIZE; i++) {cumulative_fitness += population[i].fitness;if (rand1 < cumulative_fitness && parent1_index == -1) {parent1_index = i;}if (rand2 < cumulative_fitness && parent2_index == -1) {parent2_index = i;}}parents[0] = population[parent1_index];parents[1] = population[parent2_index];}void crossover(Individual* parents, Individual* offspring) {// 交叉操作,采用单点交叉算法生成两个子代个体int crossover_point = rand() % GENE_LENGTH;for (int i = 0; i < crossover_point; i++) {offspring[0].genes[i] = parents[0].genes[i];offspring[1].genes[i] = parents[1].genes[i];}for (int i = crossover_point; i < GENE_LENGTH; i++) {offspring[0].genes[i] = parents[1].genes[i];offspring[1].genes[i] = parents[0].genes[i];}offspring[0].fitness = calculate_fitness(&offspring[0]);offspring[1].fitness = calculate_fitness(&offspring[1]);}void mutation(Individual* individual) {// 变异操作,以一定概率翻转基因位上的值for (int i = 0; i < GENE_LENGTH; i++) {if (rand() / (double)RAND_MAX < MUTATION_RATE) {individual->genes[i] = individual->genes[i] == '0' ? '1' : '0'; }}individual->fitness = calculate_fitness(individual);}void replace(Individual* population, Individual* offspring) {// 替换操作,将两个子代个体中适应度更高的一个替换掉种群中适应度最低的一个个体int worst_index = -1;double worst_fitness = INFINITY;for (int i = 0; i < POPULATION_SIZE; i++) {if (population[i].fitness < worst_fitness) {worst_index = i;worst_fitness = population[i].fitness;}}if (offspring[0].fitness > worst_fitness || offspring[1].fitness > worst_fitness) {if (offspring[0].fitness > offspring[1].fitness) {population[worst_index] = offspring[0];} else {population[worst_index] = offspring[1];}}}。
一个简单实用地遗传算法c程序
一个简单实用的遗传算法c程序(转载)c++ 2009-07-28 23:09:03 阅读418 评论0 字号:大中小这是一个非常简单的遗传算法源代码,是由Denis Cormier (North Carolina State University)开发的,Sita S.Raghavan (University of North Carolina at Charlotte)修正。
代码保证尽可能少,实际上也不必查错。
对一特定的应用修正此代码,用户只需改变常数的定义并且定义“评价函数”即可。
注意代码的设计是求最大值,其中的目标函数只能取正值;且函数值和个体的适应值之间没有区别。
该系统使用比率选择、精华模型、单点杂交和均匀变异。
如果用Gaussian变异替换均匀变异,可能得到更好的效果。
代码没有任何图形,甚至也没有屏幕输出,主要是保证在平台之间的高可移植性。
读者可以从,目录 coe/evol中的文件prog.c中获得。
要求输入的文件应该命名为‘gadata.txt’;系统产生的输出文件为‘galog.txt’。
输入的文件由几行组成:数目对应于变量数。
且每一行提供次序——对应于变量的上下界。
如第一行为第一个变量提供上下界,第二行为第二个变量提供上下界,等等。
/************************************************************************** //* This is a simple genetic algorithm implementation where the *//* evaluation function takes positive values only and the *//* fitness of an individual is the same as the value of the *//* objective function *//************************************************************************** /#include <stdio.h>#include <stdlib.h>#include <math.h>/* Change any of these parameters to match your needs */#define POPSIZE 50 /* population size */#define MAXGENS 1000 /* max. number of generations */#define NVARS 3 /* no. of problem variables */#define PXOVER 0.8 /* probability of crossover */#define PMUTATION 0.15 /* probability of mutation */#define TRUE 1#define FALSE 0int generation; /* current generation no. */int cur_best; /* best individual */FILE *galog; /* an output file */struct genotype /* genotype (GT), a member of the population */{double gene[NVARS]; /* a string of variables一个变量字符串 */ double fitness; /* GT's fitness适应度 */double upper[NVARS]; /* GT's variables upper bound 变量的上限*/ double lower[NVARS]; /* GT's variables lower bound变量的下限 */ double rfitness; /* relative fitness 相对适应度*/double cfitness; /* cumulative fitness 累计适应度*/};struct genotype population[POPSIZE+1]; /* population */struct genotype newpopulation[POPSIZE+1]; /* new population; */ /* replaces the *//* old generation *//* Declaration of procedures used by this genetic algorithm */ void initialize(void);double randval(double, double);void evaluate(void);void keep_the_best(void);void elitist(void);void select(void);void crossover(void);void Xover(int,int);void swap(double *, double *);void mutate(void);void report(void);/***************************************************************/ /* Initialization function: Initializes the values of genes */ /* within the variables bounds. It also initializes (to zero) */ /* all fitness values for each member of the population. It */ /* reads upper and lower bounds of each variable from the *//* input file `gadata.txt'. It randomly generates values */ /* between these bounds for each gene of each genotype in the */ /* population. The format of the input file `gadata.txt' is */ /* var1_lower_bound var1_upper bound */ /* var2_lower_bound var2_upper bound ... */ /***************************************************************/ void initialize(void){FILE *infile;int i, j;double lbound, ubound;if ((infile = fopen("gadata.txt","r"))==NULL){fprintf(galog,"\nCannot open input file!\n");exit(1);}/* initialize variables within the bounds */for (i = 0; i < NVARS; i++){fscanf(infile, "%lf",&lbound);fscanf(infile, "%lf",&ubound);for (j = 0; j < POPSIZE; j++){population[j].fitness = 0;population[j].rfitness = 0;population[j].cfitness = 0;population[j].lower[i] = lbound;population[j].upper[i]= ubound;population[j].gene[i] = randval(population[j].lower[i], population[j].upper[i]);}}fclose(infile);}/***********************************************************//* Random value generator: Generates a value within bounds *//***********************************************************/ double randval(double low, double high){double val;val = ((double)(rand()%1000)/1000.0)*(high - low) + low;return(val);}/*************************************************************/ /* Evaluation function: This takes a user defined function. */ /* Each time this is changed, the code has to be recompiled. */ /* The current function is: x[1]^2-x[1]*x[2]+x[3] */ /*************************************************************/ void evaluate(void){int mem;int i;double x[NVARS+1];for (mem = 0; mem < POPSIZE; mem++){for (i = 0; i < NVARS; i++)x[i+1] = population[mem].gene[i];population[mem].fitness = (x[1]*x[1]) - (x[1]*x[2]) + x[3]; }}/***************************************************************/ /* Keep_the_best function: This function keeps track of the *//* best member of the population. Note that the last entry in *//* the array Population holds a copy of the best individual *//***************************************************************/ void keep_the_best(){int mem;int i;cur_best = 0; /* stores the index of the best individual */for (mem = 0; mem < POPSIZE; mem++){if (population[mem].fitness > population[POPSIZE].fitness){cur_best = mem;population[POPSIZE].fitness = population[mem].fitness;}}/* once the best member in the population is found, copy the genes */ for (i = 0; i < NVARS; i++)population[POPSIZE].gene[i] = population[cur_best].gene[i];}/****************************************************************//* Elitist function: The best member of the previous generation */ /* is stored as the last in the array. If the best member of */ /* the current generation is worse then the best member of the */ /* previous generation, the latter one would replace the worst */ /* member of the current population */ /****************************************************************/ void elitist(){int i;double best, worst; /* best and worst fitness values */ int best_mem, worst_mem; /* indexes of the best and worst member */ best = population[0].fitness;worst = population[0].fitness;for (i = 0; i < POPSIZE - 1; ++i){if(population[i].fitness > population[i+1].fitness){if (population[i].fitness >= best){best = population[i].fitness;best_mem = i;}if (population[i+1].fitness <= worst) {worst = population[i+1].fitness; worst_mem = i + 1;}}else{if (population[i].fitness <= worst){worst = population[i].fitness; worst_mem = i;}if (population[i+1].fitness >= best) {best = population[i+1].fitness; best_mem = i + 1;}}}/* if best individual from the new population is better than *//* the best individual from the previous population, then *//* copy the best from the new population; else replace the *//* worst individual from the current population with the *//* best one from the previous generation */if (best >= population[POPSIZE].fitness){for (i = 0; i < NVARS; i++)population[POPSIZE].gene[i] = population[best_mem].gene[i]; population[POPSIZE].fitness = population[best_mem].fitness;}else{for (i = 0; i < NVARS; i++)population[worst_mem].gene[i] = population[POPSIZE].gene[i]; population[worst_mem].fitness = population[POPSIZE].fitness;}}/**************************************************************//* Selection function: Standard proportional selection for *//* maximization problems incorporating elitist model - makes *//* sure that the best member survives */ /**************************************************************/ void select(void){int mem, i, j, k;double sum = 0;double p;/* find total fitness of the population */for (mem = 0; mem < POPSIZE; mem++){sum += population[mem].fitness;}/* calculate relative fitness */for (mem = 0; mem < POPSIZE; mem++){population[mem].rfitness = population[mem].fitness/sum; }population[0].cfitness = population[0].rfitness;/* calculate cumulative fitness */for (mem = 1; mem < POPSIZE; mem++){population[mem].cfitness = population[mem-1].cfitness + population[mem].rfitness;}/* finally select survivors using cumulative fitness. */for (i = 0; i < POPSIZE; i++){p = rand()%1000/1000.0;if (p < population[0].cfitness)newpopulation[i] = population[0];else{for (j = 0; j < POPSIZE;j++)if (p >= population[j].cfitness &&p<population[j+1].cfitness)newpopulation[i] = population[j+1];}}/* once a new population is created, copy it back */for (i = 0; i < POPSIZE; i++)population[i] = newpopulation[i];}/***************************************************************/ /* Crossover selection: selects two parents that take part in */ /* the crossover. Implements a single point crossover */ /***************************************************************/ void crossover(void){int i, mem, one;int first = 0; /* count of the number of members chosen */ double x;for (mem = 0; mem < POPSIZE; ++mem){x = rand()%1000/1000.0;if (x < PXOVER){++first;if (first % 2 == 0)Xover(one, mem);elseone = mem;}}}/**************************************************************/ /* Crossover: performs crossover of the two selected parents. */ /**************************************************************/ void Xover(int one, int two){int i;int point; /* crossover point *//* select crossover point */if(NVARS > 1){if(NVARS == 2)point = 1;elsepoint = (rand() % (NVARS - 1)) + 1;for (i = 0; i < point; i++)swap(&population[one].gene[i], &population[two].gene[i]); }}/*************************************************************/ /* Swap: A swap procedure that helps in swapping 2 variables *//*************************************************************/ void swap(double *x, double *y){double temp;temp = *x;*x = *y;*y = temp;}/**************************************************************/ /* Mutation: Random uniform mutation. A variable selected for */ /* mutation is replaced by a random value between lower and */ /* upper bounds of this variable */ /**************************************************************/ void mutate(void){int i, j;double lbound, hbound;double x;for (i = 0; i < POPSIZE; i++)for (j = 0; j < NVARS; j++){x = rand()%1000/1000.0;if (x < PMUTATION){/* find the bounds on the variable to be mutated */ lbound = population[i].lower[j];hbound = population[i].upper[j];population[i].gene[j] = randval(lbound, hbound);}}}/***************************************************************//* Report function: Reports progress of the simulation. Data *//* dumped into the output file are separated by commas *//***************************************************************/void report(void){int i;double best_val; /* best population fitness */double avg; /* avg population fitness */double stddev; /* std. deviation of population fitness */ double sum_square; /* sum of square for std. calc */double square_sum; /* square of sum for std. calc */ double sum; /* total population fitness */sum = 0.0;sum_square = 0.0;for (i = 0; i < POPSIZE; i++){sum += population[i].fitness;sum_square += population[i].fitness * population[i].fitness; }avg = sum/(double)POPSIZE;square_sum = avg * avg * POPSIZE;stddev = sqrt((sum_square - square_sum)/(POPSIZE - 1));best_val = population[POPSIZE].fitness;fprintf(galog, "\n%5d, %6.3f, %6.3f, %6.3f \n\n", generation, best_val, avg, stddev);}/**************************************************************/ /* Main function: Each generation involves selecting the best */ /* members, performing crossover & mutation and then */ /* evaluating the resulting population, until the terminating */ /* condition is satisfied *//**************************************************************/ void main(void){int i;if ((galog = fopen("galog.txt","w"))==NULL){exit(1);}generation = 0;fprintf(galog, "\n generation best average standard \n"); fprintf(galog, " number value fitness deviation \n"); initialize();evaluate();keep_the_best();while(generation<MAXGENS){generation++;select();crossover();mutate();report();evaluate();elitist();}fprintf(galog,"\n\n Simulation completed\n");fprintf(galog,"\n Best member: \n");for (i = 0; i < NVARS; i++){fprintf (galog,"\n var(%d) = %3.3f",i,population[POPSIZE].gene[i]);}fprintf(galog,"\n\n Best fitness = %3.3f",population[POPSIZE].fitness);fclose(galog);printf("Success\n");}/***************************************************************/链接库文件?这个简单一般的第三方库文件有2种提供方式 1.lib静态库,这样必须在工程设置里面添加。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
/********************************************************** ********/
/* 基于基本遗传算法的函数最优化SGA.C */
/* A Function Optimizer using Simple Genetic Algorithm */
/* developed from the Pascal SGA code presented by David E.Goldberg */
//********************************************************** ********/
#include
#include
#include
#include "graph.c"
/* 全局变量*/
struct individual /* 个体*/
{
unsigned *chrom; /* 染色体*/
double fitness; /* 个体适应度*/
double varible; /* 个体对应的变量值*/
int xsite; /* 交叉位置*/
int parent[2]; /* 父个体*/
int *utility; /* 特定数据指针变量*/
};
struct bestever /* 最佳个体*/
{
unsigned *chrom; /* 最佳个体染色体*/
double fitness; /* 最佳个体适应度*/
double varible; /* 最佳个体对应的变量值*/
int generation; /* 最佳个体生成代*/
};
struct individual *oldpop; /* 当前代种群*/ struct individual *newpop; /* 新一代种群*/ struct bestever bestfit; /* 最佳个体*/
double sumfitness; /* 种群中个体适应度累计*/ double max; /* 种群中个体最大适应度*/ double avg; /* 种群中个体平均适应度*/ double min; /* 种群中个体最小适应度*/
float pcross; /* 交叉概率*/
float pmutation; /* 变异概率*/
int popsize; /* 种群大小*/
int lchrom; /* 染色体长度*/
int chromsize; /* 存储一染色体所需字节数*/ int gen; /* 当前世代数*/
int maxgen; /* 最大世代数*/
int run; /* 当前运行次数*/
int maxruns; /* 总运行次数*/
int printstrings; /* 输出染色体编码的判断,0 -- 不输出, 1 -- 输出*/ int nmutation; /* 当前代变异发生次数*/
int ncross; /* 当前代交叉发生次数*/
/* 随机数发生器使用的静态变量*/
static double oldrand[55];
static int jrand;
static double rndx2;
static int rndcalcflag;
/* 输出文件指针*/
FILE *outfp ;
/* 函数定义*/
void advance_random();
int flip(float);rnd(int, int);
void randomize();
double randomnormaldeviate();
float randomperc(),rndreal(float,float);
void warmup_random(float);
void initialize(),initdata(),initpop();
void initreport(),generation(),initmalloc();
void freeall(),nomemory(char *),report();
void writepop(),writechrom(unsigned *);
void preselect();
void statistics(struct individual *);
void title(),repchar (FILE *,char *,int);
void skip(FILE *,int);
int select();
void objfunc(struct individual *);
int crossover (unsigned *, unsigned *, unsigned *, unsigned *); void mutation(unsigned *);
void initialize() /* 遗传算法初始化*/
{
/* 键盘输入遗传算法参数*/
initdata();
/* 确定染色体的字节长度*/
chromsize = (lchrom/(8*sizeof(unsigned)));
if(lchrom%(8*sizeof(unsigned))) chromsize++;
/*分配给全局数据结构空间*/
initmalloc();
/* 初始化随机数发生器*/
randomize();
/* 初始化全局计数变量和一些数值*/ nmutation = 0;
ncross = 0;
bestfit.fitness = 0.0;
bestfit.generation = 0;
/* 初始化种群,并统计计算结果*/ initpop();
statistics(oldpop);
initreport();
}
void initdata() /* 遗传算法参数输入*/
{
char answer[2];
setcolor(9);
disp_hz16("种群大小(20-100):",100,150,20); gscanf(320,150,9,15,4,"%d", &popsize);
if((popsize%2) != 0)
{
fprintf(outfp, "种群大小已设置为偶数\n"); popsize++;
};。
