分子标记辅助选择
14分子标记辅助选择育种

A Chromosome Map of Tomato
1、作图群体的建立 (1)亲本的选配 亲本选择的原则 亲本间的DNA多态性丰富; 亲本纯度高; 杂交后代可育。
(2)群体的类型
P1
×
P2
F2群体
F1
F2
BC1群体
重组近交系群体 (recombinant inbred lines ,RIL ) RIL群体是杂交后代经过多代 自交而产生的一种作图群体, 通常从F2代开始,采用单粒的 方法建立。常自交6-7代。
CAPs标记的优点 ①引物与限制酶组合非常多 ② CAPS标记呈共显性 ③所需DNA量极少 ④结果稳定可靠 ⑤操作简便、快捷
7、STS(Sequence-tagged Sites) 序列标签位点 简称序标位 指基因组中长度为200—500bp,且核苷酸顺序已知 的单拷贝序列,可采用PCR技术将其专一扩增出来。 RFLP单拷贝的探针序列 引物来源 微卫星序列 表达基因序列 YAC或cosmid插入末端序列
2、基于PCR的DNA标记 1)单引物PCR标记 2)双引物选择性扩增的PCR标记 3)通过克隆、测序来构建特殊双引物的PCR标记。
3、基于PCR与限制性内切酶技术相结合的DNA标记
分为两类 限制性酶切片段的选择性扩增 如AFLP PCR扩增片段的限制性酶切 如CAPs
4、基于单核苷多态性的DNA标记 单核苷酸多态性 Single nucleotide polymorphism, 简称SNP
限制性酶
酶切频率较高的限制性酶(frequent cutter), 产生易于扩增基因组DNA 酶切频率较低的限制性酶(rare cutter) 限制扩增模板DNA片段的数量
AFLP扩增数量由酶切频率较低的限制酶在基因组 中的酶切位点数量决定。
分子标记辅助选择步骤及所需药品配方

附录1:实验程序及所用溶液配方实验程序1:DNA小量提取法(SDS小量提取法)1. 取每个水稻样本新鲜叶片2cm左右于1.5 eppondorf管中,加入液氮磨碎。
2. 加入700ul已预热至650C 的SDS抽取液,迅速搅匀后置于650C水中,温浴30min。
3. 加入200ul 5MKAc混合液,颠倒充分混匀,冰浴30min后,40C 8000转离心5min,将上层液倒入另一新的1.5ml eppondorf管中。
4. 加入等体积的异丙醇,置于-200C 30min,40C 10000转离心4min。
5. 弃上清,加入70% 乙醇清洗,晾干。
6. 将风干的DNA溶于100ul TE溶液中,存于40C备用。
实验程序2:DNA大量提取法(SDS大量提取法)1.准备工作:将DNA提取液放于650C的水浴锅中预热,将异丙醇放于-200C冰箱中预冷,选好50 ml的离心管灭菌。
2.每个水稻样本新鲜叶片取1-3g,剪碎,放入研钵,加入液氮磨碎。
3.将研磨好的叶片转于50ml的离心管中,加入20ml已预热至650C 的SDS抽取液,迅速搅匀后置于650C水中,温浴20min,以使反应充分完全。
4.取出离心管,加入5 ml 5MKAc混合液,颠倒充分混匀,冰浴20min,冰浴期间要将离心管上下颠倒数次。
5.加入15ml氯仿,嫩叶片会起泡沫,老叶片则变成墨绿或变黑。
室温下以3000(10000 rmp的速度离心 3min),将上层液倒入另一新的50 ml离心管中。
6.加入等体积的异丙醇,轻轻摇晃直至形成DNA絮状沉淀。
7.待DNA结块,用玻棒挑出DNA,用75%乙醇清洗两次,将乙醇到掉,晾干。
8. 将风干的DNA溶于500ul TE溶液中,存于40C备用。
SDS抽提液1M Tris-Hcl 100ml Ph 8.00.5MEDTA 100ml pH 8.05MNaCl 100ml10%SDS (十二烷基硫酸钠)) 125ml加蒸馏水定容至1000ml。
第七章 分子标记辅助选择 PPT课件

一、质量性状的标记辅助选择 二、数量性状的标记辅助选择 三、标记辅助选择的应用研究 四、标记辅助选择的发展策略
➢选择是指在一个群体中选择符合要求的基因型。
➢传统育种通过表现型间接对基因型进行选择, 存在许多缺陷。
➢分子标记为实现对基因型的直接选择提供了可 能,通过对分子标记基因型的检测,就能获知目 标基因的基因型。
直接依据个体的基因型进行选择,即对每个 目标QTL利用其两侧相邻标记或单个紧密连锁 的标记进行选择,这是才真正的标记辅助选择。
数量性状QTL标记辅助选择的困难:初级定 位,效应小的QTL未被检测。
用3个相邻的连锁标记进行跟踪选择(保证QTL位于 目标区段内)
QTL位于染色体中部
QTL位于染色体末端
6、可提高回交育种效率
利用传统回交方法将一个野生种的优良基因转移 到栽培品种中,回交20代以上还有可能带有100个以 上的其它非期望基因。如果是数量性状位点(QTL) 的转移,由于上位效应问题和连锁累赘更为复杂,将 更加困难。
利用分子标记可以允许选择出那些含有重组染色 体(打破了连锁累赘)的个体,从而帮助减小不需要 的染色体片断,从而提高育种效率至少10倍以上。另 外,对隐性性状可以进行不间断的回交(传统回交中 是隔代回交),从而提高基因的回交转移速度。
2)、双标记选择
❖ 同时用两侧相邻的两个标记对目标基因
进行跟踪选择,可大大提高选择的正确
率。
M1 Q M2 ╳ m1 q m2
M1 Q M2 亲本型:比例高
M1 q M2 双交换型:比例低
在单交换间无干扰的情况下,在F2代通过选择 标记基因型MlM2/MlM2而获得目标基因型Q/Q的概 率为:
在两标记间的图距固定的情况下,r1=r2(亦即目标 基因正好位于两标记之间的中点)为最坏的情形,这 时的选择正确率为最小。 在实际情况中,单交换间一般总存在干扰,使得双交 换概率更小,因而双标记选择的正确率要比理论期望 值更高。
分子标记辅助选择讲解

第十七章分子标记辅助选择育种传统的育种主要依赖于植株的表现型选择(Phenotypieal selection)。
环境条件、基因间互作、基因型与环境互作等多种因素会影响表型选择效率。
例如抗病性的鉴定就受发病的条件、植株生理状况、评价标准等影响;品质、产量等数量性状的选择、鉴定工作更困难。
一个优良品种的培育往往需花费7~8年甚至十几年时间。
如何提高选择效率,是育种工作的关键。
育种家在长期的育种实践中不断探索运用遗传标记来提高育种的选择效率与育种预见性。
遗传标记包括形态学标记、细胞学标记、生化标记与分子标记。
棉花的芽黄、番茄的叶型、抗TMV的矮黄标记、水稻的紫色叶鞘等形态性状标记,在育种工作中曾得到一定的应用。
以非整倍体、缺失、倒位、易位等染色体数目、结构变异为基础的细胞学标记,在小麦等作物的基因定位、连锁图谱构建、染色体工程以及外缘基因鉴定中起到重要的作用,但许多作物难以获得这类标记。
生化标记主要是利用基因的表达产物如同工酶与贮藏蛋白,在一定程度上反映基因型差异。
它们在小麦、玉米等作物遗传育种中得到应用。
但是它们多态性低,且受植株发育阶段与环境条件及温度、电泳条件等影响,难以满足遗传育种工作需要。
以DNA多态性为基础的分子标记,目前已在作物遗传图谱构建、重要农艺性状基因的标记定位、种质资源的遗传多样性分析与品种指纹图谱及纯度鉴定等方面得到广泛应用,尤其是分子标记辅助选择(molecular marker-as—sisted selection,MAS)育种更受到人们的重视。
第一节分子标记的类型和作用原理一、分子标记的类型和特点按技术特性,分子标记可分为三大类。
第一类是以分子杂交为基础的DNA标记技术,主要有限制性片段长度多态性标记(Restriction fragment length polymorphisms,RFLP标记);第二类是以聚合酶链式反应(Polymerase chain reaction,PCR反应)为基础的各种DNA指纹技术。
分子标记辅助选择课件

以DNA多态性为基础的分子标记,目前已在作 物遗传图谱构建、重要农艺性状基因的标记定位、 种质资源的遗传多样性分析与品种指纹图谱及纯度 鉴定等方面得到广泛应用,尤其是分子标记辅助选 择(molecular marker-assisted selection , MAS )育种更受到人们的重视。
3
– Greatest potential advantage over phenotypic selection for traits with low penetrance or low heritability
May reduce population sizes needed for phenotypic selection
8
Pyramid genes for a single trait that could not otherwise be distinguished at the phenotypic level
– Accumulating multiple quantitative trait loci (QTL) for disease resistance may provide a higher level of resistance and/or more durable resistance to changes in the pathogen population
with a long generation time – Reduce number of generations in a backcrossing program by
selecting for recovery of the recurrent parent genome as well as genes of interest from the donor parent
分子标记辅助选择的有关问题与发展策略

分子标记辅助选择的有关问题与发展策略黎裕(中国农科院作物品种资源研究所,北京100081)植物育种项目包括两方面的重要工作,首先是确定育种材料中是否存在有用的遗传变异,其次是采用有效的方法把目标基因转移到品种中。
利用易于鉴定的遗传标记来辅助选择是提高选择效率和降低育种的盲目性的常用手段。
但是在常规育种过程中基本上还是应用的是形态学标记,这类标记往往受环境影响,不利于直接选择,并且标记数目有限。
近一、二十年来迅速发展起来的基于DNA的分子标记技术给育种提供了崭新的途径,这就是所谓的“分子标记辅助选择”(marker-assisted selection,缩写为MAS)。
但在这里它不包括利用分子标记进行优异种质的鉴定筛选、亲本的确定、遗传材料的鉴定和分析、亲本的遗传多样性和亲缘关系分析等,而是指把分子标记技术应用于育种过程之中,通过分析与目的基因紧密连锁的分子标记的基因型来进行育种,从而达到提高育种效率的目的(Dudley, 1993;Lee, 1995)。
分子标记辅助选择是分子育种的一部分(分子育种还包括利用基因工程手段育种)。
一、分子标记辅助选择的优越性有很多理论方面的研究都发现MAS比以表现型为基础的选择更有效,如Knapp (1998)发现在分离群体中,如果目标是从最好的1-2%基因型中选出一个基因型,考虑到成本则MAS是最有效的。
MAS不仅针对主基因有效,针对数量性状位点(QTL)也有效;不仅针对异交作物有效,针对自花授粉作物也有效(特别是在QTL存在显性等位基因并处于相引相时效率更高)(van Berloo Ralph & Stam, 1998)。
MAS的优越性可以体现在以下方面:1.可克服性状基因型鉴定的困难:以下三方面的原因使基因型的鉴定带来不便,即等位基因的外在表现不明显,为隐性等位基因,或与其它基因或环境之间存在互作。
尤其对多基因控制的性状(数量性状)来说,环境变异会使不同基因型表现为部分或全部相同的表型,这使基因型的鉴定更加困难。
第17章 分子标记辅助选择

Efficient data tracking, management, and integration with phenotypic data
Decision support tools for breeders
– optimal design of selection strategies – accurate selection of genotypes
Marker technologies provide the potential to understand the underlying causes of epistasis and GXE, which could greatly improve selection efficiency
Paradox of MAS
目的基因与标记连锁(交换值为r)
亲本中的标记带型
mM RS
×
F1中的标记带型 F2群体中3种标记带型
RR (1-r)2 0.9025
RS 2r(1-r) 0.095
SS r2 0.0025
当 r=0.05 时 , 根 据 标 记 基 因 型 mm选择目的基因型RR,选错的 概率约为0.10
Definitions
第三节分子标记辅助选择
(3)控制单一性状的多个(等位)基因的利用:在不 同育种材料中,影响同一性状(如抗病、品质)的基 因可能有多个,特别是在同一位点上存在不同(复) 等位基因,利用表型是很难鉴定出这些等位基因的。
(4)允许同时选择多个性状:育种目标性状往往需要 综合考虑,也就是说,当选单株或品系不仅要在单一 的抗病、抗虫、品质、产量等方面表现优良,而且在 综合性状上应该比较优良,为此,需要对育种世代群 体的每个目标性状一一作鉴定筛选。
第三节 分子标记助选择
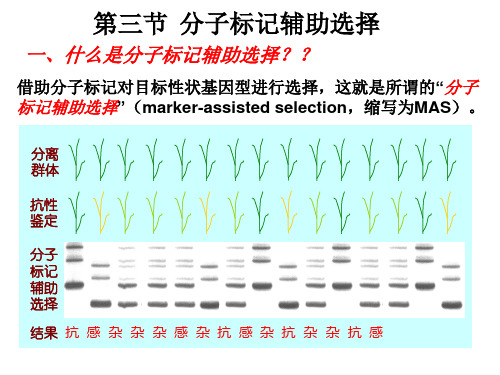
一、什么是分子标记辅助选择??
借助分子标记对目标性状基因型进行选择,这就是所谓的“分子 标记辅助选择”(marker-assisted selection,缩写为MAS)。
分离 群体
抗性 鉴定
分子 标记 辅助 选择
结果 抗 感 杂 杂 杂 感 杂 抗 感 杂 抗 杂 杂 抗 感
广义而言,分子标记辅助选择不仅包括利用分子标 记进行优异种质的鉴定筛选、(自交系)亲本间的 亲缘关系分析、遗传多样性的保持和利用,而且包 括利用分子标记选择、转移、聚合目标基因等育种 的其它诸多环节,如植物系统发育关系分析、品种 注册、专利保护、体细胞杂种鉴定、新品种权保护 等。
二、分子标记辅助选择的优越性
等位基因从一个亲本导入到优良亲本中时,常规表型育 种往往是通过选择保留含有隐形基因的杂合基因型,作 进一步回交,而杂合基因型鉴定通常是通过考察自交后 代分离状况来判断,而且当选择的是抗病性状时,还要 借助繁琐的接种鉴定等,这样会延迟育种进程。分子标 记辅助选择则不受上述限制条件的影响。
在传统育种中,选择的依据通常是表现型而非基因型。 这是因为人们无法直接知道个体的基因型,只能从表现型 加以推断,也就是说,传统育种是通过表现型间接对基因 型进行选择的。理论上,利用分子标记可从DNA水平上直 接鉴定基因型的差异,因此,避免了表现型推断基因型不 准确等缺点。MAS的优越性可以体现在以下方面:
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
第六章分子标记辅助选择选择是育种中最重要的环节之一。
所谓选择,是指在一个群体中选择符合要求的基因型。
但在传统育种中,选择的依据通常是表现型而非基因型,这是因为人们无法直接知道个体的基因型,只能从表现型加以推断。
也就是说,传统育种是通过表现型间接对基因型进行选择的。
这种选择方法对质量性状而言一般是有效的,但对数量性状来说,则效率不高,因为数量性状的表现型与基因型之间缺乏明确的对应关系。
即使是质量性状,有的也可能会因为表型测量难度较大或误差较大而造成表型选择的困难。
另外,在个体发育过程中,每一性状都有其特定的表现时期。
许多重要的性状(如产量和品质)都必须到发育后期或成熟时才得以表现,因而选择也只能等到那时才能进行。
这对于那些植株高大、占地多、生长季长的作物,特别是果树之类的园艺作物,显然是非常不利的。
总之,传统的基于表型的选择方法存在许多缺点,效率较低。
要提高选择的效率,最理想的方法应是能够直接对基因型进行选择。
分子标记为实现对基因型的直接选择提供了可能,因为分子标记的基因型是可以识别的。
如果目标基因与某个分子标记紧密连锁,那么通过对分子标记基因型的检测,就能获知目标基因的基因型。
因此,我们能够借助分子标记对目标性状的基因型进行选择,这称为标记辅助选择(MAS)。
这是分子标记在育种中应用的最主要方面。
第一节质量性状的标记辅助选择如前所述,传统的表型选择方法对质量性状一般是有效的,86因为质量性状的表现型与基因型之间通常存在清晰可辨的对应关系。
因此,在多数情况下,对质量性状的选择无须借助于分子标记。
但对于以下三种情况,采用标记辅助选择可提高选择效率:(1)当表现型的测量在技术上难度很大或费用太高时;(2)当表现型只能在个体发育后期才能测量,但为了加快育种进程或减少后期工作量,希望在个体发育早期(甚至是对种子)就进行选择时;(3)除目标基因外,还需要对基因组的其它部分(即遗传背景)进行选择时。
另外,有些质量性状不仅受主基因控制,而且还受到一些微效基因的修饰作用,易受环境的影响,表现出类似数量性状的连续变异。
许多常见的植物抗病性都表现为这种遗传模式。
这类性状的遗传表现介于典型的质量性状和典型的数量性状之间,所以有时又称之为质量-数量性状。
不过,育种上感兴趣的主要还是其中的主基因,因此习惯上仍把它们作为质量性状来对待。
这类性状的表型往往不能很好地反映其基因型,所以按传统育种方法,依据表型对其主基因进行选择,有时相当困难,效率很低。
因此,标记辅助选择对这类性状就特别有用。
一个典型的例子是大豆孢囊线虫病抗性的标记辅助育种(Young 1999)。
本节首先介绍质量性状标记辅助选择的基本方法(前景选择和背景选择),然后介绍它们在育种上的应用(基因聚合和基因转移)。
一、前景选择对目标基因的选择称为前景选择(foreground selection; Hospital and Charcosset 1997),这是标记辅助选择的主要方面。
前景选择的可靠性主要取决于标记与目标基因间连锁的紧密程度。
若只用一个标记对目标基因进行选择,则标记与目标基因间的连锁必须非常紧密,才能够达到较高的正确率。
假设某标记座位(M/m)与目标基因座位(Q/q)连锁,重组率为r,F1代基因型为MQ/mq,其中Q为目标等位基因,亦即要选择的对象。
由于M87与Q连锁在一起,因此在后代中可通过M来选择Q。
由第五章式(5.3)可知,在F2代通过选择标记基因型M/M而获得目标基因型Q/Q的概率(即单株选择的正确率)为:2=(6.1)p-)1(r从图6.1可以看出,选择正确率随重组率的增加而迅速下降。
若要求选择正确率达到90%以上,则标记与目标基因间的重组率必须不大于0.05。
当重组率超过0.10时,选择正确率已降到80%以下。
不过,如果我们并不要求中选的所有单株都是正确的,而只要求在选中的植株中至少有一株是具有目标基因型的,那么,即使标记只是松弛地与目标基因连锁的,对选择仍然会很有帮助。
如果要求至少选到一株目标基因型的概率为P,则必须选择具有标记基因型M/M的植株的最少数目为:log pPn--=(6.2)1()1(log)图6.2给出了要求P= 0.99时,所要求的最少株数与重组率的关系。
由图可见,即使重组率高达0.3,也只需选择7株具有基因型M/M的植株,就有99%的把握能保证其中有1株为目标基因型;而如果不用标记辅助选择(相当于标记与目标基因间无连锁,重组率为0.5),则至少需选择16株。
同时用两侧相邻的两个标记对目标基因进行跟踪选择,可大大提高选择的正确率。
假设有两个标记座位(M1/m1和M2/m2)各位于目标基因座位(Q/q)的一侧,与目标基因间的重组率分别为r1和r2,F1代的基因型为M1QM2/m1qm2。
那么,F1产生的标记基因型为M1M2的配子具有两种类型,一种包含目标等位基因(M1QM2),为亲本型,另一种包含非目标等位基因(M1qM2),为双交换型。
由于双交换发生的概率很低,因此双交换型配子的比例很小,绝大部分应为亲本型配子。
所以,在后代中通过同时跟踪M1和M2来选择目标等位基因Q,正确率必然很高。
在单交8889换间无干扰的情况下,可以推得,在F 2代通过选择标记基因型M 1M 2/M 1M 2而获得目标基因型Q /Q 的概率为:221212221])1)(1[()1()1(r r r r r r p +----= (6.3) 从式(6.3)可知,在两标记间的图距固定的情况下,r 1 = r 2(亦即目标基因正好位于两标记之间的中点)为最坏的情形,这时的选择正确率为最小。
图6.1和6.2分别显示r 1 = r 2时选择正确率以及P = 0.99时所要求的最少株数与r 1(或r 2)的关系。
可以看出,双标记选择的正确率确实比单标记选择高得多。
需要指出的是,在实际情况中,单交换间一般总是存在相互干扰的,这使得双交换的概率更小,因而双标记选择的正确率要比上述理论期望值更高。
图6.1 标记与目标基因间的重组率与F 2群体中标记辅助选择正确率的关系1020304050607080901001100.00.10.20.30.40.5重组率选择正确率(%)90图6.2 标记与目标基因间的重组率与F 2群体中标记辅助选择最小应选株数的关系二、背景选择对基因组中除了目标基因之外的其它部分(即遗传背景)的选择,称为背景选择(background selection; Hospital and Charcosset 1997)。
与前景选择不同的是,背景选择的对象几乎包括了整个基因组,因此,这里牵涉到一个全基因组选择的问题。
在分离群体(如F 2群体)中,由于在上一代形成配子时同源染色体之间会发生交换,因此每条染色体都可能是由双亲染色体重新组装成的嵌合体。
所以,要对整个基因组进行选择,就必须知道每条染色体的组成。
这就要求用来选择的标记能够覆盖整个基因组,也就是说,必须有一张完整的分子标记连锁图。
当一个个体中覆盖全基因组的所有标记的基因型都已知时,就可以推测出各个标记座位上等位基因的可能来源(指来自哪个亲本),进而可以推测出该个2468101214161800.10.20.30.40.5重组率最小应选株数体中所有染色体的组成。
考虑一条染色体,如果两个相邻标记座位上的等位基因来自不同的亲本,则说明在这两个标记之间的染色体区段上发生了单交换或更高的奇数次交换;如果两标记座位上的等位基因来自同一个亲本,则可近似认为这两个标记之间的染色体区段也来自这个亲本,因为在这种情况下,该区段上只可能发生偶数次交换,而即使是最低的偶数次交换(即双交换),其发生的概率也是很小的。
这样,根据两个相邻的标记,就能够推测出它们之间的染色体区段的来源和组成。
将这个原理推广到所有的相邻标记,就可以推测出一个反映全基因组组成状况的连续的基因型,这种连续的基因型能直观地用图形表示出来,称为图示基因型(graphic genotype;Young and Tanksley 1989a)。
目前已有一些专门用于绘制图示基因型的计算机软件(沈利爽等2000)。
图6.3给出了一个栽培番茄与野生番茄杂交的F2个体的图示基因型。
图6.3一个栽培番茄×野生番茄的F2个体的图示基因型. 共12对染色体, 白色表示来自栽培番茄的区段, 黑色表示来自野生番茄的区段, 灰色表示发生了单交换的区段, 横杠表示标记所在位置. 每对染色体左边数字表示标记的基因型, 1为栽培番茄基因型, 2为杂合基因型,3为野生番茄基因型. 染色体10有两种可能的图示基因型(引自Young and Tanksley 1989a, 并作修改)91图示基因型使人们对每一个体的基因组组成情况一目了然,大大方便了对遗传背景的选择。
在标记辅助选择中,根据图示基因型,可以同时对前景和背景进行选择。
由于目标基因是选择的首要对象,因此一般应首先进行前景选择,以保证不丢失目标基因,然后再对中选的个体进一步进行背景选择,以加快育种进程。
三、基因聚合基因聚合(gene pyramiding)就是将分散在不同品种中的有用基因聚合到同一个基因组中。
这在抗病育种中是一个重要的育种目标。
植物抗病性分为垂直抗性和水平抗性两种,其中垂直抗性受主基因控制,抗性强,效应明显,易于利用。
但垂直抗性一般具有小种特异性,所以易因致病菌优势小种的变化而丧失抗性。
如果能将抵抗不同生理小种的抗病基因聚合到一个品种中,那么该品种就具有抵抗多种生理小种的能力,亦即具有多抗性,这样就不容易因致病菌优势小种的变化而丧失抗性。
多抗性还可指一个品种具有抵抗多种病害的能力,这同样也牵涉到聚合不同抗性基因的问题。
抗性鉴定需要人工接种,必须在一定的发育时期进行,并要求严格控制接种条件,因此往往比较麻烦。
特别是,在基因聚合过程中,必须对不同的抗性基因分别进行鉴定,更增加了实际操作上的难度。
有时还可能因手头缺乏某种所需的致病菌菌株而使抗性鉴定难以进行。
用标记辅助选择方法进行基因聚合则避免了上述困难。
在进行基因聚合时,通常只关注目标抗性基因,即只进行前景选择,暂时可不理会遗传背景。
下面给出一个通过标记辅助选择聚合水稻抗稻瘟病基因的实际例子(Zheng et al. 1995)。
首先是应用分子标记技术将3个抗稻瘟病基因(Pi-2、Pi-1和Pi-4)在水稻第6、11和12号染色体上进行定位(图6.4),然92图6.43个抗稻瘟病基因Pi-2、Pi-1和Pi-4在水稻第6、11和12号染色体上的定位(引自Zheng et al. 1995, 并作修改)图6.5 利用分子标记聚合3个抗稻瘟病基因Pi-2、Pi-1和Pi-4的试验方案(引自Zheng et al. 1995, 并作修改)93后利用连锁标记将这3个抗性基因聚合起来。
