关于GO分析
go 功能富集分析

go 功能富集分析许多分子生物学研究都在研究某些特定的生物学机制,但是这些研究却缺乏综合性和宏观视角,往往无法看到跨之间的关系和整体趋势。
为了解决这个问题,在生物信息学中出现了功能富集分析(GO),这是一种可以从宏观角度理解生物信息的有效工具。
GO,也称为全域功能富集分析,是一种可以从宏观角度研究生物信息的有效工具。
它最初由李纳斯-霍夫曼博士发明,是一种将基因和细胞生物学特性形象地表示出来的系统术语。
GO旨在将一些特定的功能放在一起,它主要有三个类别:生物过程(BP),细胞组分(CC)和分子功能(MF)。
它是由UniProt/Swiss-Prot Group、Gene Ontology Consortium(GOC)和The EBI(European Bioinformatics Institute)等多个组织建立的一系列结构化术语组成。
GO将与分子和细胞相关的一些特定的功能进行分类,它包含了一系列的术语,这些术语可以根据功能的不同分为生物过程,细胞组分和分子功能三大类。
GO的主要目的在于更好地理解生物系统的大型结构,它可以帮助研究人员找出不同基因之间的关系,以及不同基因在不同生物过程中的作用。
GO功能富集分析有很多优点,它可以及时地发现一些特定的生物学机制;它也可以帮助我们挖掘一些潜在的基因网络;它还可以帮助研究人员更好地了解某些特定的基因的功能;它还可以帮助研究人员快速和准确地发现未知的基因与已知的基因之间的相互作用关系。
此外,GO功能富集分析还有一些缺点,例如,缺乏实验证据这一问题,由于GO被设计成一个宏观的系统,它可能会出现一些不准确和不完整的信息,甚至可能会忽略一些重要的生物学机制,从而降低它的准确性。
另外,由于GO是一种宏观的系统,它的建模非常复杂,要学习和使用它还需要大量的时间和经验。
总的来说,GO功能富集分析是一种有效的工具,能够从宏观的角度研究生物信息。
它有助于发现一些特定的生物学机制,并可以帮助研究人员快速和准确地发现未知的基因与已知的基因之间的相互作用关系。
展示空间案例分析

虚空
展示L空O间GO分析
第一部分
空间布局与构成
曲折的实线又被叠上一条笔直的虚线。但这种空间 游客很难感觉出来,实线就是散乱的展示空间,虚 线就是六个大挑空。
虚无空间、阶梯、流亡花园的关系
展示空间分析 — 10 —
展示L空O间GO分析
第一部分
空间布局与构成
Path of salvation
自救之路
上海博物馆是一座大型的古代艺术博物馆,创建于1952年。 50年来,藏有12万件珍贵文物,设有中国古代青铜馆、中国古代陶 瓷馆、中国历代书法馆、中国历代绘画馆、中国历代印章馆、中国 古代雕塑馆、中国古代玉器馆、中国明清家具馆、中国历代钱币馆、 中国少数民族工艺馆、暂得楼陶瓷馆等11个专馆和3个展览厅。上 海博物馆已成为对外文化交流的窗口,是全国青少年爱国主义教育 基地,是市民高层次文化享受场所。
第二部分
人流动线分析——三层
展示空间分析 — 22 —
展示L空O间GO分析
第二部分
人流动线分析——四层
历 少数民族工艺馆
代
古
钱
代
币
玉
馆
器 馆
古代家居馆
展示空间分析 — 23 —
展示L空O间GO分析
第二部分
展示方式
实物场景展示
实物展示
展示空间分析 — 24 —
展示L空O间GO分析
第二部分
色彩
展示空间分析 — 25 —
第一部分
空间布局与构成
博物馆的内部的空间隐喻主要建立在虚空(void)对在场与不在场的 包容性混合之上。
展示空间分析 — 9 —
在建筑中贯穿的与“之”字型整体走向对应的另一道线实际上是由主 体内部被掏出的连续竖向空间组成。在这些竖向的空间中,活动仅仅 只能停留在底层,留给人们的是大段高耸而不能企及的界域。
GO法原理和改进的定量分析方法_沈祖培

5 /35 15~ 19
文 摘 GO 法是以成功 为导向 ,与 故障树方 法完全不同 的 一种全新概念的可靠性分析方法。该文研究 GO 法的原理及 其 应用 ,系统归 纳了 GO 法 ,阐明 了 GO 法的原 理。 通过 对 GO 法的定 量分析 ,提出 了引 入信号 流状 态累 积概率 ,导 出 了新的定量计算公式 ,并提出了共有信号的处理方法。 避免 了复杂的状态组合的概率计算 ,从而可直接进行系统状 态概 率的计算 ,易于编程 ,适于工程应用。 根据文 章提出的方法 , 开发了 GO程序 ,给出了算例。结果表明 ,提出 的改进方法是 有效的 ,有利于 GO 法的发展和应用。 关键词 GO 法 ; 可靠性 ; 系统分析 ; 故障概率
N
P ( 0) , P ( 1) , … , P ( N ) , 满足∑ P ( i ) = 1. 0- N i= 0
状态值是系统状态的代表 ,如不同的流量值 ,不同的 浓度值等。对于有时序的系统 , 0- N 状态值可以称 为时间点 ,用以代表一系列给 定的具体的时间值。 GO 文献中常用∞代表故障状态 ,本文采用最大状 态值 N 表示故障状态 ,更为方便。
AR ( i ) = AS1 ( i ) [PC ( 0)+ AS 2 ( i ) PC ( 1) ] , i = 0,… , N - 1.
类型 7: 有动作信号而关断的元件 除主输入 信号 S1 外 ,还有动作信号 S2 , 当动作信号输入使元 件动作 ,且动作成功 ,或元件提前动作时 ,主输入信
AR ( i ) = PC ( 0)+ AS ( i ) PC ( 1) , i= 0,… , N - 1.
17
类型 6: 有动作信号而导通的元件 除主输入 信号 S1 外 ,还有动作信号 S2 , 当动作信号输入使元 件动作 ,且动作成功 ,或元件提前动作时 ,主输入信
表达谱数据的 GO分析和聚类分析

表达谱数据的 GO分析和聚类分析王琼萍上海交通大学GO(gene ontology)是基因本体联合会(Gene Ontology Consortium)所建立的数据库。
GO 是多种生物本体语言中的一种,旨在建立一个能阐释各种物种的基因以及基因产物。
这个数据库最开始起源于三个模式生物的数据库:果蝇基因组数据库(Drosophila)、酵母基因组数据库(Saccharomyces Genome Database,SGD)、小鼠基因组数据库(Mouse GenomeDatabase,MGD)。
在这之后,在基因本体联合会成员的努力下,将GO 数据库扩展到了植物、动物、微生物等世界范围内各个主要的数据库。
GO 数据库建立了具有三层结构的定义方式来描述基因及其产物的生物学过程、细胞组分及分子功能,对不同信息源的信息进行整合,以DAG(有向无环图)结构组织起来作为多个分支,节点的高低也代表了每个节点的意义的广泛程度。
每个父项(parent terms)下包含若干子项(children terms),分支越远,匹配的GO 条目就越具体。
在这个层级结构中,一个生物学注释可以由一个基因集表示。
这个数据库的建立为基因功能数据挖掘提供了新的思路。
一套基因本体,其实也就是一套基因的树状结构。
GO 数据库及其序列分析程序的问世,使得差异基因的功能分析变得更加高效、准确。
目前,已经有很多可以供畜牧研究者免费使用的GO资源,如AmiGo,它可以分析一个基因的GO 术语,也可以分析多个基因。
另外,还有Onto express、DAVID、Gostat 等。
差异基因的GO 分析关键在于利用统计学方法进行基因富集,常用的方法是Fisher 的精确概率法或卡方检验。
Fisher 的精确概率法利用超几何分布(hypergeometric distribution)的原理推断每个基因集中差异表达基因的比例是否与整个基因芯片上差异表达基因的比例相同。
基因本体数据库与GO富集分析

注释系统中每一个结点都是基因或蛋白的一种描述,结点之间保持严格的关 系,即“is a”或“part of”。
(细胞质)
(细胞器)
(线粒体)
(细胞器膜)
is a· is a → is a
(线粒体)
(胞内细胞器)
part of·part of → part of
(线粒体)
(细胞质)
(细胞器)
part of·is a → part of 与is a·part of → part of
谢 谢
基因功能富集分析
P值的生物学意义决定于所提交的基因列表,例如, 如果列表中均为上调基因而某功能条目显著,则认为 此实验因素作用可能使这个功能激活;相反如果为下 调基因并且条目显著,则认为实验因素作用可能使这 个功能抑制。
基因功能富集分析
控制FDR(False Discovery Rate)值:在进行差异基因挑选时,整个 差异基因筛选过程需要做成千上万次假设检验,导致假阳性率的累积增 大。对于这种多重假设检验带来的放大的假阳性率,需要进行纠正。 FDR用比较温和的方法对p值进行了校正,在假阳性和假阴性间达到平衡, 将假/真阳性比例控制到一定范围之内。
提供目前可获得的基因或基因产物的功能。
涵盖了基因的细胞组分(cellular component)、分子功能 (molecular function)、生物学过程(biological process)。
GO注释是对某个特定基因功能的描述,每一条 GO注释,由一个基因和相应的GO term组成。
基因本体的基本单位是term,每个term都对应 一个属性。
从整体上来看GO注释系统是一个“有向无环 图”。
GO term之间关系表述
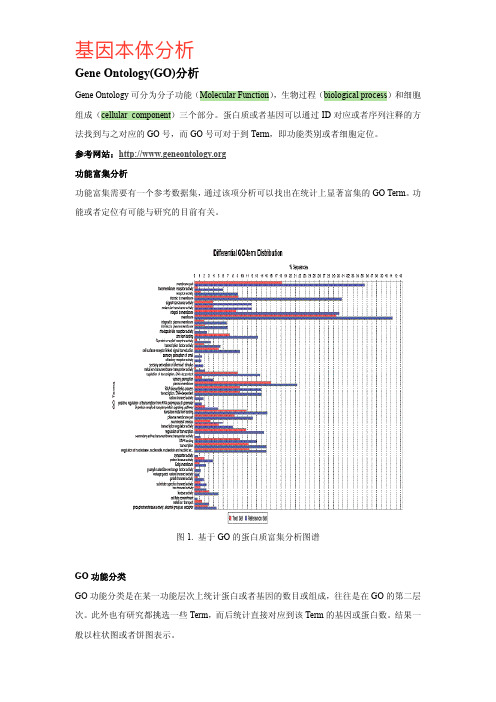
go分析

GO 分析Gene Ontology可分为分子功能(Molecular Function),生物过程(biologicalprocess)和细胞组成(cellularcomponent)三个部分。
蛋白质或者基因可以通过ID对应或者序列注释的方法找到与之对应的GO号,而GO号可对于到Term,即功能类别或者细胞定位。
功能富集分析: 功能富集需要有一个参考数据集,通过该项分析可以找出在统计上显著富集的GOTerm。
该功能或者定位有可能与研究的目前有关。
GO功能分类是在某一功能层次上统计蛋白或者基因的数目或组成,往往是在GO的第二层次。
此外也有研究都挑选一些Term,而后统计直接对应到该Term的基因或蛋白数。
结果一般以柱状图或者饼图表示。
1.GO分析根据挑选出的差异基因,计算这些差异基因同GO 分类中某(几)个特定的分支的超几何分布关系,GO 分析会对每个有差异基因存在的GO 返回一个p-value,小的p 值表示差异基因在该GO 中出现了富集。
GO分析对实验结果有提示的作用,通过差异基因的GO分析,可以找到富集差异基因的GO分类条目,寻找不同样品的差异基因可能和哪些基因功能的改变有关。
2.Pathway分析根据挑选出的差异基因,计算这些差异基因同Pathway 的超几何分布关系,Pathway 分析会对每个有差异基因存在的pathway返回一个p-value,小的p 值表示差异基因在该pathway 中出现了富集。
Pathway分析对实验结果有提示的作用,通过差异基因的Pathway 分析,可以找到富集差异基因的Pathway条目,寻找不同样品的差异基因可能和哪些细胞通路的改变有关。
与GO 分析不同,pathway分析的结果更显得间接,这是因为,pathway 是蛋白质之间的相互作用,pathway 的变化可以由参与这条pathway 途径的蛋白的表达量或者蛋白的活性改变而引起。
利用 agriGO 网络服务进行 GO 富集分析

利用agriGO网络服务进行GO富集分析苏震,徐文英,杜舟,周鑫1.分析目的随着生命科学的发展,越来越多的基因功能被实验验证或者预测推导,但如何规范地注释这些基因是一个难题。
基因本体论(Gene Ontology,GO)是一个在生物信息学领域中广泛使用的本体,应用于基因的功能注释和富集化分析。
GO是一个国际标准化的基因功能分类体系,提供了一套动态更新的标准词汇表,由Gene Ontology组织(/)开发并且维护。
并且,GO是对基因属性特征的客观描述,独立于任何物种或者细胞类型。
因此,我们利用GO,可以对不同物种、不同细胞类型下的基因功能进行规范的描述,避免了沟通上的不便,也可以将隐藏在文献中的基因功能信息更加有效地提取出来。
在动植物功能基因组的研究中,高通量技术的使用产生了海量的组学数据,比如在不同发育期、不同逆境处理下的转录组数据集可以多至上千个表达谱,如何分析和解释这些数据成为摆在生物学家面前的一个难题,而使用GO对基因功能注释进行富集分析,是一套较好的解决方案。
agriGO(GO Analysis Toolkit and Database for Agricultural Community)是一个专注农业物种(以植物物种为主)的GO功能注释与分析的网络数据库与在线分析平台。
agriGO采用的是一套具有完整结构的控制词汇集,使得对该系统可以更好地用于统计和运算,为生物信息学、生物统计学的研究带来了很大的便利。
2.分析工具Gene Ontology富集分析工具agriGO,网址:/agriGO//agriGOv2/参考文献:Zhou Du, Xin Zhou, Yi Ling, Zhenhai Zhang, and Zhen Su. (2010) agriGO: a GO analysis toolkit for the agricultural community. Nucleic Acids Research 38: W64-W70.Tian Tian, Yue Liu, Hengyu Yan, Qi You, Xin Yi, Zhou Du, Wenying Xu, Zhen Su; (2017) agriGO v2.0: a GO analysis toolkit for the agricultural community, 2017 update. Nucleic Acids Research. doi: 10.1093/nar/gkx3823.操作步骤采用agriGO平台提供的实例,练习agriGO中主要的分析工具(见/agriGO/analysis.php):Singular Enrichment Analysis (SEA) 、Parametric Analysis of Gene Set Enrichment (PAGE) 和Cross comparison of SEA (SEACOMPARE)。
GeneOntology(GO)分析
Gene Ontology
现今的生物学家们浪费了太多的时间和精力在搜寻生物信息上。这种情况归结为生物学上定 义混乱的原因:不光是精确的计算机难以搜寻到这些随时间和人为多重因素而随机改变的定 义,即使是完全由人手动处理也无法完成。举个例子来说,如果需要找到一个用于制抗生素 的药物靶点,你可能想找到所有的和细菌蛋白质合成相关的基因产物,特别是那些和人中蛋 白质合成组分显著不同的。但如果一个数据库描述这些基因产物为“翻译类”,而另一个描述 其为“蛋白质合成类”,那么这无疑对于计算机来说是难以区分这两个在字面上相差甚远却在 功能上相一致的定义。 Gene Ontology (GO)项目正是为了能够使对各种数据库中基因产物功能描述相一致的努力结 果。这个项目最初是由 1988 年对三个模式生物数据库的整合开始:: FlyBase (果蝇数据库 Drosophila),t Saccharomyces Genome Database (酵母基因组数据库 SGD) and the Mouse Genome Database (小鼠基因组数据库 MGD)。从那开始,GO 不断发展扩大,现在已包含数 十个动物、植物、微生物的数据库。 GO 的定义法则已经在多个合作的数据库中使用,这使在这些数据库中的查询具有极高的一 致性。这种定义语言具有多重结构,因此在各种程度上都能进行查询。举例来说,GO 可以 被用来在小鼠基因组中查询和信号转导相关的基因产物,也可以进一步找到各种生物地受体 酪氨酸激酶。这种结构允许在各种水平添加对此基因产物特性的认识。 GO 的结构包括三个方面:分子生物学上的功能、生物学途径和在细胞中的组件作用。当然, 它们可能在每一个方面都有多种性质。如细胞色素 C,在分子功能上体现为电子传递活性, 在生物学途径中与氧化磷酸化和细胞凋亡有关,在细胞中存在于线粒体质中和线粒体内膜 上。下面,将进一步的分别说明 GO 的具体定义情况。 基因产物 基因产物和其生物功能常常被我们混淆。例如,“乙醇脱氢酶”既可以指放在 Eppendorf 管里 的基因产物,也表明了它的功能。但是这之间其实是存在差别的,一个基因产物可以拥有多 种分子功能,多种基因产物也可以行使同一种分子功能。比如还是“乙醇脱氢酶”,其实多种 基因产物都具有这种功能,而并不是所有的这些酶都是由乙醇脱氢酶基因编码的。一个基因 产物可以同时具有“乙醇脱氢酶”和“乙醛歧化酶”两种功能,甚至更多。所以,在 GO 中,很 重要的一点在于,当使用“乙醇脱氢酶活性”这种术语时,所指的是功能,并不是基因产物。 许多基因产物会形成复合物后执行功能。这些“基因复合物”有些非常简单(如血红蛋白由血
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
【引用】关于GO分析2011-07-13 17:19:27| 分类:默认分类| 标签:|字号大中小订阅Gene Ontology现今的生物学家们浪费了太多的时间和精力在搜寻生物信息上。
这种情况归结为生物学上定义混乱的原因:不光是精确的计算机难以搜寻到这些随时间和人为多重因素而随机改变的定义,即使是完全由人手动处理也无法完成。
举个例子来说,如果需要找到一个用于制抗生素的药物靶点,你可能想找到所有的和细菌蛋白质合成相关的基因产物,特别是那些和人中蛋白质合成组分显著不同的。
但如果一个数据库描述这些基因产物为“翻译类”,而另一个描述其为“蛋白质合成类”,那么这无疑对于计算机来说是难以区分这两个在字面上相差甚远却在功能上相一致的定义。
Gene Ontology (GO)项目正是为了能够使对各种数据库中基因产物功能描述相一致的努力结果。
这个项目最初是由1988年对三个模式生物数据库的整合开始:: FlyBase (果蝇数据库Drosophila),t Saccharomyces Genome Database (酵母基因组数据库SGD) and the Mouse Genome Database (小鼠基因组数据库MGD)。
从那开始,GO不断发展扩大,现在已包含数十个动物、植物、微生物的数据库。
GO的定义法则已经在多个合作的数据库中使用,这使在这些数据库中的查询具有极高的一致性。
这种定义语言具有多重结构,因此在各种程度上都能进行查询。
举例来说,GO可以被用来在小鼠基因组中查询和信号转导相关的基因产物,也可以进一步找到各种生物地受体酪氨酸激酶。
这种结构允许在各种水平添加对此基因产物特性的认识。
GO发展了具有三级结构的标准语言(ontologies),如表所示。
根据基因产物的相关分子功能,生物学途径,细胞学组件而给予定义,无物种相关性。
本体论内容分子功能本体论基因产物个体的功能,如与碳水化合物结合或ATP水解酶活性等生物学途径本体论分子功能的有序组合,达成更广的生物功能,如有丝分裂或嘌呤代谢等细胞组件本体论亚细胞结构、位置和大分子复合物,如核仁、端粒和识别起始的复合物等基本来说,GO工作可分为三个不同的部分:第一,给予和维持定义;第二,将位于不同数据库中的本体论语言、基因和基因产物进行联系,形成网络;第三,发展相关工具,使本体论的标准语言的产生和维持更为便捷。
本体论(The ontologies)GO 的结构包括三个方面?D?D分子生物学上的功能、生物学途径和在细胞中的组件作用。
当然,它们可能在每一个方面都有多种性质。
如细胞色素C,在分子功能上体现为电子传递活性,在生物学途径中与氧化磷酸化和细胞凋亡有关,在细胞中存在于线粒体质中和线粒体内膜上。
下面,将进一步的分别说明GO的具体定义情况。
基因产物基因产物和其生物功能常常被我们混淆。
例如,“乙醇脱氢酶”既可以指放在Eppendorf管里的基因产物,也表明了它的功能。
但是这之间其实是存在差别的?D?D一个基因产物可以拥有多种分子功能,多种基因产物也可以行使同一种分子功能。
比如还是“乙醇脱氢酶”,其实多种基因产物都具有这种功能,而并不是所有的这些酶都是由乙醇脱氢酶基因编码的。
一个基因产物可以同时具有“乙醇脱氢酶”和“乙醛歧化酶”两种功能,甚至更多。
所以,在GO中,很重要的一点在于,当使用“乙醇脱氢酶活性”这种术语时,所指的是功能,并不是基因产物。
许多基因产物会形成复合物后执行功能。
这些“基因复合物”有些非常简单(如血红蛋白由血红蛋白基因产物α-球蛋白、β-球蛋白和小分子的亚血红素组成),有些非常复杂(如核糖体)。
现在,小分子的描述还没有包括在GO中。
在未来,这个问题可望由和现在的Klotho和LIGAND等小分子数据库联合而解决。
分子功能分子功能描述在分子生物学上的活性,如催化活性或结合活性。
GO分子功能定义功能而不是整体分子,而且不特异性地指出这些功能具体的时空信息。
分子功能大部分指的是单个基因产物的功能,还有一小部分是此基因产物形成的复合物的功能。
定义功能的义项包括催化活性、转运活性、结合活性等,更为狭窄的定义包括腺苷酸环化酶活性或钟形受体结合活性等。
生物学途径生物学途径是由分子功能有序地组成的,具有多个步骤的一个过程。
举例来说,较为宽泛的是细胞生长和维持、信号传导。
一些更为具体的例子包括嘧啶代谢或α-配糖基的运输等。
一个生物学途径并不是完全和一条生物学通路相等。
因此,GO 并不涉及到通路中复杂的机制和所依赖的因素。
细胞组件细胞中的位置指基因产物位于何种细胞器或基因产物组中(如糙面内质网,核或核糖体,蛋白酶体等)。
GO的形式GO 定义的术语有着直接非循环式(directed acyclic graphs (DAGs)的特点,而并非是传统的等级制(hierarchy)定义方式(随着代数增加,下一级比上一级更为具体)。
举个例子来说,生物学途径中有一个定义是己糖合成,它的上一级为己糖代谢和单糖合成。
当某个基因被注解为“己糖合成活性”后,它自动地获得了己糖代谢和单糖合成地注解。
因为在GO中,每个术语必须遵循“真途径“法则,即如果下一代的术语可以用于描述此基因产物,其上一代术语也可以适用。
GO的注释(Annotation)那么,GO中的术语如何和相对应的基因产物相联系的呢?这是由参与合作的数据库来完成的,它们使用GO的定义方法,对它们所包含的基因产物进行注解,并且提供支持这种注解的参考和证据。
每个基因或基因产物都会有一个列表,列出与之相关的GO术语。
每个数据库都会给出这些基因产物和GO术语的联系数据库,并且也可以在GO的ftp站点上和WEB方式查询到。
并且,GO联合会提供了简化的本体论术语(GO slim),这样,可以在更高级的层面上研究基因组的功能。
比如,粗略地估计哪一部分的基因组与信号传导、代谢合成或复制有关。
GO对基因和蛋白的注释阐明了基因产物和用于定义他们的GO术语之间的关系。
基因产物指一个基因编码的RNA或蛋白产物。
因为一个基因可能编码多个具有很不相同性质的产物,所以GO推荐的注释是针对基因产物的而不是基因的。
一个基因是和所有适用于它的术语联系在一起的。
一个基因产物可以被一种本体论定义的多种分支或多种水平注释。
注释需要反映在正常情况下此基因产物的功能,生物途径,定位等,而并不包括其在突变或病理状态下的情况。
GO联合会的各个数据库成员采用手动或自动的方式生成注释,这两种方式共有的原理是:一.所有的注释都需要有来源,可以是文字、另一个数据库或是计算机分析结果;二.注释必须提供支持这种基因产物和GO术语之间联系的证据。
GO文件格式GO的所有数据都是免费获得的。
GO数据有三种格式:flat(每日更新)、XML(每月更新)和MySQL(每月更新)。
这些数据格式都可以在GO ftp的站点上下载。
XML 和MySQL 文件是被储存于独立的GO数据库中。
如果需要找到与某一个GO术语相关的基因或基因产物,可以找到一个相应表格,搜寻到这种注解的编号,并且可以链接到与之对应的位于不同数据库的基因相关文件。
GO浏览器和修改器(browser and editor)GO 术语和注释使用了多种不同的工具软件,它们都可以在web方式的“GO 浏览器”下“GO software page”中找到。
大多数GO浏览器都是web模式的,允许你直观的看到术语和其相关信息,如定义、同义词和数据库参考等。
有些GO浏览器如AmiGO和QuickGO,可以看到每个术语的注释。
而可下载的DAG-Edit 编辑器,一样可以离线地显示注释和所有本体论定义的信息。
对于每一个浏览器来说,都可以选择最适用于你要求的工具软件。
常见的三种浏览器AmiGO from BDGP 在AmiGO中,可以通过查询一个GO术语而得到所有具有这个注释的基因产物,或查询一个基因产物而得到它所有的注释关系。
还可以浏览本体论,得到术语之间的关系和术语对应的基因产物数目。
AmiGO直接连接GO下的MySQL。
MGI GO Browser MGI GO的功能类似于AmiGO,所不同的在于它所得到的基因为小鼠基因。
MGI GO浏览器直接连接GO下的MGI数据库。
QuickGO at EBI QuickGO,整合在EBI下的InterPro中,可以通过查询一个GO术语而得到它的定义与关系描述、在SWISS-PROT中的定位、在酶分类学(EC)和转运分类学(TC)中的定位和InterPro中的定位等。
其他还有一些特殊的浏览GO的浏览器,其中括号中为建立机构和主要特色:EP GO Browser(EBI,基因表达情况),、GoFish (Harvard,Boolean查询、GenNav(NLM, 图像化展示)、GeneOntology@RZPD (RZPD,UniGene)、ProToGO (Hebrew University,GO的亚图像化)、CGAP GO Browser (癌症基因组解剖工程,癌症)、GOBrowser (Illuminae,perl.、TAIR Keyword Browser (TAIR,拟南芥)、PANDORA (Hebrew University,非一致化蛋白)。
修改器GO 术语和本体论结构可以由任何可以读入GO平板文件的文本修改器进行编辑,但是这需要对平板文件非常熟悉。
因此,DAG-Edit是被推荐使用的,它是为GO特别设计的,能够保证文件的句法正确。
GO注释可以被多种数据库特异性的工具所编辑,如TIGR的Manatee和EBI的Talisman tool。
但是GO数据库中写入新的注释是需要通过GO认证的管理员方可进行的,如果想提出新的注释或对本体论的建议,可以联系GO。
主要修改器为DAG-Edit和COBrA。
DAG-Edit基于Java语言,提供了能浏览、查询、编辑具有DAG数据格式的GO数据界面。
在SourceForge可以免费下载,伴随着帮助文件。
COBrA能够编辑和定位GO 和OBO本体论。
它一次显示两个本体论,因此可以在不同的水平相应定位。
(如组织和细胞类型水平)优点在于可以综合几种本体论,支持的文件格式多,包括GO平板文件、GO RDF和OWL格式等。
如图为DAG-Edit的界面,可以分为四个部分:1)定义编辑面板(term editor panel)显示当下的本体论。
也是主要的编辑本体论结构的工具,可以通过点击和拖动术语来修改本体论的从属关系。
2)文本编辑面板(text editor panel)修改术语中的内容。
在修改多个术语时,会出现一个选择菜单,可以选中后逐个修改。
3) DAG浏览器DAG浏览器是一个插件,能够以图形的方式展示具有复杂的从属关系的术语。
