手把手教你设计引物
设计引物的方法

设计引物的方法
首先,确定目标序列是进行引物设计的第一步。
目标序列的选
择应该考虑到所需扩增的DNA片段的长度和特点,以及引物的位置
和方向。
在确定了目标序列之后,就可以根据目标序列的特点来选
择合适的引物设计方法了。
一种常用的引物设计方法是基于序列比对的方法。
通过对目标
序列与相关物种的序列进行比对,可以找到共同的保守区域,从而
设计出具有较高特异性的引物。
这种方法适用于目标序列与已知序
列高度保守的情况,可以有效地提高引物的特异性和扩增效率。
另一种常用的引物设计方法是基于引物特性的方法。
在这种方
法中,可以根据引物的特性来设计引物,例如引物的长度、GC含量、熔解温度等。
通过调整这些引物特性,可以提高引物的特异性和扩
增效率。
同时,还可以利用一些引物设计软件来辅助设计引物,这
些软件可以根据一定的算法和模型来预测引物的性能,提高引物设
计的准确性和效率。
除了以上介绍的两种方法外,还有一些其他的引物设计方法,
例如基于结构的方法、基于引物库的方法等。
这些方法都有各自的
优缺点,可以根据实际情况来选择合适的方法进行引物设计。
总的来说,设计引物的方法是一个复杂而又重要的过程。
在进行引物设计时,需要考虑到目标序列的特点、引物的特性以及实验条件等多个因素。
通过选择合适的引物设计方法,可以设计出具有较高特异性和扩增效率的引物,从而提高PCR扩增和测序结果的准确性和可靠性。
希望本文介绍的引物设计方法能够对研究人员在实验中有所帮助。
引物设计——手把手教新手

②Analyze中,第一项为Key info,点击Selected primers,会给出两条引物的概括性信息,其中包括引物的Tm值,此值Oligo是采用nearest neighbor method计算,会比Primer5中引物的Tm值略高,此窗口中还给出引物的Delta G和3’端的Delta G.3’端的Delta G过高,会在错配位点形成双链结构并引起DNA聚合反应,因此此项绝对值应该小一些,最好不要超过9。
⑤在Primer5窗口中,若觉得某一对引物合适,可以在搜索结果窗口中,点击该引物,然后在菜单栏,选择File-Print-Current pair,使用PDF虚拟打印机,即可转换为Pdf文档,里面有该引物的详细信息。
3、用Oligo验证评估引物
①在Oligo软件界面,File菜单下,选择Open,定位到目的cDNA序列(在primer中,该序列已经被保存为Seq文件),会跳出来两个窗口,分别为Internal Stability(Delta G)窗口和Tm窗口。在Tm窗口中,点击最左下角的按钮,会出来引物定位对话框,输入候选的上游引物序列位置(Primer5已经给出)即可,而引物长度可以通过点击Change-Current oligo length来改变。定位后,点击Tm窗口的Upper按钮,确定上游引物,同样方法定位下游引物位置,点击Lower按钮,确定下游引物。引物确定后,即可以充分利用Analyze菜单中各种强大的引物分析功能了。
引物设计方法及说明
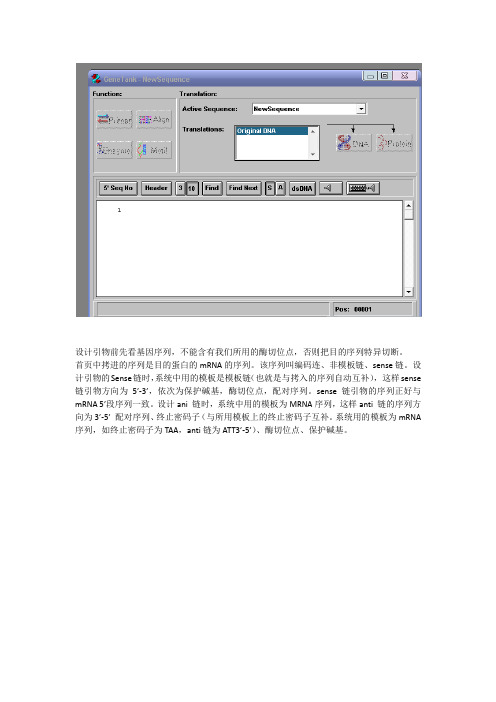
设计引物前先看基因序列,不能含有我们所用的酶切位点,否则把目的序列特异切断。
首页中拷进的序列是目的蛋白的mRNA的序列。
该序列叫编码连、非模板链、sense链。
设计引物的Sense链时,系统中用的模板是模板链(也就是与拷入的序列自动互补),这样sense 链引物方向为5’-3’,依次为保护碱基,酶切位点,配对序列。
sense链引物的序列正好与mRNA 5’段序列一致。
设计ani 链时,系统中用的模板为MRNA序列,这样anti 链的序列方向为3’-5’配对序列、终止密码子(与所用模板上的终止密码子互补。
系统用的模板为mRNA 序列,如终止密码子为TAA,anti链为ATT3’-5’)、酶切位点、保护碱基。
PCR使用说明引物设计技巧

PCR使用说明引物设计技巧PCR(聚合酶链反应)是一种常用的分子生物学技术,可用于扩增DNA片段以及进行基因分型、疾病诊断和DNA克隆等应用。
在PCR实验中,引物的设计是非常关键的步骤之一,合理的引物设计可以确保PCR反应的特异性和高效性。
以下是一些PCR引物设计的技巧和原则。
1.引物长度:引物长度应该在18到30个核苷酸对之间,一般来说,较短的引物可以提高反应的特异性,但也容易导致非特异性扩增。
较长的引物可以提高特异性,但也会降低PCR反应的效率。
2.引物的碱基组成:引物的G+C含量应在40%到60%之间,避免过高或过低的含量,以确保引物的熔解温度适中。
3.引物之间的互补性:引物之间不应有任何互补性,以避免引物之间的杂交和产生非特异性扩增。
4. 引物的熔解温度:引物的熔解温度(Tm)应该相近,通常设计为60℃至70℃之间。
可以使用一些在线工具来计算引物的Tm,例如NCBI的Primer-BLAST。
5.引物的位点选择:引物应该选择在目标序列上独特的位点,避免引物在其他不需要扩增的区域上产生扩增。
可以使用序列比对工具,如BLAST,来确定引物的特异性。
6.引物的末端设计:引物的末端应该避免酶切位点,以防止引物被酶切和降解。
此外,末端的碱基对的GC含量应保持平衡,以确保引物的稳定性。
7.引物的序列结构:引物的序列中应避免重复和倒序重复的碱基序列,因为这些序列容易形成引物间的二级结构和非特异性扩增。
8.引物的交叉反应:引物的序列应该经过认真筛选,避免与其他非目标序列发生交叉反应。
在引物设计前,可以先使用基因序列比对工具,如BLAST,来检查引物是否会与其他区域发生交叉反应。
9.引物的引导方向:引物的引导方向应与目标序列的末端互补,以确保正确的扩增方向。
总而言之,PCR引物的设计应遵循特异性、高效性和可重复性的原则。
合理设计的引物对PCR实验的成功至关重要,可以提高扩增产物的特异性和产量,并避免非特异性扩增和交叉反应的发生。
引物设计的详细步骤

引物设计的详细步骤详细步骤如下:步骤一:了解引物设计的基本原理引物设计是指为特定的DNA序列设计一对合适的引物,以便在PCR反应中扩增目标DNA序列。
引物是PCR反应的关键组成部分,引物的选择和设计对于PCR扩增的成功率和特异性非常重要。
因此,了解引物设计的基本原理对于有效设计合适的引物至关重要。
步骤二:确定PCR反应的目标序列在设计引物之前,我们需要确定PCR反应的目标序列,即我们需要扩增的DNA区域。
这个目标序列可以是已知的基因序列,也可以是未知的区域。
确定目标序列后,我们可以继续设计引物。
步骤三:确定引物的一些基本参数在设计引物之前,我们需要确定一些基本的参数,以便帮助我们选择合适的引物。
这些参数包括引物的长度、GC含量、Tm值以及避免二聚体形成等。
引物长度:通常来说,引物的长度应在18-25个核苷酸之间。
过长的引物可能导致不特异的扩增产物的形成,而过短的引物则可能导致低扩增效率。
GC含量:引物的GC含量对于引物的稳定性和特异性有影响。
在正常情况下,引物的GC含量应在40%-60%之间。
Tm值:引物的Tm值是指引物在PCR反应中的解离温度。
Tm值过低可能导致非特异的扩增产物的形成,而Tm值过高则可能导致低扩增效率。
避免二聚体形成:在设计引物时,我们还需要考虑引物之间的互补性以及避免引物形成二聚体。
引物之间的互补性可能导致引物形成二聚体,从而降低PCR反应的效率和特异性。
步骤四:选择合适的引物设计工具目前有很多在线引物设计工具可供选择,例如NCBI Primer-BLAST、OligoAnalyzer等。
这些工具可以根据输入的目标序列帮助我们快速选择合适的引物。
此外,还可以使用一些商业引物设计软件,如Primer Premier等。
步骤五:进行引物特异性分析设计好引物后,我们需要进行引物特异性分析,确保引物只扩增目标序列而不扩增其他非特异性产物。
这可以通过BLAST或其他相似性工具来完成。
特异性分析的目的是排除可能存在的非特异性扩增产物,以确保PCR反应的准确性和特异性。
Primer5设计引物------图文并茂一步步教你
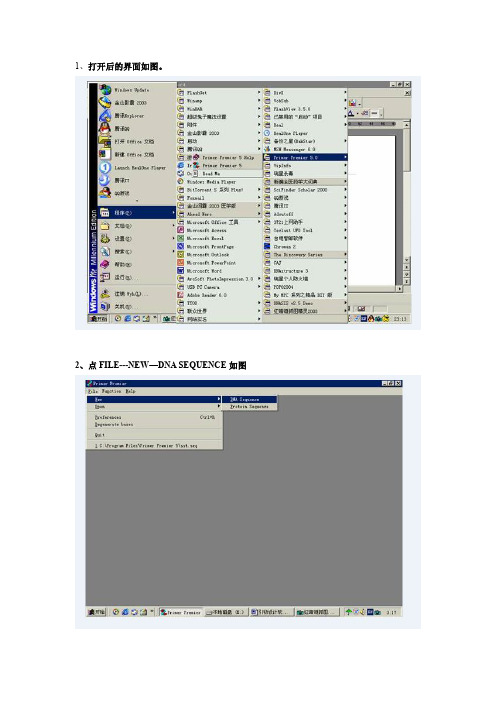
1、打开后的界面如图。
2、点FILE---NEW—DNA SEQUENCE如图3、输入目的基因片段,可以复制后用ctrl+V键拷贝到栏内,后应加数个N以备后续设计时加酶切位点及保护碱基,如图所示。
输入目的基因片段,可以复制后用ctrl+V键拷贝到栏内,后应加数个N以备后续设计时加酶切位点及保护碱基,如图所示。
4、此主题相关图片如下:选中enzyme图标,将所选质粒上的多克隆酶切位点加入左栏此主题相关图片如下:选中OK键,5、分析目的基因中所含的酶切位点,选插入位点时就应排除这些酶此主题相关图片如下:6、选中primer图标,点S图标,edit primer,开始设计正义链。
此主题相关图片如下:7、软件默认引物为二五个碱基此主题相关图片如下8、可将鼠标点在设计框的3端从右向左删除7-9个碱基,保留16-18个配对即可此主题相关图片如下:9、在引物的5端加入选好的酶切位点并在其左侧加3个保护碱基,入该图加入HIND III 酶切位点及TTA保护碱基,完成后点analyze,认为可以后点OK。
此主题相关图片如下:10、选中左上角A图标,用鼠标拉动滑块将待选引物放至目的基因末端。
11、从3端删除7-9个碱基同正义链。
此主题相关图片如下:12、将酶切位点加在5端,应将产品目录所示的酶切位点序列从右至左加入(注意不要加反)如图加入BamH I酶切位点及CGC3个保护碱基。
完成后点analyze,认为可以后点OK。
此主题相关图片如下:13、最后分析结果如图,反义链的FALSE PRIMING可以不考虑,RATING表示引物评分也可以不考虑,主要看Tm值正义链和反义链相差不应超过3度。
GC含量不应超过60%此主题相关图片如下:14、该软件有个缺点,不能保存分析结果,只能选择打印此主题相关图片如下:15、如果设计RT-PCR检测的引物就如下所示,同上输入目的基因片段,选SEARCH图标,选择参数,一般选PCR primers---both—100至250个碱基,引物长短20+/-2,search parametere 中的参数可以不选,为默认设置。
引物设计——手把手教新手

引物设计——手把手教新手引物设计是指在实验室中用于特定目的的DNA序列,用于扩增特定的DNA片段。
引物设计在分子生物学研究和遗传工程方面起着至关重要的作用。
对于新手来说,如何设计合适的引物是一个基础且重要的技能。
下面将以一种扩增人类基因的引物设计为例,手把手地教新手进行引物设计。
首先,我们需要确定扩增的目标基因。
假设我们的目标基因是人类TNF-α基因。
TNF-α是一种调节免疫反应的细胞因子,与一些疾病如炎症性疾病、自身免疫性疾病等密切相关。
因此,深入研究TNF-α基因的功能和表达是非常重要的。
第二步是获取TNF-α基因的序列。
我们可以在公共数据库如NCBI (National Center for Biotechnology Information)中人类TNF-α基因的序列。
在NCBI的网站上,可以输入关键词“human TNF-alpha gene sequence”,并选择合适的结果进行。
我们可以找到人类TNF-α基因的RefSeq基因组序列。
然后,我们需要对所获得的基因序列进行分析,以确定扩增的目标区域。
在TNF-α基因中,选择合适长度的外显子或启动子区域作为目标区域进行扩增。
在选择时,需要考虑该区域的特异性和扩增效率。
接下来,我们需要使用一些在线工具来进行引物设计。
有许多免费的引物设计工具可供选择,如Primer3、NCBI Primer-BLAST等。
在这里,我们将使用Primer3进行引物设计。
在Primer3的网站上,可以将TNF-α基因的目标区域序列粘贴到“SEQUENCE INPUT”输入框中。
然后,可以设置一些参数,如引物长度、Tm值、GC含量等。
对于引物长度,通常选择18-25个核苷酸;Tm值通常在55-65°C之间;GC含量通常在40-60%之间。
设置好参数之后,点击“PICK PRIMERS”按钮,即可获得计算结果。
得到计算结果后,将得到一对引物的序列和其他相关信息,如引物的Tm值、GC含量、特异性等。
设计引物的方法

设计引物的方法
首先,我们需要明确引物的特性。
引物是一种短链的DNA或RNA序列,通常用于PCR扩增、实时荧光定量PCR、测序等实验中。
因此,引物的选择需要考虑
到引物的长度、GC含量、配对温度等特性。
一般来说,引物的长度应在18-25个
碱基对之间,GC含量应在40%-60%之间,配对温度应在50-65摄氏度之间。
这些
特性的选择将直接影响到引物的扩增效率和特异性。
其次,针对目标序列的特点,我们需要选择合适的引物设计方法。
对于已知的
序列,我们可以利用计算机软件进行引物设计,如Primer3、Beacon Designer等。
这些软件可以根据用户输入的序列信息,自动给出符合要求的引物设计方案。
对于未知序列,我们可以采用引物库筛选的方法,从引物库中挑选符合要求的引物进行实验。
此外,实验的具体要求也是引物设计的考量因素之一。
在进行引物设计时,我
们需要考虑到实验的目的、样本的特点、实验条件等因素。
比如,在进行实时荧光定量PCR实验时,我们需要选择特定的引物和探针,以确保实验的准确性和灵敏度。
在进行测序实验时,我们需要选择特定的引物,以确保测序结果的可靠性和准确性。
综上所述,设计引物的方法需要考虑引物的特性、目标序列的特点以及实验的
具体要求。
在进行引物设计时,我们可以利用计算机软件进行设计,也可以采用引物库筛选的方法。
同时,我们需要根据实验的具体要求,选择合适的引物设计方案。
希望以上内容能够帮助大家更好地进行引物设计,提高实验的效率和准确性。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
手把手教你设计引物(图文并茂)
关键词:引物设计2014-05-14 10:33来源:丁香园点击次数:9911
不知不觉几年下来自己也快毕业了,感谢丁香园这些年来的帮助。
没有什么可回报的东西,就发个帖教教新人如何设计引物吧,尽量做到手把手的教,图文并茂。
引物设计的帖子不少,以前很多战友会推荐Oligo、PrimerPremier、DNA man等等软件。
这些软件设计完最后还是要去BLAST比对下,所以我教大家一种易懂实用的在线设计方法,觉得好的话请投个票。
就以人的PTEN基因为例,首先你要找到他的基因序列,如果你要用的是cDNA,就找它的mRNA序列。
如果你要做的是DNA,就找DNA的序列。
以cDNA为例,普遍的一种方法是上PUBMED中GENE栏搜索找到cDNA那栏,但PUBMED 导出序列不太方便,我介绍个网站 /index.html
1. 输入目的基因,进入
2.在左侧栏选择TRANSCRIPT,选择后进入
3. 选择PTEN-001中的TRANSCRIPT进入,点击左侧cDNA
4. 然后点击CONFIGURE THIS PAGE进入设置你要显示的内容
5. 除了第一栏SHOWEXONS选择YES外,其他的都选择NO,然后取个名字保存SAVE CONFIGURATION AS
6. 然后在左侧栏点击DOWNLOADVIEW AS RTF可下载你要的cDNA序列,这个文件可以用WORD打开,不同的颜色代表一个外显子间断
下载后打开的WORD
7. 然后根据可以根据你感兴趣的序列设计引物了,比如我在分别在第6和第7外显子分别设计上下游引物。
选取并复制第6和第7外显子序列
8. 登陆 /tools/primer-blast/,粘贴这段序列,设置好RANGE和PCR产物的大小,然后在下面点击GET PRIMERS,可以在线设计并比对引物
9.最后选择一个比较特异性的引物,条带大小要尽量单一,其他的基因序列尽量不要比对到。
