SWISS-MODEL_蛋白质结构预测教程
5.5计算方法预测三级结构-02-同源建模法SWISS-MODEL

《生物信息学》第五章:蛋白质结构预测与分析(第二部分) 计算方法预测三级结构:同源建模法SWISS-MODEL预测蛋白质三级结构的首选方法是同源建模法(homolog modeling)。
该方法基于原理:相似的氨基酸序列对应着相似的蛋白质结构。
比如三个蛋白质,它们在序列水平上十分相似,解析出的结构也十分相似。
第四个蛋白质的序列和前面三个也高度相似,那么就可以比着前三个结构的样子“画”出第四个的样子。
所以同源建模法的关键就是找到一个好的模板。
好的模板要求,在序列水平上模板(template)要与目标(target)蛋白质具有超过30%的一致度。
同源建模法操作流程如下(图1):图1. 同源建模法操作流程1. 确定模板:找到与目标蛋白质同源的已知蛋白质结构作为模版(目标序列与模版序列间的一致度要≥30%)。
2. 序列比对:为目标序列与模板序列创建序列对比。
模板可以选取多个,通过做多序列比对,各取所长,让模板序列中与目标序列相似的片段尽可能多的覆盖整个目标序列,同时要尽量避免没有模板参考的断口。
3. 计算模型:通过序列比对,将目标序列里的氨基酸替换到模板结构里对应的氨基酸所在的空间位置上。
这一步通过同源建模软件来实现。
4.换模板或修正序列比对,重新构建模型,再次评估。
SWISS-MODEL()它能帮助完成上述步骤中从模板选取到创建序列比对,再到计算模型,以及最后的质量评估的全部过程。
你需要做的只是:输入目标序列,点Build Model(创建模型)(图2左)。
大约三到五分钟之后就会返回结果。
如果这种自动挡模式不能满足你的要求,可以通过点击Search For Templates切换成手动挡,以便指定模板。
也可以直接把做好的目标序列与模板序列之间的序列比对按照指定格式黏贴到输入框里,再点击Build Model(创建模型)(图2右)。
这时,SWISS-MODEL会根据输入的特定格式的序列比对,识别出哪个是目标序列,哪个是模板,并自动从PDB数据库下载模板结构,最后根据输入的比对计算结构模型。
蛋白质三级结构预测(swiss-model同源建模)
利用同源建模预测蛋白质的三级结构首先声明一下,以下纯属个人观点,方法步骤仅供参考,不可作为规范标准,结果出来之后请自行分析结果。
我用的是SWISS-MODEL同源建模的方法进行的蛋白质高级结构预测,其实这个方法是有限制条件的,不过作为一个选修课作业,我们不用深入探究,所以有时不够严谨,大家知道就行!对于一个未知结构的蛋白质,白质建立结构模型。
那么,我们首先要做的就是找到和我们空格和“—”的氨基酸序列,例如:【字母大小写没有影响】vlqdsigyirilsmmdpvvdefdrayqqvkdfpdlmvdvrengggnsgngkkiceylihkpqphcvspdweiiprkd)同源的、相似度最高的、已知三级结构的蛋白质作为模版。
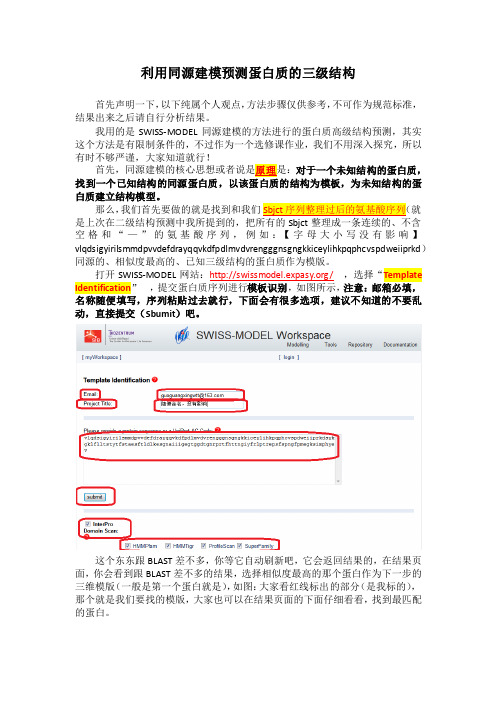
打开SWISS-MODEL网站:/,选择“Template Identification,提交蛋白质序列进行模板识别,如图所示,注意:邮箱必填,名称随便填写,序列粘贴过去就行,下面会有很多选项,建议不知道的不要乱动,直接提交(Sbumit)吧。
这个东东跟BLAST差不多,你等它自动刷新吧,它会返回结果的,在结果页面,你会看到跟BLAST差不多的结果,选择相似度最高的那个蛋白作为下一步的三维模版(一般是第一个蛋白就是),如图:大家看红线标出的部分(是我标的),那个就是我们要找的模版,大家也可以在结果页面的下面仔细看看,找到最匹配的蛋白。
这里还有一点要作说明,就是上图标出的代码是PDB编号,前四个表示PDB- Code,最后一位表示Chain-ID,具体什么意思,大家有兴趣就去了解一些吧。
接下来,去NCBI串串门吧,在NCBI中搜索上面查到的蛋白的PDB号,一般输入前四位就行啦,注意:搜索蛋白库(Protein)。
找到以后,以FASTA格式显示。
接下来,我们再回到SWISS-MODEL,接下来就是重点和难点啦,在线提交序列进行同源建模分析,这个在线提交不是大家想象的那么容易,这个耗费了我大部分的时间,说到这里我就想画个圈圈诅咒它,大家注意啦~~~~~~~~~~~SWISS-MODEL 是一个自动化的蛋白质比较建模服务器,该服务器提供用户三种模式可选择:Automatic mode(简捷模式): 用于建模的氨基酸序列或是Swiss-Prot/TrEMBL (/sprot )编目号(accession)可以直接通过web界面提交。
SWISS-MODEL_蛋白质结构预测教程

SWISS-MODEL 蛋白质结构预测SWISS-MODEL是一项预测蛋白质三级结构的服务,它利用同源建模的方法实现对一段未知序列的三级结构的预测。
该服务创建于1993年,开创了自动建模的先河,并且它是讫今为止应用最广泛的免费服务之一。
同源建模法预测蛋白质三级结构一般由四步完成:1. 从待测蛋白质序列出发,搜索蛋白质结构数据库(如PDB,SWISS-PROT等),得到许多相似序列(同源序列),选定其中一个(或几个)作为待测蛋白质序列的模板;2. 待测蛋白质序列与选定的模板进行再次比对,插入各种可能的空位使两者的保守位置尽量对齐;3. 建模:调整待测蛋白序列中主链各个原子的位置,产生与模板相同或相似的空间结构——待测蛋白质空间结构模型;4. 利用能量最小化原理,使待测蛋白质侧链基团处于能量最小的位置。
最后提供给用户的是经过如上四步(或重复其中某几步)后得到的蛋白质三级结构。
SWISS-MODEL工作模式SWISS-MODEL服务器是以用户输入信息的最小化为目的设计的,即在最简单的情况下,用户仅提供一条目标蛋白的氨基酸序列。
由于比较建模程序可以具有不同的复杂性,用户输入一些额外信息对建模程序的运行有时是有必要的,比如,选择不同的模板或者调整目标模板序列比对。
该服务主要有以下三种方式:∙First Approach mode(简捷模式):这种模式提供一个简捷的用户介面:用户只需要输入一条氨基酸序列,服务器就会自动选择合适的模板。
或者,用户也可以自己指定模板(最多5条),这些模板可以来自ExPDB 模板数据库(也可以是用户选择的含坐标参数的模板文件)。
如果一条模板与提交的目标序列相似度大于25%,建模程序就会自动开始运行。
但是,模板的可靠性会随着模板与目标序列之间的相似度的降低而降低,如果相似度不到50%往往就需要用手工来调整序列比对。
这种模式只能进行大于25个残基的单链蛋白三维结构预测。
∙Alignment Interface(比对界面):这种模式要求用户提供两条已经比对好的序列,并指定哪一条是目标序列,哪一条是模板序列(模板序列应该对应于ExPDB模板数据库中一条已经知道其空间结构的蛋白序列)。
生物信息学 实验六 蛋白质高级结构预测
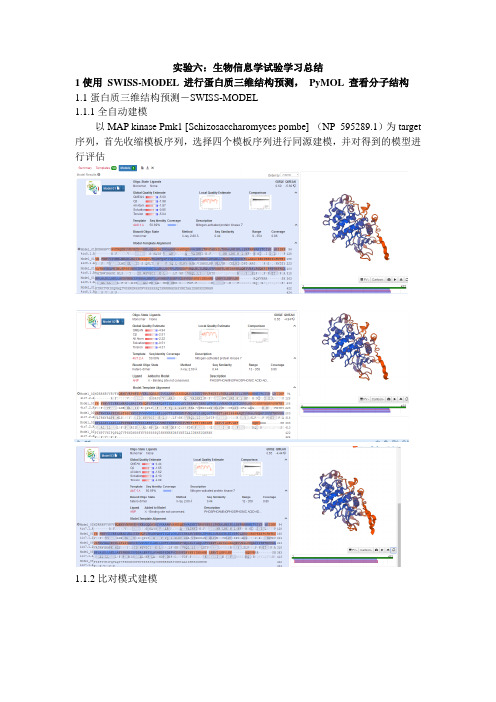
实验六:生物信息学试验学习总结1使用SWISS-MODEL 进行蛋白质三维结构预测,PyMOL 查看分子结构1.1蛋白质三维结构预测-SWISS-MODEL1.1.1全自动建模以MAP kinase Pmk1 [Schizosaccharomyces pombe] (NP_595289.1)为target 序列,首先收缩模板序列,选择四个模板序列进行同源建模,并对得到的模型进行评估1.1.2比对模式建模模型评估:GMQE(全局模型质量评估)是一种质量评估,它结合了目标-模板对齐和模板搜索方法的属性。
由此产生的GMQE分数表示为0到1之间的数字,反映了用该校准和模板构建的模型的预期精度。
较高的数字表明更高的可靠性。
一旦建立了模型,在这个特定的情况下,GMQE(1)就会得到更新,同时考虑到获得的模型的q 平均值,从而提高质量评估的可靠性。
QMEANQMEAN平均数(Benkert等)是基于不同几何属性的复合得分函数,并提供了全局的(即:对于整个结构)和局部(即每个残余物)绝对质量的估计是基于一个单一模型的。
z分数(2)提供了对模型中观察到的结构特征的“本土程度”的估计,并指出该模型是否具有与实验结构相似的质量。
较高的q均值z分数表明模型结构与相似尺寸的实验结构之间的一致性较好。
得分在0-4.0或以下的是一个质量很低的模型,这一点也可以通过在分数旁边的“拇指向下”符号的变化来突出显示。
QMEAN由四个单独的术语组成。
全球q平均值质量分数的四个单独术语也列在上面。
在巴图的白色区域(数值接近于零)表明这一特性与实验结构中所观察到的相似。
实证值表明,该模型平均得分高于实验结构,负数表明该模型平均得分低于实验结构。
q均值z分数本身显示在顶部。
单独的z分数比较了Cbeta原子之间的相互作用势,所有的原子,溶解势和扭转角度的潜力。
“局部质量”图显示了模型的每一个剩余部分(在x轴上报告),期望与本机结构(y轴)的相似性。
Swissmodel蛋白三维结构预测

Swissmodel蛋白三维结构预测swiss-model蛋白三维结构预测蛋白三维结构模型预测LHCGR基因NM_000233,c.547G>A,p.G183R蛋白质三维结构预测方法概览比较建模法比较建模又称同源建模,原理简单,是基于计划相关的序列具有相似的三维结构且进化过程中三维结构比序列保守的原理,利用计划相关模板结构信息建模。
基本步骤:1)将目标序列作为查询序列,在已知蛋白结构数据中搜索,确定和识别出一个同源模板。
2)将目标序列和模板结构进行比对。
3)以模板结构骨架为模型,建立目标蛋白质骨架模型。
4)对模型结果进行评价,确定模板的实用性。
查找蛋白序列LHCGR基因,NM_000233;exon14;c.547G>A(p.G183R);699aaNCBI数据库:https://w/查找蛋白序列LHCGR基因,NM_000233;exon14;c.547G>A(p.G183R);699aaSWISS-MODEL同源建模法--swiss-model:目前应用最广泛的比较建模法蛋白结构预测工具https://查询到的氨基酸序列复制到输入框,开始建模输入蛋白全序列构建模型,共2个结果页面解读:(1)首先看搜到的模板是否覆盖当前变异位点;(2)检查搜到的模板与输入序列一致度是否>30%(2)如果可用,再看swissmodel评分高低情况。
GMQE:可信度范围为0-1,值越大表明质量越好QMEAN4:区间-4-0,越接近0,评估待测蛋白与模板蛋白的匹配度越好。
模型下载下载后的蛋白结构预测模型可以用Swiss-pdbviewer查看ATCG用Swiss-pdbviewer打开pdb模型文件,在controlpanel里去掉氨基酸骨架和side显示,添加ribn显示在controlpanel的colR里选择添加ribbon(colR->ribbon)。
Color栏下选择SecondaryStructureSuccession根据二级机构进行着色进行着色。
蛋白质结构与功能预测

酸)、Met(甲硫氨酸)、Phe(苯丙氨酸)、Trp(色 氨酸)、Cys(半胱氨酸)、Ala(丙氨酸)、Pro(脯 氨酸)和Gly(甘氨酸) • 胞内-外分界区:Tyr(络氨酸)、 Trp(色氨酸)和Phe(苯丙氨酸) • 胞内末端:Lys(赖氨酸)和Arg(精氨酸)
数据: C:\ZCNI\shixi4\protein.txt
7
一、蛋白质基本理化性质分析
蛋白质理化性质是蛋白质研究的基础
蛋白质的基本性质:
相对分子质量 氨基酸组成
等电点(pI) 消光系数
半衰期
不稳定系数
总平均亲水性 ……
实验方法:
• 相对分子质量的测定、等电点实验、沉降实验 • 缺点:费时、耗资
基于实验经验值的计算机分析方法
算法,还考虑到蛋白质结构 分类信息
http://bioweb.pasteur.fr/seqanal/interfaces/pre 预测时考虑了氨基酸残基间
dator-simple.html
的氢键
29
蛋白质二级结构分析工具(续)
工具 PredictProtein
Prof PSIpred
SOPMA SSPRED
实习4:蛋白质结构与功能分析
阮陟
陈晓龙 胡杰峰 刘秋香
浙江加州国际纳米技术研究院(ZCNI)
实习课程内容
实习一
基因组数据注释和功能分析
基因组学
实习二
核苷酸序列分析
系
统
实习三
芯片的基本数据处理和分析
转录组学
生
物
实习四
蛋白质结构与功能分析
蛋白质结构预测

Bioinformatics
Liaoning University
SWISS-MODEL:基于同源建模法与PDB数据库已知 结构的蛋白质序列比对,使用同源建模方法预测蛋白质 结构的在线服务器。 网址:/
Bioinformatics
Liaoning University
Liaoning University
同源建模法的理论基础:序列相似性大于20%,长度大 于80个氨基酸的蛋白质具有相似的三维结构。
同源建模是目前最为成功且实用的蛋白质结构预测方法, 建模的前提是已知一个或多个同源蛋白质的结构。
同源建模的大体过程分为4个步骤:1鉴定结构模板;2 目标序列和模板结构比对;3建立模型;4模型质量评估。
Bioinformatics
Liaoning University
PSIPRED预测二级结构
粘贴蛋白质序列fasta
输入Email 输入一个名称 点击Predict预测
Bioinformatics
Liaoning University
pmRab7二级结构预测结果
/psipred/result/a728be72-d6b6-11e3-9c06-00163e110593 Bioinformatics
CATH
SCOP
Birsity
拜氏梭菌的黄素氧还蛋白在SCOP中的分类情况
• Root SCOP • Class a and b protein (a/b) • Fold Flavodoxin-like – Three layers, a/b/a; paraelle b-sheet of vie strands, order 21345 • Superfamily Flavoproteins • Family Flavodoxin-related. Binds FMN (flavin monoucleotide) • Protein Flavodoxin • Species Clostridium bejerinckii
献给初学者:手把手教你在线预测蛋白质结构「免费送PyMOL安装包」
献给初学者:手把手教你在线预测蛋白质结构「免费送PyMOL安装包」蛋白质是生命活动的体现者,其结构决定着功能。
由线性氨基酸组成的蛋白质需要折叠成特定的空间结构才具有相应的生理活性和生物学功能。
解析蛋白质的空间结构对于认识蛋白质的功能、功能的执行、生物大分子间的相互作用,以及医学和药学的发展「如药物靶点的设计等」具有重要意义。
为了更快速地了解蛋白质功能,不能只等待蛋白质的测定结果,尤其是对未知蛋白质开展研究之前,通过对蛋白质结构进行预测具有明显的优势。
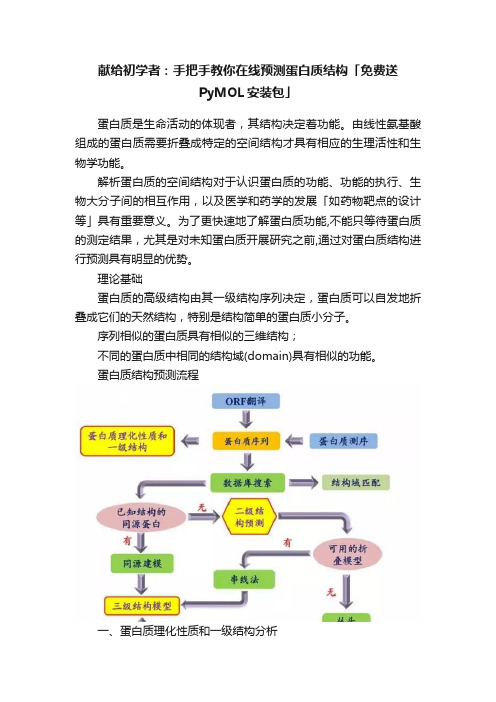
理论基础蛋白质的高级结构由其一级结构序列决定,蛋白质可以自发地折叠成它们的天然结构,特别是结构简单的蛋白质小分子。
序列相似的蛋白质具有相似的三维结构;不同的蛋白质中相同的结构域(domain)具有相似的功能。
蛋白质结构预测流程一、蛋白质理化性质和一级结构分析1. 分析蛋白质的 pI、Mw、氨基酸组成、消光系数、稳定系数等「1」进入 Expasy 主页:「2」点击Resource A…...Z「3」查找「ProtParam」「protein physical and chemical parameters」「4」粘贴序列进行分析。
2. 分析蛋白质的亲水性和疏水性「1」进入 Expasy 主页:「2」点击Resource A…...Z「3」查找「ProtScale」「protein profile computation and representation」「4」粘贴序列,选择分析方法,进行分析。
「5」蛋白质的亲水和疏水性分析结果,图形显示「亲水用负值表示,疏水用正值表示」。
3. 分析蛋白质的跨膜区「1」Tmpred 法分析蛋白质的跨膜区:基于对 Tmbase 数据库的统计分析来预测蛋白质跨膜区和跨膜方向。
「2」TMHMM 法分析蛋白质的跨膜区:基于 HMM 方法的蛋白质跨膜区预测工具。
4. 信号肽预测 SignalP预测的是分泌型的信号肽,而不是参与细胞内信号传递的蛋白。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
SWISS-MODEL 蛋白质结构预测
SWISS-MODEL是一项预测蛋白质三级结构的服务,它利用同源建模的方法实现对一段未知序列的三级结构的预测。
该服务创建于1993年,开创了自动建模的先河,并且它是讫今为止应用最广泛的免费服务之一。
同源建模法预测蛋白质三级结构一般由四步完成:
1. 从待测蛋白质序列出发,搜索蛋白质结构数据库(如PDB,SWISS-PROT等),得到许多相似序列
(同源序列),选定其中一个(或几个)作为待测蛋白质序列的模板;
2. 待测蛋白质序列与选定的模板进行再次比对,插入各种可能的空位使两者的保守位置尽量对齐;
3. 建模:调整待测蛋白序列中主链各个原子的位置,产生与模板相同或相似的空间结构——待测蛋白
质空间结构模型;
4. 利用能量最小化原理,使待测蛋白质侧链基团处于能量最小的位置。
最后提供给用户的是经过如上四步(或重复其中某几步)后得到的蛋白质三级结构。
SWISS-MODEL工作模式
SWISS-MODEL服务器是以用户输入信息的最小化为目的设计的,即在最简单的情况下,用户仅提供一条目标蛋白的氨基酸序列。
由于比较建模程序可以具有不同的复杂性,用户输入一些额外信息对建模程序的运行有时是有必要的,比如,选择不同的模板或者调整目标模板序列比对。
该服务主要有以下三种方式:
∙First Approach mode(简捷模式):这种模式提供一个简捷的用户介面:用户只需要输入一条氨基酸序列,服务器就会自动选择合适的模板。
或者,用户也可以自己指定模板(最多5条),这些模板可以来自ExPDB 模板数据库(也可以是用户选择的含坐标参数的模板文件)。
如果一条模板与提交的目标序列相似度大于25%,建模程序就会自动开始运行。
但是,模板的可靠性会随着模板与目标序列之间的相似度的降低而降低,如果相似度不到50%往往就需要用手工来调整序列比对。
这种模式只能进行大于25个残基的单链蛋白三维结构预测。
∙Alignment Interface(比对界面):这种模式要求用户提供两条已经比对好的序列,并指定哪一条是目标序列,哪一条是模板序列(模板序列应该对应于ExPDB模板数据库中一条已经知道其空间结构的蛋白序列)。
服务器会依据用户提供的信息进行建模预测。
∙Project mode(工程模式):手工操作建模过程:该模式需要用户首先构建一个DeepView工程文件,这个工程文件包括模板的结构信息和目标序列与模板序列间的比对信息。
这种模式让用户可以控制许多参数,例如:模板的选择,比对中的缺口位置等。
此外,这个模式也可以用于“first approach mode简捷模式”输出结果的进一步加工完善。
此外,SWISS-MODEL还具有其他两种内容上的模式:
∙Oligomer modeling(寡聚蛋白建模):对于具有四级结构的目标蛋白,SWISS-MODEL提供多聚模板的模式,用于多单体的蛋白质建模。
这一模式弥补了简捷模式中只能提交单个目标序列,不能同时预测两条及以上目标序列的蛋白三维结构的不足。
∙GPCR mode(G蛋白偶联受体模式):是专门对7次跨膜G蛋白偶联受体的结构预测。
First Approach mode(简捷模式)的使用在SWISS-MODEL主页中进行如下操作:
选择所需域值:
这里小数点后零的位数越多,在蛋白库中搜索时得到的结果与提交序列的同源性就越高。
SWISS-MODEL服务器的所有结果都将通过邮件的形式反馈用户,反馈结果的形式由用户自己选择,如图所示:
SWISS-MODEL服务器可以反馈给用户以下结果选项,spdbv模式、普通模式和简短模式。
这里,建议使用SWISS-MODEL默认的spdbv模式,这种模式可以用Swiss-PDBViewer程序打开,可以根据需要对结果进行多种操作。
结果:
以如下序列为例:
NIDRPKGLAFTDVDVDSIKIAWESPQGQVSRYRVTYSSPEDGIHELFPAPDGEEDTAELQ GLRPGSEYTVSVVALHDDMESQPLIGTQSTAIPA
域值选择:0.0000000001
结果选项:Swiss-PdbViewer mode
根据前面选项不同,可能会收到多封邮件。
