Bioconductor基因芯片数据分析系列(一):数据的读取
bioconductor系列教程之一分析基因芯片上
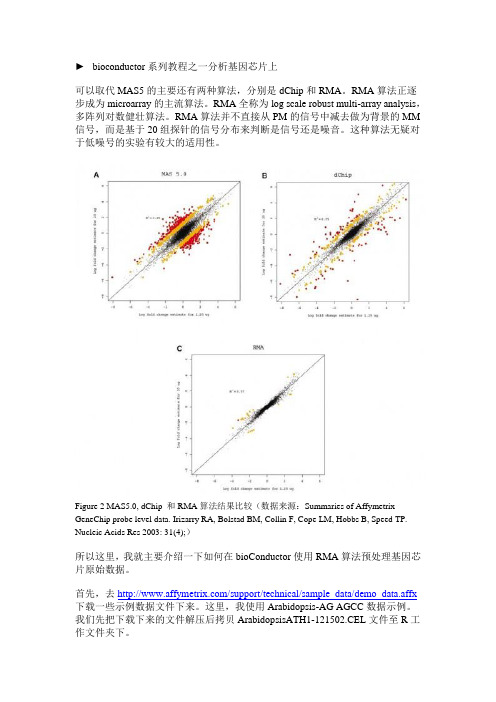
►bioconductor系列教程之一分析基因芯片上可以取代MAS5的主要还有两种算法,分别是dChip和RMA。
RMA算法正逐步成为microarray的主流算法。
RMA全称为log scale robust multi-array analysis,多阵列对数健壮算法。
RMA算法并不直接从PM的信号中减去做为背景的MM 信号,而是基于20组探针的信号分布来判断是信号还是噪音。
这种算法无疑对于低噪号的实验有较大的适用性。
Figure 2 MAS5.0, dChip 和RMA算法结果比较(数据来源:Summaries of Affymetrix GeneChip probe level data. Irizarry RA, Bolstad BM, Collin F, Cope LM, Hobbs B, Speed TP. Nucleic Acids Res 2003: 31(4);)所以这里,我就主要介绍一下如何在bioConductor使用RMA算法预处理基因芯片原始数据。
首先,去/support/technical/sample_data/demo_data.affx 下载一些示例数据文件下来。
这里,我使用Arabidopsis-AG AGCC数据示例。
我们先把下载下来的文件解压后拷贝ArabidopsisATH1-121502.CEL文件至R工作文件夹下。
首先是一个快速上手教程:1 2 3 4 5 6 7 8 910111213141516171819202122232425262728293031323334353637 > library(affy)##加载库文件Loading required package: BiobaseWelcome to BioconductorVignettes contain introductory material. To view, type'openVignette()'. To cite Bioconductor, see'citation("Biobase")'and for packages 'citation(pkgname)'.> Data <- ReadAffy()##读取工作目录下的CEL文件> eset <- rma(Data)##用RMA算法预处理数据,这时它会自动下载CDF文件,所以需要联网。
基因芯片的数据分析

基因表达谱芯片的数据分析基因芯片数据分析就是对从基因芯片高密度杂交点阵图中提取的杂交点荧光强度信号进行的定量分析,通过有效数据的筛选和相关基因表达谱的聚类,最终整合杂交点的生物学信息,发现基因的表达谱与功能可能存在的联系。
然而每次实验都产生海量数据,如何解读芯片上成千上万个基因点的杂交信息,将无机的信息数据与有机的生命活动联系起来,阐释生命特征和规律以及基因的功能,是生物信息学研究的重要课题[1]。
基因芯片的数据分析方法从机器学习的角度可分为监督分析和非监督分析,假如分类还没有形成,非监督分析和聚类方法是恰当的分析方法;假如分类已经存在,则监督分析和判别方法就比非监督分析和聚类方法更有效率。
根据研究目的的不同[2,3],我们对基因芯片数据分析方法分类如下。
(1)差异基因表达分析:基因芯片可用于监测基因在不同组织样品中的表达差异,例如在正常细胞和肿瘤细胞中;(2)聚类分析:分析基因或样本之间的相互关系,使用的统计方法主要是聚类分析;(3)判别分析:以某些在不同样品中表达差异显著的基因作为模版,通过判别分析就可建立有效的疾病诊断方法。
1 差异基因表达分析(difference expression, DE)对于使用参照实验设计进行的重复实验,可以对2样本的基因表达数据进行差异基因表达分析,具体方法包括倍数分析、t检验、方差分析等。
1.1倍数变化(fold change, FC)倍数分析是最早应用于基因芯片数据分析的方法[4],该方法是通过对基因芯片的ratio值从大到小排序,ratio 是cy3/cy5的比值,又称R/G值。
一般0.5-2.0范围内的基因不存在显著表达差异,该范围之外则认为基因的表达出现显著改变。
由于实验条件的不同,此阈值范围会根据可信区间应有所调整[5,6]。
处理后得到的信息再根据不同要求以各种形式输出,如柱形图、饼形图、点图等。
该方法的优点是需要的芯片少,节约研究成本;缺点是结论过于简单,很难发现更高层次功能的线索;除了有非常显著的倍数变化的基因外,其它变化小的基因的可靠性就值得怀疑了;这种方法对于预实验或实验初筛是可行的[7]。
生物信息学讲义——基因芯片数据分析

生物信息学讲义——基因芯片数据分析生物信息学是指运用计算机技术和统计学方法来解析和理解生物领域的大规模生物数据的学科。
基因芯片数据分析是生物信息学研究的一个重要方向,通过对基因芯片数据进行分析,可以揭示基因在生物过程中的功能和调节机制。
本讲义将介绍基因芯片数据的分析方法和应用。
一、基因芯片数据的获取与处理基因芯片是一种用于检测和测量基因表达水平的高通量技术,可以同时检测上千个基因的表达情况。
获取基因芯片数据的第一步是进行基因芯片实验,如DNA芯片实验或RNA芯片实验。
实验得到的数据一般为原始强度值或信号强度值。
接下来,需要对这些原始数据进行预处理,包括背景校正、归一化和过滤噪声等步骤,以消除实验误差和提高数据质量。
二、基因表达分析基因芯片数据的最主要应用之一是进行基因表达分析。
基因表达分析可以揭示在不同条件下基因的表达模式和差异表达基因。
常用的基因表达分析方法包括差异表达分析、聚类分析和差异共表达网络分析等。
差异表达分析常用来寻找在不同条件下表达差异显著的基因,如差异表达基因的筛选和注释;聚类分析可以将表达模式相似的基因分为一组,如聚类分析可以将不同样本中的基因按照表达模式进行分类;差异共表达网络分析可以找到一组在差异表达样本中共同表达的基因,揭示潜在的功能模块。
三、功能富集分析对差异表达基因进行功能富集分析可以帮助我们理解这些基因的生物学功能和参与的生物过程。
功能富集分析可以通过对差异表达基因进行GO(Gene Ontology)注释,找到在特定条件下富集的生物学过程、分子功能和细胞组分等。
另外,功能富集分析还可以进行KEGG(Kyoto Encyclopedia of Genes and Genomes)富集分析,找到差异表达基因在代谢通路和信号传导通路中的富集情况。
四、基因调控网络分析基因调控网络分析可以帮助我们揭示基因间的调控关系和寻找关键调控基因。
基因调控网络是基于差异表达数据构建的,它可以包括转录因子-靶基因调控网络和miRNA-mRNA调控网络等。
bioconductor分析芯片数据教程

bioconductor分析芯片数据教程wangpeng905 2017.04.21 09:21* 这是我在The Bioinformatics Knowledgeblog 上看到的一篇教程,原文在这里,教程条理清晰,对我理解芯片数据分析流程帮助很大,就把它翻译了过来。
介绍芯片数据分析流程有些复杂,但使用R 和Bioconductor 包进行分析就简单多了。
本教程将一步一步的展示如何安装R 和Bioconductor,通过GEO 数据库下载芯片数据,对数据进行标准化,然后对数据进行质控检查,最后查找差异表达的基因。
教程示例安装的各种依赖包和运行命令均是是在Ubuntu 环境中运行的(版本:Ubuntu 10.04,R 2.121),教程的示例代码和图片在这里。
安装R 和Bioconductor 包打开命令终端,先安装R 和Bioconductor 的依赖包,然后安装R.$ sudo apt-get install r-base-core libxml2-devlibcurl4-openssl-dev curl$ R之后在R 环境中安装Bioconductor 包> # 下载Bioconductor 的安装程序> source("/biocLite.R")> # 安装Bioconductor 的核心包> biocLite()> # 安装GEO 包> biocLite("GEOquery")如果你没有管理员权限,你需要将这些包安装到你个人库目录中。
安装Bioconductor 需要一段时间,GEOquery 包也需要安装,GEOquery 是NCBI 存储标准化的转录组数据的基因表达综合数据库GEO 的接口程序。
下载芯片数据本教程中我们使用Dr Andrew Browning 发表的数据集GSE20986。
生物信息学讲义——基因芯片数据分析资料

生物信息学讲义——基因芯片数据分析资料基因芯片是一种高通量的技术,可以用于同时检测和量化数以千计的基因在一个样本中的表达水平。
通过分析基因芯片数据,我们可以获得大量的基因表达信息,并进一步了解基因在不同条件和疾病状态下的调控和功能。
下面是一份关于基因芯片数据分析的讲义。
一、基因芯片数据的处理与预处理1.数据获取与质控-从基因芯片实验中获取原始数据(CEL文件)。
-进行质控,包括检查芯片质量、样本质量和数据质量。
2.数据预处理-背景校正:去除背景信号,减小非特异性杂音。
-样本标准化:对样本间进行标准化处理,消除技术变异和样本间差异。
-基因过滤:去除低表达和不变的基因,减少多重检验问题。
二、差异基因分析1.统计分析-基于统计学的差异表达分析方法,如t检验、方差分析(ANOVA)等。
-根据差异分析结果,获取差异表达的基因列表。
2.功能注释与生物学解释-对差异表达的基因进行功能注释,包括富集分析、通路分析和基因功能类别分析等。
-通过生物学数据库查询和文献阅读,解释差异表达基因的生物学意义和可能的调控机制。
三、基因共表达网络分析1.相关性分析-计算基因间的相关系数,筛选出相关性较高的基因对。
-构建基因共表达网络,通过网络可视化方式展示基因间的关系。
2.模块发现和功能注释-使用聚类算法将基因分组成不同的模块,每个模块表示一组具有相似表达模式的基因。
-对每个模块进行功能注释,了解模块内基因的共同功能或通路。
四、基因云图和热图分析1.基因云图-使用基因注释信息和基因表达水平,绘制基因表达的云图。
-通过颜色和大小表示基因的表达水平、功能注释等信息。
2.热图分析-根据基因表达水平计算基因间的相似性,将相似性转换为颜色,绘制热图。
-热图可用于显示基因表达模式的相似性和差异。
五、整合分析与生物信息学工具1.基因集富集分析-将差异表达的基因列表输入基因富集分析工具,寻找与特定通路、功能或疾病相关的基因集。
2.数据可视化工具- 使用生物信息学工具和软件,如R、Bioconductor、Cytoscape等,进行数据可视化和交互式分析。
基因表达谱芯片数据分析及其Bioconductor实现

基因表达谱芯片数据分析及其Bioconductor实现1.表达谱芯片及其应用表达谱DNA芯片(DNA microarrays for gene expression profiles)是指将大量DNA片段或寡核昔酸固定在玻璃、硅、塑料等硬质载体上制备成基因芯片,待测样品中的mRNA被提取后,通过逆转录获得cDNA,并在此过程中标记荧光,然后与包含上千个基因的DNA芯片进行杂交反应30min~20h后,将芯片上未发生结合反应的片段洗去,再对玻片进行激光共聚焦扫描,测定芯片上个点的荧光强度,从而推算出待测样品中各种基因的表达水平。
用于硏究基因表达的芯片可以有两种:①cDNA芯片;② 寡核昔酸芯片。
cDNA芯片技术及载有较长片段的寡核昔酸芯片采用双色荧光系统:U前常用Cy3—dUTP (绿色)标记对照组mRNA, Cy5—dUTP (红色)标记样品组mRNAUl。
用不同波长的荧光扫描芯片,将扫描所得每一点荧光信号值自动输入计•算机并进行信息处理,给出每个点在不同波长下的荧光强度值及其比值(ratio值),同时计算机还给出直观的显色图。
在样品中呈高表达的基因其杂交点呈红色,相反,在对照组中高表达的基因其杂交点呈绿色,在两组中表达水平相当的显黄色,这些信号就代表了样品中基因的转录表达情况⑵。
基因芯片因具有高效率,高通量、高精度以及能平行对照研究等特点,被迅速应用于动、植物和人类基因的研究领域,如病原微生物毒力相关基因的。
基因表达谱可直接检测mRNA的种类及丰度,可以同时分析上万个基因的表达变化,来揭示基因之间表达变化的相互关系。
表达谱芯片可用于研究:①同一个体在同一时间里,不同基因的表达差异。
芯片上固定的已知序列的cDNA或寡聚核昔酸最多可以达到30 000多个序列,与人类全基因组基因数相当,所以基因芯片一次反应儿乎就能够分析整个人的基因⑶。
②同一个体在不同时间里,相同基因的表达差异。
③不同个体的相同基因表达上的差异。
基因芯片原理及数据分析01

基因芯片数据分析流程
生物学问题 实验设计 芯片实验 图像采集和处理(图像分析) 预处理和标准化 聚类分析 差异表达基因分析 判别分析 基因网络分析
生物学解释和验证
基因芯片数据分析
基因芯片数据的预处理是一个十分关键的步
骤,通过数据过滤获取需要的数据、数据转 换满足正态分布的分析要求、缺失值的估计 弥补不完整的数据、数据归一化纠正系统误 差等处理为后续分析工作做准备,预处理分 析的重要性并不亚于基因芯片的后续分析, 它将直接影响后续分析是否能得到预期的结 果 ,Arraytools
基因芯片原理及数据分析
杨德印 生物信息学系
参考教材和资料
《基因芯片数据分析与处理》李瑶 化学工业出版社 2006年 《生物芯片分析》 [美]M.谢纳 著 科学出版社 《DNA芯片技术的方法与应用》 马文丽 郑文岭 广东科 技出版社 《生物芯片技术》 邢婉丽 程京 清华大学出版社 《生物芯片技术》 陈忠斌 化学工业出版社 《基因芯片与功能基因组》 李瑶 化学工业出版社 Google,ncbi,endnote:网络资源,文章(Paper) 相关关键词microarray,gene chip,gene expression
数据
数据表示:常用矩阵表示,即行列表示
含义 主要基因芯片数据库 smd,Geo(www.ncbi,/geo),EBI ArrayExpress
Outline
得到矩阵后?
芯片数据:众多基因的时空表达情况 基因表达模式------聚类 差异表达基因筛选(疾病相关基因筛选) 疾病类型识别 网络分析:通过芯片数据找出基因之间的 相互作用 基因注释 其他
内容
基因芯片技术(概念、制作过程、应用等) 基因芯片数据分析一般流程和主要内容
基因芯片数据处理流程与分析介绍

基因芯片数据处理流程与分析介绍关键词:基因芯片数据处理当人类基因体定序计划的重要里程碑完成之后,生命科学正式迈入了一个后基因体时代,基因芯片(microarray)的出现让研究人员得以宏观的视野来探讨分子机转。
不过分析是相当复杂的学问,正因为基因芯片成千上万的信息使得分析数据量庞大,更需要应用到生物统计与生物信息相关软件的协助。
要取得一完整的数据结果,除了前端的实验设计与操作的无暇外,如何以精确的分析取得可信数据,运筹帷幄于方寸之间,更是画龙点睛的关键。
基因芯片的应用基因芯片可以同时针对生物体内数以千计的基因进行表现量分析,对于科学研究者而言,不论是细胞的生命周期、生化调控路径、蛋白质交互作用关系等等研究,或是药物研发中对于药物作用目标基因的筛选,到临床的疾病诊断预测,都为基因芯片可以发挥功用的范畴。
基因表现图谱抓取了时间点当下所有的动态基因表现情形,将所有的探针所代表的基因与荧光强度转换成基本数据(raw data)后,仿如尚未解密前的达文西密码,隐藏的奥秘由丝丝的线索串联绵延,有待专家抽丝剥茧,如剥洋葱般从外而内层层解析出数千数万数据下的隐晦含义。
要获得有意义的分析结果,恐怕不能如泼墨画般洒脱随兴所致。
从raw data取得后,需要一连贯的分析流程(图一),经过许多统计方法,才能条清理明的将raw data整理出一初步的分析数据,当处理到取得实验组除以对照组的对数值后(Iog2 ratio),大约完成初步的统计工作,可进展到下一步的进阶分析阶段。
Rosetta profile error model calculation2Sqweeze replicated probes^Normalize intensities (exclude flagged ^nd wontroldata) with median scaling"Basic statistic plot and Pearson correlationcoefficient^Combine tech nicar repeatPairwise ratid calculation图一、整体分析流程。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Bioconductor基因芯片数据分析系列(一):R包中数据的读取
R软件的Bioconductor包是分析芯片数据的神器,今天小编打算推出芯片数据的系列教程。
首先讲数据读取,以CLL数据包中的数据为例。
打开R studio。
#安装所需的R包以及CLL包,注意大小写,一般函数都是小写的
source("/biocLite.R");
biocLite(“CLL”)
图1.显示已经安装好Bioconductor了,版本为3.4
#打开CLL包
library(CLL)
图2.显示打开CLL成功
图3.右侧栏内可见看到目前载入的程序包
data(CLLbatch)
#调用RMA算法对数据预处理
CLLrma<-rma(CLLbatch)
#读取处理后所有样品的基因表达值
e<- exprs(CLLrma)
#查看数据
e
我们可以看到,CLL数据集中共有24个样品(CLL10.CEL, CLL11.CEL, CLL12.CEL, 等),此数据集的病人分为两组:稳定组和进展组,采用的设计为两组之间的对照试验(Control Test)。
从上面的结果可知,Bioconductor具有强大的数据预处理能力和调用能力,仅仅用了6行代码就完成了数据的读取及预处理。
Bioconductor基因芯片数据分析系列(二):GEO下载数据CEL的读取首先得下载一个数据,读取GEO的CEL文件采用如下命令:
登陆pubmed,找到一个你感兴趣的数据库
在底下栏目下载CEL文件
打开R软件
#安装所需的R包以及CLL包,注意大小写,一般函数都是小写的
source("/biocLite.R");
biocLite(“CLL”)
>library(affy)
>affybatch<- ReadAffy(celfile.path = "GSE36376_RAW")
请注意目录的路径,在window下,反斜杠‘\’要用转义字符“\\”表示。
然后可以使用RMA或者MAS5等方法对数据进行background.correction, normaliztion, pm.correct等等一系列处理。
如果你一切用默认参数,则可以使用如下命令:
>eset<- rma(affybatch),or eset<- mas5(affybatch)
>exp<- exprs(eset)
exp就是数字化的表达谱矩阵了
请注意,rma只使用匹配探针(PM)信号,exp数据已经进行log2处理。
mas5综合考虑PM和错配探针(MM)信号,exp数据没有取对数。
下一期就得等到2017年春节期间啦,敬请期待~
另外一种是直接利用GEO上面的GEO2R按钮里面的R script下载文件:
# Version info: R 3.2.3, Biobase 2.30.0, GEOquery 2.40.0, limma 3.26.8
# R scripts generated Mon Dec 26 06:54:42 EST 2016 Server:
Query:
acc=GSE36376&platform=GPL10558&type=txt&groups=&color s=&selection=XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX XXXXXXXXXXXXXXXXXXXXXX&padj=fdr&logtransform=auto&col umns=ID&columns=adj.P.Val&columns=P.Value&columns=F&c
olumns=Gene+symbol&columns=Gene+title&num=250&annot=n cbi
# Unable to generate script analyzing differential expression.
# Invalid input: at least two groups of samples should be selected.
##################################################### ###########
# Boxplot for selected GEO samples
library(Biobase)
library(GEOquery)
# load series and platform data from GEO
gset<- getGEO("GSE36376", GSEMatrix =TRUE,
getGPL=FALSE)
if (length(gset) > 1) idx<- grep("GPL10558", attr(gset, "names")) else idx<- 1
gset<- gset[[idx]]
# set parameters and draw the plot
dev.new(width=4+dim(gset)[[2]]/5, height=6)
par(mar=c(2+round(max(nchar(sampleNames(gset)))/2),4, 2,1))
title<- paste ("GSE36376", '/', annotation(gset), " selected samples", sep ='')
boxplot(exprs(gset), boxwex=0.7, notch=T, main=title, outline=FALSE, las=2)。
