真核生物基因结构的预测分析方法(软件)
蛋白质结构与功能分析
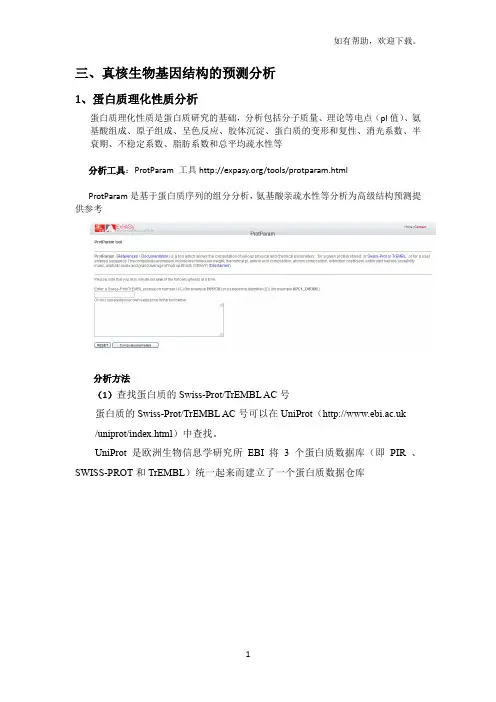
三、真核生物基因结构的预测分析1、蛋白质理化性质分析蛋白质理化性质是蛋白质研究的基础,分析包括分子质量、理论等电点(pI值)、氨基酸组成、原子组成、呈色反应、胶体沉淀、蛋白质的变形和复性、消光系数、半衰期、不稳定系数、脂肪系数和总平均疏水性等分析工具:ProtParam 工具/tools/protparam.htmlProtParam是基于蛋白质序列的组分分析,氨基酸亲疏水性等分析为高级结构预测提供参考分析方法(1)查找蛋白质的Swiss-Prot/TrEMBL AC号蛋白质的Swiss-Prot/TrEMBL AC号可以在UniProt( /uniprot/index.html)中查找。
UniProt是欧洲生物信息学研究所EBI 将3个蛋白质数据库(即PIR 、SWISS-PROT和TrEMBL)统一起来而建立了一个蛋白质数据仓库在搜索框输入蛋白质名称(如Pichia pastoris Agglutinin-like protein 3)→Find(2)如果需要分析的蛋白是SWISS-PROT和TrEMBL数据库中已收录的蛋白质,则在输入蛋白质的Swiss-Prot/TrEMBL AC号(accession number)→点击“Compute parameters”(3)如果需要分析的是未知序列,则需在搜索框中粘贴氨基酸序列,返回结果即可得出结果分析:2、跨膜区分析使用工具:TMpredTMpred,它依靠一个跨膜蛋白数据库Tmbase(Hofmann和Stoffel,1993)。
Tmbase来源与Swiss-Prot库,并包含了每个序列的一些附加信息:跨膜结构区域的数量、跨膜结构域的位置及其侧翼序列的情况。
Tmpred利用这些信息并与若干加权矩阵结合来进行预测。
分析方法Tmpred的Web界面十分简明。
用户将单字符序列输入查询序列文本框,并可以指定预测时采用的跨膜螺旋疏水区的最小长度和最大长度。
研究miRNA,这些数据库你必须得知道!常用数据库汇总

研究miRNA,这些数据库你必须得知道!常⽤数据库汇总miRNA是近年来在多种真核细胞及病毒中发现的⼀类来源内源性染⾊体上的⾮编码单链RNA,长度为21~25nt的短序列,在进化上具有⾼度的保守性,能够通过与靶mRNA特异性的碱基互补配对,引起靶mRNA降解或者抑制其翻译,从⽽对基因进⾏转录后的表达调控。
miRNA由⼀段具有发夹环结构的长度为70~80个核苷酸的miRNA前体(pre-miRNA)剪切后⽣成。
它通过与其⽬标mRNA分⼦的3′端⾮编码区域(3-untranslated region, 3′ UTR)互补导致该mRNA分⼦的翻译受到抑制。
最先发现的miRNAs是线⾍中控制发育时序的lin-4和let-7基因。
现已发现miRNA⼴泛地存在哺乳动物、线⾍、果蝇和植物等⽣物中。
除了lin-4和let-7基因外,其他miRNAs现在统⼀⽤miR-#表⽰miRNA,同⼀物种内相同或极相近似的miRNA可以使⽤相同的数字,只是进⼀步在数字之后加数字或字母作为后缀以区别其基因在序列上只有微⼩的差别。
尽管miRNA基因不编码蛋⽩质,但其编码的RNA在⽣物的整个⽣命过程中发挥着重要作⽤。
miRBasemiRBase数据库是⼀个提供包括已发表的miRNA序列数据、注释、预测基因靶标等信息的全⽅位数据库,是存储miRNA信息最主要的公共数据库之⼀。
该数据库于2014年6⽉更新为最新版本V21.0,包含223个物种的35828个成熟的miRNA序列。
该数据库提供便捷的⽹上查询服务,允许⽤户使⽤关键词或序列在线搜索已知的miRNA和靶标信息(仅包含已有的靶标信息,所以会出现部分miRNA靶标信息⽆的现象)。
该数据库⽤于miRNA信息查询较多,靶关系预测较少。
TargetScanTargetScan数据库是⼤家⽐较常⽤的预测miRNA靶基因数据库,主要通过搜索和每条miRNA种⼦区域匹配的保守的8mer和7mer位点来预测靶基因。
基因数据分析的主流软件

基因数据分析的主流软件在过去的几年中,许多生物的基因组完成了测序工作,如何对如此庞大的原始序列信息进行分析和应用,正是现在最为棘手的问题。
大量的基因预测软件和在线工具应运而生。
如何广泛而深入地了解并能有的放矢地利用这些工具,已经成为21世纪分子生物学家的必修课。
随着大规模EST和cDNA序列信息的获取,那些基于表达序列同源范围的程序,在基因组注释中的作用日益显著。
即使在稀少基因或组织特异性表达的基因中,基因组序列的相关性信息也颇具参考价值。
所以利用基因组序列的比对来扩充基因的信息是不可获缺的。
特别是在对人类基因组做注释时,与那些相对完整的脊椎动物基因组,如小鼠和鱼类的基因组比较是必不可少的步骤。
许多基因组测序计划正在进行之中,尽管仍存在急需解决的问题,比较基因组学方法(comparative genome approach)被认为是最有应用前景的方法。
该方法不仅在基因预测中举足轻重,而且在鉴定调控基因、探索垃圾基因(junk gene)等方面的作用也不容忽视。
基因预测软件的用户应该认识到,软件预测结果的可靠性和置信水平都有较大程度的提升。
但这些毕竟是预测的结果,分子生物学家,总是试图证明真实存在的蛋白质,及其功能和在组织中的表达状态。
当前,已有超过60种真核基因组测序计划在进程之中。
然而生物学方面的相关注释还远不能匹配如此汹涌而至的原始序列数据。
当务之急是,研发出更多的准确而快速的分析工具,特别在寻找基因、确定其准确功能等应用方面。
许多基因预测程序都可以免费共享。
当前,几乎没有一个完美的程序可以解决用户们的所有问题。
这就需要用户最大程度地利用主流程序的整合优势。
基因数目预测的主流软件10年前,研究人员开始预测人类基因的数目,这个数目在很长时间没有明显改变。
几年前,最多的预测是100,000;当人类基因组完成测序时,这个数目降至30,000。
现在有降至20,000左右。
研究人员相信:充分考虑人类的基因组序列和其它生物的基因组序列,可以做出近似的估计。
(完整word版)生物信息学填空题(个人整理)

(完整word版)生物信息学填空题(个人整理)1、BLAST教案所程序中,哪个方法是不存在的?(D)A:BLASTP B:BLASTN C:BLASTX D:BLASTQ2、下列哪个软件不是常用来观察蛋白质结构视图的?(D)A:AVS B:Chimera C:MICE D:HMM3、下列哪个不是点突变的类型?(A)A:染色体畸变 B:错义突变 C:无义突变 D:移码突变4、基因突变的效应不包括:(C)A:有利突变 B:中性突变 C:移码突变D:遗传多态现象5、人类基因组的结构特点不包括:(A)A:基因进化 B:基因数目 C:基因重复序列 D:基因组复制6、世界上三大数据库不包括:(B)A:NCBI B:BLAST C:UCSC D:Ensembl7、常用序列比对方法错误的是:(C)A:编辑距离 B:点阵描图 C:局部比对 D:记分模式8、下列哪个不是蛋白质结构模型?(D)A:同源性模型B:折叠识别C:ab initio折叠D:MoLScript 结构9、下列哪个选项不是微阵列实验设计的内容?(A)A:贝叶斯网络法 B:对照组的选择 C:重复样本的使用 D:随机化原则10、构建序列进化树的一般步骤不包括:(A)A:建立DNA文库 B:建立数据模型 C:建立取代模型 D:建立进化树11、下列中属于一级蛋白质结构数据库的是:(C)A. EMBLB. DDBJC. PDBD.SWISS-PROT12.蛋白质结构预测分为:(B)A.一级和三级结构预测 B. 二级和空间结构预测C. 三级和空间结构预测D. 二级和三级结构预测13.数据挖掘的四个步骤不包括下列哪个:(C)A. 数据选择B. 数据转换C. 数据记录D. 结果分析14.下列哪项不是生物学研究必备的工具:(A)A.数据分析B.数据统计C.因素分析D.多元回归分析15.Linux中rmdir 命令的功能是:(D)A.改变工作目录 B.删除工作目录C. 创建目录D.删除空目录16.BLAST教案所程序中,哪个方法是不存在的?(D)A:BLASTP B:BLASTN C:BLASTX D:BLASTQ17.下列哪个不是蛋白质结构模型?(D)A:同源性模型B:折叠识别C:ab initio折叠D:MoLScript 结构18.人类基因组的结构特点不包括:(A)A:基因进化 B:基因数目 C:基因重复序列 D:基因组复制19、下列哪个选项不是微阵列实验设计的内容?(A)A:贝叶斯网络法 B:对照组的选择 C:重复样本的使用 D:随机化原则20、构建序列进化树的一般步骤不包括:(A)A:建立DNA文库 B:建立数据模型 C:建立取代模型 D:建立进化树三、填空题1、数据格式的建立、数据的准确性和质量控制、方便的数据搜寻方式以及数据的及时更新是数据库建立和维护中的重要问题。
核酸序列分析软件介绍

核酸序列分析1、核酸序列检索可通过NCBI使用Entrez系统进行检索,也可用EBI的SRS服务器进行检索。
在同时检索多条序列时,可通过罗逻辑关系式按照GenBank接受号进行批量检索。
如用“AF113671 [ac] OR AF113672 [ac]”可同时检索这两条序列。
其中“[ac]”是序列接受号的描述字段。
2、核酸序列的基本分析(1)分子质量、碱基组成、碱基分布分子质量、碱基组成、碱基分布可通过一些常用软件等直接获得。
如:BioEdit(/BioEdit/bioedit.html),DNAMAN()。
(2)序列变换进行序列分析时,经常需要对DNA序列进行各种变换,例如反向序列、互补序列、互补反向序列、显示DNA双链、转换为RNA序列等。
这些用DNAMAN软件可很容易实现,这些功能集中在Sequence→Display,从中可选择不同的序列变换方式对当前通道的序列进行转换。
(3)限制性酶切分析该方面最好的资源是限制酶数据库(Restriction Enzyme Database,REBASE)。
REBASE数据库(,/rebase)中含有限制酶的所有信息,包括甲基化酶、相应的微生物来源、识别序列位点、裂解位点、甲基化特异性、酶的商业来源及公开发表的和未发表的参考文献。
其它资源还有:WebGene:/~tjyin/WebGene/RE.html,/personal/tyin.htmlWebCutter2:http://www//firstmarkert/firstmarket/cutter/cut2.html同时,很多软件也能够识别REBASE限制酶数据库。
强烈推荐使用集成化的软件如BioEdit和DNAMAN等。
所得出的结果给出指定DNA序列的酶切位点信息,为克隆鉴定和亚克隆提供了重要信息。
在实际进行分子生物学实验中,有时需要对多条相关序列(如发生突变的一批序列)同时进行酶切分析,以便为后续的克隆鉴定提供参考。
生物信息学题库

■一、选择题:1.以下哪一个是mRNA条目序列号: A. J01536■. NM_15392 C. NP_52280 D. AAB1345062.确定某个基因在哪些组织中表达的最直接获取相关信息方式是:■. Unigene B. Entrez C. LocusLink D. PCR3.一个基因可能对应两个Unigene簇吗?■可能 B. 不可能4.下面哪种数据库源于mRNA信息:■dbEST B. PDB C. OMIM D. HTGS5.下面哪个数据库面向人类疾病构建: A. EST B. PDB ■. OMIM D. HTGS6.Refseq和GenBank有什么区别: A. Refseq包括了全世界各个实验室和测序项目提交的DNA序列B. GenBank提供的是非冗余序列■. Refseq源于GenBank,提供非冗余序列信息D. GenBank源于Refseq7.如果你需要查询文献信息,下列哪个数据库是你最佳选择: A. OMIM B. Entrez ■PubMed D. PROSITE8.比较从Entrez和ExPASy中提取有关蛋白质序列信息的方法,下列哪种说法正确:A. 因为GenBank的数据比EMBL更多,Entrez给出的搜索结果将更多B. 搜索结果很可能一样,因为GenBank和EMBL的序列数据实际一样■搜索结果应该相当,但是ExPASy中的SwissProt记录的输出格式不同9.天冬酰胺、色氨酸和酪氨酸的单字母代码分别对应于:■N/W/Y B. Q/W/Y C. F/W/Y D. Q/N/W10.直系同源定义为:■不同物种中具有共同祖先的同源序列B. 具有较小的氨基酸一致性但是有较大的结构相似性的同源序列C. 同一物种中由基因复制产生的同源序列D. 同一物种中具有相似的并且通常是冗余的功能的同源序列11.下列那个氨基酸最不容易突变: A. 丙氨酸B. 谷氨酰胺 C. 甲硫氨酸■半胱氨酸12.PAM250矩阵定义的进化距离为两同源序列在给定的时间有多少百分比的氨基酸发生改变: A. 1% B. 20%■. 80% D. 250%13.下列哪个句子最好的描述了两个序列全局比对和局部比对的不同:A. 全局比对通常用于比对DNA序列,而局部比对通常用于比对蛋白质序列B. 全局比对允许间隙,而局部比对不允许C. 全局比对寻找全局最大化,而局部比对寻找局部最大化■全局比对比对整体序列,而局部比对寻找最佳匹配子序列14.假设你有两条远源相关蛋白质序列。
2012生物信息学题库(1)(2)
■一、选择题:1.以下哪一个是mRNA条目序列号: A. J01536■. NM_15392 C. NP_52280D. AAB1345062.确定某个基因在哪些组织中表达的最直接获取相关信息方式是:■. Unigene B.Entrez C. LocusLink D. PCR3.一个基因可能对应两个Unigene簇吗?■可能 B. 不可能4.下面哪种数据库源于mRNA信息:■dbEST B. PDB C. OMIM D.HTGS5.下面哪个数据库面向人类疾病构建: A. EST B. PDB ■. OMIMD. HTGS6.Refseq和GenBank有什么区别: A. Refseq包括了全世界各个实验室和测序项目提交的DNA序列B. GenBank提供的是非冗余序列■. Refseq源于GenBank,提供非冗余序列信息D. GenBank源于Refseq7.如果你需要查询文献信息,下列哪个数据库是你最佳选择: A. OMIM B. Entrez■PubMed D. PROSITE8.比较从Entrez和ExPASy中提取有关蛋白质序列信息的方法,下列哪种说法正确:A. 因为GenBank的数据比EMBL更多,Entrez给出的搜索结果将更多B. 搜索结果很可能一样,因为GenBank和EMBL的序列数据实际一样■搜索结果应该相当,但是ExPASy 中的SwissProt记录的输出格式不同9.天冬酰胺、色氨酸和酪氨酸的单字母代码分别对应于:■N/W/Y B. Q/W/YC. F/W/YD. Q/N/W10.直系同源定义为:■不同物种中具有共同祖先的同源序列B. 具有较小的氨基酸一致性但是有较大的结构相似性的同源序列C. 同一物种中由基因复制产生的同源序列D. 同一物种中具有相似的并且通常是冗余的功能的同源序列11.下列那个氨基酸最不容易突变: A. 丙氨酸 B. 谷氨酰胺 C. 甲硫氨酸■半胱氨酸12.PAM250矩阵定义的进化距离为两同源序列在给定的时间有多少百分比的氨基酸发生改变: A. 1% B. 20%■. 80% D. 250%13.下列哪个句子最好的描述了两个序列全局比对和局部比对的不同:A. 全局比对通常用于比对DNA序列,而局部比对通常用于比对蛋白质序列B. 全局比对允许间隙,而局部比对不允许C. 全局比对寻找全局最大化,而局部比对寻找局部最大化■全局比对比对整体序列,而局部比对寻找最佳匹配子序列14.假设你有两条远源相关蛋白质序列。
大学分子生物学考试(试卷编号191)
大学分子生物学考试(试卷编号191)1.[单选题]指导蛋白质进入细胞核的信号序列称为A)核定位信号B)核输出信号:C)分子伴侣D)转录因子答案:A解析:2.[单选题]“下 列哪种修饰通常作为真核生物胞内蛋白质被蛋白酶体选择性降解的先兆A)泛酰化B)甲基化C)乙酰化D)糖基化答案:A解析:3.[单选题]()在产生遗传多样性的过程中非常重要。
A)翻译B)转录C)重组D)转化答案:C解析:4.[单选题]拓扑异构酶不能A)参与DNA复制B)改变DNA的一级结构C)改变DNA的空间构象D)释放DNA中额外的张力答案:B解析:5.[单选题]G.riffith在 肺炎双球菌试验中,加热杀死的S型菌中什么物质使R型菌变得具有致死性?A)DNAB)RNAC)蛋白质D)其他大分子6.[单选题]关于断裂基因,以下说法正确的是A)含有内含子的基因称为不连续基因或断裂基因B)只有 真核生物的基因是以断裂基因的形式存在的C)基因的编码区 称为内含子D)在编码序列中插有与编码氨基酸无关的间隔区,称为外显子答案:A解析:7.[单选题]关于真核生物结构基因的转录,正确的说法是A)产物多为多顺反子RNAB)产物多为单顺反子RNAC)不连续转录D)对称转录答案:B解析:8.[单选题]含有稀有碱基最多的RNA是下列哪种RNA:A)tRNAB)mRNAC)rRNAD)siRNA答案:A解析:9.[单选题]下列不属于复制起始区的特征的是( )A)由多个短的重复序列组成B)常富含AT序列C)能够被特定的复制起始区结合蛋白识别并结合D)原核生物中-10区影响DNA复制的频率答案:D解析:10.[单选题]通常,DNA的复制起始区富含______碱基。
A)GCB)AGC)CTD)AT答案:D解析:B)乙酰化C)泛酰化D)生物素化答案:B解析:12.[单选题]在细菌翻译系统中,起始密码子的识别依赖于( ) 5’端SD序列和 ( )3’端反SD序列之间的相互作用。
生物数据库介绍——NCBI
⽣物数据库介绍——NCBINCBI(National Center for Biotechnology Information,美国国家⽣物技术信息中⼼)除了维护GenBank核酸序列数据库外,还提供数据分析和检索资源。
NCBI资源包括Entrez、Entrez编程组件、MyNCBI、PubMed、PudMed Central、PubReader、Gene、the NCBI Taxonomy Browser、BLAST、Pimer-Blast、COBALT、RefSeq、UniGene、HomoloGene、ProtEST、dbMHC、dbSNP、dbVar、Epigenomics、the Genetic Testing Registry、Genome和相关⼯具、⽐对查看器、跟踪存档、Sequence Read Archive、BioProject、BioSample、ClinVar、MedGen、HIV-1/⼈类蛋⽩质相互作⽤数据库、Gene Expression Omnibus、Probe、Online Mendelian Inheritance in Animals、the Molecular Modeling Database、the Conserved Domain Database、the Conserved Domain Architecture Retrieval Tool、Biosystem、Protein Clusters and thePubChem suite of small molecule databases,所有这些资源可以在NCBI主页找到。
Databases⼀个提供有关基因组组装结构,装配名称和其他元数据,统计报告以及基因组序列数据链接等信息的数据库。
⼀个有关培养物、动植物样本和其他⾃然样本的精选元数据集。
记录显⽰样本状态,有关馆藏的机构的信息,以及NCBI中相关数据链接。
蛋白质结构及功能预测的方法和软件
蛋白质结构及功能预测的方法和软件蛋白质是生命体内的重要组成部分,在细胞的生命活动中发挥着不可替代的作用。
因此,研究蛋白质的结构和功能具有重要的科学意义。
有很多蛋白质的结构和功能还未被解析出来,这给科学家带来了巨大的挑战。
为了更好地研究蛋白质,科学家发展了一些蛋白质结构及功能预测的方法和软件。
一、蛋白质结构预测1. 基于序列的结构预测蛋白质的结构决定了其功能,但是实验测定蛋白质的结构是非常昂贵和耗时的。
因此,研究人员发展了基于序列的结构预测方法来识别蛋白质的结构。
这种方法可以从氨基酸序列中预测蛋白质的3D结构,进而了解蛋白质的结构、功能、稳定性、抗原性等。
2. 基于比对的结构预测基于比对的结构预测方法则是通过利用已知结构的同源蛋白质比对来预测目标蛋白质的结构。
这种方法可以用于识别蛋白质的结构域、模拟蛋白质的功能分子机制、预测蛋白质的亚细胞位置等。
二、蛋白质功能预测1. 基于结构的功能预测蛋白质的功能通常与其结构有很大关系。
因此,研究人员可以通过预测蛋白质的结构来预测其功能。
利用蛋白质3D的结构信息,研究人员可以设计用于高通量筛选和分析蛋白质功能的药物分子,以及预测蛋白质的膜靶、蛋白质-蛋白质相互作用、信号传递等。
2. 基于序列的功能预测基于序列的功能预测方法则是通过分析蛋白质序列中的特定特征,来预测蛋白质的功能。
这种方法通常包括基于局部特征、亚细胞结构和功能预测等。
三、蛋白质结构及功能预测软件研究人员发展了很多软件来预测蛋白质的结构和功能。
其中最著名的包括Rosetta、I-TASSER、SWISS-MODEL、Phyre2、HHPred、ESyPred3D、ProtoNet等。
1. RosettaRosetta是著名的蛋白质结构预测软件。
它基于声学优化理论和免疫遗传算法,可以高效地预测蛋白质的结构。
利用Rosetta可以快速地研究蛋白质的折叠和稳定性等。
2. I-TASSERI-TASSER是一种全自动蛋白质结构预测软件,可以用于从氨基酸序列中预测蛋白质的3D结构、功能域、拓扑结构等。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
/genomes/MICROBES/gli Maryland mmer_3.cgi /software/glimmer
/berry.phtml?topic=fgenes& Softberry group=programs&subgroup=gfind
蛋白质组学
2
基因组序列 cDNA序列
基因组功能分析
翻译
编码区预测 蛋白质序列
蛋白质理化性质 二级结构预测 结构域分析 重要信号位点分析 三级结构预测
基因结构分析
序列比对 功能注释
Codon bias
选择性剪切
GC Content 转录调控因子
限制性酶切位点
KEGG
GO 系统发育树
3
真核生物基因的主要结构
/genomescan.html
MIT
/Wise2/
EBI
/grailexp/
ORNL
通用 真核
脊椎、拟南芥、玉米 人、小鼠、拟南芥、酵母 真核(基因结构)
原核 原核
人(基因结构)
同源比 对信息
预测结果的氨基酸序列
12
GenomeScan输出结果:图形
13
课堂练习
• 1使用GENESCAN预测序列中可能的ORF。 • 2使用GENOMESCAN预测序列中可能的
ORF。
• 练习用的序列文件在c:\zcni\shixi2文件下, 名字为clone.fasta,使用写字板打开查看。
• ORF 是潜在的蛋白质编码区
6
ORF Finder BestORF
GENSCAN Gene Finder FGENESH
GeneMark GLIMMER
Fgenes
FgeneSV
Generation FGENESB
GenomeScan GeneWise2
GRAIL
基因开放阅读框/基因结构分析识别工具
病毒
原核 细菌(基因结构)
脊椎、拟南芥、玉米 人 人、小鼠、拟南芥、7果蝇
选择物种类型
ORF识别:GENSCAN
/GENSCAN.html
是否显示非最优外显子 序列名称(可选) 显示氨基酸或CDS序列
提交序列文件
运行GENSCAN
提交序列
结果返回到邮箱(可选)
Web
CpGPlot
/emboss/cpgplot/index. html
Web
/berry.phtml?topic=c CpG finder pgfinder&group=programs&subgroup=pro Web
14
转录调控序列分析
CpG岛、转录终止信号和启动子区域的预测
15
CpG岛的预测
CpG岛
常位于真核生物基因转录起始位点,GC含>50% , 长度>200bp的一段DNA序列。
16
CpG Island 分析常用软件
CpG Island
/cpgislands2/cpg.asp x
MIT
/tools/genefinder/
Zhang lab
/berry.phtml?topic=fgenesh Softberry &group=programs&subgroup=gfind
/GeneMark/eukhmm.cgi GIT
8
GENSCAN输出结果:文本
9
GENSCAN输出结果:图形
10
ORF识别: GenomeScan
/genomescan.html
运行GenomeScan
提交待分析序列
提交同源蛋白质序列
11
GenomeScan输出结果:文本
预测外显子位置、可 信度等信息
/berry.phtml?topic=virus&gr Softberry oup=programs&subgroup=gfindv
/generation/
ORNL
/berry.phtml?topic=fgenesb Softberry &group=programs&subgroup=gfindb
4
基因结构分析常用软件
开放读码框
CpG岛 转录终止信号
基因结构分析 启动子/转录起始位点
密码子偏好分析
mRNA剪切位点
选择性剪切
GENSCAN GENOMESCAN
CpGPlot POLYAH PromoterScan CodonW NETGENE2
Spidey ASTD
5
开放读码框的识别
• 开放读码框(open reading frame, ORF) 是一段起始密码子和终止密码子之间的碱基序列
moter
CpGi130 /CpG130.do web
CpGproD
http://pbil.univlyon1.fr/software/cpgprod_query.html
实习二 真核生物基因结构的预 测分析
浙江加州国际纳米技术研究院 2010年11月
苏锟楷 楼小燕 韩 序 蒋 琰
1
课程内容
实习一
基因组数据注释和功能分析
基因组学
实习二
真核生物基因结构的预测分析
系
统
实习三
芯片的基本数据处理和分析
转录物组学
生 物
实习四
蛋白质结构与功能分析
学
实习五 实习六
蛋白质组学数据分析 系统生物学软件实习
/gorf/gorf.html
NCBI
/berry.phtml?topic=bestorf& Softberry group=programs&subgroup=gfind
/GENSCAN.html
