基于微阵列的比较基因组分析
生物大数据分析中的表达量差异分析方法

生物大数据分析中的表达量差异分析方法在生物学研究中,表达量差异分析是一种常用的方法,用于比较不同生物样品中基因或蛋白质的表达水平的差异。
这种分析可以帮助研究人员识别潜在的生物标记物,并了解基因表达与各个生物过程之间的关系。
随着高通量测序技术的快速发展,生物大数据分析在表达量差异分析中扮演着重要的角色。
本文将介绍几种常见的生物大数据分析中的表达量差异分析方法。
首先,常用的差异表达基因分析方法是RNA-seq(转录组测序)。
RNA-seq是一种通过测序RNA分子来分析其转录产物数量和结构的方法。
在RNA-seq实验中,首先提取RNA样品,然后进行cDNA合成,接着进行文库构建和测序。
通过比对测序数据到参考基因组或转录组,可以计算基因的表达量,进而比较不同样品之间的表达量差异。
一般采用的分析工具包括DESeq2、edgeR和limma等,通过这些工具可以识别差异表达基因,并进行差异表达基因的注释和功能分析。
其次,基于微阵列芯片技术的差异表达分析方法也是常见的。
微阵列芯片是一种高通量的基因表达分析的方法,通过固定在平台上的探针检测目标DNA或RNA 的水平。
在实验中,首先提取RNA样品,然后进行反转录和标记,接着进行芯片杂交,并进行扫描和数据分析。
常用的分析方法有SAM(Significant Analysis of Microarrays)和limma等。
这些方法可以通过比较不同样品之间的信号强度,识别差异表达基因,并进行差异表达基因的功能注释和通路分析。
此外,对于一些非常规的生物样品(如:单个细胞)的表达量差异分析,常常采用单细胞测序技术。
单细胞测序技术允许研究人员在单个细胞的水平上进行转录组测序,从而可以发现罕见细胞类型和子群,以及细胞间的差异。
在单细胞测序中,首先对细胞进行分离和取材,然后进行单细胞测序文库构建和测序。
常用的分析软件包括scater、Seurat和scRNA-Seq等,可以对单个细胞的基因表达进行聚类、可视化和差异表达分析。
2014年染色体微阵列分析技术在产前诊断中的应用专家共识

染色体微阵列分析技术在产前诊断中的应用专家共识染色体微阵列分析技术在产前诊断中的应用协作组目前,G 显带染色体核型分析技术仍然是细胞遗传学产前诊断的“金标准”,但该技术具有细胞培养耗时长、分辨率低以及耗费人力的局限性。
包括荧光原位杂交(fluorescence in situ hybridization,FISH) 技术在内的快速产前诊断技术的引入虽然具有快速及特异性高的优点,但还不能做到对染色体组的全局分析。
染色体微阵列分析(chromosomal mlcroarray analysis,CMA) 技术又被称为“分子核型分析”,能够在全基因组水平进行扫描,可检测染色体不平衡的拷贝数变异(copy number variant,CNV),尤其是对于检测染色体组微小缺失、重复等不平衡性重排具有突出优势。
根据芯片设计与检测原理的不同,CMA 技术可分为两大类:基于微阵列的比较基因组杂交(array- based comparative genomic hybridization ,aCGH) 技术和单核苷酸多态性微阵列(single nucleotide polymorphism array,SNP array) 技术。
前者需要将待测样本DNA 与正常对照样本DNA 分别标记、进行竞争性杂交后获得定量的拷贝数检测结果,而后者则只需将待测样本DNA 与一整套正常基因组对照资料进行对比即可获得诊断结果。
通过aCGH 技术能够很好地检出CNV,而SNP array 除了能够检出CNV 外,还能够检测出大多数的单亲二倍体(uniparental disomy,UPD) 和三倍体,并且可以检测到一定水平的嵌合体。
而设计涵盖CNV+SNP 检测探针的芯片,可同时具有CNV 和SNP 芯片的特点。
2010 年,国际细胞基因组芯片标准协作组(lntemational Standards for Cytogenomic Arrays Consortium,ISCA Consortium) 在研究了21698 例具有异常临床表征,包括智力低下、发育迟缓、多种体征畸形以及自闭症的先证者的基础上,发现aCGH 技术对致病性CNV 的检出率为 12.2%,比传统G 显带核型分析技术的检出率提高了10%。
肺癌专题多发性肺癌的基因组异质性
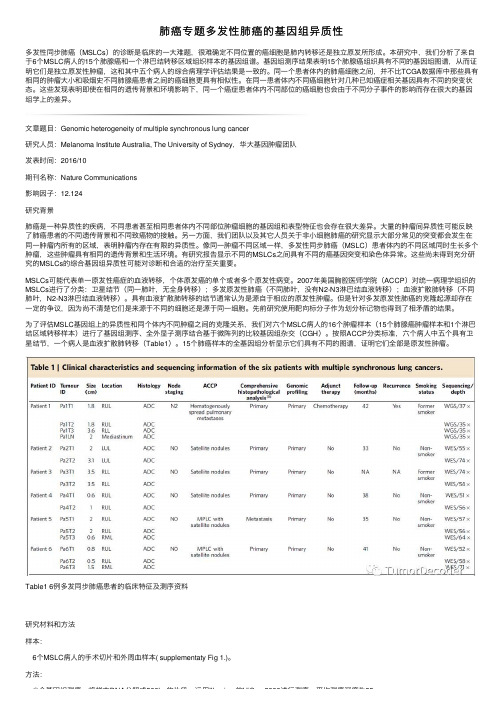
肺癌专题多发性肺癌的基因组异质性多发性同步肺癌(MSLCs)的诊断是临床的⼀⼤难题,很难确定不同位置的癌细胞是肺内转移还是独⽴原发所形成。
本研究中,我们分析了来⾃于6个MSLC病⼈的15个肺腺癌和⼀个淋巴结转移区域组织样本的基因组谱。
基因组测序结果表明15个肺腺癌组织具有不同的基因组图谱,从⽽证明它们是独⽴原发性肿瘤,这和其中五个病⼈的综合病理学评估结果是⼀致的。
同⼀个患者体内的肺癌细胞之间,并不⽐TCGA数据库中那些具有相同的肿瘤⼤⼩和吸烟史不同肺腺癌患者之间的癌细胞更具有相似性。
在同⼀患者体内不同癌细胞针对⼏种已知癌症相关基因具有不同的突变状态。
这些发现表明即使在相同的遗传背景和环境影响下,同⼀个癌症患者体内不同部位的癌细胞也会由于不同分⼦事件的影响⽽存在很⼤的基因组学上的差异。
⽂章题⽬:Genomic heterogeneity of multiple synchronous lung cancer研究⼈员:Melanoma Institute Australia, The University of Sydney,华⼤基因肿瘤团队发表时间:2016/10期刊名称:Nature Communications影响因⼦:12.124研究背景肺癌是⼀种异质性的疾病,不同患者甚⾄相同患者体内不同部位肿瘤细胞的基因组和表型特征也会存在很⼤差异。
⼤量的肿瘤间异质性可能反映了肺癌患者的不同遗传背景和不同致癌物的接触。
另⼀⽅⾯,我们团队以及其它⼈员关于⾮⼩细胞肺癌的研究显⽰⼤部分常见的突变都会发⽣在同⼀肿瘤内所有的区域,表明肿瘤内存在有限的异质性。
像同⼀肿瘤不同区域⼀样,多发性同步肺癌(MSLC)患者体内的不同区域同时⽣长多个肿瘤,这些肿瘤具有相同的遗传背景和⽣活环境。
有研究报告显⽰不同的MSLCs之间具有不同的癌基因突变和染⾊体异常。
这些尚未得到充分研究的MSLCs的综合基因组异质性可能对诊断和合适的治疗⾄关重要。
微阵列比较基因组杂交技术在产科领域推广说明

(3)曾生育出生缺陷的夫妻再生第二胎的遗传咨询。
定位:为妇产科医生提供最全面精细的遗传诊断方法,为不孕不育诊断指明方向。 策略: 优先推荐首选aCGH技术,配合外周血染色体核心分析使用。 提供后期“辅助生殖技术”绿色通道等增值支持。 市场培育 :各级学术活动铺开,主要目标客户。
市场细分、目标市场选择、市场定位策略
流产组织是最快上量的切入点。
可用CytoScan750K芯片,收费相对较低。 发病率高,可推范围广(二三级医院)。 阳性率很高:流产组织中有一半存在染色体异常。 阳性后的遗传咨询:可带动夫妻同做,带动三个标本。
谢谢
微阵列比较基因组杂交技术 (aCGH)在产科领域的推广
总部市场部 伍世兴
产品概况
1、产品属性
定义:通过对所有染色体中的基因进行扫描,检测其中片段的拷贝数目变异(CNV) 的技术,有别于细胞核型分析的分子核型分析。
染色体核型分析 技术原理 检测范围 分辨率 检测效能 显带技术 46条染色体 5-10Mb 全部染色体 aCGH 基因芯片技术 46条染色体 低于50Kb 全基因组覆盖
4、aCGH技术不能检测出平衡易位,若怀疑夫妻双方存在平衡易位,建议加做外周血
染色体核型分析。
建议检测流程
原发性不孕不育、曾生育过出生缺陷、需进行辅助生殖技术的夫妻
夫妻双方同做 aCGH(最好采用 HD芯片) 夫妻双方结果 遗传咨询
流产组织
流产组织做 aCGH(最 好采用 750K芯片) 流产物异常 结果 夫妻双方同 做 夫妻双方结 果 遗传咨询
市场细分、目标市场选择、市场定位策略
第二类目标市场——延伸导入
科室:三级医院的妇产科/不孕不育专科/遗传咨询专科/男科 数量:1043家
微阵列—比较基因组杂交技术及其在肿瘤研究中的应用

微阵列—比较基因组杂交技术及其在肿瘤研究中的应用微阵列技术是一种高通量的基因表达分析方法,它通过比较基因组杂交技术实现对大量基因表达水平的同时检测和分析。
本文将介绍微阵列技术的原理和应用,并重点探讨其在肿瘤研究中的应用。
一、微阵列技术原理微阵列技术是基于比较基因组杂交的原理实现的,其基本步骤包括样本准备、RNA提取和标记、芯片杂交和信号检测四个主要环节。
1. 样本准备:首先需要提取研究对象的RNA样本,例如从肿瘤组织或正常组织中提取RNA。
为了获得可靠的数据,研究者需要大量重复样本。
2. RNA提取和标记:首先将提取的RNA逆转录成cDNA,然后利用核酸杂交和扩增技术,将样本RNA与反义RNA标记物杂交。
标记物可以是荧光标记的核酸分子或生物素等,以便后续的检测。
3. 芯片杂交:将标记的RNA样本加入到微阵列芯片上,通过杂交反应使得标记物与芯片中的探针片段互相结合。
4. 信号检测:利用激光扫描仪扫描芯片上的标记物,获取荧光信号,并根据信号的强度和密度来定量分析基因的表达水平。
二、微阵列技术在肿瘤研究中的应用微阵列技术在肿瘤研究中具有广泛的应用前景,主要体现在以下几个方面:1. 基因表达谱的分析:通过微阵列技术可以同时检测和分析大量的基因表达水平,从而了解肿瘤发生发展的分子机制。
比较正常组织与肿瘤组织的基因表达谱差异,可以发现潜在的肿瘤标志物或靶向治疗的新靶点。
2. 肿瘤分类与诊断:肿瘤是一类异质性很强的疾病,通过微阵列技术可以将肿瘤分子分型和个性化治疗相结合,实现精准医疗。
通过分析肿瘤细胞的基因表达谱,可以准确地判断肿瘤类型和预测患者的预后。
3. 药物研发与耐药机制研究:利用微阵列技术可以筛选出特异性作用于肿瘤的新药物。
通过比较药物敏感性和耐药性细胞系的基因表达差异,可以揭示耐药机制,并寻找新的治疗策略。
4. 分子靶向治疗的预测:微阵列技术能够评估患者对靶向治疗的敏感性和预测疗效,从而帮助医生制定个体化的治疗方案。
aCGH

康成生物aCGH芯片技术服务DNA拷贝数变化(CNV)在许多人类疾病(如癌症、遗传性疾病、心血管疾病及糖尿病)的发生发展中起重要作用,但因为CNV 在健康个体间也普遍存在,因此理解特定疾病的分子机制的关键在于鉴定出正常和畸变染色体间的差异。
比较基因组杂交(CGH)是检测基因组DNA的片段扩增或缺失的有效方法,而基于微阵列技术的比较基因组杂交(aCGH)通过在一张芯片上用标记不同荧光素的样品(肿瘤样品和对照样品)同时进行杂交可检测样本基因组和对照基因组间DNA拷贝数变化(CNV),常用于肿瘤及遗传性疾病全基因组CNV检测,直观地表现出肿瘤及遗传性疾病基因组DNA在整个染色体组的缺失或扩增,对肿瘤而言缺失部位可能包含抑癌基因,而扩增片段则可能存在致癌基因。
●什么是拷贝数的变异(CNV)?人类的基因组是由包括60亿个化学碱基(或者称为核苷酸)的DNA组成的,并被包装到23对染色体中,每对染色体都是一条来自父代,一条来自母代。
这些DNA编码大约30,000个基因。
过去通常认为基因在基因组中是以2个拷贝的形式存在,然而目前的发现已经表明:有大量的DNA片段在拷贝数上有很大的变异,这些DNA片段的大小从数千到数百万碱基不等。
这些拷贝数的变异(简称CNVs)包含拷贝数改变的基因,比方说,那些过去认为每对染色体上存在2个拷贝的基因,现在发现有时是1个拷贝,有时是3个甚至3个以上,在少数罕见的情况下这些基因还会一起缺失。
●为什么说CNVs很重要?染色体上DNA序列的不同决定了我们个体的独特性,包括对疾病的易感性等等。
过去认为DNA的单核苷酸的改变(称为SNPs单核苷酸多态性)是基因变异的主要形式,最近的研究发现CNVs的发生率至少是SNPs的3倍。
因为CNVs中经常包含一些在人类疾病发生和对药物的反应中起着重要的作用的基因,所以理解CNV形成机制能够帮助我们更好地理解人类基因组的进化。
●新的CNV图谱有什么帮助?新的CNV图谱能够在三个领域对医学研究有帮助:首先也是最重要的,就是能够帮助寻找与常见疾病相关的基因,到目前为止,还没有真正认识到这些基因的CNVs在人类健康中所起的作用,我们正试图证明这一点;第二,CNV图谱正被用来研究家族遗传病;第三,数千种严重的发育缺陷正是由于染色体的重排引起的,CNV图谱被用来排除在未受影响个体上变异的发生,帮助研究人员确定可能涉及到的目标区域,整个数据的生成要归功于一个更加精确及完整的人类基因组参考序列。
cma基因芯片

cma基因芯片
CMA基因芯片是一种常规的染色体微阵列芯片,用于检测和分析染色体异常。
它基于Comparative Genomic Hybridization (比较基因组杂交技术),可以同时检测大量的目标序列和参照序列。
CMA基因芯片通常用于研究遗传性疾病、先天性缺陷和肿瘤的基因组结构异常。
它可以检测插入、缺失、重复和平衡性易位等染色体异常,并帮助诊断和研究这些疾病的底层机制。
使用CMA基因芯片进行分析时,研究者将待测样品和参照样品一起杂交到芯片上,然后测量荧光信号的强度来判断目标序列与参照序列之间的拷贝数差异。
通过比较信号的强度,可以确定目标序列的变异情况。
总之,CMA基因芯片是一种高通量、高分辨率的技术,可以帮助研究人员快速、准确地检测和分析染色体异常。
它在临床诊断和基础研究领域具有广泛的应用前景。
基于表型以及微阵列数据的基因(型)分类技术研究的开题报告

基于表型以及微阵列数据的基因(型)分类技术研究的开题报告一、研究背景随着生物技术的不断发展,生物信息学领域不断涌现出新的技术和方法。
其中,基于表型以及微阵列数据的基因(型)分类技术已成为当前生物信息学领域的热点。
随着人类基因组计划的完成,大量的基因序列数据被公布,包括人类和其他种类的基因组数据。
这些数据的分析和利用,有助于我们更好地了解基因和表型之间的关系。
在这个过程中,基因分类技术起着重要的作用。
二、研究目的本研究旨在开发一种基于表型以及微阵列数据的基因(型)分类技术,探索该技术在生物信息学领域中的应用价值,为基于表型信息的相关研究提供技术支持。
具体来说,本研究将通过以下几个方面进行探索:1.基于表型数据建立基因分类模型对大量的表型数据进行分析和处理,提取出对基因分类有意义的特征,通过建立基因分类模型实现基因分类。
2.基于微阵列数据建立基因分类模型对微阵列技术进行学习和应用,对大量的微阵列数据进行分析和处理,提取出对基因分类有意义的特征,通过建立基因分类模型实现基因分类。
3.将表型和微阵列数据相结合进行基因分类通过将表型和微阵列数据结合使用,利用两者之间的相关性进行基因分类,以提高分类准确率。
三、研究方法1.数据预处理将表型数据进行标准化、降维处理等操作,将微阵列数据进行质量控制、数据预处理、正则化等操作。
2.特征选择对预处理后的数据进行特征选择,挑选出对基因分类有重要意义的特征。
3.模型建立基于选定的特征,结合机器学习等算法建立基因分类模型。
4.模型评价对建立的模型进行评价和优化,评价指标包括分类准确率、灵敏度、特异度等。
5.模型应用将建立好的模型应用到实际数据中进行基因分类,并与其他基因分类方法进行比较分析,验证该技术的可行性和有效性。
四、研究意义本研究将有助于加深我们对基因和表型之间关系的理解,探索基于表型信息的基因分类技术,为相关领域的研究提供新的思路和方法。
同时,本研究所开发的技术具有很高的实用价值,可应用于医学诊断、生物生产、动物育种等领域。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
微阵列芯片(Microarray)以高密度阵列为特征。
其基础研究始于20世纪80年代末,本质上是一种生物技术,主要是在生物遗传学领域发展起来的。
微阵列分为cDNA微阵列和寡聚核苷酸微阵列.微阵列上"印"有大量已知部分序
列的DNA探针,微阵列技术就是利用分子杂交原理,使同时被比较的标本(用同位素或荧光素标记)与微阵列杂交,通过检测杂交信号强度及数据处理,把他们转化成不同标
本中特异基因的丰度,从而全面比较不同标本的基因表达水平的差异.微阵列技术是一种探索基因组功能的有力手段.
其发展契机主要来自于现代遗传学的一些重要发现,并直接收益于该领域的某些重要研究成果,即在载体上固定寡核苷酸的基础上以杂交法测序的技术。
因此发展早期,微阵列芯片有时被通俗的称为“生物芯片(Biochip)”,目前媒体和科普读物中仍然常用该名称。
微阵列芯片经过近十年的主要发展期,国内外学术界渐渐采用名称Microarray(微阵列芯片),而Biochip(生物芯片)由于这名称容易混淆微阵列芯片和微流控芯片,渐渐该领域用的越来越少了。
比较基因组杂交技术
比较基因组杂交(comparative genomic hybridization,CGH)是自1992年后发展起来的一种分子细胞遗传学技术,它通过单一的一次杂交可对某一肿瘤整个基因组的染色体拷贝数量的变化进行检查。
其基本原理是用不同的荧光染料通过缺口平移法分别标记肿瘤组织和正常细胞或组织的DNA制成探针,并与正常人的间期染色体进行共杂交,以在染色体上显示的肿瘤与正常对照的荧光强度的不同来反映整个肿瘤基因组DNA表达状况的变化,再借助于图像分析技术可对染色体拷贝数量的变化进行定量研究。
CGH技术的优点:1.实验所需DNA样本量较少,做单一的一次杂交即可检查肿瘤整个基因组的染色体拷贝数量的变化。
2.此法不仅适用于外周血、培养细胞和新鲜组织样本的研究,还可用于对存档组织的研究,也可用于因DNA量过少而经PCR扩增的样本的研究。
CGH技术的局限性:CGH技术所能检测到的最小的DNA扩增或丢失是在3-5Mb,故对于低水平的DNA扩增和小片段的丢失会漏检。
此外在相差染色体的拷贝数量无变化时,CGH技术不能检测出平等染色体的易位。
