微卫星多重PCR扩增技术
多重PCR技术

Part 03 多重PCR技术的前景
在硅玻璃芯片中完成系列核酸扩增反应 的多重PCR生物芯片技术 荧光定量多重PCR、AFLP多重PCR、多 重逆转录PCR、实时多重PCR技术、巢 式多重PCR
用于基因诊断&基因治疗.etc.
小结
多重PCR技术
由于多重PCR技术的巨大商业价值和广阔应用前景,很 多生物技术公司致力于多重PCR试剂盒以及相关技术的 开发
NoNoNo~
难点在于“对多对引物的设 计”
多对引物之间不形成引物二聚体;引物与 目标模板区域之间具有高度特异性.etc.
Part 02 多重PCR技术的优点
高效性
在同一PCR反应管内同时检出多种病原微生物,或对有 多个型别的目的基因进行分型
系统性
经济简便性
节省时间,节省试剂,节约经费开支,为临床提供更多 更准确的诊断信息
多重PCR技术
-multiplex PCR-
CONTENTS
1 什么是多重PCR技术 2 多重PCR技术的优点 3 多重PCR技术的前景
01 P
A R
强大的多重PCR技术
T
在同一PCR反应体系里加上二对以上
引物,同时扩增出多个核酸片段的
PCR反应
反应原理、反应试剂、操作过 程与一般PCR技术相同
多重PCR技术造福人类
具有节省时间、降低成本、提高效率的优点 ,特别是节省珍贵的实验样品
THANKS!
COMPANY
水工53 董祺祯
Examples
乳病毒
01 肝炎病毒、
的分型; 03
梅毒病毒
特殊培养
的同时检
的无芽胞
测
厌氧菌,
如脆弱杆
菌、艰难
微卫星检测方法

微卫星DNA简单重复序(SSR)也称微卫星DNA,也可称为SSRP(Simple Sequence Repeat Polymorphisms),STMS (Sequence-tagged microsatellites)。
其串联重复的核心序列为1一6 bp,其中最常见是双核昔酸重复,即(CA) n和(TG) n每个微卫星DNA的核心序列结构相同,重复单位数目10一60个,其高度多态性主要来源于串联数目的不同。
SSR标记的基本原理:根据微卫星序列两端互补序列设计引物,通过PCR反应扩增微卫星片段,由于核心序列串联重复数目不同,因而能够用PCR的方法扩增出不同长度的PCR产物,将扩增产物进行凝胶电泳,根据分离片段的大小决定基因型并计算等位基因频率。
SSR具有以下一些优点:(l)一般检测到的是一个单一的多等位基因位点;(2)微卫星呈共显性遗传,故可鉴别杂合子和纯合子;(3)所需DNA量少。
显然,在采用SSR技术分析微卫星DNA多态性时必须知道重复序列两端的DNA序列的信息。
如不能直接从DNA数据库查寻则首先必须对其进行测序。
1 微卫星DNA的构成及特点微卫星又称简单序列重复(Simple Se-quence Repeats,SSR)。
一般以1-6个碱基为核心序列,首尾相连组成的串联重复序列。
这种序列存在于几乎所有真核生物的基因组中,含量丰富,且呈随机均匀分布。
它们不仅大量分布于基因的间隔区和内含子中,而且还分布于基因的外显子和调控区(如启动子、增强子),真核生物平均每50-150 kb就存在一个微卫星位点,如在人类基因组中每6 kb就有1个微卫星,禽类基因组中约89 kb出现1个微卫星。
微卫星DNA 数目巨大,人类基因组中约有5×104-1×105个(CA)n重复序列,重复次数一般为15-60次,重复单位相同,其长度一般小于200 bp,但也有的更长。
每个特定位点的微卫星DNA均由两部分构成:中间的核心区和外围的侧翼区。
多重荧光PCR-毛细管电泳进行微卫星不稳定性分析中的常见问题
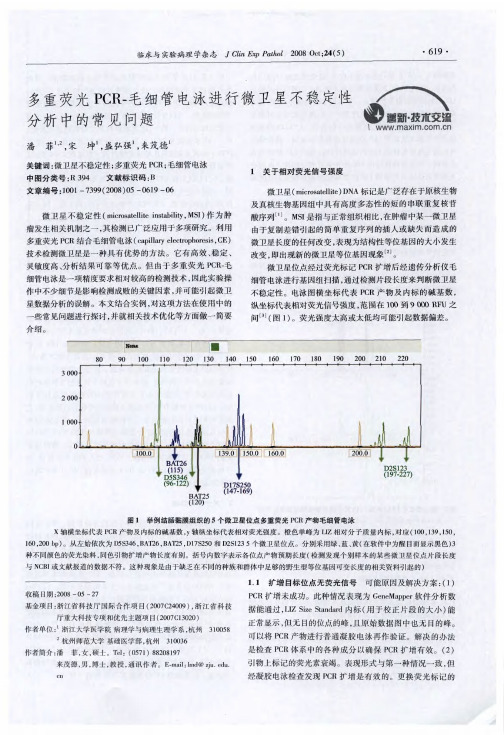
PCR
一
扩增有效 种情况
一
(2 )
,
来茂 德 男 博 士 教 授 通 讯 作 者
, ,
,
E
—
m a
il
: m
l
d@
z
沁
e
du
.
引物 上 标 记 的荧 光 素衰竭
经 凝 胶 电泳 检 查 发 现
PCR
表 现 形 式 与第
扩增 是 有效 的
。
致 但
P n
更换荧光标记 的
620
临 床 与 实验 病 理 学 杂 志
1 l
,
扩 增 目标 位 点 无 荧 光 信 号
可 能 原 因及 解 决 方 案 : ( 1 )
Mappe
r
3 收稿 1 期
2 : 00 8
—
05
—
27
,
PCR
扩增 未 成 功
,
。
此 种情 况 表 现 为 G e n e
e
软件 分 析数
基 金 项 目 :浙 江 省 科 技 厅 国 际 合 作 项 目 ( 2 0 0 7 C2 4 0 0 9 ) 浙 江 省 科 技 厅 重 大 科技专 项 和 优先 主 题 项 目 ( 2 0 0 7 C 1 3 0 2 0 )
—
0 6 19
—
06
te
llite
) D N A 标 记 是 广泛 存在 于 原 核 生 物
及 真 核 生 物 基 因组 中 具 有 高 度 多 态 性 的 短 的 串 联 重 复 核 苷
微卫 星 不 稳定性 ( m
ic
ro s a
te
ll i t e
in
微卫星标记技术原理

微卫星标记技术原理
微卫星标记技术是一种基于PCR (Polymerase Chain Reaction) 扩增的分子标记技术。
微卫星是基因组中较短、高度可变重复序列(Simple Sequence Repeats, SSRs),在不同个体或同一基因组中的
不同位置均有差异。
微卫星标记技术通过引物对位于微卫星序列的两
端的flanking序列进行PCR扩增,再以电泳方法进行分离和检测。
具体步骤包括:1. DNA提取,取得待检测组织的DNA;2. 引物
设计,设计与微卫星重复序列两侧的flanking序列可以特异性结合的
引物;3. PCR扩增,将待检测的DNA模板按设定条件进行PCR扩增;4. 电泳分离,将扩增产物以电泳方式分离出来;5. 图谱显示和分析,通
过显示分离出来的电泳带,分析出样品间和亲缘关系的差异以及基因
型信息。
微卫星标记技术具有高度多态性、重复性好、易于检测、特异性
强等优点。
它已广泛应用于动植物的遗传多样性、种间亲缘关系研究、基因定位及遗传距离测定等方面。
MLVA分型技术和实验操作步骤

MLVA分型技术和实验操作步骤MLVA(多重定量PCR分型技术)是一种基于多重PCR扩增的分型技术,它可以用来对细菌进行分型和鉴定,并用于疫情的追踪和溯源。
下面是MLVA分型技术的实验操作步骤:1.提取细菌基因组DNA:从细菌菌落或液体培养物中提取基因组DNA。
可以使用商业化的基因组DNA提取试剂盒来进行提取,按照说明书中的步骤进行操作。
2.设计引物和预扩增:根据待测菌株的基因组序列,设计适用于目标DNA片段的引物。
引物应该选择在基因组中具有高度保守性的区域,以确保反应的特异性。
将设计好的引物与提取到的DNA进行预扩增。
预扩增是将样品中的目标片段DNA扩增到足够的浓度,以确保后续的扩增反应。
3. MLVA引物的选择:根据待测菌株基因组中的微卫星重复序列,设计MLVA引物。
微卫星重复序列(也称作SSR或简单重复序列)是一种相对较短(1-6bp)的DNA序列,在基因组中重复出现。
这些引物将扩增微卫星重复序列及其周围的DNA区域。
4.MLVA引物的扩增:将设计好的MLVA引物与预扩增的DNA模板混合,进行PCR扩增。
PCR扩增反应中的引物浓度、缓冲液组成、降低PCR抑制物质(如血红素和其他化合物)对扩增反应的成功至关重要。
5.扩增产物的测定和分析:将PCR扩增产物进行凝胶电泳,可以用琼脂糖凝胶电泳、毛细管电泳或自动测序进行分析。
分析产物的大小差异,可以判断不同菌株的差异。
总结起来,MLVA分型技术是一种基于PCR扩增的分型技术,它通过设计特异的引物,扩增目标DNA片段,并通过凝胶电泳等方法对扩增产物进行分析和解释,从而实现对细菌的分型和鉴定。
这项技术在疫情追踪和溯源中具有重要的应用价值。
基因扩增技术在种群遗传学中的应用

基因扩增技术在种群遗传学中的应用随着科学技术的不断发展,基因扩增技术在种群遗传学中得到了广泛应用。
基因扩增技术是一种将DNA扩增成大量同一序列的方法,主要包括PCR、RFLP、RAPD、AFLP和Microsatellite等。
这些技术已经成为了种群遗传学中不可或缺的分子工具,对于研究种群之间的遗传差异、基因流动、分化和进化等方面都具有重要作用。
一、微卫星技术在种群遗传学中的应用微卫星是一种长度在1-6bp之间、重复序列数目在2-50之间的DNA序列,特别是在非编码区域中。
微卫星技术因其灵敏度高、稳定性好、有多态性,尤其适用于种群间遗传差异的研究。
使用微卫星技术又称SSR(Simple Sequence Repeat)技术,其主要思想是利用PCR扩增多个微卫星位点,并检测多样性。
在分子进化、弥补遗传数据的缺失等方面,微卫星技术都取得了重要的研究突破,尤其在动植物的遗传分化、种间关系、人类祖先进化等领域尤为重要。
二、RAPD技术在种群遗传学中的应用RAPD技术是一种以随即引物为基础,利用PCR技术扩增核酸片段的方法,依托于有效的PCR反应和快速的生物信息统计,RAPD技术具有快速、灵敏、具有广泛可用性等特点。
其主要优点是在从多个参考种间关系的分析过程中能够同时检测多个位点,其结果能够为数目较多的研究提供遗传基础。
RAPD技术作为种群遗传学领域中的重要技术手段,既可以用于检测种群内遗传多态性,也可以判断种群之间的遗传分化程度,并揭示生物不同种群之间的遗传关系。
三、AFLP技术在种群遗传学中的应用AFLP(Amplified Fragment Length Polymorphism)技术是近年来发展起来的一种新的分子标记技术。
AFLP方法首先通过引物选择筛选系统酶切后的DNA片段,然后通过PCR扩增产生的片段选取、分析、测序等一系列处理,得到有效的遗传标志,最终得出一组与DNA序列相关的AFLP分子标记。
j9i利用微卫星多重PCR和带型分析鉴别歧视

利用微卫星多重PCR和带型分析鉴别歧视酿酒酵母菌株Enrico Vaudano_, Emilia Garcia-Moruno摘要我们提出了一个快速的利用微卫星多重PCR分析方法鉴定酵母菌菌株。
不使用苯酚的简单的DNA提取,通过快速PCR的优化程序扩增SC8132X位点,YOR267C 位点和SCPTSY7位点,带型分析产生的琼脂糖碎片,聚丙烯酰胺凝胶电泳,这些可以使我们区分30个小组的测试的商业酒株。
该方法成功地在生态研究中发现两个菌株之间的优势发酵温度是在两个温度:15℃和20℃。
这个方法无论是在酒和酵母生产行业等日常行业和低预算的歧视酵母菌株生产是有用的。
关键词:微卫星;聚合酶链式反应;酵母菌株;葡萄酒,发酵介绍用酒精发酵菌种选择酿酒现在是很普遍的。
在酒的品质方面,酵母交互作用的影响变得清晰,特别是他们的贡献的味道。
(LambrechtsPretorius,2000;Swiegers等,2005)。
现代酿酒学家都意识到极致味道不同品系的酒会产生不同的特点。
这导致了工业生产大量的选择具有不同的应用的不同的特征。
目前,市场提供了数百株,几乎所有的物种的酵母。
使酵母菌快速鉴定方法明确的并行兴趣在快速发展。
在葡萄酒酿造,应用程序包括核查野生株中占主导地位的菌株的必须接种,发酵过程中的动态应变,从酵母生产商的观点来看,在应变生产中控制产品质量,以及检测欺诈行为。
生态研究提供了最有趣的应用之一。
在葡萄酒酿造,选定菌株接种必须预防丰富的野生酵母菌和非酵母菌株的存在。
快速应变歧视的方法应该允许酵母种群的动态变化,选定应变能力优势的菌株和影响酵母生长的环境因素。
在葡萄酒发酵,温度是影响酵母动态的主要因素之一(Fleet andHeard,1993). 生态研究表明酵母株具有不同的认知和行为学(Epifanio et al., 1999; Torijaet al., 2003).事实上,由酿酒师要求的突出特点之一是能够在一定温度下进行好发酵。
微卫星DNAPCR扩增结果示例

对于人类来说,SNP的意义在于: 1、致病SNP 2、疾病易感性SNP 3、具有诊断价值的SNP 4、疾病的SNP谱 5、人表型相关SNP 6、药物作用与不良反应的SNP 7、药物剂量SNP
目前SNP研究的两个热点: 1、cDNA上的SNP(cSNP) 2、启动子上的SNP
常用检查方法:DNA测序、芯片杂交等。
Genetics
NTMC
Genetics
染色体FISH探针
整条染色体涂染探针 染色体重复序列探针 染色体专一位点探针
基本过程: 1、染色体标本制备 2、探针制备 3、杂交 4、显微观察(染色体分带)
NTMC
Genetics
整条染色体涂染
NTMC
Genetics
通过整条染色体涂染判断易位染色体
NTMC
变性(92-95C,30s)
(25-35)
复性
(40-60C,30s)
延伸
(72C,30-60s)
总延伸
(72C,7m)
经过30次的循环, 扩增的DNA片段可达100万倍以上。
NTMC
Genetics
等位基因特异性寡核苷酸杂交(allelespecific oligonucleotide,ASO )
PCR可结合ASO,即PCR-ASO技术,即先将 含有突变点的基因有关片段进行体外扩增,然后 再与ASO探针作点杂交,这样大大简化了方法, 节约了时间,而且只要极少量的基因组DNA就可
NTMC
Genetics
正常探针 GAG
点杂交
异常探针 TAG
点杂交
正常人 DNA
患者 DNA
正常人 DNA
患者 DNA
DNA以指数形式扩增(2n),其中长片段以线性形式扩增 (2n+2),最终主产物片段长度在两个引物之间。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
10
• 银染步骤: • 1. 取下凝胶用去离子水冼胶2 min; • 2. 0.1% 硝酸银染色10-15 min; • 3. 弃去染色液, 蒸馏水洗胶30 s;
• 4. 显色液显色15秒, 换用新的显色液, 显清条带为止;
• 5. 显色后用固定液(冰乙酸)终止显色,2 min; • 6. 用去离子水洗涤2遍。
200 ul PCR管,Taq酶,dNTPs,引物,PCR缓冲液,石蜡
油,电泳所需试剂,溴酚蓝,琼脂糖,硼酸,Tris碱,蒸 馏水,离心管。
3
实验步骤
• PCR反应液配置-PCR扩增-产物检测-程序优化 -PCR扩增-产物检测-结果分析
多重PCR扩增体系为20 μl:包括100 ng基因组 DNA,1 × PCR 缓冲液,Mg2+ (1.5 mmol/L),1 U Taq 酶, dNTPs 0.2 mmol/ L,多重PCR扩增体系内各位点上、
产物大小 290-320 200-250 140-170 360-400 290-330 200-250 140-180
4-5
• PCR程序:
• • • • • •
95 C 95 C Ta 72 C 72 C 4C
3 min 30 s 30 s 32 cycles 45 s 5 min for ever
鱼类育种学实验
内容:微卫星多重PCR扩增技术
实验目的
• 掌握SSR分子标记技术的原理与分析方法;
• 学习多重PCR技术,了解体系优化的方法;
• 掌握聚丙烯酰胺凝胶电泳的方法;
• 掌握银染的实验方法。
2
实验一 PCR反应及体系优化
• 实验材料与仪器 • 材料:团头鲂DNA模板(浓度:100 ng/μl ) • 仪器用具:PCR仪,制冰机,紫外反射投射检测仪,微量 移液器,冰箱,天平,电泳仪,电泳槽,烧杯,量筒,
注:对Ta进行梯度优化,设置4个温度梯度-50,54,58,62。
7
聚丙烯酰胺凝胶电泳
• 溶液配置:
• 5×TBE:54 g Tris 碱,27.5 g硼酸,20 ml 0.5
mol/L EDTA (pH 8.0),加ddH2O定容至1000 ml 。 • 40%丙烯酰胺:380 g 丙烯酰胺,20 g甲叉-丙烯酰 胺,加ddH2O定容至1000 ml。
11
• 作业 • 完成实验报告,分析检测步骤和结果,探索 微卫星多重PCR优化程序的控制因素对反应 的影响,分析SSR带型。
12
13
8
• 0.5 mol/L EDTA (pH 8.0):186.1 g EDTA•Na•2H2O,800 ml ddH2O,NaOH调pH至8.0 (about 20 g NaOH),加
ddH2O定容至1000 ml,高压灭菌。
• 10% APS:1 g APS,加ddH2O 定容至10 ml,置于4 C。 • 8% non-denaturing PAGE gel: • 40% 丙烯酰胺 • ddH2O 14 ml 41 ml
•
65 μl
9
• 10% APS • TEMED
• 染色液-0.1% AgNO3:0.5 g AgNO3,加ddH2O 定容至 500 ml,置于4 C。 • 显色液:10 g NaOH,0.2 g NaCO3, ddH2O 定容至500
ml,置于4 C保存,用时加 2 ml 37% formaldehyde。
下游引物分别为0. 25 μmol/L
4
体积 试剂 三重PCR体系 (μl) H2O Buffer Mg2+ 13.7 2.0 1.2 四重PCR体系(μl) 13.2 2.0 1.2
dNTP
Primer-1 (F/R) Primer-2 (F/R) Primer-3 (F/R) Primer-4 (F/R) Taq 酶 DNA (100 ng/μl)
0.4
0.25+0.25 0.25+0.25 0.25+0.25 -0.2 1
0.4
0.25+0.25 0.25+0.25 0.25+0.25 0.25+0.25 0.2 1
Total
20 μl
20 μl
5
体系名称 3-7
引物组合 Mam_EST811 Mam_EST129 Mam_EST64 Mam_EST179 Mam_EST11 Mam_EST90 Mam_EST37
