分子机制-核酸检测-DNase I足迹分析
DNase I(RNase Free)
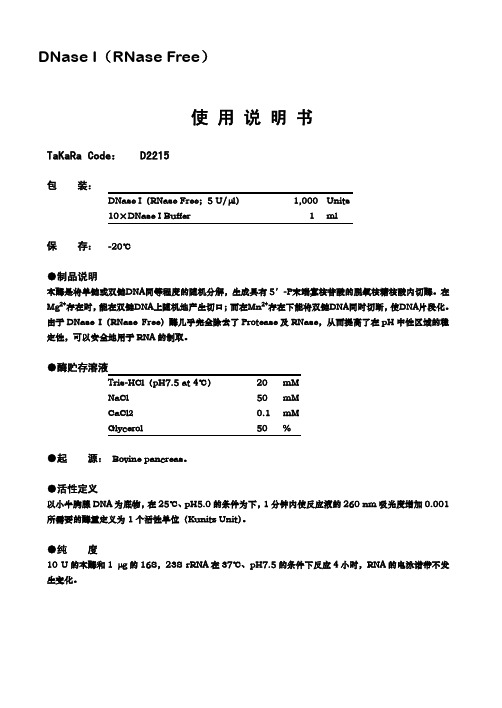
DNase I(RNase Free)使 用 说 明 书TaKaRa Code: D2215包 装:DNase I(RNase Free;5 U/μl) 1,000 Units10×DNase I Buffer 1 ml保 存: -20℃●制品说明本酶是将单链或双链DNA同等程度的随机分解,生成具有5′-P末端寡核苷酸的脱氧核糖核酸内切酶。
在Mg2+存在时,能在双链DNA上随机地产生切口;而在Mn2+存在下能将双链DNA同时切断,使DNA片段化。
由于DNase I(RNase Free)酶几乎完全除去了Protease及RNase,从而提高了在pH中性区域的稳定性,可以安全地用于RNA的制取。
●酶贮存溶液Tris-HCl(pH7.5 at 4℃) 20 mMNaCl 50 mMCaCl2 0.1 mMGlycerol 50 %●起 源:Bovine pancreas。
●活性定义以小牛胸腺DNA为底物,在25℃、pH5.0的条件为下,1分钟内使反应液的260 nm吸光度增加0.001所需要的酶量定义为1个活性单位(Kunitz Unit)。
●纯 度10 U的本酶和1μg的16S,23S rRNA在37℃、pH7.5的条件下反应4小时,RNA的电泳谱带不发生变化。
●用 途1)用T7或SP6 RNA Polymerase进行体外转录(In vitro transcription)时,合成RNA后,不必 更换Buffer,使用DNase I(RNase Free)即可将模板DNA降解。
2) 与DNA Polymerase I一起用于切口平移(Nick translation)。
3) 在Mn2+存在的条件下,为鸟枪法测序(Shot gun sequencing)制作DNA文库。
4) 用于足迹法(Foot printing)分析DNA-蛋白质相互作用。
●添附Buffer组成(保存:-20℃)10×DNase I BufferTris–HCl(pH7.5) 400 mMMgCl280 mMDTT 50 mM●使用例除去RNA中的基因组DNA1. 在微量离心管中配制下列反应液,全量50μl。
基因工程简答题

(1)修复反应,制备平末端
可用Klenow酶修复限制性内切核酸酶或其他方法产生的5'或3'突出末端,制备平末端,这样可以使原来具有不相容的黏性末端的DNA片段通过平末端重组。如在反应系统中加入放射性同位素标记的脱氧核苷酸,用这种末端填补的方法可以制备3'末端标记的探针。
答:
基因型是lac-.原因是在该密码子中插入了两个碱基,造成了移码突变,所以Lac的基因是缺陷的
27.在基因工程中,为了在细菌细胞中表达真核生物的基因产物,为什么通常要用cDNA
而不用基因组DNA?为什么要在cDNA前加上细菌的启动子?
答:
这是因为细菌没有内含子剪接系统,并且不能识别真核生物的启动子之故。
10. 列举质粒载体必须具备的4个基本特性。
.答:
(1)独立复制; (2)有选择标记; (3)有独特的酶切位点; (4)能转化但不扩散。
11. 什么叫穿梭载体?
答:
含有细菌质粒和克隆的真核生物DNA片段的杂种质粒,有两个复制起点和既能在细菌又能在真核细胞中进行选择的选择标记,所以,很容易从一宿主转到另一个宿主(来回穿梭)。
条件的改变会出现第二活性的酶的右上角加一个星号表示,因此第二活性又称为星
活性。
概括起来,诱发星活性的因素有如下几种:(1)高甘油含量(>5%, v/v);(2)限制性
内切核酸酶用量过高(>100U/ugDNA);(3)低离子强度(<25 mmol/L);(4)高pH(8.0
如果你的杂交探针是双链的,可能因为在加人杂交混合物之前忘记将探针变性而得到空白的放射自显影结果。
23.切口移位(nick translation)标记探针的主要步骤有哪些?
某理工大学《现代分子生物学》考试试卷(630)

某理工大学《现代分子生物学》课程试卷(含答案)__________学年第___学期考试类型:(闭卷)考试考试时间:90 分钟年级专业_____________学号_____________ 姓名_____________1、分析题(5分,每题5分)1. 写出原核表达实验步骤。
答案:原核表达实验步骤如下:(1)按上述步骤提取该组织的RNA,反转录为cDNA。
(2)以cDNA为模板,用上述引物扩增出带酶切位点的A基因片段。
(3)用BamHI和EcoRI分别切割目的片段和载体,连接酶将A 基因和载体连接起来构成重组质粒。
(4)将重组质粒转入感受态的大肠杆菌BL21中,涂布,根据载体所携带的抗性标记筛选出稳定遗传重组质粒的单克隆。
(5)摇菌、扩大培养,待菌液生长到对数生长期,收集菌体,裂解、收集蛋白质。
(6)将总蛋白制备成溶液通过镍柱,由于A蛋白携带有His标签,可以被吸附在镍柱上,然后加缓冲液将吸附在镍柱上的A蛋白洗脱下来,即可得到A蛋白溶液。
解析:2、判断题(80分,每题5分)1. 动物细胞中线粒体有自己的基因组,其编码基因可以在线粒体中完成转录和翻译过程。
()答案:错误解析:2. B型双螺旋是DNA的普遍构型,而Z型则被确定为仅存在于某些低等真核细胞中。
()答案:错误解析:B型双螺旋是DNA的普遍构型,而Z型在高等真核生物中也有发现,往往与基因表达调控有关。
3. 细胞内DNA复制后的错配修复不需要消耗能量。
()答案:错误解析:细胞内DNA复制后的错配修复需要水解ATP提供能量。
4. 一个基因就是一个转录单位。
()答案:错误解析:转录单位是一段可被 RNA聚合酶转录成一条连续的mRNA的DNA,包括转录起始和终止信号。
转录单位不同于基因,转录单位可能同时包含一个基因,也可能同时包含几个基因,另外,转录单位还包含启动子,非编码序列等。
所以,转录单位包含的DNA范围比基因广。
5. tRNA转录后加工有剪接反应,它是由蛋白质催化的。
生物化学及分子生物学(人卫第八版)-第22章-基因结构与功能分析

目录
第一节 基因结构分析技术
目录
一、基因一级结构解析技术
基因的一级结构是指脱氧核苷酸的排列 顺序,解析一级结构最精确的技术就是DNA
测序(DNA sequencing)。
目录
目录
3. 用原位杂交进行RNA区域定位 原位杂交( ISH )是通过设计与目标 RNA
碱基序列互补的寡核苷酸探针,利用杂交原理
在组织原位检测 RNA 的技术,其可对细胞或组 织中原位表达的 RNA 进行区域定位,同时也可 作为定量分析的补充。
目录
(二)用PCR技术检测RNA表达水平
1. 用逆转录PCR进行RNA的半定量分析 逆转录 PCR ( RT-PCR )一般用于 RNA 的 定性分析;如果设置阳性参照,则可对待测 RNA样品进行半定量分析。 2. 用实时定量PCR进行RNA的定量分析 实时定量 PCR 是定量分析 RNA 的最通用、 最快速、最简便的方法,该方法是对 PCR 反应 进行实时监测,具有很高的灵敏度和特异性。
目录
2. 预测启动子的其他结构特征
启动子区域的其他结构特征包括 GC 含量、 CpG 比 率、转录因子结合位点、碱基组成及核心启动子元件等。 用 于 启 动 子 预 测 的 数 据 库 : EPD ( eukaryotic promoter databases )数据库,主要预测真核 RNA 聚合
目录
(1)用核酸酶进行足迹分析
酶 足 迹 法 ( enzymatic footprinting ) 是利用 DNA 酶处理 DNA蛋白质复合物,然后通过 电泳分析蛋白质结合序列。 常用的酶有 DNA 酶 I ( DNase I )和核酸外切 酶III。
武汉大学分子生物学考研真题

武汉大学2001年攻读硕士学位研究生入学考试试题一、解释概念(20分,每个4分)1.Satellite DNA-卫星DNA,又称串联重复序列,由2~6个核苷酸组成的重复单位组成的串联重复。
2.Replisome-复制体,是指由多种复制相关蛋白组成的复合物。
3.逆转座子,指另一类能够通过逆转酶和整合酶共同作用介导转座的转座子4.反式激活因子,一类能结合到顺式作用元件上形式激活作用的蛋白质5.衰减子,原核生物操纵子上一段能够显著减弱或终止转录作用的核苷酸序列衰减作用,指原核生物中根据底物浓度变化来调控基因转录进程的中断与否的一种调控机制。
三、问答题(50分)1.说出双链DNA复制起始有关的五种重要的酶或蛋白并简述它们的功能。
(15分)2.简述增强子的特点和性质及作用机制。
(10分)3.简述真核RNA聚合酶II的转录起始复合物装配过程和转录起始(15分)4.DNA限制性内切酶EcoRI是人们熟悉的常用内切酶,它是在大肠杆菌(E.coli)R株中发现的,它被广泛用于分子克隆操作和DNA分析。
pUC质粒是常用克隆载体之一,它的多克隆位点上有EcoRI、BamHI、KpnI、HindIII等酶切点。
假如要你把一段由EcoRI切割产生的外源DNA段克隆到pUC质粒中,并把重组质粒转化大肠杆菌R株来扩增,已知条件是所用的R 菌株中只有EcoRI一种限制性内切酶,你设计如何做才能确保成功?为什么?(10分)武汉大学2002年攻读硕士学位研究生入学考试试题三、问答:1、简述(或绘图说明)真核细胞RNA聚合酶II转录的起始需要哪些基本转录因子及其装配过程(15分)2、简述(或绘图说明)色氨酸操纵子弱化的机制(15分)3、在讨论基因家庭时经常提到胚胎、胎儿和成体形成的蛋白质,这些述语是指什么现象?可用什么术语来描述这一类基因家族(5分)4、你正在进行Southern blot分析,并刚刚完成凝胶电泳部分,下一步是将胶浸泡在NaOH溶液中使DNA变性为单链,为了节约时间,你跳过这一步,直接把DNA从胶上转到硝酸纤维素膜上,你将标记好的探针与膜杂交,却发现放射自显影结果是一片空白,哪里错了呢?(5分)武汉大学2003年攻读硕士学位研究生入学考试试题一、名词翻译,并简要解释(共10小题,每题4分,共40分)1.Domains and motifs结构域和结构基序,分别指的是反式作用因子中与蛋白质和DNA分子相互作用的两种蛋白质亚基。
基因工程原理与技术思考题

Chapter I Introduction1)什么是基因?基因有哪些主要特点?基因是一段可以编码具有某种生物学功能物质的核苷酸序列。
①不同基因具有相同的物质基础。
②基因是可以切割的。
③基因是可以转移的。
④多肽与基因之间存在对应关系.⑤遗传密码是通用的.⑥基因可以通过复制把遗传信息传递给下一代。
2)翻译并解释下列名词genetic engineering遗传工程gene engineering基因工程:通过基因操作,将目的基因或DNA片段与合适的载体连接转入目标生物获得新的遗传性状的操作。
gene manipulation基因操作:对基因进行分离、分析、改造、检测、表达、重组和转移等操作的总称.recombinant DNA technique重组DNA技术gene cloning基因克隆:是指对基因进行分离和扩大繁殖等操作过程,其目的在于获得大量的基因拷贝,在技术上主要包括载体构建、大肠杆菌遗传转化、重组子筛选和扩大繁殖等环节。
molecular cloning分子克隆3)什么是基因工程?简述基因工程的基本过程?p2 p44)简述基因工程研究的主要内容?p55)简述基因工程诞生理论基础p2和技术准备有哪些p3?6)基因表达的产物中,氨基酸序列相同时,基因密码子是否一定相同?为什么?否,密码子简并性7)举例说明基因工程技术在医学、农业、工业等领域的应用。
医学:人胰岛素和疫苗农业:抗虫BT农药工业:工程酿酒酵母Chapter ⅡThe tools of trade1)什么是限制性核酸内切酶?简述其主要类型和特点?是一种核酸水解酶,主要从细菌中分离得到.类型特点p112)II型核酸内切酶的基本特点有哪些p12—14?简述影响核酸内切酶活性的因素有哪些p14?3)解释限制酶的信号活性?抑制星号活性的方法有哪些?4)什么是DNA连接酶p15?有哪几类p16?有何不同p16?5)什么叫同尾酶、同裂酶p12?在基因工程中有何应用价值?同裂酶:识别位点、切割位点均相同,来源不同.在载体构建方面往往可以取得巧妙的应用.应用较多的同裂酶比如Sma1和Xma1,它们均识别CCCGGG,但前者切后产生钝同尾酶:来源各异,识别序列各不相同,但切割后产生相同的粘性末端。
核酸检测篇6-DNase I足迹分析
编号:2-6主题:DNase I足迹试验概述:DNase I足迹法于1978年引入科研领域,其原理与DNA的化学测序法相似,首先将待测双链DNA片段中一条链的一端选择性地进行标记,然后加入适当浓度的DNaseI,使在DNA链上形成缺口,经过变性后电泳分离,放射自显影,即形成以相差一个核苷酸为梯度的DNA条带。
但当DNA片段与相应的序列特异性DNA结合蛋白结合后,DNA结合蛋白可保护相应的DNA序列不受DNase I 的攻击,因此在放射自显影图谱上,DNA梯度条带在相应的DNA结合蛋白的结合区域中断,从而形成一空白区域,恰似蛋白质在DNA上留下的足迹,因而被形象地称作足迹法。
如果同时进行DNA化学测序,即可判断出结合序列的精确顺序,该技术至今仍然是最常用的方法,但是在实际应用中,首先应将序列特异蛋白进行一定程度的纯化,如硫酸铵分步沉淀,离子交换层析,凝胶过滤层析等,否则很难得到满意的结果。
因此在该足迹法的基础上又发展了固相DNaseI足迹法,它具有如下优点: (1)采用生物素进行末端标记,避免放射性同位素的掺人,减少危害; (2)省略了有机抽提、沉淀等蛋白纯化步骤,操作简便,效率高; (3)使用范围广,可适用于未经纯化的核蛋白粗提物内特异性DNA结合蛋白的研究。
DNase I足迹法常与EMSA法结合共同用于体外DNA-蛋白质相互作用的鉴定,但二者的侧重点不同. EMSA主要用于与特异性DNA结合的目标蛋白的检测,而DNase I足迹法在此基础上进一步证明了DNA元件和目标蛋白的特异结合,并能告知与该蛋白结合的相应DNA元件序列。
目的:鉴别RNA聚合酶等蛋白质在DNA上的结合位点,找到与特异性DNA结合的目标蛋白,而且能告知目标蛋白结合在哪些碱基部位。
原理:将含有特定顺式元件的双链DNA片段进行单链单末端标记,与初步纯化的DNA结合蛋白在体外行结合作用,然后加入DNase I对DNA-蛋白质复合物进行随机切割,产生一系列不同长度的DNA片段,其相邻片段只相差一个核苷酸。
分子机制-核酸检测-DNaseI足迹分析
分⼦机制-核酸检测-DNaseI⾜迹分析主题:DNase I⾜迹试验概述:DNase I⾜迹法于1978年引⼊科研领域,其原理与DNA的化学测序法相似,⾸先将待测双链DNA⽚段中⼀条链的⼀端选择性地进⾏标记,然后加⼊适当浓度的DNaseI,使在DNA链上形成缺⼝,经过变性后电泳分离,放射⾃显影,即形成以相差⼀个核苷酸为梯度的DNA条带。
但当DNA⽚段与相应的序列特异性DNA结合蛋⽩结合后,DNA结合蛋⽩可保护相应的DNA序列不受DNase I 的攻击,因此在放射⾃显影图谱上,DNA梯度条带在相应的DNA结合蛋⽩的结合区域中断,从⽽形成⼀空⽩区域,恰似蛋⽩质在DNA上留下的⾜迹,因⽽被形象地称作⾜迹法。
如果同时进⾏DNA化学测序,即可判断出结合序列的精确顺序,该技术⾄今仍然是最常⽤的⽅法,但是在实际应⽤中,⾸先应将序列特异蛋⽩进⾏⼀定程度的纯化,如硫酸铵分步沉淀,离⼦交换层析,凝胶过滤层析等,否则很难得到满意的结果。
因此在该⾜迹法的基础上⼜发展了固相DNaseI⾜迹法,它具有如下优点: (1)采⽤⽣物素进⾏末端标记,避免放射性同位素的掺⼈,减少危害; (2)省略了有机抽提、沉淀等蛋⽩纯化步骤,操作简便,效率⾼; (3)使⽤范围⼴,可适⽤于未经纯化的核蛋⽩粗提物内特异性DNA结合蛋⽩的研究。
DNase I⾜迹法常与EMSA法结合共同⽤于体外DNA-蛋⽩质相互作⽤的鉴定,但⼆者的侧重点不同. EMSA主要⽤于与特异性DNA结合的⽬标蛋⽩的检测,⽽DNase I⾜迹法在此基础上进⼀步证明了DNA元件和⽬标蛋⽩的特异结合,并能告知与该蛋⽩结合的相应DNA元件序列。
⽬的:鉴别RNA聚合酶等蛋⽩质在DNA上的结合位点,找到与特异性DNA结合的⽬标蛋⽩,⽽且能告知⽬标蛋⽩结合在哪些碱基部位。
原理:将含有特定顺式元件的双链DNA⽚段进⾏单链单末端标记,与初步纯化的DNA结合蛋⽩在体外⾏结合作⽤,然后加⼊DNase I 对DNA-蛋⽩质复合物进⾏随机切割,产⽣⼀系列不同长度的DNA⽚段,其相邻⽚段只相差⼀个核苷酸。
现代分子生物学
生物医学工程1202班林可慧研究DNA-蛋白质相互作用的实验方法主要包括:一、DNase 1 足迹实验二、甲基化干扰实验三、凝胶阻滞实验四、体内足迹实验五、拉下实验一、DNase 1足迹实验1、概念:足迹实验(foot-printing assay),是一种用来检测被特定转录因子蛋白质特异性结合的DNA 序列的位置及其核苷酸序列结构的专门实验方法。
2、原理:当DNA分子中的某一区段同特异的转录因子结合之后便可以得到保护而免受DNaseI 酶的切割作用,而不会产生出相应的切割分子,结果在凝胶电泳放射性自显影图片上便出现了一个空白区,俗称为“足迹”。
3过程:(1).将待检测的双链DNA分子在体外用32P作5‘末端标记,并用适当的限制性内切酶切出其中的一个末端,于是便得到了一条单链末端标记的双链DNA(2).在体外同细胞蛋白质提取物(细胞核提取物也可以)混合,形成DNA-蛋白质复合体(3).在反应混合物中加入少量的DNase I,并控制用量使之达到平均每条DNA链,只发生一次磷酸二酯键的断裂:a、如果蛋白质提取物中不存在与DNA结合的特定蛋白质,使DNase I消化之后,便会产生出距离放射性标记末端1个核苷酸,2个核苷酸,3个核苷酸------等等一系列前后长度均相差一个核苷酸的不间断的连续的DNA片段梯度群体;b、如果DNA分子同蛋白质提取物中的某种转录因子结合,被结合部位的DNA就可以得到保护免受DNase I酶的降解作用;(4).除去蛋白,加样在20%序列胶上进行电泳分离,实验分两组:a、实验组:DNA+蛋白质混合物b、对照组:只有DNA,未与蛋白质提取物进行温育(5).最后进行放射性自显影,分析实验结果。
4、结果判断:实验组凝胶电泳显示的序列,出现空白的区域表明是转录因子蛋白质结合部;与对照组序列比较,便可以得出蛋白质结合部位的DNA区段相应的核苷酸序列。
5、其他的足迹实验方法:除了DNase1足迹试验之外,目前还发展出了若干种其他类型的足迹实验,例如:a、自由羟基足迹实验;b、菲咯啉铜足迹实验;c、DMS(硫酸二甲酯)足迹实验DMS(硫酸二甲酯)足迹实验的原理DMS能够使DNA分子中裸露的鸟嘌呤(G)残基甲基化,而六氢吡啶又会对甲基化的G 残基作特异性的化学切割。
6-生物信息学-转录调控分析
出现的概率相互独立。 矩阵每一列表示模体相应位置上四种碱基 出现的概率。 对于长度为n的模体,碱基i(i={A, C, G, T})在模体第j 个位置上出现的频率为q i,j,则整个模体用矩阵M表示如下:
q A,1 q A,2 ∙∙∙ q A,n q C,1 q C,2 ∙∙∙ q C,n
G,1
REDUCE 算法:以模体出现的次数作为自变量
来进行简单线性回归
MatrixREDUCE算法:用位置频率矩阵的打分作
为自变量进行回归
MARSMotif-M算法:多变量适应回归模型
转录因子结合位点分析可利用网络资源
Category Single motif discovery Program MobyDick YMF Consensus MEME Gibbs Sampler URL /mobydick/ /software.html /software.html /meme/intro.html /gibbs/gibbs.html
High-throughput Techniques in Transcriptional Regulation Analysis
一、ChIP技术
创立者:
20世纪80年代末
Alexander Varshavsky等人
(Cell. 1988,53(6): 937-947 )
基本实验过程: 甲醛交联,稳定蛋白质-DNA复合物 裂解细胞,分离蛋白质-DNA复合物 加入特异性抗体,沉淀蛋白质-DNA复合物 去交联,纯化DNA 应用PCR技术,特异性扩增目的DNA片段
M= q
q G,2 ∙∙∙ q G,n
q T,1 q T,2 ∙∙∙ q T,n
(三)序列标识图(sequence logo)
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
主题:DNase I足迹试验
概述:
DNase I足迹法于1978年引入科研领域,其原理与DNA的化学测序法相似,首先将待测双链DNA片段中一条链的一端选择性地进行标记,然后加入适当
浓度的DNaseI,使在DNA链上形成缺口,经过变性后电泳分离,放射自显影,即形成以相差一个核苷酸为梯度的DNA条带。
但当DNA片段与相应的序列特异性DNA结合蛋白结合后,DNA结合蛋白可保护相应的DNA序列不受DNase I 的攻击,因此在放射自显影图谱上,DNA梯度条带在相应的DNA结
合蛋白的结合区域中断,从而形成一空白区域,恰似蛋白质在DNA上留下的
足迹,因而被形象地称作足迹法。
如果同时进行DNA化学测序,即可判断出
结合序列的精确顺序,该技术至今仍然是最常用的方法,但是在实际应用中,
首先应将序列特异蛋白进行一定程度的纯化,如硫酸铵分步沉淀,离子交换层析,凝胶过滤层析等,否则很难得到满意的结果。
因此在该足迹法的基础上又
发展了固相DNaseI足迹法,它具有如下优点: (1)采用生物素进行末端标记,避免放射性同位素的掺人,减少危害; (2)省略了有机抽提、沉淀等蛋白纯化步骤,操作简便,效率高; (3)使用范围广,可适用于未经纯化的核蛋白粗提物内
特异性DNA结合蛋白的研究。
DNase I足迹法常与EMSA法结合共同用于体外DNA-蛋白质相互作用的鉴定,但二者的侧重点不同. EMSA主要用于与特异性DNA结合的目标蛋白的检测,而DNase I足迹法在此基础上进一步证明了DNA元件和目标蛋白的特异结合,并能告知与该蛋白结合的相应DNA元件序列。
目的:
鉴别RNA聚合酶等蛋白质在DNA上的结合位点,找到与特异性DNA结合的
目标蛋白,而且能告知目标蛋白结合在哪些碱基部位。
原理:
将含有特定顺式元件的双链DNA片段进行单链单末端标记,与初步纯化的DNA结合蛋白在体外行结合作用,然后加入DNase I对DNA-蛋白质复合物进行随机切割,产生一系列不同长度的DNA片段,其相邻片段只相差一个核苷酸。
DNA结合蛋白与其特异序列结合后,由于空间位阻等多种效应,DNase I 不能对其进行切割。
步骤:
1.探针制备及纯化:
构建含启动子片段的质粒,双酶切质粒,同位素标记,回收标记后的启动子片段。
2.DNaseⅠ足迹分析样品处理:
蛋白质与DNA混合,等两者结合后,加入适量的DNase I,消化DNA分子,控制酶的用量,使之达到每个DNA分子只发生一次磷酸二酯键断裂,并列设置未加蛋白质的对照.
3.G+A化学测序反应和电泳:
从DNA上除去蛋白质,将变性的DNA加样在测序凝胶中作电泳和放射性自显影,与对照组相比后解读出足迹部位的核苷酸序列。
流程图:。
