spm8-fMRI数据处理
fMRI数据分析处理原理及方法

fMRI数据分析处理原理及方法一、功能图像数据的性质功能磁共振数据包括解剖(结构)像和功能像两类。
解剖像采用高分辨的T1、T2及FSPGR三维成像方式。
功能像的处理是fMRI数据处理的关键。
因为脑皮层活动瞬息变化,相应要求足够快的成像序列对某一个刺激任务造成的皮层活动进行记录,并且要有对脑血氧代谢的产物——脱氧血红蛋白产生的T2*缩短效应敏感,EPI(Echo planar Imaging)、FLASH(Fast Low Angle Shot)等序列可以满足这两个条件,现在大都采用EPI序列采集fMRI功能像。
EPI于频率编码上采用一系列反向梯度,通过一次激发产生建成一幅MR图像的所有信号,基于小角度激发的GRE-EPI(Gradient echo- Echo planar Imaging)技术,在很短的TR时间内得到一系列(数幅至数十幅)图像。
每次采集得到的图像组成一个脑体积(V olume),相应要求在fMRI实验组块(Epoch/block Paradigm)设计时,每个组块的时间必须为TR时间的整数倍。
实际的血流动力相应是一个缓慢的过程,任务激发后信号经过一个小的下降期开始上升,4-8秒达到高峰然后缓慢下降,11-14秒恢复。
在事件相关设计(Event-related Paradigm)时,如果不考虑两(次)任务间的相互作用,需要保证间隔时间大于一次响应时间。
但也有研究显示短的刺激间隔时间对统计结果并无多大影响。
(见图1)。
EPI序列以极快的采集速度,在一个数分钟的实验(Session)中,产生数百至数千幅图像,几十个不同时间的脑体积成为EPI图像的时间序列(Time-series Image)。
快速以牺牲图像的分辨率为代价,典型的EPI图像采集矩阵为64×64,提高采集矩阵会延长采样时间并且导致更严重的图像几何变形。
除此之外,EPI 序列图像对外在磁场环境的影响十分敏感,微弱的BOLD信号会伴有大量的干扰成分。
VBM8处理流程

图像平滑
SPM Smooth Images to Smooth X:选择要进行平滑的图像 FWHM:设定高斯平滑参数,常用范围是 8-12mm. Data Type: 使用默认参数即可。 Filename Prefix: 使用默认参数即可。 File Save Batch: 保存 batch。 FileRun Batch:执行 batch。
2.
3. 统计分析
1. 2.
3.
3. VBM 分析流程详细描述
组织分割与标准化
VBM8 Estimate and write Volumes X :输入解剖图像,一般为 T1W 图像。由于在后续分割中,需要和 MNI 先验模 板对齐,所以这里输入数据最好能和先验 MNI 模板方向大致相同。若图像和模板方向差 异较大,可以使用 SPM 的 Display 和 Check Reg 按钮进行手动调整。 Estimation Options :使用默认参数即可。这里若不采用 SPM 自带的组织先验模板 TPM, 则可选择自己定制的模板。 Extended Options :使用默认参数即可。若要尽可能清除非大脑组织,可更换“Clean up any partitions”为 “Thorough Clean up”。也可以尝试改变两类降噪方法的权重,ORNLM 的最优权重是 0.7。MRF 的权重不需要调整。当不使用某个降噪方法时,可直接把其权重 设为 0。 Writing Options :使用默认参数即可。 默认的“Modulated normalized-non linear only”:仅对非线性变换带来的体积改变进行 调制后的图像,voxel 值是经过 brain size 校正后的局部组织相对体积。 A bias corrected image volume: 磁场不均匀性校正后的图像。可使用期与不校正的原 始对象进行比较,验证图像质量。 A partial volume effect (PVE) label image volume:该 volume 中的值是对每个 voxel 局 部容积效应的估计。 Jacobian determinant:每个 voxel 值表示 MNI 模板上该位置变换到被试空间时,体积 变化大小。
SPM8数据预处理
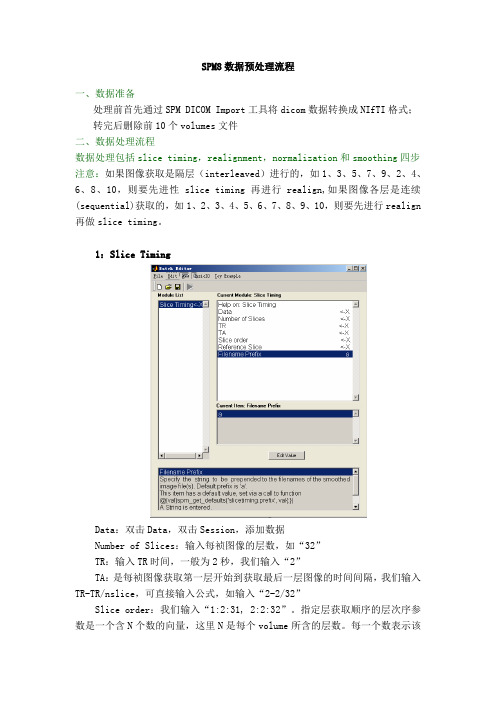
SPM8数据预处理流程一、数据准备处理前首先通过SPM DICOM Import工具将dicom数据转换成NIfTI格式;转完后删除前10个volumes文件二、数据处理流程数据处理包括slice timing,realignment,normalization和smoothing四步注意:如果图像获取是隔层(interleaved)进行的,如1、3、5、7、9、2、4、6、8、10,则要先进性slice timing 再进行realign,如果图像各层是连续(sequential)获取的,如1、2、3、4、5、6、7、8、9、10,则要先进行realign 再做slice timing。
1:Slice TimingData:双击Data,双击Session,添加数据Number of Slices:输入每祯图像的层数,如“32”TR:输入TR时间,一般为2秒,我们输入“2”TA:是每祯图像获取第一层开始到获取最后一层图像的时间间隔,我们输入TR-TR/nslice,可直接输入公式,如输入“2-2/32”Slice order:我们输入“1:2:31, 2:2:32”。
指定层获取顺序的层次序参数是一个含N个数的向量,这里N是每个volume所含的层数。
每一个数表示该层在图像(volume)中的位置。
向量内的数字排列顺序是这些层的获取时间顺序。
如行向量 [1 3 5 7 9 11 13 15 17 19 21 23 25 2 4 6 8 10 12 14 16 18 20 22 24](在Matlab中可表示为[1:2:25,2:2,25])各种扫描类型和输入的层顺序如下:ascending 升序扫描(first slice=bottom): [1:1:nslices];descending 降序扫描(first slice=top): [nslices:-1:1];interleaved 间隔扫描(middle-top):for k = 1:nslices,round((nslices-k)/2 + (rem((nslices-k),2) * (nslices - 1)/2)) + 1,end; interleaved (bottom -> up): [1:2:nslices 2:2:nslices],如[1:2:25,2:2,25];interleaved (top -> down): [nslices:-2:1, nslices-1:-2:1]Reference Slice:我们输入“31”。
全面讲解spm8,教你如何fMRI数据处理(最新整理)

目录SPM简介和安装 (1)一、数据准备(先设置数据输入和输出目录,再转换数据格式) (2)二、数据预处理流程 (2)0、预处理的workflow (2)1、Slice Timing时间层校正 (3)2、Realignment 头动校正 (3)3、Coregister 配准 (5)4、Segment 分割 (6)5、Normalize 空间标准化 (6)6、Smooth 平滑 (8)三、GLM模型和Specify 1st-level (9)四、实例:任务态数据预处理和一阶分析的批处理 (13)SPM简介和安装SPM,即统计参数图,也是这个软件的最终输出,它是由英国伦敦大学的Friston教授等人在通用数学软件包Matlab基础上开发的软件系统,其统计功能非常强大,设计这个软件包的初衷是采用统计的方法来处理fMRI,PET和SPECT的数据。
用SPM进行数据处理分析过程主要分为两大部分:预处理过程和统计分析过程。
需要注意的是,静息态、任务态和DTI数据的预处理大致框架一致,但具体步骤不同,本教程主要讲任务态数据处理。
数据预处理主要包括三个大框架:(1)Convert dicom files to hdr files and img images;(2) Temporal processing,即Slice Timing;(3)Spatial processing,包括 Realign、Normalize和Smooth。
具体步骤下面有讲。
统计分析过程包括:个体分析和组分析。
使用Specify 1st level 做单个被试(single subject)分析;使用Specify 2nd level做组分析(group analysis)。
先写一下SPM8的安装:有必要说一下SPM实际不是一个独立的软件,它相当于一个用Matlab程序编写的工具箱,必须依赖Matlab的环境完成其功能。
言归正传,首先将下载好的SPM8程序包导入Matlab,复制整个spm8文件夹到MATLAB的安装路径:MATLAB\R2009a\toolbox\下。
ASL数据处理教程

选择MASK,根据图像 dimension大小选择MASK 输出目录 Let`s go!
谢谢观赏 !
标准化
进入标准化界面
Normalize
T1分割后“seg-sn” 文件 ASL功能像图像
双击,按下图所示修改数值
平滑
进入平滑界面
Smooth
标准化后的功能像
双击,修改数值如图所示
统计分析
预处理已经完成
进行统计分析,本文选用配对T检验
打开rest,进入统计分析界面
Paired T-test
3D-ASL数据预处理
所需软件PM fmri
配准
进入配准界面
Coregister
ASL CBF图像(功能像)
T1图像(结构像)
T1分割
进入分割界面 分割结构像
Segment
配准后的T1结构像
两项均选择 “Native + Modulate Normalize”
fMRI数据处理

T2
90 100 300 - 84
T1WI
PDWI
T2WI
脑功能成像技术
Deoxy-Hb Decrease
BOLD原理
合氧血红蛋白和脱氧血红蛋白的比率变化
SPM软件包简介
主要的脑功能分析软件:
①SPM:/spm 运行平台:MATLAB (WINDOWS & Linux) 数据格式:Analyze,MINC,ecat T 注:在最新的SPM5\spm8中将只采用NIFTI格式
6 信号和图像处理(高等数学)
要有对信号和图像处理方法的基本了解,特别是 对傅里叶分析的理解。(傅里叶分析啊....总是在 papers中看到...可惜不懂......)
精心钻研十年!才能成 为一个领域的专家!在 脑功能成像(fMRI)分 析领域尤是如此!
fMRI研究框架
科学问题 结果解释
实验假设
Slice Timing
运行matlab, 执行命令:spm fmri,这样将打 开spm8的操作界面。点击“Slice Timing” .
在spm8,每一步处理都采用了直观的 “树形结构”面板, 如果一个分支项右 面有 <-X号,你必须为之指定选项(否 则不能运行该tree),分支项的选项在 其右侧面板指定,而帮助信息则在下 面的面板中显示。
一般功能面板
SPM软件包简介
SPM的处理流程:
校准
标准化
一般线形 模型
平滑
统计参数图
数据处理前准备
数据格式转换。 脑图的左右显示问题。
spm_flip_analyze_images.m中flip = 1 or 0 Spm以前的版本,spm_defouts.m文件中
defaults.analyze.flip = 1or 0
SPM任务态fMRI数据分析培训

Matlab数据处理常用命令
变量赋值: a = 14; b = [2 5 8]; c = [1 2; 3 4; 5 6; 7 8]; d = c; e = d'; clear e d c
改变路径: cd cd .. cd ../.. pwd dir ls
Matlab界面的基本组成部分
和“我的电脑” 一样,这里可以 进入文件夹,创建/ 删除/复制/粘帖 等等……
命令在这里输入, Matlab输出信息在这里看, 报错信息(红字)在这里找 跑程序的话,把命令行粘帖在这里 运行即可!
电脑内存里 的变量都存 在这里,
可以对变量 进行删除, 查看,作图 等操作,
SPM数据分析基本流程
预处理部分
模型构建与 参数估计
常用工具与 参数设置
预处理流程
Slice timing是什么?
SPM 假定每一个scan是同时获取的,但是……
头顶:3
0
TR
RAW CORRECTED
…
颅底: 1
TA
0
3
6
time in seconds
为什么要进行slice timing?
Template
Normalised Image
可以采用哪几种方法标准化?
1. mean EPI----- EPI template 2. T1 coreg with mean EPI, T1 ------ T1 template, Write
normalize 3. T1 coreg with mean EPI, T1 segment, Write normalize 4. DARTEL using SPM Batch Editor
SPMFDR校正

SPMFDR校正SPM8允许两种FDR校验。
⼀个是voxel-wise FDR,另⼀个是topological FDR。
如果要做voxel-wise FDR 校验,就把spm_defaults⾥的68⾏的defaults.stats.topoFDR 改为0。
(有很多朋友下载下来的spm在处理结果是只显⽰FWE矫正和NOR,但是FWE矫正⼜太严格,我们往往需要FDR矫正来进⾏数据处理。
spm的设置是在spm根⽬录下的spm_defaults这个⽂件。
需要再matlab中调⽤才能编辑。
在matlab中输⼊edit spm_defaults,在68⾏有defaults.stats.topoFDR = 1; 你把1改成0即可。
当然你也可以对默认阈值、默认设置的激活体素⼤⼩进⾏设置。
)此时SPM8⾥的FDR按钮会出现。
在SPM⾥点results,选FDR校验即可。
如果要做topological FDR 校验,就把defaults.stats.topoFDR设为1(默认是1)。
⾸先在SPM8⾥点results——None(⽆校验)——T threshold或p value(⾃⼰随意设,默认的0.001也可以)——extent threshold(默认0即可)。
然后在左下侧窗⼝点whole brain会得到results table。
找到FDRc对应的值。
然后再次点Results——None——T threshold或p value(输⼊上⼀次的值,例如上⼀次是0.001,这⼀次还是0.001)——extent threshold(输⼊FDRc对应的值)。
这样得到的激活图就是cluster-wise FDR校验的结果,也就是topological FDR校验的结果。
SPM⾥⾯有peak-level FDR,见result table⾥⾯的FDRp,⽤FDRp对应的值作为height threshold得到的结果就是peak-level FDR的结果。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
6)Smooth
7)Detrend
8)Filte
1
Slice Timing用来校正1个volume中层与层之间获取(采集)时间的差异,对事件相关设计的实验尤为重要。我们在按钮窗口中的预处理面板中点击“Slice Timing”,将出现一个对话框,修改其中参数:
Data:New:Session
Session:选择你要处理的数据,如文件夹data中的所有数据
SPM
SPM,即统计参数图,也是这个软件的最终输出,它是由英国伦敦大学的Friston教授等人在通用数学软件包Matlab基础上开发的软件系统,其统计功能非常强大,设计这个软件包的初衷是采用统计的方法来处理fMRI,PET和SPECT的数据。用SPM进行数据处理分析过程主要分为两大部分:预处理过程和统计分析过程。需要注意的是,静息态、任务态和DTI数据的预处理大致框架一致,但具体步骤不同,本教程主要讲任务态数据处理。
过程:
在spm8中选择Coregister(Estimate):
Reference Image---[选择头动校正后生成的mean*.img文件]
Source Image---[选择3D文件,即data3D文件夹中的.img文件]
其余选项采用默认设置,点击上方绿色的三角开始运行。
说明:Source image与Reference image的关系,可以认为是将结构像向以mean开头的功能像里估计,估计结束后就可以将旋转矩阵写入到精度更高的3D文件当中,最后做出的图像的分辨率就会很高。(结构像比功能像清晰很多)
其余选项采用默认设置,点击上方绿色的三角开始运行。
注意:很多研究者容易将时间校准和空间校准顺序颠倒,一般的观点是如果图像获取是隔层(interleaved)进行的,如1、3、5、7、9、2、4、6、8、10,则要先进行slice timing再进行realign,如果图像各层是连续(sequential)获取的,如1、2、3、4、5、6、7、8、9、10,则要先进行realign再做slice timing。
Number of Slices:我们输入每祯图像的层数,如“32”(可以在spm8->Display->data:.img->done->Dimensions:64*64*32中查看)
TR:我们输入重复时间,一般为2秒,我们输入“2”
TA:是每祯图像获取第一层开始到获取最后一层图像的时间间隔,TR-TR/nslice,可直接输入公式,如我们输入“2-2/32”
头动范围(Check Realign):平动≤2.0mm and旋转≤2.0degree[严老师观点]
我们在预处理面板校准选项中选择“Realign(Est&Res)”,出现一个参数设置对话框。
过程:
realign:estimate+reslice
Data:New:Session,然后选中data下出现的“Session”选项。点击“Specify Files”,用spm文件选择器选择刚做完时间校准的全部图像(a*.img)。
5)Normalize:①Normalize by using EPI templates,即使用公共的EPI模版来进行空间标准化,这样的话就用不到T1结构像,也就不需要对T1像进行Coregister和Segment;
②Normalize by using T1 image unified segmentation,即用T1像来进行空间标准化,这样的话需要用到T1结构像,所以需要在normalize前先对T1像进行Coregister和Segment,换句话说就是用coregisteredand segmented T1像来进行空间标准化。另外,如果以后希望把功能激活图像叠加到结构像上,那么结构像也需要做一次空间标准化。Parameters files和功能像的normalize一样,也选择在segment中生成的空间标准化参数文件(批处理中选择Subj→MNI)。Images to write选择在segment中场强校正后的结构像。这里的voxel size要设置结构像的大小,也就是[1,1,1]。
Filename Prefix:默认为r
其余选项采用默认设置,点击上方绿色的三角开始运行。
做完这一步,能给出该序列中被试的头动情况,以作为是否放弃该数据的依据,如果头动超过1个voxel(功能图像扫描矩阵一般是64*64,则体素的大小为(FOV/64)*(FOV/64)*(层厚+层间距)),则要考虑放弃该时间点数据。该程序利用最小二乘法(least squares approach)原理和含6个参数(刚体模型)的空间变换,对从一个被试获取的时间序列进行校正。用户可指定某个volume作为随后volumes的参考。可以是第1个volume,也可选择比较有代表性的volume(更明智的选择),例如选择磁场相对稳定的第4个volume。校正信息(头动信息)将在结果窗口(Graphics Window)显示。每个Session的校正信息将存储为rp*.txt,其中*为Session数据集名称。另外,头动校正信息将以plot图形显示。如下图:translation图表示被试头部在X,Y,Z三个方向的平移,分别用蓝,绿,红三种颜色表示。rotation图表示被试头部在实验过程当中绕X(L-R),Y(A-P),Z(S-I)三条轴的转动角度。横坐标代表这个序列所采集的所有图像,纵坐标表示的是偏移量和偏转角度,分别以毫米和度为单位。采用SPM8,头动信息和空间标准化的图形文件将以spm_“data”.ps的形式保存于matlab的工作目录下,如我们是2009年4月30日处理的数据,则将以spm_2009Apr30.ps文件存于matlab的work目录下。当然也可以将生成的图像保存成.jpg格式:File->Save As->.jpg
统计分析过程包括:个体分析和组分析。使用Specify 1st level 做单个被试(single subject)分析;使用 Specify 2nd level做组分析(group analysis)。
先写一下SPM8的安装:有必要说一下SPM实际不是一个独立的软件,它相当于一个用Matlab程序编写的工具箱,必须依赖Matlab的环境完成其功能。言归正传,首先将下载好的SPM8程序包导入Matlab,复制整个spm8文件夹到MATLAB的安装路径:MATLAB\R2009a\toolbox\下。然后运行Matlab,在其主窗口选择File->set path->Add with Subfolders->spm8->save->close。设置完成后在Matlab中执行命令>>spm fmri。这样出现了spm8的操作界面(如下图),这也表示spm8安装成功!我们称左上侧的窗口为按钮窗口(button window),左下侧的窗口为输入窗口(input window),右侧大窗口为树形结构窗口或图形窗口(Tree Building Window or the graphics window)。
4
目的:要将被试的结构像配到功能像里,就需要将结构像进行分割。一般分割为灰质、白质和脑脊液三部分。
过程:
在spm8中选择Segment
Data---[选择配准后的3D图像,即data3D文件夹中的.img文件]
.Clean up any partitions----Light clean
Affine Regularisation---[选择欧洲人或东亚人大脑模板]如:ICBMspace template-European brains
说明:①matlab中如何查看头动范围
打开rp_af*.txt文件,前3列为平动数据、后3列为旋转数据;
在matlab中输入命令:b=load(‘rp_af*.txt’); %----[载入头动数据文件]
c=max(abs(b)); %---[取b值的绝对值的最大值,表示找出每列的最大值]
c(4:6)=c(4:6)*180/pi %---[4-6列为转动,将以弧度为单位的数值转化为以角度为单位的值,pi表示π]
数据预处理主要包括三个大框架:(1)Convert dicom files to hdr files and img images;(2)Temporal processing,即Slice Timing;(3)Spatial processing,包括Realign、Normalize和Smooth。具体步骤下面有讲。
②运行结束后将生成一对mean*(.img和.hdr)文件(平均脑)、一个rp_*.txt文件(头动参数文件)及若干对r*(.img和.hdr)文件。
3
对于单个被试的研究,往往是通过在高分辨率结构图像上叠加激活区来精确定位功能区,也就是coregistration。
目的:上述头动校正的求解参数仅对同一被试的同一种成像方法(或成像模态modality)有效,对于同一被试的不同成像方法(功能像/结构像)所得图像,由于它们之间没有足够的可比性,不可以直接用头动校正的方法来求解参数,这时需要用图像配准的方法来做空间校正。具体为将所有的图像同一个volume对齐,对功能像与结构像做一个信息的变换。为什么选择Coregister里面的(Estimate)呢?因为我们相信对于被试,功能像与结构像是线性相关的平动与转动,而不是扭曲的。由功能像向结构像去配,对于结构像中的hdr文件存有一个矩阵,而这个矩阵就包含了功能像的信息。只需要将旋转的矩阵写入到hdr文件中,不需要生成新的文件,也就是对3D文件做一个刚体的变换,变换到功能像空间里。
