ebsco西文数据库使用说明
EBSCO外文数据库使用方法

EBSCO外文数据库 EBSCO外文数据库
信息开发部制作
进入哈尔滨商业大学图书馆
/
点击进入
选择数据库
可直接点击进入
可勾选后再继续
ASP检索界面
BSP检索界面
基本检索(以ASP为例)
常用限定选项
全文 :只检索有全文的文章 只检索有全文的文章 有参考: 有参考:只检索有参考文献的文章 学术(同行评审 期刊: 同行评审)期刊 学术 同行评审 期刊:有专家评审的期刊中的全文 出版日期: 出版日期:限定出版时间 出版物: 出版物:在限定的出版物中检索 出版物类型:期刊、报纸、 出版物类型:期刊、报纸、书等 页数:全文页数。 页数:全文页数。如:1-3; 10-; -5 附带图像的文章: 附带图像的文章:检索有图片的文章
图像检索(以ASP为例)
检索词输入框
限定条件选项
图像检 索结果
公司检索(BSP特有)
检索词输入框
查看公司概况
查看公司报表
公司概 况
报表结果 显示
辞典检索(BSP特有)
添加后生成检索词 检索词输入框
勾选项后添加
辞典检 索结果
我的EBSCOhost
点击登录
新用户注册
用户登录
可将结果添加到文件(ASP特有)
添加后生成检索词 检索词输入框
浏览方式选择
点击
勾选后再添加
点击
主题词 检索结 果
索引检索(以ASP为例)
索引输入框
浏览字段选择
勾选后再添加
点击
索引检 索结果
参考文献检索(以ASP为例)
检索词输入框
参考文 献检索 结果
检索有图片的文章检索词输入框数据库选择框限定条件选项检索结果细览检索结果处理方式高级检索高级检索以以aspasp为例检索词输入框检索字段限定条件选项数据库选择框检索结出版物检索出版物检索以以aspasp为例浏览方式选择添加后生成检索词检索词输入框勾选后再添加出版物检索结出版物细览点击出版物检索结果2主题词检索主题词检索aspasp特有特有浏览方式选择添加后生成检索词检索词输入框点击主题词检索结索引检索索引检索以以aspasp为例浏览字段选择索引输入框勾选后再添加索引检索结果参考文献检索参考文献检索以以aspasp为例检索词输入框参考文献检索结果图像检索图像检索以以aspasp为例限定条件选项检索词输入框图像检索结果公司检索公司检索bspbsp特有特有检索词输入框查看公司概况查看公司报表报表结果显示辞典检索辞典检索bspbsp特有特有添加后生成检索词检索词输入框勾选项后添加辞典检索结果我的ebscohostebscohost点击登录新用户注册用户登录文件夹的内容我的ebscohostebscohost功能功能保存文献检索结果保存图像检索结果保存检索过程中的历史检索记录检索过程中可建立检索快讯在出版物浏览过程中可建立期刊快报图书馆信息开发部电话
EBSCO数据库使用指南

EBSCO数据库使用指南1、登陆网址。
链接地址:,在我校IP范围内按地址自动登录。
2、检索方法2.1 检索界面基本检索界面,如图1高级检索界面,如图22.2 检索途径2.2.1 关键词检索点击关键词检索键,进入关键词检索界面。
在检索框中输入检索式。
基本检索和高级检索均默认为关键词检索。
(1)有关项目说明检索式检索式可以是一个词,也可以由检索字段标识符、检索词、逻辑算符以及位置算符等联合构成。
关键词关键词可以是一个完整的词,也可以使用通配符和截词符。
通配符主要用于拼写多变的单词,用问号“?”表示,只代替一个字符,可以输在词中或词尾,但不能用在词首。
截词符主要用于词根多变的词,以便扩大检索范围。
用星号“*”表示,可以代替一个或多个字符。
字段代码字段代码用两个字母表示,用于限定关键词检索的字段,输在检索词的前面。
缺省状态下在全文范围内检索。
其代码含义如下:AU为责任者、TI为题名、SU为主题词、AB为文摘、AN为登记号、IS为国际标准刊号、SO为刊名、AS作者自作文摘等。
根据数据库的不同,有的还有其它字段代码。
在高级检索界面下检索指导框的下拉菜单中都列了出来,可以参看。
逻辑算符该检索系统支持“and”、“or”、“not”三种逻辑运算,也可以用括弧括起来进行优先运算。
位置算符位置算符用于计算检索词之间能容纳的最大单词数。
它由一个字母和一个数字组成。
字母共有两个:N和W。
N是near的缩写,只限定检索词之间的单词数,而不限制检索词的顺序;W是within的缩写,既限定检索词之间的单词数,又保证检索词的顺序与输入的完全一致。
输入时放在检索词之间,如:taxn3reform或taxw5reform。
双引号“” 使用双引号,表示检索结果要与引号内的检索词完全匹配。
即检中的文献包含引号内的检索词,并且词序、词的位置都不变。
(2)逻辑关系的输入和选定在高级检索窗口下,逻辑关系可以直接输入,或者在检索指导框中选定逻辑算符,再点击检索指导框后的添加即可。
EBSCO(外文期刊数据库)使用介绍

EBSCO(外文期刊数据库)使用介绍 •EBSCOhost 由EBSCO Publishing出版•覆盖的学科范围包括:生物科学、工商经济、咨询科技、通讯传播、工程、教育、艺术、医药学等•检索结果为文献的题录、文摘信息。
其中部分文献提供全文(PDF 格式(查看PDF格式的全文要下载并安装Adobe Acrobat Reader软件,图书馆主页提供下载)、HTML格式)访问方式•任何一台接入校园网的计算机均可通过Web方式检索•进入EBSCOhost界面后,首先需要选择数据库,然后选择检索方式 •EBSCOhost主要检索方式为基本检索(basic search)和高级检索(advanced search)两种如果选择在单个数据库内检索,还有参考文献检索(Cited References)、图像检索(Images)、刊名浏览(Publications)、叙词浏览(Subject Terms)、索引浏览(Indexes)、等辅助检索工具基本检索基本检索是在篇名、作者、主题词、文摘中进行检索可做如下扩展–全文检索(在文章全文中进行检索)–逻辑“与”运算(如有两个及两个以上的检索词,自动进行“与”—and 的运算)–检索相关词还可做如下限定–全文限定(有全文)–参考文献限定(有参考文献)–学术性期刊限定(专家评定的期刊)–出版日期限定(可选择一个时间段)–出版物名称限定(在特定出版物中进行检索)–出版物类型限定(期刊论文、报纸、图书、其它文献)–页数限定(文章最多不超过多少页)–带图像的文章(所有、PDF格式、文本附图限定检索结果。
可以在如下的选项中进行选择:有全文、有参考文献、专家评定的期刊、出版日期、在特定出版物中检索、出版物类型、文献最多不超过多少页、是否附图高级检索•高级检索可以检索所有的字段,可以使用布尔逻辑运算符确定检索词之间的关系。
检索结果比基本检索更为精确•还可以扩展检索范围和对检索结果做限定•可以保存检索策略、回顾检索历史、组配检索、进行电子通告服务尔逻辑算符确定检索词间的关系,点击执行检索。
EBSCO外文全文数据库检索使用方法
加的检索结果
浙江财经学院图书馆数字资源介绍之二
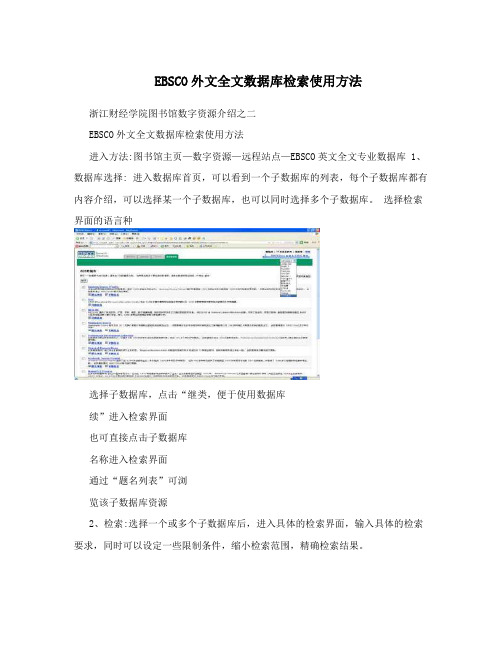
选择子数据库,点击“继类,便于使用数据库
续”进入检索界面
也可直接点击子数据库
名称进入检索界面
通过“题名列表”可浏
览该子数据库资源
2、检索:选择一个或多个子数据库后,进入具体的检索界面,输入具体的检索要求,同时可以设定一些限制条件,缩小检索范围,精确检索结果。
选择检索字段输入检索词 可定一些限制条件,精确检索结果
EBSCO外文全文数据库检索使用方法
浙江财经学院图书馆数字资源介绍之二
EBSCO外文全文数据库检索使用方法
进入方法:图书馆主页—数字资源—远程站点—EBSCO英文全文专业数据库 1、数据库选择: 进入数据库首页,可以看到一个子数据库的列表,每个子数据库都有内容介绍,可以选择某一个子数据库,也可以同时选择多个子数据库。 选择检索界面的语言种
浙江财经学院图书馆数字资源介绍之二
3、检索结果:可以选择需要的文献查看全文,也可以将需要的文献存放在“我的EBSCOhost”文件夹里,以后再次需要时可直接到文件夹中查看,无须再次检索。需要说明的是该数据库不是所有文献都有全文,如在文献列表中没有“PDF全文”字样就表示该文献没有全文,因此建议在检索时设定附加检索条件为“全文”。该数据库文献采用PDF格式,在浏览全文前请先下载安装PDF阅读器。
注册或登陆我的文件夹,
可存放检索到的文献
点击打开已经存入
文件夹的检索结果检索结果按文
献类型分类
检索结果列表,点击“PDF
全文”可查看全文
将该文献添加至
我的文件夹
点击添加到“我
的文件夹
检索结果按主题分类
4、“我的文件夹”管理:先注册一个“我的EBSCO”,将检索结果进行添加,添加后将显示“我的文件夹中有对象”,下次需要利用这些检索结果时,不用重新检索,直接打开“我的EBSCO”或点击“我的文件夹中有内容”即可。
EBSCO数据库的使用方法

EBSCO数据库的使用方法EBSCO商业数据库包括学术期刊数据库(Academic Search Elit,简称ASE)、商业资源数据库(Business Source Premier,简称BSP)等60多种数据库,是EBSCO公司的网络版数据库。
数据库将二次文献与一次文献整合在一起,为最终用户提供文献检索一体化服务。
数据库检索途径包括关键词keywords检索,主题subjects检索,出版物publications检索,公司检索company profiles,图片images检索。
由于数据库内容的差异,各个数据库检索途径不完全相同,如ASE数据库不提供公司检索。
下面结合ASE和BSP两个数据库介绍数据库的检索方法。
1关键词检索1.1简单检索(Basic Search)Find检索框中可以输入若干检索词,对输入检索词的处理方法有四种选项,选中任意一种的单选框,则按照相应方式进行检索,每种选项的含义如表1:表1 EBSCO数据库检索系统对不同选项的检索处理方式除了上述数据检索系统予设的四个选项操作,还可以在检索框中输入“and”、“or”、“not”来连结检索关键词,分别进行逻辑“与”、“或”、“非”的运算。
例:在BSP数据库中的检索实例(表2)表2 不同选项的检索结果比较表假如表中检索式“Computer business”and software改为Computer business and software,则Exact Phrase的检索结果为1条记录,而Standard Search的检索结果不变。
1.2 高级检索(Advanced Search)高级检索的检索关键词输入方法及选项和简单检索的相同,高级检索的优势在于增加了限制和扩展的功能,可以在 Search Option中选择附加条件或扩展条件,以约束检索结果,或扩展检索范围(表3)。
表3 高级检索中的各项功能解释2 主题检索(Subjects)数据库中所有文献按照人物、产品、出版物名、公司名、学科等特征分为若干主题。
EBSCO数据库使用说明

EBSCO数据库使用说明1) Academic Search Premier提供了近 4,700 种出版物全文,其中包括 3,600 多种同行评审期刊。
它为 100 多种期刊提供了可追溯到 1975 年或更早年代的 PDF 回溯资料,并提供了 1000 多个题名的可检索参考文献。
此数据库通过 EBSCOhost 每日进行更新。
2) Business Source Premier该库是行业中使用最多的商业研究数据库,提供 2,300 多种期刊的全文,包括1,100 多种同行评审标题的全文。
此数据库提供的全文可追溯至 1886 年,可搜索引文参考可追溯至 1998 年。
Business Source Premier 相比同等数据库的优势在于它对所有商业学科(包括市场营销、管理、MIS、POM、会计、金融和经济)都进行了全文收录。
此数据库通过 EBSCOhost 每日更新。
3) Communication SourceCommunication Source大众传播学暨语言学、语言沟通教学全文数据库,合并高质量的传播与语言沟通研究全文数据库(Communication & Mass Media Complete,CMMC)以及传播研究文摘数据库( Communications Abstracts, 以往为Sage出版)。
该数据库收录内容包括:超过670本全文期刊(比原CMMC增加161种全文期刊),超过1,000本核心期刊的索引与摘要,含括4,528笔首选词汇及8,528笔非首选词汇的主题分类与同义词辞典,包含了93%的CIOS’sComAbstracts所拥有的过刊(backfile),内容可回溯最早至1915年。
4) EconLitEconLit(美国经济协会电子数据库)是世界上最重要的经济学文献参考来源。
该数据库包含从 1969 年至今的 630,000 多条记录。
EconLit 实际上涵盖了与经济相关的各个范畴,EBSCO平台的EconLit还提供全文链接,有近65%的文献可以直接链接到EBSCO其他全文数据库中的全文。
EBSCO数据库使用指南

EBSCO数据库使用指南一、数据库简介EBSCO数据库由美国EBSCO公司出版, 内容涉及自然科学、社会科学、人文和艺术等多种学术领域。
我馆购买的全文数据库:Academic Search Premier(学术资源数据库ASP)Business Source Premier(商业资源数据库BSP)1.ASP是专门为学术研究机构提供的全文数据库,是全球最大的多学科的数据库,收录有关社科、人文、教育、计算机科学、工程、物理、化学、语言、文学、艺术、医学、种族研究等领域的4700多种全文期刊。
100多种期刊可追溯至1975年或更早年代。
2.BSP是专门为商学院和图书馆提供的全文数据库,收录了有关商业(包括市场营销、管理、MIS、POM、会计、金融和经济)和管理各领域的7400多种全文期刊,是行业中使用最多的商业研究数据库。
收录年限:1990年至今,最早回溯至1975年。
更新频率:数据库每日进行更新。
二、数据库登陆直接登陆EBSCOhost Web(),不须输入用户名和密码。
三、检索方法选择要检索的数据库,单击数据库旁边的小方框,点击“继续”进入检索页面。
1.基本检索界面可以在检索输入框中输入关键词或词组,选择限制结果,点击“检索”。
2.高级检索界面在各检索框中根据需要选择检索字段,输入检索词,使用逻辑运算符(and, or, not)进行逻辑组配,选择限制结果,点击“检索”。
3. 多种检索途径可选择关键字、主题词、出版物、索引、图像、参考文献等检索途径进行检索。
4.检索结果显示每篇文章题目的下方,一般都说明了该篇文章是否有全文和存在形式,即“HTML”格式、“PDF”格式或超级链接,有的标明了国内馆藏的情况。
5.检索结果处理可用“print/E-mail/save”各个功能按钮来处理检索结果。
6.个性化设置My EBSCOhost提供了个性化的使用功能。
7.下载阅读器显示或阅读PDF格式的全文时,要先下载“PDF阅读器”。
ebsco使用方法

ebsco使用方法
EBSCO是一种在线数据库,用于检索和访问数百种出版物,包括期刊、报纸、杂志和学术著作。
以下是EBSCO使用的基本步骤:
1.访问EBSCO网站并登录:在浏览器中输入EBSCO网址,然后使用您的登录信息登录。
2.选择您要搜索的数据库:EBSCO拥有多个数据库,您可以根据您的需要选择一个或多个数据库,然后开始搜索。
3.使用关键词搜索:在EBSCO搜索栏中使用关键词搜索所需的信息。
您可以使用单个词或短语搜索。
还可以使用高级搜索选项进行更精确的搜索。
4.筛选结果:使用EBSCO的筛选工具来缩小搜索结果。
您可以根据日期、主题、作者和出版物类型等过滤器来筛选结果。
5.查看和下载结果:您可以通过单击结果中的链接来查看和下载文章。
您可以选择将结果保存到您的EBSCO文件夹中,以备将来查看和下载。
6.使用其他EBSCO功能:EBSCO还提供其他功能,如保存、导出和共享搜索结果,以及创建警报和RSS订阅等。
以上是EBSCO使用的基本步骤,根据您的需求和EBSCO提供的功能,您可以自由探索和使用。
- 1 -。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
沈阳工程学院图书馆EBSCO西文全文数据库检索指南《EBSCO西文数据库》一、数据库介绍EBSCO公司是世界上最大的提供期刊、文献定购及出版服务的专业公司之一,从1986年开始出版电子出版物,收录范围涵盖自然科学、社会科学、人文和艺术、教育学、医学等各类学科领域。
在校园网范围内,可以查询《Academic Search Premier》(学术期刊数据库)、《Business Source Premier》(商业资源数据库)、ERIC、Newspaper Source 、Professional Development Collection等数据库。
Academic Search Premier(学术期刊数据库):当今全世界最大的多学科学术期刊全文数据库,专为研究机构所设计,提供丰富的学术类全文期刊资源。
Academic Source Premier 提供了近4,700 种出版物全文,其中包括3,600 多种同行评审期刊。
它为100 多种期刊提供了可追溯至1975 年或更早年代的PDF 过期案卷,并提供了1000 多个题名的可检索参考文献。
大多数期刊有PDF格式的全文;很多PDF全文是可检索的PDF(Native PDF)和彩色PDF;1,000多种期刊提供了引文链接。
这个数据库几乎覆盖了所有的学术研究领域,包括:人文社会科学、教育、计算机科学、生物学、工程学、物理、化学等。
此数据库通过EBSCOhost 每日进行更新。
Business Source Premier (商业资源电子文献库) :为商学院和与商业有关的图书馆设计,是行业中使用最多的商业研究型数据库;所收录的各类全文出版物达8800多种;学科领域包括:管理、市场、经济、金融、商业、会计、国际贸易等;全文回溯至1965年或期刊创刊年;可检索的参考文献回溯至1998年。
以所收录的期刊排名统计,Business Source Premier数据库在各个学科领域中都优于其它同类型数据库;如:市场、经营管理、MIS(管理信息系统)、POM(生产与运营管理)、会计学、财政学、经济学等。
这个数据库还提供了许多非刊全文文献,如:市场研究报告、产业报告、国家报告、企业概况、SWOT分析等。
数据库每日更新。
注:要查看全文需安装Adobe Acrobat Reader。
二、进入数据库入口一:图书馆主页——〉数字资源——〉EBSCO西文全文数据库图书馆网址:进入数据库选择页入口二:进入网址选择“BSCOhostWeb”或“BSCOhost Text Only(速度较快)”进入数据库选择页。
进入数据库选择页三、选择数据库选择单一数据库:直接点击该数据库的链接即可。
选择多个数据库:在数据库前打勾,然后点击「继续」按钮即可。
进入检索页面单一数据库检索:直接点击数据库名称数据库详细介绍数据库期刊目录多重数据库复选框在选择数据库时,界面提供两种辅助功能,即:数据库详细介绍和期刊检索。
数据库详细介绍:数据库期刊目录:包含期刊刊名目录,还提供「期刊检索」功能。
读者可按期刊刊名、年份、卷、期检索查询,直接检索到所需的文章。
其使用步骤如下所示:刊名检索输入框字顺目录选择四、检索方法EBSCOhost 提供两大类检索方法:基本检索(Basic Search)和高级检索(Advance Search)。
其中又分别提供“关键词”(Keyword)、“主题”(Subject Term)、“出版物”(Publications)、“索引”(Indexes)、“参考文献”(References)等多种检索途径。
两大类检索方式除关键词检索功能不同外,其它检索功能均相同。
1、基本检索(1)基本检索界面(2)扩展选项:搜索相关关键词选项(Also search for related words ):勾选该项,会将检索词的同义词或单复数的文献一并检索。
如:键入“car ” EBSCOhost 会检索到car 和automobile ;或键入“policy ” EBSCOhost 会检索到policy 和policies 。
在文章全文范围内搜索选项(Also search within the fulltext of the articles ):若检索词较冷僻,可勾选该项,EBSCOhost 会检索每篇全文文章,只要该篇文章的全文中有所键入的检索词,就会被纳入检索结果清单。
自动“and ”检索词语(Include all search terms by default ):勾选该项,检索系统会在每个检索词之间自动加入“and ”逻辑运算符。
2、高级检索(1)高级检索界面可选择多个字段输入不同检索词进行组配检索。
其检索步骤示例如下: ➢ 在“查找”字段中输入欲检索的关键词;➢ 从“位于”右边的下拉式列表中选择检索字段:如:TX All Text – 全文等; ➢ 选择下方逻辑运算符;➢ 在下一个“查找”字段内键入另一个关键词;➢ 再一次从“位于”右边的下拉式列表中选择检索范围,如:SU- 主题等; ➢ 在“选择”左边下拉式列表中选择数据库; ➢ 按下“检索”按钮开始检索。
基本检索关键词检索参考文献检索图像库出版物检索主题检索索引检索输入检索词选择数据库扩展选项(2)限制检索项: 同基本检索相比,高级检索除具备了初级检索的所有功能外,还增加了“Cover Story ”,表示仅检索具有深度报道的封面故事文章。
可选择将检索范围限定于:有全文的文献,有参考的文献,学术性期刊,出版物种类,多个关键词组配选择数据库输入关键词限制检索项扩展选项文献类型,封面报导,出版日期等。
(3)扩展选项:同基本检索。
注:基本检索和高级检索的结果均可按不同主题进行二次检索,以提高检准率。
选择主题进行二次检索关键字检索| 出版物检索| Subject Terms | Indexes | Cited References | Image Collection3、其它检索途径(1)关键词检索:检索界面见基本检索、高级检索。
直接输入检索词并选择检索入口,同时可以对检索结果做限定或是扩展。
(2)出版物检索:同选择数据库时的刊名列表检索。
(3)主题(Subject Terms)检索:当检索者不能够确切选择检索词时,就可利用主题检索。
输入主题,然后浏览相关检索词,进而选择检索主题词。
点击进入主题检索输入检索词输入主题词浏览相关检索词(4)索引(Indexes)检索检索步骤如下所示:步骤1:点击“Indexes”,进入索引检索;步骤2:在下拉菜单中选择查询类型,如:“Author”;步骤3:输入检索词,如:Smith ; 步骤4:点击“浏览”,检索结果将按字母顺序列出;步骤5:点击所需要的关键词前的方框,如:SMITH ALLEN, J.,再点击“添加”按钮,使相应的关键词加入到检索栏中。
重复步骤②-④,加入所有需要的关键词。
步骤6:点击“检索”,获得想要的结果。
(5)参考文献(Cited References )检索EBSCO 的部分数据库提供参考文献检索功能。
检索步骤如下所示: 步骤1:点击“Cited References ” 进入索引检索。
步骤2:在Author 、Title 、Source 、Year 或是All 各检索栏內输入关键词,如:在 “Author ” 检索栏中输入Samuelson, Paul A.,然后点击“检索”按钮。
1.点击进入索引检索3.输入检索词2.在下拉菜单选择查询类型5.选择所需关键词 6.点击“添加”7.最后点击“检索”4.点击“浏览”(6)图像(Images Collection)检索检索步骤如下所示:步骤1:点击“Images Collection”进入图像检索。
步骤2:选择图片的类型,包括:全部、人物、自然科学、风景、历史照片、地图、旗帜等,如:Photos of people。
步骤3:在检索框內输入关键词,如:在输入Mao,(即检索毛泽东的照片)然后点击“检索”按钮。
1.点击进入参考文献检索2.在选定字段输入检索词3.最后点击“检索”1.点击进入图像库2.输入检索词3.选择检索类型4.最后点击“检索”五、检索结果处理 检索结果界面1、 查看文章详细信息点击某一文章题名,显示该文章的详细外部特征。
如下图所示:结果显示检索结果数检索结果排序选择文章题名及详细出处相关引文链接点击查看文章详细信息点击查看PDF 格式全文2、查看文章全文点击或就会显示某一文章全文。
如下图所示:PDF全文(需安装Adobe Acrobat Reader):HTML全文:出处作者题名主题词作者单位摘要存盘和打印查找文本选择显示格式选择翻页3、PDF格式转换为word格式点击图标,用鼠标选定某些PDF格式的全文,单击鼠标右键,复制,然后可以以word格式粘贴到文档中。
4、“我的EBSCOhost”——文件夹功能“我的EBSCOhost”文件夹是EBSCO数据库为读者免费提供的一个私人的存储空间,用于储存在EBSCO数据库中查阅的文献以备再次使用,再次使用时不需要重新进行检索。
只要在有权限可以访问EBSCO数据库的地方均可以使用我的EBSCOhost文件夹。
注:需先注册用户,登录后方可使用此功能。
(1)注册演示:1.点击登录至“我的EBSCOhost”2.选择“我是一个新用户”3.输入个人信息4.设置用户名和密码5.点击“提交”6.点击“确定”,完成注册(2)将检索结果添加至文件夹将检索题录保存至个人文件夹:途径1:选择“添加对象(1-10)”即可,将本页面检索结果全部保存至个人文件夹;途径2:先单击文献题录后面的勾选框,选出所需要的文献,然后单击上方的“更新”按钮,即可将所需检索结果保存至个人文件夹。
途径1途径2将文章详细信息或全文保存至个人文件夹:点击检索界面上方的按钮即可。
将结果保存至文件夹将全文保存至文件夹。
