酶切位点识别序列
takara快切酶酶切位点

takara快切酶酶切位点
Takara快切酶是一种用于分子生物学实验的酶,它能够识别特定的DNA序列并在该序列上进行切割。
酶切位点是指酶在DNA分子上识别并切割的特定序列。
Takara快切酶的切位点取决于具体使用的酶种类,不同的酶有不同的识别序列和切割方式。
从分子生物学角度来看,Takara快切酶的切位点是DNA双链上的特定序列,这些序列通常是4至8个碱基对长,具有特定的碱基配对规律。
酶切位点的选择对于DNA分子的切割和连接至关重要,因为它直接影响着DNA重组、连接和修复的效率和准确性。
在实验操作中,研究人员需要根据所使用的Takara快切酶的特性来选择合适的切位点,以确保实验能够顺利进行。
通常情况下,研究人员会根据酶的说明书或相关文献来确定切位点,然后设计合适的引物或寡核苷酸序列进行实验操作。
除此之外,Takara快切酶的切位点也与基因工程、基因编辑等领域密切相关。
在基因编辑技术中,研究人员经常利用
CRISPR/Cas9等系统来指导Takara快切酶在特定的DNA序列上进行切割,从而实现对基因组的精准编辑。
总的来说,Takara快切酶的切位点是分子生物学领域中非常重要的概念,它涉及到DNA序列的识别和切割,对于基因工程、基因编辑和分子生物学研究具有重要意义。
在实验操作中,科研人员需要根据具体的实验目的和所用酶的特性来选择合适的切位点,并进行相关的实验设计和操作。
限制酶bamhi识别序列

限制酶bamhi识别序列
BamHI是一种限制酶,它识别的DNA序列为5'-G|GATCC-3',其
中竖线表示酶切位点。
这意味着BamHI在G和A之间切割DNA链。
这个识别序列是对称的,也就是说,它在DNA的两条链上都有相同
的序列。
这种对称性使得BamHI能够在DNA双螺旋结构中切割两条链,形成突出的粘性末端。
从生物学角度来看,BamHI限制酶的识别序列在分子生物学和
基因工程领域被广泛应用。
科研人员可以利用BamHI酶切割特定的DNA序列,从而在DNA分子中创建特定的切口,为后续的DNA连接、插入或分析提供方便。
在实验室中,科研人员通常会在特定的实验条件下使用BamHI 酶,以确保其能够准确地识别和切割DNA序列。
此外,他们还会根
据实验需要选择合适的缓冲液、温度和反应时间等因素,以最大限
度地发挥BamHI酶的作用。
另外,从应用角度来看,了解BamHI限制酶的识别序列有助于
科研人员在实验设计和数据分析中更好地利用这一工具。
他们可以
根据BamHI的识别序列来设计引物或探针,进行特定基因的扩增或
检测。
同时,对BamHI的识别序列有深入的了解,也有助于科研人
员在实验过程中更好地控制酶切反应的准确性和效率。
综上所述,了解BamHI限制酶的识别序列对于分子生物学和基
因工程领域的研究具有重要意义,它不仅为科研人员提供了一种重
要的DNA操作工具,同时也为实验设计和数据分析提供了有力支持。
酶切位点汇总

酶切位点汇总
酶切位点,又称为限制性内切酶位点,是指DNA分子上特定的序列,这些序列是限制
性内切酶可以识别和切割的地方。
限制性内切酶是一种在细菌和其它生物中广泛存在的酶,能够切割或切除一个或多个DNA碱基对。
这些限制性内切酶在生物技术领域广泛应用,用
于DNA序列分析、DNA重组、基因工程等方面。
以下是常见的几种酶切位点:
1. EcoRI切割位点是5′-GAATTC-3′,这是一种广泛应用的限制性内切酶,通常用于DNA纯化、制备DNA载体等。
2. BamHI切割位点是5′-GGATCC-3′,BamHI能够切割链间,产生具有黏性末端的DNA 序列。
常被用于制备双链DNA的黏性末端。
4. PstI切割位点是5′-CTGCAG-3′,PstI是一种双切酶,可以切割成不同长度的DNA 序列,适用于构建多种不同长度的DNA分子。
总之,酶切位点及其对应的限制性内切酶在现代生物领域有着广泛的应用和重要的作用。
了解不同的酶切位点是有很大帮助的,它可以为实验设计和分子生物学研究提供基础。
同时,也让我们更好地理解限制性内切酶在DNA分子上的作用,帮助我们在生物技术领域
更加熟练地掌握其应用。
tev蛋白酶切位点的dna序列

tev蛋白酶切位点的dna序列
Tev蛋白酶切位点的DNA序列是指Tev蛋白酶可以识别并切割的DNA序列,也被称为Tev切割位点。
Tev蛋白酶是一种常用的内切酶,具有高度特异性和高效性。
Tev蛋白酶的切割位点为5'-G|TATAC-3',其中“|”所示的位置为切割位点。
因此,Tev蛋白酶切割位点的DNA序列为5'-GTTAAC-3'。
Tev蛋白酶的切割位点的序列具有较高的特异性和保守性,因此在进行基因克隆和重组DNA技术时,Tev蛋白酶切割位点的选择非常重要。
在进行DNA片段连接时,通过在DNA的末端引入Tev蛋白酶切割位点,可以使用Tev蛋白酶将DNA 片段剪切开,并通过该切口将不同的DNA片段连接起来。
需要注意的是,Tev蛋白酶切割位点的DNA序列只是其中一种常用的切割位点序列,不同种类的内切酶所识别的切割位点序列也是不同的。
因此,在进行基因克隆和重组DNA技术时,需要选择适合特定目的的内切酶切割位点序列。
基因工程名词解释

名词解释【基因工程】:在体外对不同生物的遗传物质(基因)进行剪切、重组、连接,然后插入到载体分子中(细菌质粒、病毒或噬菌体DNA),转入微生物,植物或动物细胞内进行无性繁殖,并表达出基因产物。
【限制性核酸内切酶】:是一类能够识别双链DNA分子中的某种特定核苷酸序列(4-8bp),并由此处切割DNA双链结构的核酸内切酶。
【识别序列】:限制性核酸内切酶在双链DNA上能够识别的特殊核苷酸序列被称为识别序列。
【酶切位点】:DNA在限制性核酸内切酶的作用下,使多聚核苷酸链上磷酸二酯键点开的位置被称为切割位点。
【粘性末端】:是指含有几个核苷酸单链的末端,可通过这种末端的碱基互补,使不同的 DNA片段发生退火。
【平末端】:限制酶在它识别序列的中心轴线处切开时产生的平齐的末端。
【同裂酶】:一些来源不同的但能识别位点的序列相同的限制性内切酶。
【同尾酶】:一些来源不同且识别序列不同,但能产生相同粘性末端的限制性内切酶。
【DNA的甲基化程度】:DNA被甲基化酶甲基化,识别序列中的核苷酸一旦被甲基化,就会影响内切酶的切割效率。
【位点偏爱】:对不同位置的同一个识别序列表现出不同的切割效率的现象【内切酶的star活性】:某种限制性核酸内切酶在特定条件下,可在不是原来的识别序列处切割DNA,这种现象称为star活性。
【末端转移酶】:一种能将脱氧核苷酸三磷酸(dNTP)加到某DNA片段上3’-OH基上的酶。
【DNA连接酶】:借助ATP或NAD水解提供的能量催化DNA双链,DNA片段紧靠在一起的3’-OH末端与5’-PO4末端之间形成磷酸二酯键,使两末端连接【DNA聚合酶】:以DNA为复制模板,使DNA由5'端点开始复制到3'端的酶。
【反转录酶】:与DNA聚合酶作用方式相似:5’→3’聚合,模版是mRNA,合成DNA【碱性磷酸酶】:能够催化核酸分子脱掉5’磷酸基团,从而使DNA(或RNA)片段的5’-P 末端转换成5’-OH末端。
酶切位点识别序列
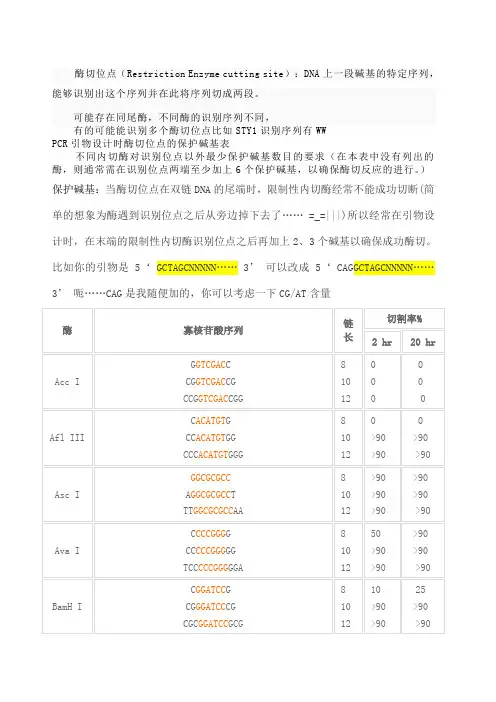
酶切位点(Restriction Enzyme cutting site):DNA上一段碱基的特定序列,能够识别出这个序列并在此将序列切成两段。
可能存在同尾酶,不同酶的识别序列不同,
有的可能能识别多个酶切位点比如STY1识别序列有WW
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
)
保护碱基:当酶切位点在双链DNA的尾端时,限制性内切酶经常不能成功切断(简单的想象为酶遇到识别位点之后从旁边掉下去了…… =_=|||)所以经常在引物设计时,在末端的限制性内切酶识别位点之后再加上2、3个碱基以确保成功酶切。
比如你的引物是5‘ GCTAGCNNNNN……3’ 可以改成5‘ CAGGCTAGCNNNNN……3’ 呃……CAG是我随便加的,你可以考虑一下CG/AT含量
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相应的
碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。
酶切位点识别序列
酶切位点识别序列 Document serial number【KKGB-LBS98YT-BS8CB-BSUT-BST108】
酶切位点(Restriction Enzyme cutting site):DNA上一段碱基的特定序列,能够识别出这个序列并在此将序列切成两段。
可能存在同尾酶,不同酶的识别序列不同,
有的可能能识别多个酶切位点比如STY1识别序列有WW
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相应的
碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。
酶切位点的特点
酶切位点的特点
酶切位点是指在DNA或RNA分子中,特定酶能够切割的位置。
以下是酶切位点的一些特点:
1. 序列特异性:每种酶都有特定的序列要求,只能识别并切割特定的核酸序列。
这些序列通常由4种碱基(腺嘌呤、胸腺嘧啶、鸟嘌呤和胞嘧啶)组成,例如常见的EcoRI酶切位点是GAATTC。
2. 对称性:大多数酶切位点是对称的,即从5'到3'方向的顺序与互补链上的同一序列相同。
例如EcoRI酶切位点的互补链也是GAATTC。
3. 切割位置:酶通常在特定的位置切割DNA或RNA分子。
切割位点可以是在切割位点序列内的特定碱基之间,也可以是切割位点序列的边缘。
4. 切割方式:不同的酶可以以不同的方式切割DNA或RNA。
一些酶会切割两条链上的碱基对称地,形成平滑的切割端;而其他酶则会产生不对称的切割,形成突起或粘性末端。
5. 应用广泛:酶切位点在分子生物学和基因工程领域应用广泛。
通过识别和利用酶切位点,可以进行DNA片段的精确切割、连接和重
组,用于构建重组DNA分子、进行基因克隆、检测基因突变等。
总之,酶切位点是特定酶能够识别和切割的DNA或RNA分子中的特定序列,具有序列特异性、对称性、切割位置和切割方式等特点。
这些特点使得酶切位点在分子生物学和基因工程中发挥着重要的作用。
酶切位点平末端
酶切位点平末端
酶切位点平末端指的是一种特定的DNA或RNA序列结构,其中两个不同的序列在同一个平面上相互平行且相对。
这种结构通常出现在DNA或RNA的限制性内切核酸酶的切割位点处,这些酶能够识别特定的序列并切割DNA或RNA分子。
在DNA或RNA分子中,酶切位点平末端的具体示例包括:
1.EcoRI酶切位点:EcoRI是一种限制性内切核酸酶,能够识别并切割DNA
分子中的GAATTC序列。
切割后产生的片段具有平末端。
2.BamHI酶切位点:BamHI是一种限制性内切核酸酶,能够识别并切割
DNA分子中的GGATCC序列。
切割后产生的片段同样具有平末端。
在生物实验中,酶切位点平末端的应用非常广泛。
例如,在构建基因克隆载体时,需要将目的基因与载体进行连接,通常需要在目的基因和载体之间进行限制性内切核酸酶的切割。
当使用平末端的限制性内切核酸酶进行切割时,可以获得两个平末端的DNA片段,通过连接这些片段可以实现目的基因与载体的连接。
总结来说,酶切位点平末端指的是一种特定的DNA或RNA序列结构,其中两个不同的序列在同一个平面上相互平行且相对。
这种结构通常出现在限制性内切核酸酶的切割位点处,这些酶能够识别特定的序列并切割DNA或RNA 分子。
在生物实验中,酶切位点平末端的应用非常广泛,例如在构建基因克隆载体时需要进行限制性内切核酸酶的切割和连接。
酶切位点识别序列
酶切位点识别序列 Last revised by LE LE in 2021
酶切位点(Restriction Enzyme cutting site):DNA上一段碱基的特定序列,能够识别出这个序列并在此将序列切成两段。
可能存在同尾酶,不同酶的识别序列不同,
有的可能能识别多个酶切位点比如STY1识别序列有WW
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相应的
碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
上一段碱基的特定序列,DNAcutting site):Restriction 酶切位点(Enzyme 序列切成两段。
限制性内切酶能够识别出这个序列并在此将DNA可能存在同尾酶,不同酶的识别序列不同,
WW 有的可能能识别多个酶切位点比如STY1识别序列有
PCR引物设计时酶切位点的保护碱基表不同内切酶对识别位点以外最少保护碱
基数目的要求(在本表中没有列出的)酶,则通常需在识别位点两端至少加上
6个保护碱基,以确保酶切反应的进行。
简DNA的尾端时,限制性内切酶经常不能成功切断(保护碱基:当酶切位点在双链所以经常在引物设计=_=|||)单的想象为酶遇到识别位点之后从旁边掉下去了……
个碱基以确保成功酶切。
比32时,在末端的限制性内切酶识别位点之后
再加上、改3'
成可以的引物是5‘
GCTAGCNNNNN……如你5‘CAGGCTAGCNNNNN……3' 呃……CAG是我随便加的,你可以考虑一下CG/AT含量
注释
1.如果要加在序列的5'端,就在酶切位点识别碱基序列(红色)的5'端加上相应的碱基(黑色),如果要在序列的3'端加上保护碱基,就在酶切位点识别碱基序列(红色)的3'端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
加保护碱基时最好选用切割率高时加的相应碱基。
3.。
