融合基因的转录检测分析
BCR-ABL融合基因检测——CML诊断“黄金标准”

BCR/ABL融合基因检测——CML诊断“黄金标准”1 1 11对照阴性杂交结果图(2R2G)BCR/ABL(DF)典型阳性图(1R1G2F)BCR/ABL(SF)典型阳性图(1R1G1F)BCR/ABL(ES)典型阳性图(2R1G1F)慢性粒细胞白血病(chronic myelogenous leukemia, CML)是一种起源于造血干细胞的克隆性骨髓增殖性肿瘤,主要累及粒细胞系,表现为持续、进行性外周血白细胞数量增加,分类中出现不同分化阶段的粒细胞,尤其以中性粒细胞增多为主,90%以上患者白血病细胞中有恒定的、特征性的Ph染色体及其分子标志BCR/ABL融合基因。
本病发病率较高,可见于各年龄组,以20~50岁多见,诊断时中位年龄为45~50岁。
CML起病缓慢,初期症状不明显,逐渐出现乏力、低热、盗汗、食欲减退及消瘦等。
其自然病程是由慢性期进展为加速期,最后发展为急变期,急变后患者常在3~5个月内死亡。
CML患者中易位类型绝大多数为t(9;22)(q34;q11)的典型易位,少数有变异易位,包括22号与非9号染色体间的简单变异易位,3条或更多条染色体间的复杂易位(隐匿易位)。
易位形成的Ph染色体是由位于9q34的ABL原癌基因断裂并易位到22q11的断裂点簇集区BCR形成,并在断点处形成BCR/ABL融合基因。
该基因可转录出一个8.5kb的异常mRNA,最终翻译成210KD蛋白质(P210)。
P210具有较强的酪氨酸蛋白激酶活性,可通过多种信号传导途径来活化癌基因和某些细胞因子,最终导致细胞的恶性转化。
典型易位(NCCN Guidelines for patients TM Version 1.2011)BCR/ABL融合基因检测意义:1.CML辅助诊断90~95%CML患者可检测出典型的t(9;22)(q34;q11)易位,约5%的CML患者通过核型分析难以检测到Ph染色体,但可检测出融合基因存在,仍被归为Ph+ CML分类。
鸡血细胞融合实验报告

鸡血细胞融合实验报告鸡血细胞融合实验报告引言细胞融合是一种重要的实验技术,通过将两种或多种细胞融合在一起,可以研究细胞间的相互作用、基因表达调控以及细胞发育等方面的问题。
在本次实验中,我们选择了鸡血细胞作为研究对象,通过融合不同类型的鸡血细胞,探究其对细胞功能和特性的影响。
实验材料与方法1. 实验材料:- 鸡血细胞样本:从鸡体内提取鲜血样本,分离出鸡血细胞。
- 细胞培养基:含有适宜的营养物质和生长因子,维持细胞生长和分裂。
- 细胞培养器具:培养皿、离心管、移液器等。
2. 实验方法:- 细胞培养与扩增:将鸡血细胞接种于含有细胞培养基的培养皿中,放置于恒温培养箱中,控制适宜的温度和湿度,使细胞能够正常生长和分裂。
- 细胞融合:选取两种不同类型的鸡血细胞,分别标记为A和B。
将细胞A和细胞B分别收集,离心沉淀后,用细胞培养基将其悬浮于一起。
利用电融合或化学融合等方法,使细胞A和细胞B融合为一个细胞群体。
- 细胞观察与分析:观察融合后细胞的形态变化、生长速率、基因表达等特性,并与原始细胞进行对比分析。
实验结果与讨论通过实验观察和数据分析,我们得到了以下结果和结论:1. 细胞融合后形态变化融合前的细胞A和细胞B在形态上存在明显的差异。
然而,经过细胞融合后,新形成的细胞群体呈现出一种中间状态,既保留了细胞A和细胞B的一些特征,又具有自身的特点。
这表明细胞融合可以导致细胞形态的重塑和变异。
2. 细胞融合后生长速率我们观察到,在细胞融合后的细胞群体中,生长速率明显高于单独培养的细胞A和细胞B。
这可能是由于融合后细胞的互补性增强,使得细胞能够更有效地利用培养基中的营养物质和生长因子。
3. 细胞融合后基因表达通过实时荧光定量PCR等技术,我们检测了融合后细胞中一些关键基因的表达水平。
结果显示,融合后细胞的基因表达模式发生了明显的变化,一些基因的表达水平显著上调或下调。
这表明细胞融合可以影响基因的转录水平,从而影响细胞的功能和特性。
临床常用基因突变及融合基因检查方法

临床常用基因突变及融合基因检查方法全文共四篇示例,供读者参考第一篇示例:临床常用基因突变及融合基因检查方法随着基因组学和分子生物学的发展,基因检测技术在临床诊断中的应用日益广泛。
基因突变和融合基因是许多疾病的发生和发展的关键因素,因此对其检测越来越受到重视。
本文将介绍一些临床常用的基因突变及融合基因检查方法,希望能够帮助读者更深入了解这一领域的知识。
1. PCR技术PCR技术是一种常用的基因检测方法,通过扩增特定基因片段,可以检测到基因的突变。
PCR技术具有高灵敏度和高特异性的优点,可以快速、准确地检测基因突变。
在临床诊断中,PCR技术经常用于检测与疾病相关的基因突变,如遗传性疾病、肿瘤等。
2. 高通量测序技术高通量测序技术是目前最流行的基因检测技术之一,可以同时测序大量基因,并快速高效地识别突变。
高通量测序技术具有高灵敏度、高准确性和高通量的优点,可以帮助医生更准确地诊断疾病,并指导个体化治疗方案的制定。
3. 质谱技术质谱技术是一种基于质量-电荷比的物质分析技术,可以用于检测DNA、RNA等生物分子。
质谱技术具有高灵敏度、高分辨率和高准确性的优点,可以用于检测基因突变、基因表达水平等,对于肿瘤的早期诊断和治疗、药物敏感性的评估等方面具有重要的意义。
1. FISH技术FISH技术是一种常用的融合基因检测方法,通过用荧光染料标记特定基因,可以直接观察到基因的融合情况。
FISH技术具有高分辨率、高灵敏度和高特异性的优点,可以帮助医生准确诊断某些特定的融合基因相关的疾病,如慢性髓性白血病、胃肠道间质瘤等。
在临床实践中,基因突变和融合基因的检测对于疾病诊断、治疗和预后评估具有重要的意义。
随着基因检测技术的不断发展和完善,相信在未来会有更多更高效、更精准的检测方法出现,为临床诊断和治疗带来更多的帮助和改善。
希望本文能为读者对基因突变及融合基因检查方法有更深入的了解和认识。
第二篇示例:临床常用基因突变及融合基因检查方法基因突变是指基因组中的某个基因发生了突变,使其序列发生了改变,导致基因的功能发生变化或失去功能。
转录组测序数据分析流程
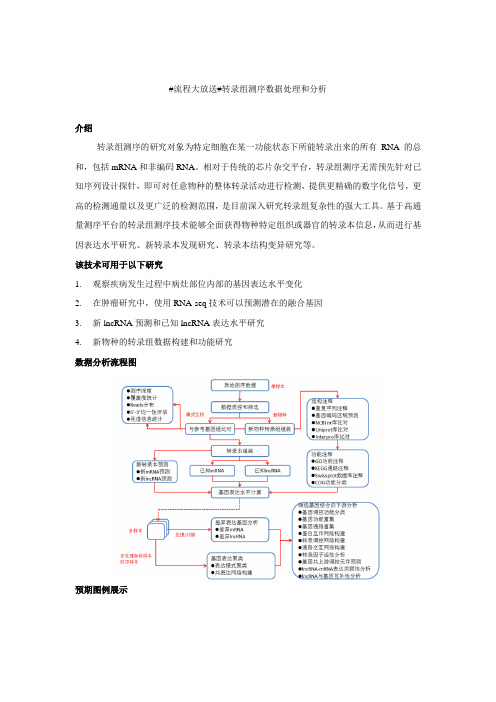
#流程大放送#转录组测序数据处理和分析
介绍
转录组测序的研究对象为特定细胞在某一功能状态下所能转录出来的所有RNA的总和,包括mRNA和非编码RNA。
相对于传统的芯片杂交平台,转录组测序无需预先针对已知序列设计探针,即可对任意物种的整体转录活动进行检测,提供更精确的数字化信号,更高的检测通量以及更广泛的检测范围,是目前深入研究转录组复杂性的强大工具。
基于高通量测序平台的转录组测序技术能够全面获得物种特定组织或器官的转录本信息,从而进行基因表达水平研究、新转录本发现研究、转录本结构变异研究等。
该技术可用于以下研究
1.观察疾病发生过程中病灶部位内部的基因表达水平变化
2.在肿瘤研究中,使用RNA-seq技术可以预测潜在的融合基因
3.新lncRNA预测和已知lncRNA表达水平研究
4.新物种的转录组数据构建和功能研究
数据分析流程图
预期图例展示
示例图1 差异表达基因筛选示例2 基因聚类分析heatmap图
示例3 差异基因互作网络图示例4 lncRNA、基因与上游共有miRNA网络图。
ret融合基因

RET融合基因1. 简介RET融合基因是一种在人类癌症中常见的突变形式。
它是由于cancer cells中的RET基因与其他基因(通常是一个partner gene)发生重排而产生的。
这种重排会导致RET基因产生不正常的融合蛋白,进而促进肿瘤的发展和进展。
2. RET基因和其功能RET(rearranged during transfection)基因是人类基因组中一个重要的酪氨酸激酶受体编码基因。
它位于第10号染色体上,包含21个外显子。
RET受体在胚胎发育过程中起着关键作用,参与多个信号转导通路,如细胞增殖、分化和存活等。
3. RET融合基因的形成机制RET融合基因的形成主要通过染色体重排事件来实现。
这种重排通常涉及到一个伴侣(partner)基因,将其与RET基因连接在一起,形成一个新的复合蛋白。
具体来说,RET与伴侣基因之间发生染色体断裂,并且两个断裂点之间发生互换。
这种互换可能是由于DNA损伤修复机制的错误引起的。
当RET和伴侣基因发生重排后,产生的融合基因会编码一个异常的融合蛋白,具有激活酪氨酸激酶活性的特点。
4. RET融合基因与癌症RET融合基因在多种肿瘤中都被发现。
最早发现的是在甲状腺乳头状癌(papillary thyroid carcinoma)中,大约有10-20%的病例中存在RET/PTC (Papillary Thyroid Carcinoma)融合基因。
此外,RET融合基因也与肺癌、结直肠癌、儿童神经母细胞瘤等多种癌症相关。
这些融合基因能够通过激活RET信号通路来促进肿瘤细胞的增殖、存活和侵袭能力。
此外,它们还可能改变细胞分化状态和抑制细胞凋亡等。
5. RET融合基因检测对于患者而言,检测其体内是否存在RET融合基因具有重要意义。
这可以帮助医生确定治疗策略,并预测疾病的预后。
目前,常用的RET融合基因检测方法主要有荧光原位杂交(FISH)和逆转录聚合酶链反应(RT-PCR)。
检测基因融合的方法

检测基因融合的方法
基因融合检测的方法有多种,目前常见的包括荧光原位杂交(FISH)、逆转录聚合酶链式反应(RT-PCR)、免疫组织化学(IHC)、二代测序技术(NGS)等。
荧光原位杂交技术(FISH)利用荧光标记的探针,在细胞内杂交特定基因或染色体区域,通过荧光显微镜观察目标基因或染色体的变化。
该方法可用于检测染色体易位、基因重排等。
逆转录聚合酶链式反应(RT-PCR)是一种将RNA逆转录为cDNA,然后
进行PCR扩增的方法。
该方法可用于检测基因表达和基因突变,例如检测
融合基因的表达。
免疫组织化学技术(IHC)利用抗体与特定抗原的结合反应,在组织或细胞
中检测蛋白质的表达。
通过使用针对融合蛋白的特异性抗体,可以检测融合基因的表达。
二代测序技术(NGS)是一种高通量测序技术,可以检测基因组的所有序列。
通过NGS技术,可以检测基因融合、基因突变和其他基因组异常。
NGS技术具有高灵敏度和高特异性,能够检测低丰度的融合基因。
除了以上几种方法,还有其他一些检测基因融合的技术,例如单细胞测序、比特流式细胞、CRISPR-Cas9和遗传图谱等。
这些技术各有特点,可以根据具体的研究目的和实验条件选择合适的方法。
白血病融合基因检测综述

白血病相关融合基因的检测及意义白血病是造血系统的恶性克隆性疾病,由于造血干细胞受损,导致克隆中的白血病细胞失去进一步分化成熟的能力而停滞在细胞发育的不同阶段。
白血病细胞具有自我更新增强、增殖失控、分化障碍、凋亡受阻等特点,患者会出现不同程度的贫血、出血、感染和浸润的临床症状,严重危害生命健康。
近年来,随着细胞生物学和分子生物学技术的发展,人们已经认识到大部分的白血病中都存在着包括缺失、重复、易位等染色体畸变,导致原癌基因或抑癌基因结构变异,原癌基因激活或抑癌基因失活,产生新的融合基因,编码融合蛋白。
现有报道的染色体畸变已有五十种以上,累及更多数目的融合基因,这些异常已经逐渐成为不同类型白血病的分子生物学特异性标志。
白血病相关融合基因的种类多样,常见的融合基因有BCR-ABL、AML1-ETO、PML-RARα、E2A-PBX1、MLL-AF4、TEL-AML1、SIL-TAL1、DEK-CAN、CBFβ-MYH11等。
BCR(breakpoint cluster region)基因是BCR-ABL融合基因的组成部分,与费城染色体(Philadelphia Chromosome)的形成有关,具有两种转录异构体。
正常的BCR基因编码产物的功能还尚未清楚,它编码的蛋白具有丝氨酸/苏氨酸激酶活性,是RAC1和CDC42的GTP酶激活蛋白。
ABL1基因是编码细胞质和细胞核蛋白酪氨酸激酶的原癌基因,与细胞分化、细胞分裂、细胞粘附、应激反应等生命活动相关。
活化的ABL1蛋白通过SH3结构域受到负调控,SH3结构域的缺失会导致ABL1基因转化为癌基因。
CDC2介导的磷酸化能够调节ABL1酪氨酸激酶的DNA结合活化过程,表明ABL1可能在细胞周期中发挥作用。
Nowell及Hungerford于1960年发现在慢性粒细胞性白血病(CML)患者外周血中有一个比G组染色体还小的近端着丝粒染色体,由于首先在美国费城(Philadelphia)发现,故命名为费城染色体。
多个基因的共同转录因子检测方法

多个基因的共同转录因子检测方法随着生物技术的不断发展,人们对基因调控的研究日益深入,共同转录因子在基因调控中扮演着至关重要的角色。
共同转录因子是一类能够同时调控多个基因转录的蛋白质,它们通过结合到多个基因的启动子区域,协同调控这些基因的表达。
发展一种可靠、高效的方法来检测多个基因的共同转录因子对于揭示基因调控网络的机制具有重要意义。
本文将介绍一些常用的多个基因的共同转录因子检测方法,并探讨它们的优缺点。
一、ChIP-seq技术ChIP-seq(Chromatin Immunoprecipitation Sequencing)技术是目前最为常用的转录因子结合位点检测方法之一。
该方法通过将特定的抗体与转录因子结合后,利用染色质免疫沉淀技术富集转录因子结合的染色质片段,再结合高通量测序技术对富集的染色质片段进行测序,从而获得转录因子结合的基因组位置信息。
在多个基因的共同转录因子检测中,研究人员可以利用ChIP-seq技术分析多个基因启动子区域上的转录因子结合情况,进而筛选出共同调控这些基因的转录因子。
ChIP-seq技术还可以通过比较不同条件下的样品来鉴定共同转录因子的动态结合情况,进一步揭示基因调控的机制。
ChIP-seq技术也存在部分缺点,如对实验条件的要求较高、数据分析复杂等。
二、RNA-seq技术除了ChIP-seq技术外,RNA-seq技术也可以用于检测多个基因的共同转录因子。
RNA-seq技术是一种利用高通量测序技术对RNA进行定量和质量分析的方法,可以全面、准确地检测基因的表达情况。
研究人员可以利用RNA-seq技术分析在不同条件下多个基因的表达模式,通过寻找共同上调或下调的基因来筛选可能存在的共同转录因子。
RNA-seq 技术还可以通过分析基因的剪接异构体来揭示共同转录因子对于基因的剪接调控作用。
RNA-seq技术在检测转录因子结合位点以及动态结合情况方面相对ChIP-seq技术来说存在局限性。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
這是較接 近的敘述
融合基因的轉錄檢測分析 我們進行GUS分析以揭開啟動子是否具轉錄的活性並測量不同菌種
中具轉錄的活性之啟動子活性。 為了GUS分析,將推測的啟動子由… 基因黏接至pAL280表現載體
上。
區別分析與概念
融合基因的轉錄檢測分析 我們進行G轉U錄S的分融析合以分揭析開,啟其動中子將是推否測具的轉啟錄動的子活與性β並-g測lu量cu不ron同id菌ase種
中報具導轉基錄因的(g活usA性)的之開啟放動讀子架活融性合。。以此方式測量GUS活性以揭開啟動子是 否具轉錄為的了活G性U並S分測析量,它將於推不測同的菌啟種動中子之由活…性基。因黏接至pAL280表現載體 上。 為了GUS分析,將推測的啟動子由… 基因黏接至pAL280表現載體 上。
不ee好k!!
為得到一個gentamicin抗藥株的轉位體,我們自pMSR1質體分離出這個轉位 子。
避免使用實驗室黑話
為了確認轉位體是否隨意插入基因體中,所有經質體救援之菌種均經定 序…
避免使用實驗室黑話
為了確認轉位體是否隨意插入基因體中,所有經質體救援之菌種均經定 序…
咦?
避免使用實驗室黑話
上。
區別分析與概念
GUS分析是工具 非概念
分析…啟動子活性 為測量野生與突變菌種的AN12其…啟動子活性,我們進行GUS分析。
GUS = enzyme gusA = gene
這是較接
近的敘述
但它漏掉正文 中的一點
融合基因的轉錄檢測分析
我們進行GUS分析以揭開啟動子是否具轉錄的活性並測量不同菌種
中具轉錄的活性之啟動子活性。
為了確認轉位體是否隨意插入基因體中,所有經質體救援之菌種均經定 序…
咦?
為了確認轉位體是否已隨意插入基因體中,我們檢查轉位子所插入之基因 體序列。為進行此項工作,我們首先以質體救援的流程 (參閱材料與方法一 節)分離出每個轉位子及一部份與其相鄰的基因體DNA。定序以此法分離出 的DNA顯示…
避免使用實驗室黑話
這些質體也以前置引子定序轉位體,上游區域經BLAST與已知序列比較其 相似性。
什麼的上 游?
咦?
這些質體也以前置引子定序轉位體,於轉位子終端外的序列以BLAST分析 法 (Altschul et al., 1990) 檢測以確認是否與GenBank中序列相似。
留意不清楚的解釋
…這質體藉著與nimB 及 ORF5486基因的相似性重組而插入基因體中。一個 可能剔除質體的純種培養實驗顯示插入後再丟失質體的機率是很低的。
區別分析與概念
分析…啟動子活性 為測量野生與突變菌種的AN12其…啟動子活性,我們進行GUS分析。
融合基因的轉錄檢測分析 我們進行GUS分析以揭開啟動子是否具轉錄的活性並測量不同菌種
中具轉錄的活性之啟動子活性。 為了GUS分析,將推測的啟動子由… 基因黏接至pAL280表現載體
上。
區別分析與概念
GUS分析是工具 非概念
分析…啟動子活性 為測量野生與突變菌種的AN12其…啟動子活性,我們進行GUS分析。
融合基因的轉錄檢測分析 我們進行GUS分析以揭開啟動子是否具轉錄的活性並測量不同菌種
中具轉錄的活性之啟動子活性。 為了GUS分析,將推測的啟動子由… 基因黏接至pAL280表現載體
上。
區別分析與概念
GUS分析是工具 非概念
為了GUS分析,將推測的啟動子由… 基因黏接至pAL280表現載體
上。
區分分析與概念
分析…啟動子活性 為測量野生與突變菌種的AN12其…啟動子活性,我們進行G融U合S基分因析的。轉錄 分析-將…基因的啟動子與β-glucuronidase 報導基因 (gusA)的轉譯序列融合。
融合基因的轉錄檢測分析 我們進行GUS分析以揭開啟動子是否具轉錄的活性並測量不同菌種
勿過度使用個人的代名詞
為得到一個gentamicin抗藥株的轉位體,我們自pMSR1質體分離出我們的轉 位子。
勿過度使用個人的代名詞
為得到一個gentamicin抗藥株的轉位體,我們自pMSR1質體分離出我們的轉 位子。
不ee好k!!
勿過度使用個人的代名詞
為得到一個gentamicin抗藥株的轉位體,我們自pMSR1質體分離出我們的轉 位子。
留意不清楚的解釋
…這質體藉著與nimB 及 ORF5486基因的相似性重組而插入基因體中。一個 可能剔除質體的純種培養實驗顯示插入後再丟失質體的機率是很低的。
於材料與方法一節清楚解釋 「純種培養實驗」或在此含括詳細資料。
留意不清楚的解釋
…這質體藉著與nimB 及 ORF5486基因的相似性重組而插入基因體中。一個 可能剔除質體的純種培養實驗顯示插入後再丟失質體的機率是很低的。
這些質體也以前置引子定序轉位體,上游區域經BLAST與已知序列比較其 相似性。
避免使用實驗室黑話
這些質體也以前置引子定序轉位體,上游區域經BLAST與已知序列比較其 相似性。
咦?
避免使用實驗室黑話
這些質體也以前置引子定序轉位體,上游區域經BLAST與已知序列比較其 相似性。
什麼的上 游?
咦?
避免使用實驗室黑話
中具轉錄的活性之啟動子活性。 為了GUS分析,將推測的啟動子由… 基因黏接至pAL280表現載體
上。
區分分析與概念
分析…啟動子活性 為測量野生與突變菌種的AN12其…啟動子活性,我們進行G融U合S基分因析的。轉錄 分析-將…基因的啟動子與β-glucuronidase 報導基因 (gusA)的轉譯序列融合。
留意不清楚的解釋
…這質體藉著與nimB 及 ORF5486基因的相似性重組而插入基因體中。一個 可能剔除質體的純種培養實驗顯示插入後再丟失質體的機率是很低的。
我應該知道這 是什麼意思嗎?
留意不清楚的解釋
…這質體藉著與nimB 及 ORF5486基因的相似性重組而插入基因體中。一個 可能剔除質體的純種培養實驗顯示插入後再丟失質體的機率是很低的。
GUS分析是工具 非概念
分析…啟動子活性 為測量野生與突變菌種的AN12其…啟動子活性,我們進行GUS分析。
這是較接
近的敘述
但它漏掉正文 中的一點
融合基因的轉錄檢測分析来自我們進行GUS分析以揭開啟動子是否具轉錄的活性並測量不同菌種
中具轉錄的活性之啟動子活性。
為了GUS分析,將推測的啟動子由… 基因黏接至pAL280表現載體
