ReadMe
readme的todolist

对于Readme中的Todo List,可以按照以下步骤进行编写:
1. 确定Todo List的目的和用途:在编写Todo List之前,需要明确其目的和用途。
是为了记录项目中的待办事项、任务还是为了展示未来可能的功能特性?
2. 确定受众:明确Todo List的受众是谁,例如是开发者、用户还是其他利益相关者。
根据受众的不同,编写的内容和风格也会有所不同。
3. 制定任务列表:根据项目需求和目标,列出所有需要完成的任务或待办事项。
确保每个任务都是具体、可衡量和可完成的。
4. 优先级排序:根据任务的紧急程度和重要性,对任务进行优先级排序,以便确定任务的执行顺序。
5. 编写说明:对于每个任务,编写简要的说明,包括任务的描述、目标、预期结果和完成情况等信息。
确保说明清晰、简洁且易于理解。
6. 使用适当的标记语言:为了使Todo List更加清晰和易于阅读,可以使用适当的标记语言来突出显示任务的状态、优先级等信息。
例如,使用Markdown的加粗、斜体或列表等语法。
7. 更新和同步:在项目开发过程中,及时更新Todo List以反映任务的完成情况和进度。
同时,确保与团队成员或其他利益相关者保持同步,以便他们了解项目的最新进展。
8. 放置在适当的位置:将Todo List放置在项目文档中适当的位置,以便相关人员能够轻松地访问和查看。
可以考虑将其放在Readme文件的开头或结尾,或者在专门的文档章节中列出。
通过以上步骤,你可以编写一个清晰、有用和易于理解的Todo List,为项目的管理和协作提供便利。
githubapi获取readme

githubapi获取readme使用GitHub API获取README文件是一个简单且常用的操作。
在以下步骤中,我们将使用Python编程语言来实现这个任务。
首先,我们需要先安装必需的库。
我们可以使用`pip`来安装`requests`库,它可以帮助我们发送HTTP请求并获取响应。
```pythonpip install requests```接下来,我们需要导入`requests`库并定义一个函数来获取README文件的内容。
```pythonimport requestsdef get_readme_content(owner, repo):url = f"/repos/{owner}/{repo}/readme"response = requests.get(url)response_json = response.json()readme_content = response_json["content"]return readme_content```上面的代码定义了一个名为`get_readme_content`的函数,它接受两个参数`owner`和`repo`,分别表示所有者和仓库的名称。
函数首先构造了一个API请求的URL,然后使用`requests.get`发送GET请求并获取响应JSON。
最后,我们从响应JSON中提取`content`字段,该字段包含了README文件的Base64编码内容。
使用这个函数,我们可以获取README文件的内容。
以下是一个示例:```pythonowner = "github_username"repo = "repository_name"readme_content = get_readme_content(owner, repo)print(readme_content)```在上面的代码中,将`github_username`替换为你的GitHub用户名,将`repository_name`替换为仓库的名称。
Readme(安装参考方法二)

8.打开License Server Administrator,启动服务。
FEATURE ArcView3 ARCGIS 10.0 01-jan-0000 2048 ADBC8EF1F28B9DC18F96 \
vendor_info=TXDH42L7EY6Z4X4JE221 ck=174
FEATURE ArcView31 ARCGIS 10.0 01-jan-0000 2048 FDCC4EB18017443D6297 \
vendor_info=H2T570H3D291F1TGH254 ck=55
FEATURE ArcReader ARCGIS 10.0 01-jan-0000 2048 0D9CBE710E5695C37E6E \
vendor_info=758Y1G82MC96F90LD184 ck=78
4.如果你只想授权几个功能就选择相应的功能按添加,每添加一个就增加一个功能的授权(按移除可以移除上一个授权,按清除可清除所有已添加的授权),如果你想授权所有功能,按所有即可。
5生成了许可文件内容后,全选框内的文字复制这些内容,新建一个文本文档,粘贴这些文字,另存文件为service.txt。
6.也可将下面上下------------之间内容copy至service.txt,就不用注册机了,这个是长久注册的,要修改“此处为计算机名”内容为你的计算机名
github readme 格式

github readme 格式一、概述GitHub是一个流行的代码托管平台,用于存储、共享和协作开发代码。
在GitHub上,每个项目都有自己的仓库,其中包含代码、文档、图片等资源。
为了更好地展示项目信息和与他人的协作,通常会编写一个Readme文件,该文件位于项目根目录下,用于介绍项目的基本信息、功能、依赖项、安装和使用方法等。
二、格式规范1. 标题:使用加粗字体,例如**标题**。
标题应简明扼要地描述项目的主要特点或目的。
2. 项目描述:用一段简短的文字介绍项目的主要功能和用途。
可以包括项目的应用场景、主要解决的问题等。
3. 依赖项:列出项目所依赖的库、框架或其他项目,以及它们的版本要求。
确保所有依赖项都已正确安装并更新到最新版本。
4. 安装方法:提供项目的安装指南,包括操作系统、软件包管理器、命令行指令等。
确保安装过程清晰明了,易于操作。
5. 文件结构:简要描述项目文件和文件夹的结构,包括公共文件和子文件夹。
6. 使用方法:提供如何使用项目的基本指南,包括如何创建实例、调用方法、配置参数等。
提供示例代码和截图,以帮助读者更好地理解项目的使用方式。
7. 常见问题:列出在使用项目过程中可能遇到的问题及解决方案,以便用户快速找到帮助。
8. 贡献指南:说明如何参与项目的开发、提交补丁和参与讨论。
提供所需的开发工具和环境要求,以及代码规范和贡献流程。
9. 许可证:明确项目的许可证信息,确保用户在使用项目时遵守相关规定。
三、示例以下是一个简单的GitHub Readme示例:介绍一个简单的Python程序库,用于处理数字计算。
该库依赖于Python标准库和其他常用库(如numpy和matplotlib),可以通过pip安装。
* 项目描述:该库提供了一些基本的数学函数和计算工具,用于处理数字计算问题。
* 依赖项:依赖Python标准库、numpy和matplotlib。
* 安装方法:使用pip安装该库:`pip install simple_math`* 文件结构:库文件位于一个名为`simple_math`的文件夹中,包含一些Python模块和脚本文件。
readme(客户端使用须知)
CUMCM客户端使用须知
注意事项:
1.本客户端是一个绿色软件,无需安装,下载的是一个压缩文件,但需要解压后使用,如因未解压造成使用造成不能上传或相关问题请自行负责
2.第一次登录客户端会与远程服务器进行绑定,然后会自动退出,请在退出后重新启动并登录使用即可
3.本客户端仅限于WINDOWS操作系统环境下使用,当前主流的WINDOWS版本(如win7/8/10/xp)均已测试运行正常,但不保证某些版本的情况中出现不能运行或异常情况
4.使用过程中如弹出现有提示框信息,请务必认真阅读,以免影响上传和其他操作
5.建议将本客户端解压到自己的移动硬盘中,或建立本参赛队专属的目录夹
6.一个参赛队,请勿同时使用多个客户端
一些约定:
1.允许上传的参赛作品文件格式为doc、docx、pdf、wps,支撑材料格式为rar、zip
2.允许上传的文件大小规定最大值为20M,即20971520字节
可能出现的问题:
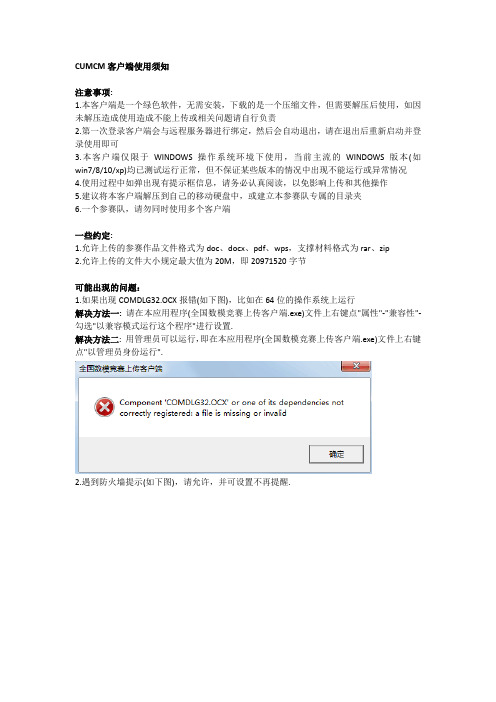
1.如果出现COMDLG3
2.OCX报错(如下图),比如在64位的操作系统上运行
解决方法一:请在本应用程序(全国数模竞赛上传客户端.exe)文件上右键点"属性"-"兼容性"-勾选"以兼容模式运行这个程序"进行设置.
解决方法二: 用管理员可以运行,即在本应用程序(全国数模竞赛上传客户端.exe)文件上右键点"以管理员身份运行".
2.遇到防火墙提示(如下图),请允许,并可设置不再提醒.。
github readme模板

github readme模板在GitHub上创建一个readme文件时,可以使用以下模板来提供项目的重要信息和说明。
# 项目名称简短的项目描述## 目录- [特色](#特色)- [使用方法](#使用方法)- [技术栈](#技术栈)- [贡献者](#贡献者)- [许可证](#许可证)## 特色在这里介绍项目的特色和亮点。
可以列出几个项目的主要功能或特性。
## 使用方法提供如何使用项目的简要说明,例如安装、配置和运行项目的步骤。
```bash# 示例代码或命令```## 技术栈列出项目使用的主要技术、工具和库。
- 技术1- 技术2- 技术3## 贡献者感谢所有参与该项目的贡献者。
可以列出贡献者的GitHub用户名,并对其进行感谢。
## 许可证在这里说明项目的许可证类型和约束条件。
例如,MIT许可证。
```版权所有 (c) 年份项目作者根据MIT许可证授权,任何人都可以获得此软件和相关文档文件(“软件”)的副本,以处理若干限制亦非此达的目的,包括但不限于:本软件的复制、修改、合并、发布、发布、再许可、分发和/或销售副本的许可和授权,以及为软件被提供的提供支持的人员,但本软件的所有支持。
上述版权声明和本许可声明应包含在本软件的所有副本或实质性部分中。
软件按“原样”提供,不提供任何明示或暗示的保证,包括但不限于适销性、特定目的的适用性和非侵权性。
在任何情况下,作者或版权所有者均不对由本软件引起的任何索赔、损害或其他责任承担责任,无论是因合同、侵权还是其他方式产生的,除非根据适用的法律或书面同意,软件所要求的授权。
```使用此模板来创建您的GitHub readme文件,可以提供清晰的项目信息和指导,帮助他人了解和使用您的项目。
同时,根据自己的项目需求,可以根据以上模板进行灵活修改和扩展。
Gitee中README.md文件的编辑和使用

Gitee中README.md⽂件的编辑和使⽤最近对它的README.md⽂件颇为感兴趣。
便写下这贴,帮助更多的还不会编写README⽂件的同学们该⽂件⽤来测试和展⽰书写README的各种markdown语法。
开始编辑README当没有README⽂件的时候我们可以添加⼀个README.me⽂件关于标题规范的README⽂件开头都写上⼀个标题,这被称为⼤标题⽐⼤标题低⼀级的是中标题,也就是显⽰出来⽐⼤标题⼩点。
⼤标题===效果如图:在⽂本下⾯加上等于号 = ,那么上⽅的⽂本就变成了⼤标题。
等于号的个数⽆限制,但⼀定要⼤于0个哦。
⽐⼤标题低⼀级的是中标题,也就是显⽰出来⽐⼤标题⼩点中标题-------效果如图:在⽂本下⾯加上下划线 - ,那么上⽅的⽂本就变成了中标题,同样的下划线个数⽆限制。
除此之外,你也会发现⼤,中标题下⾯都有⼀条横线,没错这就是 = 和 - 的显⽰结果。
如果你只输⼊了等于号=,但其上⽅⽆⽂字,那么就只会显⽰⼀条直线。
如果上⽅有了⽂字,但你⼜只想显⽰⼀条横线,⽽不想把上⽅的⽂字转义成⼤标题的话,那么你就要在等于号=和⽂字直接补⼀个空⾏。
补空⾏:是很常⽤的⽤法,当你不想上下两个不同的布局⽅式交错到⼀起的时候,就要在两种布局之间补⼀个空⾏。
如果你只输⼊了短横线(减号)-,其上⽅⽆⽂字,那么要显⽰直线,必须要写三个减号以上。
不过与等于号的显⽰效果不同,它显⽰出来时虚线⽽不是实线。
同减号作⽤相同的还有星号*和下划线_,同样的这两者符号也要写三个以上才能显⽰⼀条虚横线。
除此以外,关于标题还有等级表⽰法,分为六个等级,显⽰的⽂本⼤⼩依次减⼩。
不同等级之间是以井号 # 的个数来标识的。
⼀级标题有⼀个 #,⼆级标题有两个# ,以此类推。
# ⼀级标题## ⼆级标题### 三级标题#### 四级标题##### 五级标题###### 六级标题效果如图:实际上,前⽂所述的⼤标题和中标题是分别和⼀级标题和⼆级标题对应的。
readme

0x0051BA9A Thunder.exe[0011BA9A] :(7FFD5000,0012FFD4,77B3B3F5,7FFD5000 [00 50 FD 7F D4 FF 12 00 F5 B3 B3 77 00 50 FD 7F])
0x00413671 Thunder.exe[00013671] :(0026184E,00000113,00000001,00000000 [4E 18 26 00 13 01 00 00 01 00 00 00 00 00 00 00])
0x00410D03 Thunder.exe[00010D03] :(01526A74,00000113,00000001,00000000 [74 6A 52 01 13 01 00 00 01 00 00 00 00 00 00 00])
thunder5(迅雷5) 5.9.20.1418
Process(PID:DD8) : C:\Program Files\Thunder Network\Thunder\Program\Thunder.exe
Microsoft Windows 7 [Build 6.1.7600]
Architecture : x86,ProcessorNum : 2
0x2611D686 XLGUIPlatform.dll[0001D686] :(00000000,01526A70,00000001,0012FCF0 [00 00 00 00 70 6A 52 01 01 00 00 00 F0 FC 12 00])
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
1result/|-- 00.RawData/ 【原始下机数据和原始拼接后数据】 | |-- Sample_Name/ 【每个样品对应的原始下机数据和原始拼接后数据】 | | |--*_1.fq.gz 【reads1去除barcode 和primer 后得到的序列】 | | |--*_2.fq.gz【reads2去除barcode 和primer 后得到的序列】| | |--*.raw_1.fq.gz 【reads1原始下机序列,包含barcode 和primer 】 | | |--*.raw_2.fq.gz 【reads2原始下机序列,包含barcode 和primer 】 | | `--*.extendedFrags.fastq 【原始下机的reads 拼接后的序列】| |-- SampleSeq_info.xls 【所有样品的barcode 和primer 信息】 | `-- assemble_stat.xls【所有样品的序列拼接信息统计列表】|-- 01.CleanData/ 【质控后可用于后续分析的有效数据】 | |-- Sample_Name/ 【每个样品的质控结果】| | |-- *.fastq 【质控后序列的FASTQ 格式文件】 | | |-- *.fna 【质控后序列的FASTA 格式文件】 | | `-- histograms.txt 【质控后的序列长度分布统计表】| `-- QCstat.xls【数据预处理统计及质控信息表】|-- 02.OTUanalysis/【OTUs 聚类和物种注释结果】 | |-- OTUs.fasta【OTUs 代表序列】| |-- OTUs.tax_assignments.txt 【OTUs 物种注释结果】 | |-- all_rep_set_tax_assignments.krona.html 【krona 网页展示】 | |-- OTUs.tre 【OTUs 进化树文件】| |--otu_table_even.biom 【均一化处理后biom 格式的绝对丰度表】 | |-- taxa_abundance/【每个样品中物种丰度信息】| | |-- evenabs/ 【均一化处理后的绝对丰度(均一化处理使每个样品的OTUs 丰度之和相等)】2| | | |-- otu_table.absolute.xls 【OTUs 绝对丰度】| | | `-- otu_table.*.absolute.xls 【界门纲目科属种(k,p,c,o,f,g,s )水平上的绝对丰度】 | | `-- relative/【均一化处理后的相对丰度(均一化处理使每个样品的OTUs 丰度之和相等)】| | |-- otu_table.relative.xls 【OTUs 相对丰度】| | `-- otu_table.*.relative.xls 【界门纲目科属种(k,p,c,o,f,g,s )水平上的相对丰度】| |-- taxa_stat/【OTUs 分析统计结果】| | |-- Classified_stat.{png,svg} 【注释到界门纲目科属种(k,p,c,o,f,g,s )水平上的Tags 数目分布图】 | | |-- classified_stat.xls 【注释到界门纲目科属种(k,p,c,o,f,g,s )水平上的Tags 数目统计表】 | | |-- Sample_Tags-OTUs_dis.{png,svg} 【各样品的Tags 及OTUs 数目统计分布图】 | | `-- Tags_stat.xls 【各样品的Tags 及OTUs 数目统计表】 | |-- taxa_charts_html/ 【注释结果网页版展示】 | | |-- area_charts.html 【注释结果面积图展示】 | | |-- bar_charts.html 【注释结果柱形图展示】 | | |-- pie_charts.html【注释结果饼状图展示】| | |-- charts/【网页展示用到的图片】| | |-- css/ 【网页配置文件】 | | `-- js/ 【网页配置文件】 | |-- top10/【门纲目科属(p,c,o,f,g )水平上的top10物种相对丰度柱形图】 | |-- taxa_heatmap/【物种注释聚类热图】 | | |-- cluster/ 【门纲目科属(p,c,o,f,g )水平上的物种聚类热图】 | | `-- OTU_heatmap/【OTUs 注释和丰度热图】 | |-- taxa_tree/【物种分类树】| |-- *.taxtree.{png,svg} 【单个样品的物种分类树】 | `-- all.taxtree.{png,svg}【所有样品的物种分类树】| |-- GraPhlan/ 【GraPhlan 结果展示】 | | |-- graphlan.{png,pdf} 【GraPhlan 图】3| |-- phylo_tree/ 【OTUs 进化树】| | |-- OTU.cluster.tree.{png,svg} 【OTUs 进化树图】 | | `-- OTUs.selected.tre 【OTUs 进化树文件,MEGA 软件可打开】|-- 03.AlphaDiversity/ 【alpha 多样性分析结果】 | |-- alpha_rarefaction_plots/ 【alpha 多样性分析结果网页版展示】 | |-- alpha_diversity_index.xls【alpha 多样性指数表格】 | |-- observed_species.{pdf,png} 【稀释曲线图(样本)】| |-- group_observed_species.{pdf,png}【稀释曲线图(分组)】| |-- plot_observed_species.txt【稀释曲线作图数据】 | |-- rank_abundance.{pdf,png} 【等级丰度曲线图(样本)】| |-- group_rank_abundance.{pdf,png}【等级丰度曲线图(分组)】| |-- venn_figure/ 【韦恩图】 | |-- Flower_figure/【花瓣图】| |-- Specaccum/ 【物种累积曲线】 | |-- specaccum.{png,pdf} 【物种累积曲线图】 | |-- Alpha_div/ 【组间Alpha 多样性比较箱型图】| |-- ACE / | |-- ACE.{png,pdf}【ACE 指数箱型图】| |-- ACE_Tukey.txt 【ACE 指数多组间方差分析】||--ACE_wilcox.txt 【ACE 指数多组间非参数wilcox 检验】| |-- chao1/| |-- chao1.{png,pdf}【chao1指数箱型图】| |-- chao1_Tukey.txt 【chao1指数多组间方差分析】| |-- chao1_wilcox.txt 【chao1指数多组间非参数wilcox 检验】 | |-- goods_coverage /4| |-- goods_coverage.{png,png} 【goods_coverage 指数箱型图】| |-- goods_coverage _Tukey.txt 【goods_coverage 指数多组间方差分析】| |-- goods_coverage _wilcox.txt 【goods_coverage 指数多组间非参数wilcox 检验】 | |-- observed_species /| |-- observed_species.{png,png} 【observed_specie 指数箱型图】| |-- observed_species _Tukey.txt 【observed_species 指数多组间方差分析】| |-- observed_species _wilcox.txt 【observed_species 指数多组间非参数wilcox 检验】 | |-- shannon /| |-- shannon.{png,png}【shannon 指数箱型图】| |-- shannon _Tukey.txt 【shannon 指数多组间方差分析】| |-- shannon _wilcox.txt 【shannon 指数多组间非参数wilcox 检验】 | |-- simpson /| |-- simpson.{png,png}【simpson 指数箱型图】| |-- simpson _Tukey.txt 【simpson 指数多组间方差分析】| |-- simpson _wilcox.txt 【simpson 指数多组间非参数wilcox 检验】|-- 04.BetaDiversity/ 【Beta 多样性分析结果】| |-- Beta_div/ 【组间Beta 多样性比较箱型图】| |-- weighted_unifrac.{png,pdf} 【基于加权的unifrac 距离的Beta 多样性箱型图】 | |-- weighted_unifrac_TukeyHSD.txt 【基于加权的unifrac 距离的多组间方差分析】| |-- weighted_unifrac_wilcox.txt 【基于加权的unifrac 距离的多组间非参数wilcox 检验】 | |-- unweighted_unifrac.{png,pdf} 【基于非加权unifrac 距离的Beta 多样性箱型图】 | |-- unweighted_unifrac_TukeyHSD.txt 【基于非加权的unifrac 距离的多组间方差分析】| |-- un weighted_unifrac_wilcox.txt 【基于非加权的unifrac 距离的多组间非参数wilcox 检验】 | |-- beta_div_heatmap/【unifrac 距离热图】| | |-- beta_diversity.heatmap.{png,svg} 【包含两种距离的热图】5| | |-- beta_diversity.heatmap.UnW.{png,svg} 【非加权unifrac 距离热图】 | | |-- beta_diversity.heatmap.W.{png,svg} 【加权unifrac 距离热图】 | | |-- unweighted_unifrac_sorted_otu_table.txt 【非加权unifrac 距离值】 | | `-- weighted_unifrac_sorted_otu_table.txt 【加权unifrac 距离值】 | |-- PCA 【PCA 分析结果】 | | |-- PCA12_2.{png,pdf}【标有样品名称的PCA 图】| | |-- PCA12.{png,pdf} 【未标样品名称的PCA 图】 | | |-- pca.csv 【各个主成分分析结果】 | | |-- PCA_stat_correlation1.txt 【第一主成分分析结果】 | | `-- PCA_stat_correlation2.txt 【第二主成分分析结果】 | |-- PCoA/【PCoA 分析】| | |-- unweighted_unifrac/【基于非加权unifrac 距离的PCoA 分析结果】| | | |-- {*.png ,*.pdf} 【前三个主成分两两作图结果】| | | |-- unweighted_unifrac_dm.txt 【用于PCoA 分析的unweighted unifrac 距离矩阵】 | | | `-- unweighted_unifrac_pc.txt 【PCoA 分析主成分信息】 | | `-- weighted_unifrac/【基于加权unifrac 距离的PCoA 分析结果】| | |-- {*.png ,*.pdf} 【前三个主成分两两作图结果】| | |-- weighted_unifrac_dm.txt 【用于PCoA 分析的weighted unifrac 距离矩阵】 | | `-- weighted_unifrac_pc.txt 【PCoA 分析主成分信息】 | | |--binary_jaccard__dm.txt 【binary_jaccard 距离矩阵】 | | |--binary_jaccard_pc.txt 【PCoA 分析主成分信息】 | | |--bray_curtis_dm.txt 【bray_curtis 距离矩阵】 | | |--bray_curtis_pc.txt 【PCoA 分析主成分信息】 | `-- Tree/【聚类树】| |-- unweighted_unifrac/【基于非加权unifrac 距离的聚类结果】| | |-- unweighted_unifrac.{png,pdf} 【基于非加权unifrac 距离的UPGMA 聚类树图】6| | |-- sorted_otu_table_upgma.tre 【基于非加权unifrac 距离的UPGMA 聚类树文件,MEGA 软件可以打开】 | | `-- UPGMA.UnW.tree.{png,svg} 【加入门水平物种组成的UPGMA 聚类树图】 | `-- weighted_unifrac/【基于加权unifrac 距离的聚类结果】| |-- sorted_otu_table_upgma.tre 【基于加权unifrac 距离的UPGMA 聚类树文件,MEGA 软件可以打开】 | |-- weighted_unifrac.{pdf,png} 【基于加权unifrac 距离的UPGMA 聚类树图】 | `-- UPGMA.W.tree.{png,svg} 【加入门水平物种组成的UPGMA 聚类树图】 | |-- NMDS/ 【NMDS 分析结果】| | |-- NMDS.{png,pdf} 【标有样品名称的NMDS 图】 | | |-- NMDS2.{png,pdf} 【未标样品名称的NMDS 图】| | |-- NMDS_scores.txt 【各样品在前两个主成分轴上的位置坐标】 | |-- LEfSe/ 【LEfSe 分析结果】 | | |-- */ LDA.*.{png,pdf}【LDA 值分布柱状图】| | |-- */ LDA.*.tree.{png,pdf} 【LEfSe 进化分支图】 | | |-- */LDA.*.res 【LEfSe 统计结果】| | |-- */biomarkers_raw_images/ 【各biomarker 在各组样品中的相对丰度比较图】 | |-- MetaStat/【MetaStat 分析结果】| | |-- */*.test.xls【门纲目科属(p,c,o,f,g )水平上MetaStat 分析结果】| | |-- */*.psig.xls 【从MetaStat 分析结果中,筛选出的 p value<=0.05的信息】 | | |-- */*.qsig.xls 【从MetaStat 分析结果中,筛选出的 q value<=0.05的信息】 | | |-- */cluster.*.diff.{pdf,png,txt} 【具有显著性差异物种的 heatmap 热图分析结果和输入文档】 | | |-- */boxplot / 【具有显著性差异物种的箱图结果】 | | |-- */PCA/ 【具有显著性差异物种的 PCA 分析结果】 | |-- Anosim/ 【Anosim 分析结果】 | | |-- stat_anosim.txt【Anosim 分析结果】| | |-- *.{pdf,png} 【Anosim 分析箱图结果】 | |-- MRPP/ 【MRPP 分析结果】7| | |-- stat_mrpp.txt 【MRPP 分析结果】 | |-- Adonis/ 【Adonis 分析结果】 | | |-- bray_adonis.txt 【Adonis 分析结果】 | |-- Amova/ 【Amova 分析结果】 | | |--(un)weighted_unifrac/*_amova.txt 【Amova 分析结果】| |-- t.test_bar_plot/ 【组间差异显著的物种分析】| |-- */*-VS-*. {png,svg} 【门纲目科属(p,c,o,f,g )水平上的组间差异显著的物种分析条形图】 | |-- */*-VS-*.xls 【门纲目科属(p,c,o,f,g )水平上组间差异显著的物种分析结果】 | |-- */*-VS-*.psig.xls 【从组间差异显著的物种分析结果中,筛选出的 p value<=0.05的信息】 | |-- ternaryplot/ 【ternaryplot 分析结果】| |-- */*/-- ternary.{png,pdf} 【未标有样品名称的ternaryplot 图】 | |-- */*/-- ternary_1.{png,pdf} 【标有物种名称的ternaryplot 图】 | |-- Environmen_factor/【环境因子分析】| | |-- mantel_test/ 【mantel_test 分析结果】| | |-- spearman/ 【spearman 分析结果】 | | |-- VPA/ 【VPA 分析结果】| | |-- multiCCA/【CCA 分析结果】`-- 05.WebShow/ 【网页版展示内容综合,可交互式操作,同时含使用说明】。
