质粒载体的构建
质粒构建实验步骤1

质粒构建实验步骤实验一1.将合成的引物溶解(10uM),分别扩增P1P2,P3P4,2.PCR扩增条件如下:95 ℃ 10 min95 ℃ 30 s50℃ 30 s 10cycle72℃ 30 s72℃ 5min3.反应体系为:20 ul (p1p2和p3p4各两管)dNTP 2 ul10xBuffer 2ulTaq 0.4 ulPrimer P1 (P3) 1.0 ulPrimer P2 (P4) 1.0 ulH2O 13.6 ul4.将产物电泳,检测是否为目标片断(分别为A1B、A2B),证实为目的片断以后,进行后续扩增5.将扩增出的AB和CD片断等摩尔加入作为模板进行第二次PCR,并且在第二次PCR 时加入引物扩增25个循环,PCR扩增条件如下:95 ℃ 15 min95 ℃ 30 s60 ℃ 30 s 25cycle72℃ 40 s72℃ 5min6.反应体系为:100uldNTP 10 ul10xBuffer 10 ulTaq 1ulProduct P1P2 2 ulProduct P3P4 2 ulPrimer F 1 ulPrimer R 1 ulH2O 73ul实验二连接载体DNA外源DNA片段10×T4 DNA ligase bufferT4 DNA ligase 0.5μl16℃保温8-24小时。
做二组对照反应,其中对照组一只有质粒载体无外源DNA;对照组二只有外源DNA片段没有质粒载体。
实验三10.转化①取100ul感受态细胞置于冰上融化,将50ul感受态细胞加入至10ul连接产物中,冰上30min。
②42℃放置45~60s,冰上放置2~3min。
③加入37℃预温好的200ul LB液体培养基(不含Amp)④37℃振荡培养1h(160~220 rpm)11.铺板铺板之前要先将AMP抗性的LB固体培养基先预温到常温,然后取100 ul菌液涂板,37℃培养过夜(16~24h)12.不带有质粒DNA的细胞,由于无Amp抗性,不能在含有Amp的筛选培养基上成活。
质粒载体的操作和cDNA文库的构建

(一)细菌培养物的生长从琼脂平板上挑取一个单菌落,接种到培养物中(有含有行当抗生素的液体培养基中生长),然后从中纯化质粒,质粒的提纯几乎总是如此。
现在使用的许多质粒载体(如pUC系列)都能复制到很高的拷贝数,惟致只要将培养物放在标准LB 培养基中生长到对数晚期,就可以大量提纯质粒。
此时,不必造反性地扩增质粒DNA。
然而,较长一代的载体(如pBR322)由于不能如此自由地复制,所以需要在得到部分生长的细菌培养物中加入氯霉素继续培养若干小时,以便对质粒进行性扩增。
氯霉素可抑制宿主的蛋白质合成,结果阻止了细菌染色体的复制,然而,松弛型质粒仍可继续复制,在若干小时内,其拷贝数持续递增。
这样,像pBR322-类的质粒,从经氯霉素处理和未经处理的培养物中提取质粒的产量迥然不同,前者大为增高。
多年来,加入足以完全抑制蛋白质合成的氯霉素μg/ml)已成为标准的操作、用该方法提取的质粒DNA量,对于分子克隆中几乎所有想象到的工作任务。
(二)细菌的收获和裂解细菌的收获可通过离心来进行,而细菌的裂解则可以采用多种方法中的任意一种,这些方法包括用非离子型或离子型去污剂、有机溶剂或碱进行处理及用加热处理等。
选择哪一种方法取决于3个因素:质粒的大小、小肠杆菌菌株及裂解后用于纯化质粒DNA的技术。
尽管针对质粒和宿主的每一种组合分别提出精确的裂解条件不切实际,但仍可据下述一般准则来选择适当方法,以取得满意的结果。
1)大质粒(大于15kb)容易受损,故应采用漫和裂解法从细胞中释放出来。
将细菌悬于蔗糖等渗溶液中,然后用溶菌酶和EDTA进生处理,破坏细胞壁和细胞外膜,再加入SDS一类去污剂溶解球形体。
这种方法最大限度地减小了从具有正压的细菌内部把质粒释放出来所需要的作用力。
2)可用更剧烈的方法来分离小质粒。
在加入EDTA后,有时还在加入溶菌酶后让细菌暴露于去污剂,通过煮沸或碱处理使之裂解。
这些处理可破坏碱基配对,故可使宿主的线状染色体DNA变性,但闭环质粒DNA链由于处于拓扑缠绕状态而不能彼此分开。
质粒的构建

② -半乳糖苷酶Xgal显色反应: -半乳糖苷酶能把无色的化合物 Xgal分解成半乳糖和一个深蓝色的 物质5-溴-4-氯靛蓝。 -半乳糖苷酶 Xgal 半乳糖
5-溴-4-氯靛蓝
③ lacZ的肽互补 1)-肽( lacZ’ ): -半乳糖苷酶N端的一段氨基酸片断 (11-41氨基酸)。 N端的11-41aa N端的11-41aa N端的11-41aa N端的11-41aa C端大部分 C端大部分 C端大部分 C端大部分
(2)长度
4363bp
(3)选择标记 氨苄青霉素和四环素抗性。
(4)克隆位点
24个克隆位点。
其中9个会导致Tetr基因失活(如 BamH I、Hind Ⅲ、Sal I); 3个会导致Ampr基因失活(Sca I、 PvuI、Pst I)。
(5)pBR322的筛选 ①双抗菌素抗性选择标记
在EcoRⅠ和HindⅠ酶剪切位点之间插入外源基因
(2)长度
(3)克隆位点
约2.7kb
10个连续的单一限制酶切位 点,位于lacZ’基因的5’端。
ห้องสมุดไป่ตู้
pUC18/19
选择标记 Ampicillin 抗性和 lacZ的肽互补(蓝白 斑)相结合。 筛选方法
选用带有含有ampicillin和X-gal的培养基 受体菌lacZ突变(lacZ∆M15)
受体菌lacZ突变(lacZ∆M15) 受体菌基因组的-半乳糖苷酶基因的 氨基端有缺失(缺失肽),不能形成 4聚体的活性酶,不能分解Xgal 受体菌株:JM系列、TG1、TG2、 XL1-blue、XS127、XS101、KK2186、 MV1184、DH5a
质粒载体生物学特征
分类 按复制类型可分为:松弛型 严紧性 按转移性可分为转移型和非转移型
质粒构建课件

Digestion & ligation
PCR product or vector Enzyme 1 Enzyme 2 10XBuffer 100XBSA(?) ddH2O
37°C 2~3 h
PCR product Vector T4 DNA ligase 10Xligase buffer ddH2O
pCS2(GFP)
……
/vectordb/
Gene cloning
Target gene: hBex2 Vector selection Primer design Amplification, digestion & ligation Transformation & Identification
AAT TCG AAG
CGG TG
EcoR I/BamHI digestion
BamH I GAT CCA CCG
GFP
G AAT TCG AAG
insert
CTT CG A ATT C
GCG GAT CCA CCG
CGG TGG ATC CGC
GFP
Gene cloning
Target gene: hBex2 Vector selection Primer design Amplification, digestion & ligation Transformation & Identification
质粒构建
孟庆明 2019-04-09
1
1
主要内容
质粒的概念及特点 质粒构建的基本步骤和原理
一、质粒的概念
载体(vector;carrier;vehicle) 可以插入核酸片段、能携带外源核酸进入宿主细胞,并在其中进行 独立和稳定的自我复制的核酸分子。基因工程中广泛应用的载体多 来自人工改造的细菌质粒、噬菌体或病毒核酸等。多数载体是DNA 分子,但某些RNA分子也能用做载体。
重组表达质粒的构建——原核表达载体选择
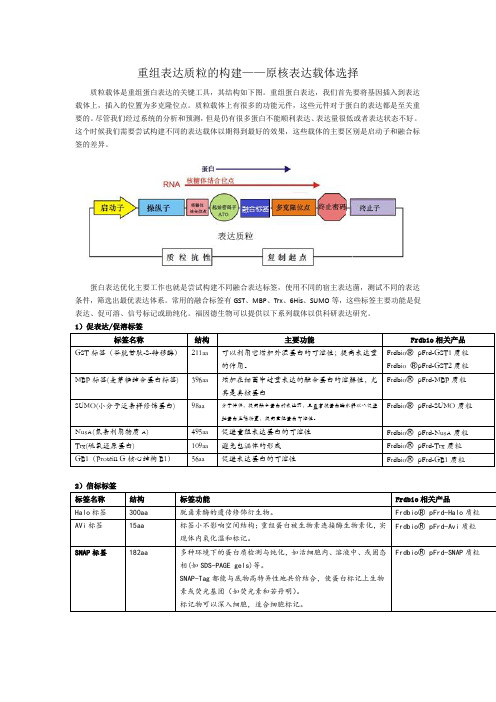
重组表达质粒的构建——原核表达载体选择质粒载体是重组蛋白表达的关键工具,其结构如下图。
重组蛋白表达,我们首先要将基因插入到表达载体上,插入的位置为多克隆位点。
质粒载体上有很多的功能元件,这些元件对于蛋白的表达都是至关重要的。
尽管我们经过系统的分析和预测,但是仍有很多蛋白不能顺利表达、表达量很低或者表达状态不好。
这个时候我们需要尝试构建不同的表达载体以期得到最好的效果,这些载体的主要区别是启动子和融合标签的差异。
蛋白表达优化主要工作也就是尝试构建不同融合表达标签,使用不同的宿主表达菌,测试不同的表达条件,筛选出最优表达体系。
常用的融合标签有GST、MBP、Trx、6His、SUMO等,这些标签主要功能是促表达、促可溶、信号标记或助纯化。
福因德生物可以提供以下系列载体以供科研表达研究。
1)促表达/促溶标签2)信标标签3)纯化标签我们选择表达载体的时候不但要考虑蛋白怎么表达成功,更要考虑蛋白怎么纯化出来,纯化的问题主要是考虑纯化标签和酶切位点的选择,下表我们列举了常见的纯化标签和酶切位点。
4)酶切位点以上为原核表达常用的标签和酶切位点,其性质也都作了简要的介绍,各专业网站或专业书籍已对此做详尽解释,科研工作者可根据具体实验设计方案,组合设计以上标签和酶切位点的使用。
特别值得注意的是,选用和设计蛋白酶切位点的时候首要考虑的是序列内部有没有蛋白酶位点,同时要考虑酶切的效率和蛋白酶试剂成本。
一般商业化载体,在标签蛋白与载体多克隆位点之间都设计有酶切位点。
标签可设计在N-端也可在C-端,设计在N-端的优势是,可通过标签高效翻译起始位点带动插入蛋白的表达,可溶性标签的高效表达更可促进蛋白的可溶性表达;同时,大部分的蛋白内切酶的切割位点在C-端,所以标签设计在N-端可将标签切割完全。
在设计标签序列与酶切位点的时候还要考虑N-端稳定性原则,也就是所谓宿主细胞的N-端规则(N-end rule),这个要避免;同时,还应该检查是否引入了可与别的蛋白相互作用的序列或者蛋白酶切位点。
载体构建介绍

5.常见问题
选择标记类型和选择 1、选择性标记类型 药物抗性(如Kan,Amp等) 营养依赖性标记(如SC-Leu 等) 2、如何选择 根据所选载体上所带的标记
5.常见问题
Gateway系统引物设计-需要对读码框
这是因为中间载体和终载体上某些编码氨基酸或者抗性 基因同目的片段共用一个起始密码子。
载体构建
戎浩 2015.12
C
ONTENTS
目录
1 2 3 4
载体简介 载体构建
注意事项 常见问题
1.载体简介
目的基因的克隆与鉴定
ห้องสมุดไป่ตู้
生理检测 纯化
载体构建
大肠转化,质粒 提取与鉴定
移栽
分子检测
继代繁殖 农杆菌的转化与活化
外植体制备
筛选
浸
染
共培养
1.1载体
载体(vector) ,能将外源DNA或基因片段携带入 宿主细胞内的一个具有自我复制能力的DNA分子。
退火温度不合适,设置梯度,选择最适退火温度
2、条带不单一
引物不特异,适当增长引物序列长度;
适当提高退火温度
5.常见问题
载体酶切的问题 1、酶切质粒浓度和纯度要好 2、酶切温度和时间
如果两个酶的最适温度不同,建议单酶切,回收后在
用另一个酶切,时间最好过夜切。 3、没有切开 可能是酶失活,建议酶切时增加阳性对照,确定酶是 否好用
5.常见问题
克隆基因的酶切位点及引物问题 1、单酶切 单酶切后进行连接,质粒自连、目的片段自连、目的
片段之间连接、目的片段和载体各种错误连接、目的片段
反向连接等等,尽量不选单酶切 2、保护碱基数目的问题。 在设计PCR引物时,引入酶切位点后,常常要加入保 护碱基,这会使得酶切效率大大提高。
质粒载体的构建

四、连接 很多公司都有快速连接试剂盒和普通试剂盒。一般都是 16 度数个小时或者过夜,或者 4 度 过夜,不一而同。曾在园子里看到一个高人的文章,认为是时间越长越好,甚至建议 4 度放 几天,我没有试过。16 度可以用 PCR 仪创造,或者找一个泡沫盒,加上冰,水搞到 15 度 左右,放入 4 度冰箱即可。我用的 TAKARA 的快速连接试剂盒,4 度过夜。 菜鸟体贴提示: 1、 通过电泳,粗略判定质粒和 DNA 的浓度比例。一般加入连接液的 DNA:质粒=9:1 2、 放入质粒和 DNA 的总量要适当,不可超过说明书,宁少不要多。
质粒载体的构建-菜鸟入门手册
dongkey 制作
本人刚刚完成质粒载体的构建,总结了一下,以方便要做这方面的菜鸟同学借鉴一下,希望 老鸟同学批评指教。 一、确定插入的基因片段。 首先要确定自己要插入载体中的基因片段,比如要做蛋白表达和功能,可以选择 CDS 区进 行插入。然后自然是如何得到这段基因片段的问题了。最常见的是 PCR 解决了。那么就涉 及到引物的设计。插入 CDS 基因片段的例子:找到 CDS,根据 CDS 设计全长引物,然后 加上内切酶的碱基片段(这要根据自己手头上有的质粒的内切酶位点来决定了),注意,前 面要加上保护碱基!然后进行 PCR。 PCR 后要跑电泳确定目的基因的长度是正确的。 用乙醇沉淀法纯化 PCR 产物(具体见分子克隆一书) 菜鸟体贴提示:要在质粒上找到两个最好不是连续在一起的内切酶,然后分别添加在两条引 物的 5‘端,当然内切酶的温度最好是一致的,而且可以能够同时切开的(双酶切)。这可 以在试剂公司的限制性内切酶的列表上找到有没有共同的 BUFFER 及其双酶切时的活性如 何(当然是越大越好了)。内切酶的公司一般可以找 NEB, TAKARA, TOYOBO 等公司,本 人用的是 TAKARA。 二、酶切 将质粒和基因片段分别进行酶切。最省事的是同时双酶切了。一般酶切温度都是 37 度。 菜鸟体贴提示: 1、 确定质粒和基因片段的量!!(宁少不多),根据说明书加样,一般是 DNA+BUFFER+
质粒构建

PCR(多聚酶链式反应)一、实验原理PCR用于扩增位于两端已知序列之间的DNA区段,即通过引物延伸而进行的重复双向DNA合成。
基本原理及过程如下:PCR循环过程中有三种不同的事件发生:(1)模板变性;(2)引物退火;(3)热稳定DNA聚合酶进行DNA合成。
1、变性:加热使模板DNA在高温下(94-95℃)变性,双链间的氢键断裂而形成两条单链,即变性阶段。
2、退火:在体系温度降至37-65℃,模板DNA与引物按碱基配对原则互补结合,使引物与模板链3’端结合,形成部分双链DNA,即退火阶段。
3、延伸:体系反应温度升至中温72℃,耐热DNA聚合酶以单链DNA为模板,在引物的引导下,利用反应混合物中的4种脱氧核苷三磷酸(dNTP),按5’到3’方向复制出互补DNA,即引物的延伸阶段。
上述3步为一个循环,即高温变性、低温退火、中温延伸3个阶段。
从理论上讲,每经过一个循环,样本中的DNA量应该增加一倍,新形成的链又可成为新一轮循环的模板,经过25~30个循环后DNA可扩增106~109倍。
典型的PCR反应体系由如下组分组成:DNA模板、反应缓冲液、dNTP、MgCl2、两条合成的DNA引物、耐热DNA Taq聚合酶。
二、按下列组份配制PCR 反应液TaKaRa LA Taq(5 U/μl)0.5 μl10×LA PCR Buffer II(Mg2+ Plus) 5 μldNTP Mixture(各2.5 mM)8 μl模板DNA(λDNA)* 2.5 ng引物1(10 μM) 2 μl引物2(10 μM) 2 μl灭菌蒸馏水up to 50 μl*【50 μl PCR反应体系中模板DNA 推荐使用量】人基因组DNA 0.1 μg~1 μg大肠杆菌基因组DNA 10 ng~100 ngλDNA 0.5 ng~5 ng质粒DNA 0.1 ng~10 ng三、PCR 反应条件。
以λDNA 为模板,扩增 1 kbp、35 kbp 的DNA片段的PCR 反应条件如下:【1 kbp】95℃ 5 min.95℃30 sec.55℃30 sec. 35 Cycles72℃ 1 min.72℃10 min.【35 kbp】94℃ 5 min.98℃10 sec.68℃15 min. 35 Cycles72℃10 min.常用循环:95℃ 5 min.95℃45 sec.55℃45 sec. 35 Cycles72℃ 2 min.72℃10 min.四、可能出现的问题与解决方案:1、假阴性,不出现扩增条带PCR反应的关键环节有①模板核酸的制备,②引物的质量与特异性,③酶的质量,④PCR循环条件。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
质粒载体的构建摘要:质粒载体的构建。
首先要获得目的DNA。
根据其目的基因序列和启动子序列设计引物,为提高目的基因产率,采用两次PCR的方法,即第一次设计引物扩增全序列基因,第二次设计带酶切位点的引物以第一次扩增产物为模板进行扩增,进而加尾连接到T-DNA上,再利用电转化的方法将连接产物转化到带有PCAMBIA1381的DH5α感受态细胞中复制表达。
关键词:质粒DNA PCR 电泳感受态转化1.引言质粒(plasmid)是细菌或细胞染色质以外的,能自主复制的,与细菌或细胞共生的遗传成分。
其特点如下:①是染色质外的双链共价闭合环形DNA(cccDNA),可自然形成超螺旋结构,不同质粒大小在2-300kb之间,<15kb的小质粒比较容易分离纯化,>15kb的大质粒则不易提取。
②能自主复制,是能独立复制的复制子。
一般质粒DNA复制的质粒可随宿主细胞分裂而传给后代。
③质粒对宿主生存并不是必需的。
某些质粒携带的基因功能有利于宿主细胞的特定条件下生存,例如,细菌中许多天然的质粒带有抗药性基因,如编码合成能分解破坏四环素、氯霉素、氨芐表霉素等的酶基因,这种质粒称为抗药性质粒,又称R质粒,带有R质粒的细菌就能在相应的抗生素存在生存繁殖。
所以质粒对宿主不是寄生的,而是共生的。
现在分子生物学使用的质粒载体都已不是原来细菌或细胞中天然存在的质粒,而是经过了许多的人工的改造。
从不同的实验目的出发,人们设计了各种不同的类型的质粒载体。
质粒载体pBR322是研究得最多,是使用最早且应用最广泛的大肠杆菌质粒载体之一。
符号质粒载体pBR322中的“p代表质粒;“BR”代表两位两位研究者Bolivar和Rogigerus姓氏的字首,“322”是实验编号。
质粒载体pBR322的大小为4361bp,相对分子质量较小的是它第一个优点。
优点之二是它带有一个复制起始位点,保证了该质粒只在大肠杆菌的细胞中行使复制的功能。
具有两种抗生素抗性基因,可供转化子的选择标记是它的第三个优点。
质粒载体pBR322的第四个优点是具有较高的拷贝数,经过氯霉素扩增以后,每个细胞中可累积1000-3000份拷贝,该特性为重组体DNA的制备提供了极大的方便。
构建质粒载体所用的方法基本上是分子克隆技术,是在分子水平上提供一种纯化和扩增特定DNA片段的方法。
常含有目的基因,用体外重组方法将它们插入克隆载体,形成重组克隆载体,通过转化与转导的方式,引入适合的寄主体内得到复制与扩增,然后再从筛选的寄主细胞内分离提纯所需的克隆载体,可以得到插入DNA的许多拷贝,从而获得目的基因的扩增。
2. 材料方法2.1目的DNA的获得2.1.1 引物设计第一次引物设计:正向引物:sinn3F 冰盒标注:P2a引物序列:5’—AAGCAAAATCTAACCGTGTAATGTA—3’引物长度:25bp反向引物:sinn3R 冰盒标注:P2b引物序列:5’—GCAAGAGCGTCGTTTGTAGTTA—3’引物长度:22bp第二次引物设计(带酶切位点):正向引物:sinn3FE 冰盒标注:P2c引物序列:5’—ACTGGATCC AAGCAAAATCTAACCGTGTAATGTA—3’引物长度:34bp反向引物:sinn3RE 冰盒标注:P2d引物序列:5’—TCACTGCAGCTCATAAGACCAAAGAACGTTTCTT—3’引物长度:34bp2.1.2 PCR扩增2.1.2.1 PCR技术的基本原理PCR即聚合酶链式反应,是指在DNA聚合酶的催化下,以母链DNA为模板,以特定引物为延伸起点,通过变性、延伸、复性等步骤,体外复制出与母链模板DNA互补的子链DNA 的过程.是一项DNA体外合成放大技术,可用于基因分离克隆,序列分析,基因表达调控,基因多态性研究等许多方面.PCR反应五要素:参加PCR反应的物质主要有五种即引物、酶、dNTP、模板和Mg2 PCR反应的基本步骤:1.变性: 高温使双链DNA解离成单链.(94℃ 30s)2.退火:低温下引物与模板DNA互补区结合形成杂交分子.(55℃ 30s)3.延伸:中温延伸. 在DNA聚合酶、dNTP、Mg2+存在下, DNA聚合酶催化以引物为起始点的DNA链5’向3’方向的延伸,合成出与模板DNA链互补的DNA子链.(70-72℃30-60s)以上三个步骤为一循环,每一循环的产物均可作为下一循环的模板,经过n次循环后,目的DNA以2n形式增加.2.1.2.2 PCR检测PCR反应扩增出了高的拷贝数,下一步检测就成了关键。
荧光素(溴化乙锭,EB)染色琼脂糖凝胶电泳是最最常用的检测手段。
电泳法检测特异性是不太高的,因此引物两聚体等非特异性的杂交体很容易引起误判。
但因为其简捷易行,成为了主流检测方法2.1.2.3 PCR反应体系第一次PCR扩增:调整PCR反应体系中各成份、组成及反应条件,经多次反复试验结果得出下面的体系可得到清晰明亮的目的DNA电泳条带(1kb): PCR反应体系2*100ul(每管25ul,共8管)Pyrobest 2ulbuffer(10x) 10ulWT4(模板) 2ulP2a(10uM) 4ulP2b(10uM) 4uldNTP(2.5x) 2ulddH2O 76ulPCR反应条件:94℃ 5min30 cycles: 94℃ 40s53℃ 40s72℃ 2min30s72℃ 10min4℃ forever将扩增产物进行琼脂糖凝胶电泳(方法见2.1.3电泳),切胶回收目的DNA条带,共切3管,分别标注A、B、C.首先用25ul约60℃的无菌蒸馏水洗脱A管一次,洗脱液标注1,分别用15ul的无菌蒸馏水洗脱B管和C管,得到的DNA洗脱液标注2,最后各用30ul的无菌蒸馏水洗脱A、B、C管,得到的DNA洗脱液分别标注a、b、c.第二次PCR扩增(带酶切位点):分别用DNA的洗脱液2、a、b、c为模板,进行第二次PCR扩增,设计反应体系(各25ul)如下:模板2 模板a 模板b 模板c Pyrobest 0.5ul 0.5ul 0.5ul 0.5ulbuffer(10x) 2.5ul 2.5ul 2.5ul 2.5ul模板 0.5ul 2ul 4ul 6ul P2a(10uM) 1ul 1ul 1ul 1ulP2b(10uM) 1ul 1ul 1ul 1ul dNTP(2.5x) 0.5ul 0.5ul 0.5ul 0.5ul ddH2O 19ul 17.5ul 15.5ul 13.5ul PCR反应条件:94℃ 5min30 cycles: 94℃ 40s54℃ 40s72℃ 2min30s72℃ 10min4℃ forever电泳结果表明:以2管和c管为模板可得到明显的目的基因条带,依据此次电泳结果,可先后再利用PCR扩增(各100ul,每管25ul,共8管)PCR反应体系2*100ul(每管25ul,共8管)模板2 模板cPyrobest 2ul 2ulbuffer(10x) 10ul 10ul模板 4ul 40ulP2a(10uM) 4ul 4ulP2b(10uM) 4ul 4uldNTP(2.5x) 2ul 2ulddH2O 74ul 38ulPCR反应条件:94℃ 5min30 cycles: 94℃ 40s54℃ 40s72℃ 2min30s72℃ 10min4℃ forever电泳条带明显而且明亮,切胶回收保存待用.。
2.1.3 电泳2.1.3.1 配胶①配制适量的电泳及制胶用的缓冲液(1X TAE Buffer)②根据制胶量和凝胶浓度,准确称量琼脂糖粉(1g),加入适当的锥形瓶中。
③加入一定量的电泳缓冲液(1X TAE Buffer 100ml)。
④熔化完全,冷却至600C左右,加入EB5-7ul,充分混匀。
⑤将溶液倒入制胶模中,之前应在适当位置插上梳子。
⑥室温下凝固,不立即使用时,可用保鲜膜将凝胶包好后放40C保存,一般可保存2-5天。
2.1.3.2 电泳取适量的PCR产物与适量的溴酚蓝混合混匀后加入凝胶槽中,另取适量的DNA Maker 加入右边槽中,开始电泳。
2.1.3.3 紫外观察电泳条带当溴酚蓝跑到适当位置时,在紫外光下观察目的基因的电泳条带(1kb处)2.1.4 切胶回收目的DNA切胶回收目的DNA的方法步骤:操作流程见右图,全套操作约需30分钟,详细说明如下。
1.使用TAE缓冲液或TBE缓冲液制作琼脂糖凝胶,然后对目的DNA进行琼脂糖凝胶电泳。
2.在紫外灯下切出含有目的DNA的琼脂糖凝胶,用纸巾吸尽凝胶表面的液体。
此时应注意尽量切除不含目的DNA部分的凝胶,尽量减小凝胶体积,提高DNA回收率。
注)切胶时请注意不要将DNA长时间暴露于紫外灯下,以防止DNA损伤。
3.切碎胶块。
胶块切碎后可以加快操作步骤6的胶块融化时间,提高DNA的回收率。
4. 称量胶块重量,计算胶块体积。
计算胶块体积时,以1 mg=1 μl 进行计算。
5. 向胶块中加入胶块融化液DR-I Buffer ,DR-I Buffer 的加量如下表:6. 均匀混合后75℃加热融化胶块(低熔点琼脂糖凝胶只需在45℃加热)。
此时应间断振荡混合,使胶块充分融化(约6~10分钟)。
注)胶块一定要充分融化,否则将会严重影响DNA 的回收率。
7. 向上述胶块融化液中加入DR-I Buffer 量的1/2体积量的DR-II Buffer ,均匀混合。
当分离小于400 bp的DNA 片段时,应在此溶液中再加入终浓度为20%的异丙醇。
8. 将试剂盒中的Spin Column 安置于Collection Tube上。
9. 将上述操作7的溶液转移至Spin Column 中,3,600 rpm离心1分钟(如Spin Column 中有液体残留,可适当提高离心速度,再离心1分钟),弃滤液。
注)如将滤液再加入Spin Column 中离心一次,可以提高DNA 的回收率。
10. 将500 μl 的Rinse A 加入Spin Column 中,3,600 rpm离心30秒,弃滤液。
11. 将700 μl 的Rinse B 加入Spin Column 中,3,600 rpm离心30秒,弃滤液。
12. 重复操作步骤11,然后12,000 rpm 再离心1分钟。
凝胶浓度DR-I Buffer 使用量 1.0%3个凝胶体积量 1.0%~1.5%4个凝胶体积量 1.5%~2.0%5个凝胶体积量13.将Spin Column安置于新的1.5 ml的离心管上,在Spin Column膜的中央处加入25 μl的60℃水或洗脱液,室温静置1分钟。
14.12,000 rpm离心1分钟洗脱DNA。
2.1.5 目的DNA加尾用25ul的无菌蒸馏水洗脱DNA,再用20ul的无菌蒸馏水洗脱,共得约40ul的洗脱液,然后抽真空使其浓缩至约8ul。
