真核生物基因结构的预测分析
真核生物的基因组结构与功能分析

真核生物的基因组结构与功能分析真核生物是指在生命进化过程中逐渐形成的一类生物,其基本特征之一是存在真核细胞核。
真核生物的基因组结构较为复杂,包含多个线性染色体和一些质粒。
对基因组结构的分析与理解,对于揭示其生物功能和进化机制是至关重要的。
一、真核生物的基因组结构真核生物的基因组大小较大,同一物种不同个体之间的基因组大小存在较大的差异。
基因组大小与细胞大小和复杂度之间存在着类似关联性。
人类基因组大小约为3亿个碱基对,其中蛋白编码基因仅占大约2%。
真核生物的基因组在基本结构上与细菌大相径庭,主要包括以下几个方面。
1. 染色体染色体是真核生物中最重要、最基本的遗传物质,是基因在生物体内的物质传递介质,是遗传信息的载体。
在精细结构上,真核细胞中存在很多复杂的染色体结构,如核小体、类固醇激素受体、平衡染色体等。
2. 基因组复制真核生物的基因组复制主要包括原核生物和真核生物的不同模式,其中原核生物中存在着DNA单线复制机制,而真核生物则采用DNA复制机器进行自我复制。
与原核生物不同的是,真核生物的DNA复制机器必须满足染色体的线性特性和复杂的三维结构,包括多个酶和蛋白质。
3. 基因只读基因只读是指通过读取基因组中的基因序列,进而达到生物高效功能表达和调节的过程。
真核生物基因组的序列阅读具有高度异质性,不同物种、不同个体之间存在大量的序列差异,这在一定程度上阻碍了对真核生物的功能研究。
二、真核生物的基因组功能分析真核生物的基因组分析主要包括以下几个方面。
1. 蛋白编码基因预测蛋白编码基因是真核生物基因组的重要组成部分,对真核生物的基因组进行蛋白编码基因预测,可以揭示其生物功能和进化机制。
目前,已经建立了多种基于序列、结构、相对位置等的蛋白编码基因预测算法与工具,如Glimmer、InterProScan、Pfam等。
2. 生物信息分析真核生物的基因组分析需要大量的计算资源和分析工具,这就需要借助生物信息学的手段来实现。
蛋白质结构与功能分析
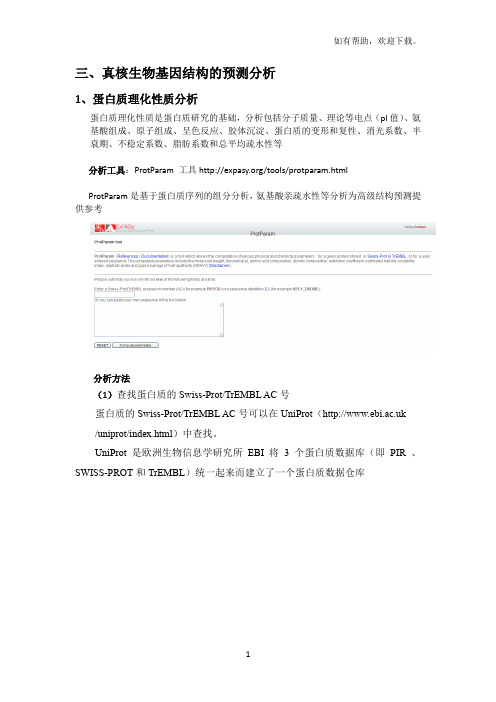
三、真核生物基因结构的预测分析1、蛋白质理化性质分析蛋白质理化性质是蛋白质研究的基础,分析包括分子质量、理论等电点(pI值)、氨基酸组成、原子组成、呈色反应、胶体沉淀、蛋白质的变形和复性、消光系数、半衰期、不稳定系数、脂肪系数和总平均疏水性等分析工具:ProtParam 工具/tools/protparam.htmlProtParam是基于蛋白质序列的组分分析,氨基酸亲疏水性等分析为高级结构预测提供参考分析方法(1)查找蛋白质的Swiss-Prot/TrEMBL AC号蛋白质的Swiss-Prot/TrEMBL AC号可以在UniProt( /uniprot/index.html)中查找。
UniProt是欧洲生物信息学研究所EBI 将3个蛋白质数据库(即PIR 、SWISS-PROT和TrEMBL)统一起来而建立了一个蛋白质数据仓库在搜索框输入蛋白质名称(如Pichia pastoris Agglutinin-like protein 3)→Find(2)如果需要分析的蛋白是SWISS-PROT和TrEMBL数据库中已收录的蛋白质,则在输入蛋白质的Swiss-Prot/TrEMBL AC号(accession number)→点击“Compute parameters”(3)如果需要分析的是未知序列,则需在搜索框中粘贴氨基酸序列,返回结果即可得出结果分析:2、跨膜区分析使用工具:TMpredTMpred,它依靠一个跨膜蛋白数据库Tmbase(Hofmann和Stoffel,1993)。
Tmbase来源与Swiss-Prot库,并包含了每个序列的一些附加信息:跨膜结构区域的数量、跨膜结构域的位置及其侧翼序列的情况。
Tmpred利用这些信息并与若干加权矩阵结合来进行预测。
分析方法Tmpred的Web界面十分简明。
用户将单字符序列输入查询序列文本框,并可以指定预测时采用的跨膜螺旋疏水区的最小长度和最大长度。
《基于序列能量和结构信息的原核生物与真核生物启动子预测》范文

《基于序列能量和结构信息的原核生物与真核生物启动子预测》篇一一、引言原核生物和真核生物作为生命世界中的两大生物类型,各自在进化、发育、代谢等许多生物学方面具有显著的差异。
特别是在基因调控领域,这两种生物类型的启动子(promoter)结构与功能存在显著的差异。
随着生物信息学和计算生物学的发展,利用序列能量和结构信息来预测启动子已成为一种有效的方法。
本文将详细介绍基于序列能量和结构信息的原核生物与真核生物启动子预测的原理和方法。
二、序列能量与启动子预测(一)序列能量分析启动子是基因转录调控的重要区域,通常由DNA序列组成,含有特定模式的信息以指导转录因子的结合。
通过计算序列的能量变化,我们可以了解序列的稳定性和转录因子的结合能力。
在启动子预测中,我们主要关注的是序列的能量分布和变化规律。
(二)启动子预测基于序列能量的分析,我们可以对启动子进行预测。
通常,具有较低能量的序列更稳定,更有可能成为启动子的一部分。
通过比较已知的启动子序列的能量分布模式,我们可以对未知序列进行预测。
此外,还可以利用机器学习等方法建立预测模型,提高预测的准确度。
三、结构信息与启动子预测(一)结构信息分析除了序列能量外,DNA的结构信息也是启动子预测的重要依据。
DNA的结构包括双螺旋结构、碱基堆积、超螺旋等,这些结构可能影响转录因子的结合和基因的表达。
通过分析DNA的结构信息,我们可以更好地理解启动子的功能和作用机制。
(二)结构信息在启动子预测中的应用结合DNA的结构信息,我们可以更准确地预测启动子的位置和功能。
例如,通过分析DNA的弯曲程度、碱基堆积等结构特征,我们可以确定转录因子结合的位点,从而预测出可能的启动子区域。
此外,还可以利用三维结构模型等手段,进一步验证和优化预测结果。
四、原核生物与真核生物启动子的预测比较(一)原核生物与真核生物启动子的差异原核生物和真核生物的启动子在结构和功能上存在显著的差异。
原核生物的启动子通常较短,含有特定的转录因子结合位点;而真核生物的启动子则较为复杂,包含多个调控元件和辅助元件。
真核生物染色体基因组的结构和功能

真核生物染色体基因组的结构和功能真核生物的基因组一般比较庞大,例如人的单倍体基因组由3×106bp硷基组成,但人细胞中所含基因总数大概会超过3万个。
这就说明在人细胞基因组中有许多DN A序列并不转录成mR NA用于指导蛋白质的合成。
研究发现这些非编码区往往都是一些大量的重复序列,这些重复序列或集中成簇,或分散在基因之间。
在基因内部也有许多能转录但不翻译的间隔序列(内含子)。
因此,在人细胞的整个基因组当中只有很少一部份(约占2-3%)的DNA序列用以编码蛋白质。
真核生物基因组有以下特点。
1.真核生物基因组DNA与蛋白质结合形成染色体,储存于细胞核内,除配子细胞外,体细胞内的基因的基因组是双份的(即双倍体,diploi d),即有两份同源的基因组。
2.真核细胞基因转录产物为单顺反子。
一个结构基因经过转录和翻译生成一个mRNA分子和一条多肽链。
3.存在重复序列,重复次数可达百万次以上。
4.基因组中不编码的区域多于编码区域。
5.大部分基因含有内含子,因此,基因是不连续的。
6.基因组远远大于原核生物的基因组,具有许多复制起点,而每个复制子的长度较小。
高度重复序列:高度重复序列在基因组中重复频率高,可达百万(106)以上。
在基因组中所占比例随种属而异,约占10-60%,在人基因组中约占20%。
高度重复顺序又按其结构特点分为三种(1)反向重复序列这种重复顺序约占人基因组的5%。
反向重复序列由两个相同顺序的互补拷贝在同一DNA链上反向排列而成。
变性后再复性时,同一条链内的互补的拷贝可以形成链内碱基配对,形成发夹式或“+”字形结构。
反向重复间可有一到几个核苷酸的间隔,也可以没有间隔。
没有间隔的又称回文结构,这种结构约占所有反向重复的三分之一。
课件第8讲 基因预测方法
组(12.1Mb)的2/5
2、绝大部分原核生物基因组由一个单一的 环状DNA分子组成; 3、原核生物的基因通常比真核生物的少;
E. coli:4000多个基因,人:~30000个
4、原核生物的基因绝大多数是连续基因, 不含间隔的内含子;基因组结构紧密,重 复序列远少于真核生物的基因组。
著名原核基因预测软件
1、GeneMark系列软件(包括最新版本GeneMarkS)
Borodovsky等,1993~2001
——Borodovsky, M. and McIninch. J. (1993) GENMARK: parallel gene recognition for both DNA strands. Comput. Chem., 17, 123-134. ——Besemer, J., Lomsadze, A. and Borodovsky, M. (2001) GeneMarkS: a self-training method for prediction of gene starts in microbial genomes genomes. Implications for finding sequence motifs in regulatory regions. Nucleic Acids Res., 29: 2607-2618.
二、 原核生物基因组中的基因预测
• 原核基因预测概述 • 预测算法举例:MED原核基因预测方法 • 预测性能的评价
• ForCon:核酸与氨基酸不同序列格式之间的 转换
3
2011/11/21
(一)原核基因预测概述
第六章 真核生物的遗传分析

链孢霉的特点是它的四分体是顺序排列的。
不仅减数分裂的四个产物在子囊中仍连在 一起,而且代表减数分裂四个染色单体的子囊 孢子是直线排列的,排列的顺序跟减数分裂中 期板上染色单体的定向相同。
因此,我们用遗传学方法可以区分每个染 色单体及其基因型,而用细胞学检查方法是办 不到的。
四分体遗传分析的特殊意义:
接着在每条产囊菌丝中都发生下列过程: ①由每种交配型的一个核共同形成子囊原始细胞, ②这两个核在伸长的细胞中融合成二倍体细胞核; ③二倍体细胞核立即进行减数分裂; ④减数分裂的四个产物再进行一次有丝分裂,在一个
子囊中形成四对子囊孢子。 同时,其他菌丝形成了一个厚壁包围着产囊菌丝,构
成长颈瓶状的子囊壳。
的特异的碱基序列(单拷贝)的长度(或核苷数)之和来表示 复杂度(的大小) 。
DNA分子中无重复的核苷酸序列的最大长度.
病毒或细菌的基因组无重复序列,其基因组的复杂度与 C值(即基因组的大小)相等。
四、真核生物基因组DNA序列的复杂度
DNA复性动力学研究结果表明,真核生物基因组序列大致 可分为3种类型: 1、单拷贝序列(非重复序列):每个基因只有1-2个 拷贝。 2、中度重复序列:平均长度300bp,重复次数10-102。 3、高度重复序列:通常为6-200bp,重复次数在106。
第二次分裂分离: + - + - +--+
-++-
-+-+
每一个第二次分裂分离的子囊是供试位点与着丝点 之间发生一次交换的结果。
根据这种特殊情况,就有可能计算某一位点和着丝点之间的重组百分率。 重组百分率的标准公式如下:
A位点和着丝点之间重组 染色单体数 染色单体总数
100
交换值 (%)
重组型配子数 总配子数
真核生物基因结构的预测分析-HE

http://pbil.univlyon1.fr/software/cpgprod_query.html
27
CpG岛的预测:CpGPlot
/emboss/cpgplot/index.html
参数选项
提交序列
基因结构及基因预测

§6.1
高等真核生物 基因结构与基因预测简介
§6.1.1 真核生物的基因结构
1 基因(gene)的概念
基因的概念随着科学的发展而不断发展,迄今为止,仍有各种 说法。
Today when we speak of a gene for some malady, a regulatory gene, a structural gene, or a gene frequency, it is entirely possible that we are deploying different gene concepts even though we are using the same term.
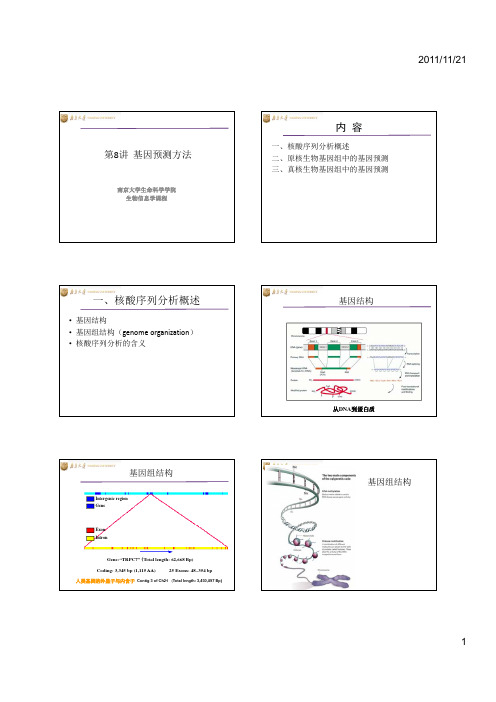
人类结构基因的结构示意图
人类结构基因的结构示意图
Contig 3 of Ch21
(Total length: 3,450,497 Bp)
Intergenic region Gene Exon Intron
Gene:“TRPC7” (Total length: 62,668 Bp)
Coding: 3,345 bp (1,115 AA)
人(Homo sapiens)的基因组:
平均每个基因包含内含子4.0个(最多的是116个),外显子5.0 个,每1kb的CDS平均含有5.3个内含子,是这10种真核生物中 内含子数目最多、长度最大的。内含子的平均长度为3413.1bp, 其中大多数为75~150bp,已知最长的内含子要大于100kb。 每1kb的CDS所包含的内含子长度为6825bp。同样地,人类基 因组外显子长度的概率分布要比内含子的概率分布要紧凑得多。
6 与转录有关的调控信号
(1)、启动子(promoter) (2)、增强子(enhancer) (3)、负性调节元件 (4)、LCR(Locus control regions)(基因座调控区) (5)、转录因子 (6)、与转录终止有关的序列: (7)、mRNA的剪接
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
10
ORF识别: GenomeScan
/genomescan.html
提交待分析序列
提交同源蛋白质序列
11
运行GenomeScan
GenomeScan输出结果:文本
预测外显子位臵、可 信度等信息
同源比 对信息
预测结果的氨基酸序列
12
GenomeScan输出结果:图形
22
DBTSS搜索结果
23
FXYD5基因的启动子区域显示
SNP位点
覆盖的cDNA序列数目 转录起始位点TSS
DBTSS图例说明
24
ALB基因的启动子区域显示
TF:转录 因子结合 位点
覆盖的cDNA序列数目
转录起始位点TSS
25
下载启动子序列
下载启动子区序列 设臵下载序列的起点、终点
需选择转录起始位点
成熟mRNA
5’
AAUAAA
CAAAAAAAAAAAAA
3’
下游作用元件:GC rich二重对称区、UUUUUU
RNA 5’
UUUUUUUUU C-G C-G G-C G-C U-A G-C G-C C-G G-C
3’
31
转录终止信号polyA预测:POLYAH
/berry.phtml?topic=polyah&group=programs &subgroup=promoter
13
课堂练习
• 1使用GENESCAN预测序列中可能的ORF。 • 2使用GENOMESCAN预测序列中可能的 ORF。
• 练习用的序列文件在c:\zcni\shixi2文件下, 名字为clone.fasta,使用写字板打开查看。
14
转录调控序列分析
15
启动子区结构
启动子(Proபைடு நூலகம்oter)
位于结构基因5’端上游,能活化RNA聚合酶,使之与模板 DNA结合并具有转录起始的特异性。 转录起始位点(Transcription start site, TSS)
PYCAPY(嘧啶)
核心启动子元件(Core promoter element) TATA box,Pribnow box (TATAA)
上游启动子元件(Upstream promoter element,UPE)
CAAT box,GC box,SP1,Otc
增强子(Enhancer)
16
Fgenes FgeneSV Generation FGENESB GenomeScan GeneWise2
Softberry Softberry ORNL Softberry MIT EBI
人(基因结构) 病毒 原核 细菌(基因结构) 脊椎、拟南芥、玉米 人 7 人、小鼠、拟南芥、果蝇
GRAIL
/grailexp/
CpG Island 分析
CpG Island CpGPlot CpG finder CpGi130 CpGproD /cpgislands2/cpg.aspx /emboss/cpgplot/index.html /berry.phtml?topic=cpgfinder& group=programs&subgroup=promoter /CpG130.do http://pbil.univ-lyon1.fr/software/cpgprod_query.html Web Web Web web web
19
PromoterScan输出结果
找到的TATA box和转录起始位点
预测可能的转录因子
转录因子在提交序列中的位臵
20
转录起始位点数据库:DBTSS
http://dbtss.hgc.jp/
21
DBTSS搜索工具条
限定物种“H. sapiens” 最新数据库版本加入Solexa测序新数据支持 限定搜索“基因名称” 搜索基因“FXYD5” 限定至少需要多少条cDNA序列覆盖
ORNL
ORF识别:GENSCAN
选择物种类型
/GENSCAN.html
是否显示非最优外显子 序列名称(可选) 显示氨基酸或CDS序列 提交序列文件
提交序列
结果返回到邮箱(可选)
8
运行GENSCAN
GENSCAN输出结果:文本
9
GENSCAN输出结果:图形
如何分析mRNA/cDNA的外显子组成?
通过对特征序列(GT-AG)的分析进行直 接的预测基因预测软件(NetGene2)
与相应的基因组序列比对,分析比对片 段的分布位臵(Spidey)
35
36
剪切位点识别:NetGene2
http://www.cbs.dtu.dk/services/NetGene2/
BLAST比对到的三条mRNA序列
40
Spidey序列提交页面
输入基因组序列或序 列数据库号
输入相似性序列 判断用于分析的序列间的差异, 并调整比对参数 比对阈值 不受默认内含子长度限 制, 默认长度:内部内含子 为35kb, 末端内含子为 100kb 输出格式选择
选择物种
41
Spidey输出结果
原核和真核生物基因转录起始位点上游区 结构
原核生物
-35 -10 +1 mRNA
TTGACA
TATAAT
A
真核生物
-110 -40 -25 +1
mRNA
GC区
增强子
CAAT区
TATAAT
PyCAPy
上游启动子元件,UPE
核心启动子元件
转录起始 位点
17
启动子结合位点分析常用软件
PromoterScan Promoser Neural Network Promoter Prediction Softberry: BPROM, TSSP, TSSG, TSSW MatInspector
:80/molbio/proscan/ /zlab/PromoSer/ /seq_tools/promoter.html /berry.phtml?topic=index&gr oup=programs&subgroup=promoter http://www.gene-regulation.de/ Web Web Web Web Web
提交序列 提交序列文件
32
POLYAH输出结果
GENESCAN预测结果 PolyA位点52398bp
polyA位臵
33
课堂练习
• 使用CpG plot预测clone.fasta中的CpG 岛。 • 使用POLYAH预测clone.fasta中的POLYA 剪切位点。
34
内含子/外显子剪切位点识别
5
开放读码框的识别
• 开放读码框(open reading frame, ORF) 是一段起始密码子和终止密码子之间的碱基序列 • ORF 是潜在的蛋白质编码区
6
基因开放阅读框/基因结构分析识别工具
ORF Finder BestORF GENSCAN Gene Finder FGENESH GeneMark GLIMMER /gorf/gorf.html /berry.phtml?topic=bestorf& group=programs&subgroup=gfind /GENSCAN.html /tools/genefinder/ /berry.phtml?topic=fgenesh &group=programs&subgroup=gfind /GeneMark/eukhmm.cgi /genomes/MICROBES/gli mmer_3.cgi /software/glimmer /berry.phtml?topic=fgenes& group=programs&subgroup=gfind /berry.phtml?topic=virus&gr oup=programs&subgroup=gfindv /generation/ /berry.phtml?topic=fgenesb &group=programs&subgroup=gfindb /genomescan.html /Wise2/ NCBI Softberry MIT Zhang lab Softberry GIT Maryland 通用 真核 脊椎、拟南芥、玉米 人、小鼠、拟南芥、酵母 真核(基因结构) 原核 原核
真核生物基因的主要结构
4
基因结构分析常用软件
开放读码框 GENSCAN GENOMESCAN CpG岛 转录终止信号 启动子/转录起始位点 CpGPlot
基因结构分 析
POLYAH PromoterScan DBTSS database NETGENE2
mRNA剪切位点 Spidey 选择性剪切 ASTD
实习二 真核生物基因结构的预 测分析
浙江加州国际纳米技术研究院 2009年11月
1
课程内容
实习一 实习二 基因组数据注释和功能分析 真核生物基因结构的预测分析
基因组学 系 统 生 物 学
实习三
实习四 实习五 实习六
芯片的基本数据处理和分析
蛋白质结构与功能分析 蛋白质组学数据分析
转录物组学
蛋白质组学
RSAT
Cister
http://rsat.ulb.ac.be/rsat/
/~mfrith/cister.shtml
Web
Web
18
启动子预测:PromoterScan
/molbio/proscan/
提交序列
系统生物学软件实习
2
基因组功能分析
基因组序列 cDNA序列
翻译
编码区预测
基因结构分析
蛋白质序列
