BLAST 和 FASTA 的应用
blast参数

blast参数1. 什么是blast参数?BLAST(Basic Local Alignment Search Tool)是一种常用的生物信息学工具,用于比对两个或多个生物序列,以寻找相似性和同源性关系。
在进行BLAST比对时,我们可以设置一些参数来调整比对的敏感性和特异性,以便更好地满足研究的需求。
2. BLAST参数的分类BLAST参数可以分为两类:搜索参数(search parameters)和输出参数(output parameters)。
2.1 搜索参数搜索参数用于控制BLAST比对的敏感性和速度。
常见的搜索参数包括:•-query:指定查询序列文件或序列字符串,可以是FASTA格式或GenBank 格式。
•-subject:指定被比对序列文件或序列字符串,可以是FASTA格式或GenBank格式。
•-task:指定比对任务的类型,如blastn、blastp、blastx等。
•-evalue:设置期望值(E-value)的阈值,用于筛选显著的比对结果。
•-word_size:设置比对时使用的单词大小,影响比对的敏感性和速度。
•-gapopen:设置开启一个gap的惩罚分数。
•-gapextend:设置扩展一个gap的惩罚分数。
2.2 输出参数输出参数用于控制BLAST比对结果的格式和内容。
常见的输出参数包括:•-outfmt:设置输出格式,如0表示默认格式,5表示XML格式。
•-out:指定比对结果的输出文件。
•-max_target_seqs:设置返回的最大比对序列数目。
•-num_threads:设置线程数,用于加速比对过程。
•-num_alignments:设置返回的最大比对结果数目。
•-max_hsps:设置每个比对结果返回的最大高分片段数目。
3. 如何选择合适的blast参数?选择合适的blast参数是进行BLAST比对的关键步骤,以下是一些选择参数的建议:3.1 根据比对任务类型选择参数不同的比对任务类型需要使用不同的参数。
生物信息学中的序列比对算法及评估指标比较

生物信息学中的序列比对算法及评估指标比较序列比对是生物信息学中非常重要的工具之一,用于分析和比较生物序列的相似性和差异。
序列比对是理解生物进化和功能注释的关键步骤,在基因组学、蛋白质学和遗传学等领域都有广泛应用。
本文将介绍序列比对的算法原理和常用的评估指标,并对几种常见的序列比对算法进行比较。
一、序列比对算法1.全局比对算法全局比对算法用于比较整个序列的相似性,常见的算法有Needleman-Wunsch 算法和Smith-Waterman算法。
这两种算法都是动态规划算法,其中Needleman-Wunsch算法用于比较两个序列的相似性,而Smith-Waterman算法用于寻找局部相似的片段。
这些算法考虑了序列的整体结构,但在处理大规模序列时计算量较大。
2.局部比对算法局部比对算法用于找出两个序列中最相似的片段,常见的算法有BLAST (Basic Local Alignment Search Tool)算法和FASTA(Fast All)算法。
这些算法以快速速度和高敏感性著称,它们将序列切割成小的段落进行比对,并使用统计模型和启发式搜索来快速找到最佳匹配。
3.多序列比对算法多序列比对算法用于比较多个序列的相似性,常见的算法有ClustalW和MAFFT(Multiple Alignment using Fast Fourier Transform)算法。
这些算法通过多次序列比对来找到共有的特征和区域,并生成多序列的一致性描述。
二、评估指标1.一致性分数(Consistency Score)一致性分数是衡量序列比对结果一致性的指标,它反映了序列比对的精确性和准确性。
一致性分数越高,表示比对结果越可靠。
常用的一致性分数有百分比一致性(Percentage Identity)和序列相似度(Sequence Similarity)。
2.延伸性(Extension)延伸性是衡量序列比对结果的长度的指标。
生物信息研究中的序列对齐与比对算法研究

生物信息研究中的序列对齐与比对算法研究序列对齐与比对算法在生物信息研究中扮演着至关重要的角色。
生物信息学是一门研究生物大分子之间的相似性和差异性的学科,它涉及到生命科学、计算机科学和统计学等多个领域的交叉。
序列对齐是生物信息学中的一项基础工作,旨在寻找和比较两个或多个生物序列(如DNA、RNA或蛋白质序列)之间的相似性和差异性。
本文将介绍序列对齐的基本原理、常用算法以及其在生物信息研究中的应用。
首先,我们来解释一下序列对齐的基本概念。
在生物学中,序列是指基因组中的碱基序列或蛋白质中的氨基酸序列。
序列对齐是将两个或多个序列进行比对,并找到它们之间的相似性和差异性的过程。
序列对齐通常分为全局对齐和局部对齐两种类型。
全局对齐旨在比较整个序列,而局部对齐则重点关注序列中的一部分区域。
序列对齐可以揭示生物分子的进化关系、功能预测以及寻找序列中的共同特征。
序列对齐的方法有多种,其中最常用的算法是Smith-Waterman算法和Needleman-Wunsch算法。
Smith-Waterman算法是一种局部序列比对算法,它通过构建一个得分矩阵,并根据得分矩阵找到两个序列中最佳的相似区域。
Needleman-Wunsch算法是一种全局序列比对算法,它通过动态规划的方法,建立一个得分矩阵,并找到两个序列中的最佳匹配。
这些算法都是基于动态规划的思想,通过寻找最优的对齐方案来确定序列的相似性。
除了Smith-Waterman和Needleman-Wunsch算法,还有一些其他的序列比对算法,如BLAST算法和FASTA算法。
BLAST算法是一种常用的快速比对算法,它通过将查询序列与数据库中的序列进行比对,找到最相似的序列。
FASTA算法也是一种常用的快速比对算法,它通过构建一个特殊的索引,加速序列的比对过程。
这些比对算法的不同之处在于其运行速度、准确性和适用范围。
序列对齐和比对算法在生物信息研究中有着广泛的应用。
首先,它们可以用来研究物种的进化关系。
BLAST使用方法

BLAST使用方法一、BLAST的安装和准备工作2.获取待比对的序列文件,可以是FASTA格式的DNA或蛋白质序列。
二、BLAST的常用参数和选项1. Program:指定使用哪种BLAST程序(如BLASTn、BLASTp等)。
2. Database:指定使用哪个数据库进行比对。
3. Query:指定待比对的序列文件。
4. E-value:期望值。
一种描述比对结果误差率的指标,值越小表示结果越可信。
通常情况下,E-value小于0.01被认为是显著结果。
5. Word size:BLAST在比对时使用的核心词的长度。
长度越大表示查全率(sensitivity)越高,但速度会减慢。
6. Gap open:允许在比对过程中插入空位(如插入一个碱基)。
Gap open参数定义了开放一个空位的惩罚分数。
7. Gap extension:允许空位的延伸。
Gap extension参数定义了延伸一个空位的惩罚分数。
三、使用BLAST进行比对1.命令行方式:-打开命令行界面,并定位到BLAST软件的安装目录。
- 输入命令,指定BLAST程序、数据库、查询文件和其他参数。
例如:blastn -db nt -query query.fasta -out output.txt -evalue 0.01-运行命令,BLAST将开始进行比对并生成结果文件。
2.网页方式(以NCBIBLAST为例):- 打开NCBI网站的BLAST页面()。
-选择需要使用的BLAST程序(如BLASTn、BLASTp等)。
-上传待比对的序列文件,或者粘贴序列文本到输入框中。
-选择适当的数据库和其他参数。
-点击“BLAST”按钮,等待比对完成。
四、解读BLAST结果1. E-value:表示在随机比对中获得与查询序列相似度更高的结果的期望概率。
E-value越小表示比对结果越显著。
2. Bitscore:用于表示比对结果的质量。
Bitscore越高表示比对结果越可信。
BLAST 和 FASTA 的应用
51
/bioinplant/
《生物信息学札记》 樊龙江
K-tuple 的大小可以变化并将间接影响搜索的速度和敏感度。然后程序要对待检 序列和序列库中的所有序列进行处理,找出字典中长度与 K-trple 相等的所有序 列段的位置。比较两个序列的字典要比比较两个序列本身快得多,可以有效地找 出小段相似区。一旦通过初始的快速检索找到一批评分最高的序列,就可以仅对 这些高分序列进行第二轮比较。第二轮的序列比对是采用 Needleman-Wunsch 算 法(1970)进行空位联配计算,得出分析的最后结论。如果 FASTA 运行后找到较好 的相似序列,有时采用较小的 K-tuple 值或换一个评分矩阵重新检索分析,也许 会有帮助。
使用 Smith-Waterman 算法联配比对
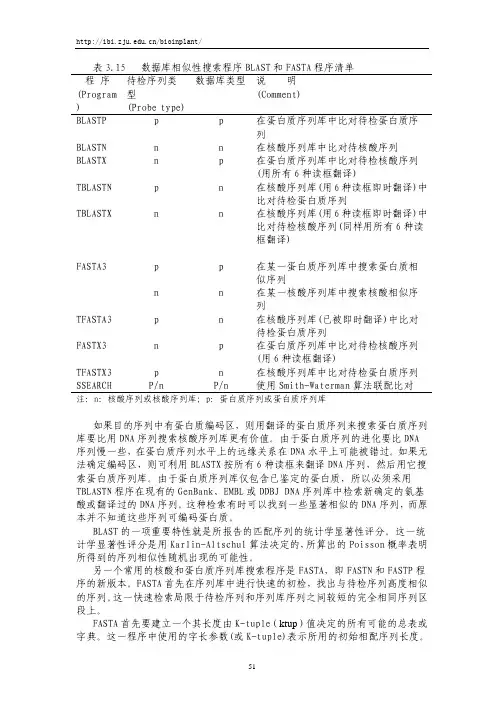
注:n:核酸序列或核酸序列库;p:蛋白质序列或蛋白质序列库
如果目的序列中有蛋白质编码区,则用翻译的蛋白质序列来搜索蛋白质序列 库要比用 DNA 序列搜索核酸序列库更有价值。由于蛋白质序列的进化要比 DNA 序列慢一些,在蛋白质序列水平上的远缘关系在 DNA 水平上可能被错过。如果无 法确定编码区,则可利用 BLASTX 按所有 6 种读框来翻译 DNA 序列,然后用它搜 索蛋白质序列库。由于蛋白质序列库仅包含已鉴定的蛋白质,所以必须采用 TBLASTN 程序在现有的 GenBank、EMBL 或 DDBJ DNA 序列库中检索新确定的氨基 酸或翻译过的 DNA 序列。这种检索有时可以找到一些显著相似的 DNA 序列,而原 本并不知道这些序列可编码蛋白质。
Blast和Fasta的应用与原理

3
生物序列的同源性
同源性: 指从一些数据中推断出的两个基因或蛋 白质序列具而共同祖先的结论,属于质的 判断。就是说A和B的关系上,只有是同 源序列,或者非同源序列两种关系。而说 A和B的同源性为80%都是不科学的。
16
Blast任务提交表单(二)
2.设置各种参数部分
设置搜索的范围,entrez关键词, 或者选择特定物种
一些过滤选项,包括简 单重复序列,人类基因 组中的重复序列等
E值上限 窗口大小 如果你对blast的命令行选项熟悉的话,可以在这里加入更多的参数
17
Blast任务提交表单(三)
3.设置结果输出显示格式 E值范围 选择需要显示的选项 以及显示的文件格式 显示数目 Alignment的显 示方式
12
两种版本的Blast比较(一)
网络版本 包括NCBI在内的很多网站都提供了在线 的blast服务,这也是我们最经常用到的 blast服务。网络版本的blast服务就有方便, 容易操作,数据库同步更新等优点。但是 缺点是不利于操作大批量的数据,同时也 不能自己定义搜索的数据库。
13
两种版本的Blast比较(二)
5
序列相似性比较和序列同源性分析
序列相似性比较: 就是将待研究序列与DNA或蛋白质序列库进行比较, 用于确定该序列的生物属性,也就是找出与此序列相似 的已知序列是什么。完成这一工作只需要使用两两序列 比较算法。常用的程序包有BLAST、FASTA等;
序列同源性分析: 是将待研究序列加入到一组与之同源,但来自不同物种 的序列中进行多序列同时比较,以确定该序列与其它序 列间的同源性大小。这是理论分析方法中最关键的一步。 完成这一工作必须使用多序列比较算法。常用的程序包 有CLUSTAL等;
BLAST 应用专题实践
BLAST 应用专题实践摘要BLAST 采用一种局部的算法获得两个序列中具有相似性的序列。
BLAST中的常用程序有BLASTP(蛋白质序列到蛋白质库的查询)、BLASTX是核酸序列到蛋白库中的一种查询、BLASTN(核酸序列到核酸库中的一种查询)、TBLASTN(蛋白序列到核酸库中的一种查询)、TBLASTX(核酸序列到核酸库中的一种查询)。
BLAST与FASTA是当今最流行的两种比对程序,在生物学中被广泛应用。
本文主要介绍DNA序列比对和蛋白质序列比对。
ABSTRACTBLAST use a partial algorithm to find the similar sequences between two sequences. There are some common used program in the BLAST : BLASTP, BLASTX, BLASTN, TBASTN, TBLASTX. Today, BLAST and FASTA is two of the most popular comparing program, which is widely used in the biology field. This paper mainly introduces the alignment of DNA sequences and protein sequeces.一、前言Blast(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
本文通过使用NCBI中的BLAST比对DNA序列和蛋白质序列,重点介绍了BLAST页面中各项参数的功能。
二、DNA序列比对2.1准备工作进入/Blast.cgi ,点击nucleotide blast 出现“Enter Query Sequence”的文本框,输入需要进行比对的序列,如选择:CCTCCCCCTTTGCTTTTTGCTCTCTTGTTAGTATATTAATTGTTTTCACTC TCTGAATCTTTTTTCCCCATTTCTTTGGCAGACATTTTTACTTGTCTTGGAAGAGTAG GTGAAGAGCTGTTTTTAGGACTCTTTGAAAGGGTACAGTATGGGTGACAGTCT>11462Query subrange 可以从需要比对的序列中截取一段,其中from和to代表起始和中指的位置。
实习报告 FastA和BLAST的使用
实习二BLAST和Fasta的使用
实习目标
1.掌握BLAST的基本功能并可熟练使用
2.了解FastA的基本功能及使用方法
实习内容
1.BLAST
(1).在NCBI中搜索人或动物任一基因的核苷酸序列。
/
(2).用NCBI中的BLAST在线工具搜索与其相匹配的序列。
(3).在EBI中做BLAST找到与所给序列相匹配的序列
/blastall/
*2.Fasta
(1).打开网址/fasta33/进入Fasta 的查询网址(2).浏览并练习Fasta 的查询方法
一个相关的网站:/pages/lab.htm
注:供参考的基因: Myoglobin肌红蛋白,HB血红蛋白,hiv等。
思考题
1.比较两种查询方法的不同与优劣
2.比较在NCBI和EBI中blast查询结果的不同
实习三序列对位显著性检验的计算机模拟
实习目标
1. 通过编写程序更深刻的理解动态规划法的原理步骤
2. 编写显著性检验的程序并输出结果来理解显著性检验的原理
实习内容
1.编写所给序列的局部对位的程序(动态规划法)
2.得出最优分并记录
3.编写显著性检验程序
(1).随机打乱所给两条序列
(2).进行局部对位
(3)反复循环(>10000) 记录结果
4. 求出p-value值并画出得分分布图
注:可能用到的VB语法及函数: 数组, 循环, 判断语句,随机数的产生,文件的读写保存等.
思考题
1.观察随机序列分值的分布情况
2.解释两条序列局部对位分值与相似性的关系。
生物信息学中的序列比对方法
生物信息学中的序列比对方法序列比对是生物信息学中一项非常重要的工具,其主要目的是将两个或更多的DNA、RNA或蛋白质序列进行比较,以找到它们之间的相似性和差异性。
这样的比对可以用来识别基因、预测蛋白质结构、推断进化关系和研究生物系统的复杂性等。
随着DNA测序技术的快速发展,越来越多的生物学家和生物信息学家开始研究序列比对方法。
序列比对是一项复杂而耗时的任务,需要对大量的序列进行计算和分析。
因此,发展高效的序列比对方法对于生物信息学的发展至关重要。
当前,生物信息学界广泛应用的序列比对方法主要包括全局比对、局部比对和多序列比对等。
一、全局比对全局比对是指将整个序列与另一个相似序列进行比对。
它的应用场景通常是在两个相对较短的序列中查找相似片段,以便在进一步的研究中进行详细的分析。
全局比对方法最常用的是Needleman-Wunsch算法和Smith-Waterman算法。
Needleman-Wunsch(NW)算法是第一个被开发出来的全局比对算法。
该算法基于动态编程的思想,通过将整个序列进行比对,计算出最佳匹配的得分和路径。
然而,这种方法的时间复杂度非常高,随着序列长度的增加,其计算成本也会呈指数级增长。
Smith-Waterman(SW)算法是一种优化的全局比对算法,其核心思想与NW算法类似。
不同之处在于SW算法将匹配的得分设置为正数,而将多余的间隔和未匹配的子序列得分设置为负数。
通过这种方式,SW算法可以得到一个全局最佳的比对结果。
然而,该算法的计算成本也比较高,因此其应用场景受到一定的限制。
二、局部比对局部比对是指在比对序列的过程中,只对部分区域进行比对。
与全局比对不同,局部比对更适用于两个序列之间只有一些片段相似的情况。
常用的局部比对方法主要包括BLAST算法和FASTA算法等。
BLAST算法是一种聚集序列算法,它将大量的搜索序列放入一个空间中,通过加速计算找到最匹配的序列。
通过BLAST算法,可以快速搜索数据库中的所有序列,并找到与目标序列相似的匹配。
多序列比对方法
多序列比对方法多序列比对是生物信息学中一个常见的分析方法,用于比较多个序列之间的相似性和差异性。
本文将介绍多序列比对的基本原理、常用方法和软件工具,以及其在生物学研究中的应用。
一、多序列比对的基本原理多序列比对是指对多个生物序列进行比较和分析。
生物序列可以是蛋白质序列、DNA序列或RNA序列等。
多序列比对的主要目的是确定序列之间的保守区域和变异区域,并发现序列之间的结构和功能相关性。
多序列比对的基本原理是通过构建序列之间的相似性矩阵,确定最佳的比对结果。
相似性矩阵用于测量两个序列之间的相似性,通常使用BLOSUM、PAM或Dayhoff矩阵等。
基于相似性矩阵和动态规划算法,可以计算序列之间的最佳比对路径,以及比对的得分。
二、常用的多序列比对方法1. 基于全局比对的方法:该方法适用于序列之间的整体相似性比较,常用的算法有Needleman-Wunsch算法和Smith-Waterman算法。
这两种算法都采用动态规划策略,通过计算各种可能的比对路径来确定最佳比对结果。
全局比对方法的主要缺点是在序列相似性较低的情况下,比对结果可能不准确。
2. 基于局部比对的方法:该方法适用于序列之间的部分相似性比较,常用的算法有BLAST和FASTA。
局部比对方法主要通过搜索局部相似片段来进行比对,可以提高比对的敏感性和准确性。
BLAST和FASTA是两种常用的快速局部比对工具,可以快速比对大规模序列数据库。
3. 基于多重比对的方法:该方法适用于多个序列之间的比较和分析,常用的算法有ClustalW和MAFFT。
多重比对方法通过构建多个序列的比对结果,可以识别序列之间的共同保守区域和变异区域,以及序列的结构和功能相关性。
ClustalW和MAFFT是两种常用的多重比对工具,具有较高的准确性和可靠性。
三、常用的多序列比对软件工具1. ClustalW:ClustalW是一个常用的多重比对软件,主要用于比对蛋白质和DNA序列。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
ATATTTTTATATTTCCTTTATGTGAATTTTTAAGCTGCAAATCTGATGGCCTTAAT
TTCCTTTTTGACACTGAAAGTTTTGTAAAAGAAATCATGTCCATACACTTTGTTGC
AAGATGTGAATTATTGACACTGAACTTAATAACTGTGTACTGTTCGGAAGGGGTTC
《生物信息学札记》 樊龙江
条序列进行常规分析来鉴定它。你可以这样进行: ①复制该序列:
AAAAGAAAAGGTTAGAAAGATGAGAGATGATAAAGGGTCCATTTGAGGTTAGGTAA
TATGGTTTGGTATCCCTGTAGTTAAAAGTTTTTGTCTTATTTTAGAATACTGTGAT
二. BLAST:核苷酸数据库搜索
BLAST 包含有 5 个子程序,它是目前运行速度最快的检索搜索程序。最初的 程序版本(Version1.4)不允许设置空位(gap),这对运行速度的提高有好处。正 如前文所述,空位直接关系到搜索结果,所以目前的 BLAST 版本(Version2.0) 均能进行空位联配。BLAST 的快速得益于它的统计算法:BLAST 使用的是快速局 部而不是缓慢、整体的联配策略。BLAST 不追求整条序列的匹配。 1.BLAST 实战操作(1)
列
BLASTN
n
n
在核酸序列库中比对待核酸序列
BLASTX
n
p
在蛋白质序列库中比对待检核酸序列
(用所有 6 种读框翻译)
TBLASTN
p
n
在核酸序列库(用 6 种读框即时翻译)中
比对待检蛋白质序列
TBLASTX
n
n
在核酸序列库(用 6 种读框即时翻译)中
比对待检核酸序列(同样用所有 6 种读
框翻译)
CTCAAATTTTTTGACTTTTTTTGTATGTGTGTTTTTTCTTTTTTTTTAAGTTCTTA
TGAGGAGGGGAGGGTAAATAAACCACTGTGCGTCTTGGTGTAATTTGAAGATTGCC
使用 Smith-Waterman 算法联配比对
注:n:核酸序列或核酸序列库;p:蛋白质序列或蛋白质序列库
如果目的序列中有蛋白质编码区,则用翻译的蛋白质序列来搜索蛋白质序列 库要比用 DNA 序列搜索核酸序列库更有价值。由于蛋白质序列的进化要比 DNA 序列慢一些,在蛋白质序列水平上的远缘关系在 DNA 水平上可能被错过。如果无 法确定编码区,则可利用 BLASTX 按所有 6 种读框来翻译 DNA 序列,然后用它搜 索蛋白质序列库。由于蛋白质序列库仅包含已鉴定的蛋白质,所以必须采用 TBLASTN 程序在现有的 GenBank、EMBL 或 DDBJ DNA 序列库中检索新确定的氨基 酸或翻译过的 DNA 序列。这种检索有时可以找到一些显著相似的 DNA 序列,而原 本并不知道这些序列可编码蛋白质。
CTATTTCTTTAGTATTAATTTTTCCTTCTGTTTTCCTCATCTAGGGAACCCCAAGA
GCATCCAATAGAAGCTGTGCAATTATGTAAAATTTTCAACTGTCTTCCTCAAAATA
AAGAAGTATGGTAATCTTTACCTGTATACAGTGCAGAGCCTTCTCAGAAGCACAGA
52
/bioinplant/
《生物信息学札记》 樊龙江
括在 SWISS-PROT 和 PIR(蛋白质)或 EMBL 和 GenBank(核酸)的所有记录,这往往 是最佳选择。但不要滥用这些资源,例如,如果你正在构建序列重叠群(contig), 则只需进行最终组合序列的 BLAST 或 FASTA 搜索即可,而不必对每个序列片段均 进行搜索。同样,为了查找克隆载体的污染序列而进行整个非冗余数据库的 BLAST 运行,也不是一个有效办法。
/bioinplant/
《生物信息学札记》 樊龙江
第三节 数据库搜索——BLAST 和 FASTA 应用
一. 数据之海与一叶轻舟
《科学》(science)杂志在 2001 年 2 月 16 日的人类基因组专刊上发表了一 篇题为“生物信息学:努力在数据的海洋里畅游”的文章,文章写到:“我们身 处急速上涨的数据海洋中…我们如何避免没顶之灾?”一条可靠的办法可能是赶 紧找到“一叶轻舟”,而且在轻舟上装上先进的电子设备,诸如卫星定位系统、 卫星信息传输系统等等……BLAST 和 FASTA 便是这样的一条“轻舟”,它们往来 穿梭,速度奇快。
图 3.6 送往EBI FASTA电子邮递服务中心(电子邮件地址:fasta@) 的一份邮件的内容。这份邮件要求用该序列对EMBL序列库中的其它哺乳动物序列 进行检索。送回的答案中包括 100 条最匹配序列和头 20 条最匹配序列的联配结 果。
不论是 FASTA 还是 TFASTA 都提供一项评分,以评价用前述 PAM250 矩阵生成 的每一对联配序列的匹配程度。但 FASTA 并不像 BLAST 程序那样给出一项显著值。 无论采用 FASTA 或 BLAST,推断相似性是否具有生物学意义都取决于研究者。要 作出决断,必须充分考虑蛋白质已知的或推断的功能,与已知活性位点或模序的 相似程度等等。
50
/bioinplant/
《生物信息学札记》 樊龙江
表 3.15 数据库相似性搜索程序 BLAST 和 FASTA 程序清单
程 序 待检序列类 数据库类型 说 明
(Program 型
(Comment)) Nhomakorabea(Probe type)
BLASTP
p
p
在蛋白质序列库中比对待检蛋白质序
因为BLAST和FASTA采用不同的算法,同时用这两种搜索引擎重新检索某一特 定序列往往是可取的。如果用其中一种找不到显著相似序列,不妨试一试另一程 序。如果BLAST和FASTA均找不到显著匹配的序列,还可以选择第 3 条比较费时的 搜索策略。一些网站允许用户使用基于Smith-Waterman算法的搜索程序,如 BLITZ。BLITZ(/searchs/blitz.html)被设计在大型并行计算机上运 行,因此使检索更灵敏。虽然运行这样的程序比较费时,但它们有时会发现一些 被BLAST和FASTA错过的勉强达到显著的联配。
TITLE A test search of the EMBL other Mammalian DNA sequences LIB EMAM WORD 4 LIST 100 ALIGN 20 SEQ tgcttggctgaggagccataggacgagagcttcctggtgaaagtgtgtttcttgaaatcagcaccaccatg gacagcaaa END
BLAST 的一项重要特性就是所报告的匹配序列的统计学显著性评分。这一统 计学显著性评分是用 Karlin-Altschul 算法决定的,所算出的 Poisson 概率表明 所得到的序列相似性随机出现的可能性。
另一个常用的核酸和蛋白质序列库搜索程序是 FASTA,即 FASTN 和 FASTP 程 序的新版本。FASTA 首先在序列库中进行快速的初检,找出与待检序列高度相似 的序列。这一快速检索局限于待检序列和序列库序列之间较短的完全相同序列区 段上。
大多数研究目前都通过国际互联网 Internet 应用 NCBI 研制的 BLAST 程序 (Basic Local Alignment Search Tool)来进行 DNA 和蛋白质序列相似性搜索。 用一组 BLAST 程序联配可以快速进行核酸和蛋白质序列库的相似性检索。采用 BLAST 的基本算法编成了若干各不同的程序,分别使用特定的序列库和用于特定 类型的输入序列。BLASTN 是在核苷酸序列库搜索核苷酸序列。BLASTP 是在蛋白 质序列库中搜索氨基酸序列。TBLASTN 则可以在核酸序列库中搜索氨基酸序列, 此时序列库在搜索之前要按所有 6 种读框即时翻译。与此相反的一项分析则由 BLASTX 来完成,它要将所输入的核酸序列按所有 6 种读框翻译,然后再以之搜 索蛋白质序列库。近期 Altschul S.F.等人(1997)提出了一个通过寻找蛋白质家 族保守序列来提高算法敏感性的 PSI-BLAST(Position-Specific Iterated BLAST) 算法,并开发了相应的软件。PSI-BLAST 可以对数据库进行多轮循环检索,每 一轮的检索速度都大约是 BLAST 的两倍,但每一轮都能提高检索的敏感性。它 是目前 BLAST 程序家族中敏感性性最高的成员。
FASTA 首先要建立一个其长度由 K-tuple(ktup)值决定的所有可能的总表或 字典。这一程序中使用的字长参数(或 K-tuple)表示所用的初始相配序列长度。
51
/bioinplant/
《生物信息学札记》 樊龙江
K-tuple 的大小可以变化并将间接影响搜索的速度和敏感度。然后程序要对待检 序列和序列库中的所有序列进行处理,找出字典中长度与 K-trple 相等的所有序 列段的位置。比较两个序列的字典要比比较两个序列本身快得多,可以有效地找 出小段相似区。一旦通过初始的快速检索找到一批评分最高的序列,就可以仅对 这些高分序列进行第二轮比较。第二轮的序列比对是采用 Needleman-Wunsch 算 法(1970)进行空位联配计算,得出分析的最后结论。如果 FASTA 运行后找到较好 的相似序列,有时采用较小的 K-tuple 值或换一个评分矩阵重新检索分析,也许 会有帮助。
由于数据库相似性搜索是生物信息学最为重要的组成部分,所以很多网站都 提供了 BLAST 和 FASTA 搜索服务。在选择何种数据源时,有很多标准可以应用。 并非所有的 BLAST 和 FASTA 均提供相同的服务,你所搜索的数据库各不相同, 这就如同我们有多种替换矩阵一样。另外,一些网站还为熟练使用者提供了特别 服务。总之,在一些非冗余序列数据库中搜索均是被允许的。这类数据库至少包
