Vector NTI Suite使用简介
常用生物学软件简介

网址:/ 1. Oligo 6是目前使用最为广泛的一款引物设计软件,除了可以简单快捷地完成各种引物和探针的设计与分析外,还具有很多其他同类软件所不具有的高级功能:a) 已知一个PC R引物的序列,搜寻和设计另一个引物的序列。
b) 按照不同的物种对MM子的偏好性设计简并引物。
c) 对环型DNA片段,设计反向PCR引物。
d) 设计多重PCR引物。
e) 为LCR反应设计探针,以检测某个突变是否出现。
f) 分析和评价用其他途径设计的引物是否合理。
g) 同源序列查找,并根据同源区设计引物。
h) 增强了的引物/探针搜寻手段。
设计引物过程中,可以“Lock”每个参数,如Tm值范围和引物3’端的稳定性等。
i) 以多种形式存储结果;支持多用户,每个用户可保存自己的特殊设置。
网址:Oligo 6.71 Demo(引物设计软件):/Soft/2006/112.htmOligo—引物设计软件电子教程(引物设计和评估)Oligo 6 Tour 主要功能介绍/2.Vector NTI Suite是一套功能最全,而且界面最美观,最友好的分子生物学应用软件包。
主要包括四个大型软件,它们分别可以对DNA、RNA、蛋白质分子进行各种分析和操作。
Vector⑴NTI:作为Vecto r NTI Suite的核心组成部分,它可以在生物研究的全过程中提供数据组织和序列编辑的软件支持。
Vector NTI 是以一种窗口形式,且支持项目组织的数据库来完成这一功能的;通过这个数据库,可以保存和组织大部分的实验数据,比如:基因结构、载体、序列片断、引物、蛋白质、多肽、电泳Markers和限制性内切酶等。
实际上,该数据库还支持对Vector NTI Suite中各种小型的绘图和结果展示工具的管理。
Vector NTI 可以按照用户要求设计克隆策略。
用户只需提供克隆载体,外源片断序列,明确载体克隆的大致位置或酶切位点,其它工作由软件完成。
设计结果以图文形式输出到屏幕;最后根据客户定制的条件进行模拟电泳。
vector nti 使用教程 多重序列比对
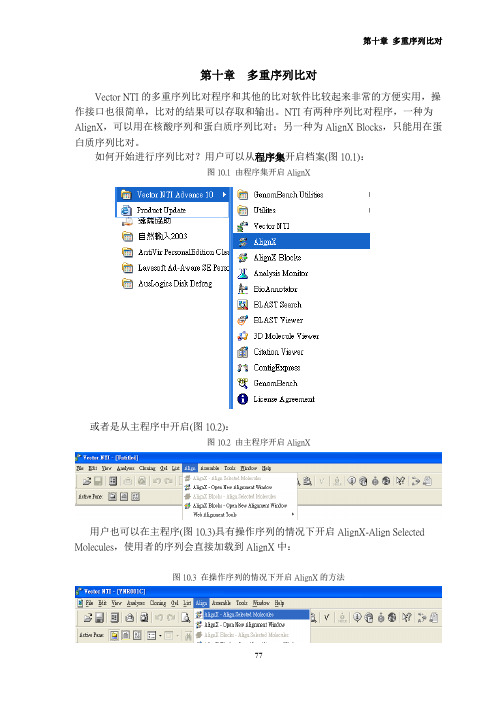
第十章多重序列比对 Vector NTI的多重序列比对程序和其他的比对软件比较起来非常的方便实用,操作接口也很简单,比对的结果可以存取和输出。
NTI有两种序列比对程序,一种为AlignX,可以用在核酸序列和蛋白质序列比对;另一种为AlignX Blocks,只能用在蛋白质序列比对。
如何开始进行序列比对?用户可以从程序集开启档案(图10.1):图10.1 由程序集开启AlignX 或者是从主程序中开启(图10.2):图10.2 由主程序开启AlignX 用户也可以在主程序(图10.3)具有操作序列的情况下开启AlignX-Align Selected Molecules,使用者的序列会直接加载到AlignX中:图10.3 在操作序列的情况下开启AlignX的方法 开启AlignX之后,使用者会见到图10.4的画面:图10.4在操作序列的情况下开启AlignX首先用户要把序列加载Vector NTI程序中,可以点选或者从左上方的Project →Add Files把序列档案加载,请注意文件名不可以过长,檔名过长会造成程序进行比对时无法完全显示文件名(图10.5):图10.5 输入的档名注意不可过长 选取档案后按下开启就可以加载程序中,若比对的序列很多时可以用鼠标圈选欲分析的序列后选择开启。
序列档案加载的时候程序会询问该序列为核酸序列或是蛋白质序列,点选好以后再点选Import就可以了(图10.6):图10.6 载入时,会询问序列的性质,核酸序列或蛋白质序列接下来程序的左上方会出现使用者加载的序列(图10.7),序列加载完成以后就可以开始进行比对的操作:图10.7 成功载入序列的画面进行比对前,先把欲比对的序列用鼠标进行圈选(图10.8):图10.8 选取欲比对之序列只要按下或是从上方Align→Align Selected Sequence(图10.9)就会进行比对运算:图10.9按下Align→Align Selected Sequence进行比对运算好以后就会出现下面的画面(图10.10);图10.10 比对完的结果 分析完成后画面(图10.11)会出现比对的相关结果,最下方是序列比对的图形,左边中间的区块所显示的图形为导引树(Guide tree),用来表示序列之间的关连性。
Vector NTI Suite使用简介

Vector NTI Suite使用简介资料来源:丁香园 Vector NTI Suite是一套功能强大、界面美观而又友好的分子生物学应用软件包。
它主要包括四个组件,分别对DNA、RNA和蛋白质进行各种分析和操作。
一、Vector NTI作为Vector NTI Suite的核心组成部分,它可以在各种分子生物学研究项目的全过程中提供数据组织、编辑和分析支持。
(一)对分子序列的操作我们以一个DNA序列为例,进行一系列的常规分析;最后将此DNA序列翻译成氨基酸序列,并对此氨基酸序列进行各种分析。
A,DNA序列为猪生长激素的cDNA序列,长为761bp。
首先使用Vector NTI的Create New命令将此序列导入到Vector NTI的数据库中:1,第一种方法:如果只知道序列时,点击Molecule才菜单中的Create New——Using Sequence Editor(DNA/RNA……);2,在出现的“New DNA/RNA Molecule”对话框中,首先在General填入导入序列的名称——PGH;3,在DNA/RNA Molecule活页中,选中Linear DNA, Animal/other Eukaryotes,Replicon Type中选Chromosome;4,Description中填入:S.Scrofa Growth hormone mRNA;5,在Sequence and Maps中点击“Edit Sequence”按钮,将DNA序列复制后,点“Paste”按钮-点“OK”-确认后就可以完成序列导入。
B,如果是一个从GenBank上下载的序列文件,则:点击“Molecule”菜单-Open-Molecule files命令,找到序列文件,在File format中选中GenBank Files;点击OK。
(二)常规操作:当序列导入完成后,在桌面出现三个窗口,上左侧的窗口中显示的是该序列的常规信息,上右侧窗口则以图形的格式展示序列的特征区及酶切图谱等。
Vector NTI的使用-文档资料

功能模块主要包括:序列编辑、引 物设计、模拟翻译、模拟电泳、序列比 对、Contig、第三方附加模块等。
4
一、本机数据模块:
5
✓ 数据库分类
✓ 核酸分子 ✓ 蛋白质分子 ✓ 内切酶 ✓ 寡核苷酸 ✓ 电泳marker ✓ 引文 ✓ BLAST搜索结果 ✓ (PCR)分析结果
Vector NTI的使用
1
软件: ✓Vector NTI 8.0 / 10.3
此课件仅是提纲,具体使用 库的维护 ✓ 限制性内切酶 ✓ DNA→Protein ✓ Contig ✓ 序列比对及多重比对 ✓ DNA/质粒图谱绘制
3
软件包含两大模块: 数据库模块和功能模块。数据库用
✓酶切片段
✓ 完全酶切 ✓ 部分酶切
21
22
部分酶切
23
部分酶切
24
部分酶切
25
✓序列比对
26
按住“Ctrl”键后,用鼠标左键可以进行复杂的选择, 然后在选择的项目上点鼠标右键,关联菜单弹出……
27
这里先做选取
28
✓Contig
✓ *.abi或*.scf文件与序列文件 ✓ 构建一个Contig ✓Contig是“contiguous sequences group”的缩
✓ 每类数据库又分为基库(MAIN)和子库(subset)
✓ 真正的数据都在基库里,子库只是用户自己建立的数据条目(item)的子集,里面 是数据的联接不是数据本身,但是可以象操作数据一样进行直接操作;
✓ 对数据条目进行“拷贝”、“粘贴”操作,只会产生一个新的联接,不会产生新 的数据;要产生数据复制,需要用“Duplicate”(数据条目上点右键);
vectornti使用教程第十六章_GenomicAnalysisI

vectornti使用教程第十六章_GenomicAnalysisI第十六章:基因组分析I本章将介绍如何进行基因组分析。
基因组分析是对一个或多个生物体的基因组进行研究和解读的过程。
该过程涉及到收集、整理和解析大量的基因组数据,以了解生物体的遗传信息、基因功能和基因组结构。
以下是一个基因组分析的一般流程:1.数据收集:基因组分析的第一步是收集所需的基因组数据。
这些数据可以是DNA序列、RNA序列、基因组注释信息等。
2.数据整理:收集到的数据需要经过一系列的处理来整理和准备,以便于后续的分析。
这个过程中可能包括数据清洗、去除冗余数据、对数据进行标准化等。
3. 基因组注释:注释是对基因组数据进行功能和结构解读的过程。
通过注释可以了解基因的功能、基因之间的关系以及可能的遗传变异等。
常用的注释工具包括Ensembl、NCBI等。
4.基因功能分析:基因功能分析是对基因组数据进行功能注释的过程。
通过比对已知的基因数据库,可以确定基因的功能以及与其他基因的关联程度。
这个过程中常用的工具有BLAST、HMMER等。
5. 基因组结构分析:基因组结构分析是对基因组序列进行结构解读的过程。
通过分析基因的启动子、外显子以及剪接位点等,可以了解基因组的结构变异以及可能的突变情况。
常用的工具包括Geneious、Cufflinks等。
6. 基因组比较:基因组比较是将不同生物体的基因组数据进行比较的过程。
通过比较不同生物体的基因组,可以了解它们之间的遗传关系、共享的功能基因以及可能的进化路径。
常用的工具包括Mauve、UCSC等。
7. 数据可视化:最后一步是将分析结果进行可视化展示。
通过可视化可以更直观地理解基因组数据,并使得结果更易于解读。
常用的可视化工具包括Cytoscape、UCSC Genome Browser等。
以上是基因组分析的大致流程,实际的分析过程可能因具体的研究目的和数据类型而有所调整。
基因组分析是生物信息学领域的重要研究方向,它对于理解生物体的遗传信息和生命过程有着重要的意义。
Vector NTI Suite在分子生物学领域的应用

V
NTI S
在 分 子 生 物 学 领 域 的 应 用
道不断拓展, 国内科研人员对各种软件的使用不断增 计、 序列的查找下载与比对、 序列拼装、 蛋白结构 预测 加, 但总体上国内科研人员对 � � � � � V NTI的使用与国 分析等一系列的应用上, 与V NTI相似的一些同类 外还有很大差距 � � � � � ( 表 1) 。通过 � � � � � 检索 � C NKI和 NC BI中 软件如 L B S 公司的 DNA M A N 、 B T 公司 涉及使用了 V 差距。
[ 1] 大量的分析和操 作, 以 及建立和 管理生 物数据 库 。
1
V
NTI: 的功能十分全面, 综合 性强, 所以 许多
生物工作者都喜欢选用此软件, 尤其对于那些 不喜欢 装备各种专业性强的软件的用 户更是首选。因此, 在 许多文献中, 特别是近几年发表的论文中我们常能 看 到V 以发现 V NTI 的使用。从 CNK I 检索 ( 2001 年以后) 可 NTI 不同功能的应用分 布状况, 其 中涉
中图分类号: TP 31 9 、 Q7
随着计算机和生物学的发展, 一系列的生物学数 2 应用现状 � � � � � � 由于早期版本不支持 P C 单机及 W 操作系 据库如核酸序列数据库、 蛋白质序列数据库、 蛋白质三 维结构数据库等, 被建立起来。如何对数据进行分析 统等原因, 早期其应用不是十分广泛, 随着其功能的增 处理是摆在我们面前的一个问题, � � � � � � 目前的数据处理软 加和对 PC 单机及 W 操作系统的支持, 其使用频 件包 括: 对资料 收集整理阶段 的有 R M 率和范围逐渐增大。在近几年所发表的论文中应用到 9.0 , E 8 .01 ; 序 列 分析、 实 验 设计 模 拟 阶段 的 � � V NTI S 的文章明显增多 (图 1 ) 。使用范围上 D NA STA R 4 .03 , V NTI S 9.0 , 0 2.0 、 D NA 涉及多个研 究方向, 例如癌症 的研究、 肝炎 病毒的 研 SIS 2.5 , DNA T B V S 4 .50 , S NTI S 5.1 ; 实验 操作和数据分析阶段的 P 20 00 等等。在众多软件中, 以其高度集成、 功能齐全、 操作简便且 NTI S 软 件的概 究、 菌种的筛选、 水稻蛋白分析、 猪瘟病的研究、 幽门螺 杆菌 C A 基因及蛋白序列多态性分析、 人金属硫蛋白 基因合成及其表达、 SA RS 冠状病毒 N 蛋白的表达及二 级结构预测分析等等。
最新Vector NTI使用介绍(PPT)

☼ Splicing exons of a gene ☼ Translated from DNA molecules
Molecule Construction vs. Molecule Design
❖ Recipient and donor fragments are defined by users.
construction parameters, including necessary terminus modifications. 4. Name, select data and describe the new molecule. 5. Verify and edit the component fragments in the Goal Molecule Definition List. 6. Initiate molecule construction.
❖ Fragment Wizard
☼ Construct fragments (define positions and termini) ☼ Design recipient/donor fragments (define termini) ☼ Add defined fragments to the Goal Molecule Definition List
❖ Restriction sites are defined by users.
❖ When required, the methods of terminus modification are defined by users.
❖ Recipient and donor fragments are selected by users.
vector-NTI_中文教程_GenomicAnalysisⅢ

第十八章 Genomic Analysis III第十八章 Genomic Analysis ⅢPromoter binding factors analysisÆ分析基因的轉錄因子結合位置:在GenomBench的功能中,使用者可以找出基因的promoter序列,promoter 位於基因的上游,使用者可以先把欲分析的範圍圈選出來:圖18.1 將欲分析的範圍圈選圈選出來的序列可以複製輸出,這一段部分即為該基因的上游區域,一般而言選取的範圍為基因上游3000-5000nt區域。
遺憾的是NTI沒有分析promoter 序列transcription factor binding site的功能,使用者只能把找到的promoter序列放到相關的分析網站進行transcription factor binding site的分析。
Exon/intron structure analysisÆ由NTI主程式分析基因體結構:使用者欲分析的序列必須存放在主程式中的Local Database(圖18.2),建議使用者將欲比對的序列存放於特定的資料夾中,詳細的操作方法請參考序列資料建立的章節。
圖18.2 將欲比對的序列放在Local Database,左方為Local Database內的檔案該資料夾可存放多條序列,程式會把該資料夾中的所有序列進行分析。
Vector NTI 教育訓練手冊接下來在主程式中開啟序列時請先將”限制酶分析”的功能關閉(圖18.3):圖18.3 關閉”限制酶分析”的功能接著選擇Analysis→GenomBench Tools(圖18.4):圖18.4 比對序列可以選擇兩種方式:SIM4或Spidey比對基因序列和基因體序列有兩種方式:SIM4和Spidey,這兩種方法的計算方式有些許的不同,使用SIM4的視窗(圖18.5)如下圖所示:第十八章 Genomic Analysis III 圖18.5使用SIM4,左上方為此序列名稱,中間為比對的基因序列左上方Sequence to analyze的欄位為目前使用者所分析的基因體序列(圖18.6);中間上方Alignment sequences為提供比對的基因序列,請把資料項目選至上述設定的資料夾:圖18.6 選擇Alignment sequence提供的比對資料右上方的Strand欄位可以選擇只針對正股、互補股或者雙股進行分析。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Vector NTI Suite使用简介资料来源:丁香园 Vector NTI Suite是一套功能强大、界面美观而又友好的分子生物学应用软件包。
它主要包括四个组件,分别对DNA、RNA和蛋白质进行各种分析和操作。
一、Vector NTI作为Vector NTI Suite的核心组成部分,它可以在各种分子生物学研究项目的全过程中提供数据组织、编辑和分析支持。
(一)对分子序列的操作我们以一个DNA序列为例,进行一系列的常规分析;最后将此DNA序列翻译成氨基酸序列,并对此氨基酸序列进行各种分析。
A,DNA序列为猪生长激素的cDNA序列,长为761bp。
首先使用Vector NTI的Create New命令将此序列导入到Vector NTI的数据库中:1,第一种方法:如果只知道序列时,点击Molecule才菜单中的Create New——Using Sequence Editor(DNA/RNA……);2,在出现的“New DNA/RNA Molecule”对话框中,首先在General填入导入序列的名称——PGH;3,在DNA/RNA Molecule活页中,选中Linear DNA, Animal/other Eukaryotes,Replicon Type中选Chromosome;4,Description中填入:S.Scrofa Growth hormone mRNA;5,在Sequence and Maps中点击“Edit Sequence”按钮,将DNA序列复制后,点“Paste”按钮-点“OK”-确认后就可以完成序列导入。
B,如果是一个从GenBank上下载的序列文件,则:点击“Molecule”菜单-Open-Molecule files命令,找到序列文件,在File format中选中GenBank Files;点击OK。
(二)常规操作:当序列导入完成后,在桌面出现三个窗口,上左侧的窗口中显示的是该序列的常规信息,上右侧窗口则以图形的格式展示序列的特征区及酶切图谱等。
下面一个窗口显示的是序列:默认状态下以双链形式出现,也可以更改为单链显示。
1.选择序列区域:在图形区域或序列区域直接拖动鼠标左键,同时在最下端的状态栏中显示出所选区域的范围。
2.删除:选中后直接点击键盘上的Delete键,确认后即可删除。
3.选中序列片段后,点击Edit菜单,用其中的命令可以完成对此片段的剪切、复制、删除、定义为新的特征区和用其它序列来代替等。
4.当点在其一特定位置时,我们也可以在此位置插入新的序列:Edit – New – Insert Sequence as5.当希望序列显示单链时,点击View – Show Both Strands(三)常规分析:1.设计PCR Sequence Primer, Hybridization, Probes:选中设计引物的模板区或点击Analyze中的相应命令即可。
需要注意的是,在设计前,首先得将序列存入数据库中,具体设计由于我们推荐使用Oligo,所以此处不详述。
2.序列基本信息分析:选中序列区段后,选Analyze – Oligo Analysis, 在Oligo Analysis对话框中,点击Analyze按钮,即可得到分子量、GC含量、Tm值、3‘端的自由能、回文结构及重复序列等基本信息。
3.酶切图谱分析点击Analyze菜单中Restriction Sites命令,出现“Restriction Map Setup”对话框,点击Add按钮,填入需要分析的位点,不需要的位点夜可以选中后点Remove按钮移除。
为了显示正确,我们可以设定超过一定位点数量的酶不显示,可以限定分析的区域等。
点击OK后程序自动完成酶切分析。
4.Motif查找点击Analyze菜单中的Motifs命令,在出现的Motifs Setup对话框中我们可以添加新的Oligo 或从Oligo Database和Oligo List中选取;选中后点击OK按钮,程序完成Motif的查找,同时给出相似性的百分比。
5.ORF查找点击Analyze菜单中的ORF命令,在出现的ORF Setup对话框中,填入ORF的最小长度(多少个密码子)以及其它一些设定后点击OK,程序自动完成ORF的查找。
6.翻译翻译前选中一个ORF或一个区域,这里我们希望把pGH cDNA基因完全翻译成蛋白质,因此选中最长的一个ORF(7-657bp)。
点击Analyze菜单中的Translate命令中的“Into New Protein”-“Direct Strand”,在出现的“New Protein Molecule”对话框中给出新蛋白质的名称后点击确定,程序完成翻译并打开一个新的窗口,显示氨基酸序列。
7.反翻译选中氨基酸序列片断,点击Analyze菜单中的BackTranslate命令,确认是“整个序列”还是“仅为选中的序列”后,即可设定简并度及组织特异性来完成反翻译。
(三)模拟电泳:模拟电泳是指对DNA片段进行酶切分析后,通过电脑模拟电泳过程,将酶切片段分离。
该功能有利于评估电泳时间,便于验证实际电泳结果的好坏。
1.点击Gel菜单– Create New命令:出现Gel Setup对话框,首先选择电泳介质的类型-“Agarose Gel”,以及胶的浓度、电压、胶长和Buffer种类。
2.点击OK后即打开一个电泳界面,左侧是对电泳情况的描述,右侧则为胶板。
3.首先配置一个Marker:点Gel菜单– Create Gel Marker, 出现New Gel Marker对话框, 首先输入Marker的名称:pGH-Marker,在Gel Marker活页中输入自己设定的片段,100,200,300,400,500,600,1000bp,点击确定。
4.样品制备:点Gel菜单– Create Gel Sample命令出现Create Gel Sample对话框,选择来源分子SSPGH,选中AvaI和SmaI两个酶,输入Sample的名称SSPGH- AS。
此时我们可以把酶切的片段直接加入到胶上,也可以加入到样品列表中或保存为Marker,我们点击Add to Gel,则在胶上出现1号样品。
5.点Marker到胶中:点击第二行工具栏中的Add Marker Lane图标,出现Choose Database Gel Marker对话框,选中pGH - Marker,点击OK,则Marker出现在2道6.电泳:用鼠标点击右侧窗口激活胶板,点击第二栏工具栏中的双箭头,开始电泳,同时在旁边显示电泳时间,再次点击双箭头,电泳停止。
7.计算两条带分开时间:选中没有分开的条带,点工具栏中的计算器Calculate Seperation Time,即可得到所需信息。
8.导出胶图:激活胶板,点击工具栏中的Camera工具,出现Camera对话框,选中需要导出的选项,结果可以到剪贴板,也可以保存成文件。
到剪贴板后就可以在Word等文档编辑器中粘贴。
(四)图形操作对于图形展示的序列信息,我们可以对图形进行各种修饰和改动,并最终导出需要的图形,如果序列是质粒,则可得到质粒图谱。
我们还是以SSPGH序列为例:1.激活图形栏,点击工具栏中的Edit Picture。
此时,点击任意一个需要改动的组件,则鼠标变为四方向箭头,按住左侧则可以任意拖动其位置。
2.点击左键,选中Properties命令,则在Properties对话框中可以改变文字,字体,连线的粗细和颜色。
3.对于特征序列,我们还可改变其填充方式,箭头方向等。
4.加入注释:点击工具栏中的回形针按钮,出现Annotation对话框,输入注释文字(支持中、英文),如:“这是一个测试Sequence”。
点击确定后,文字就出现在图示窗口中,同样可以改变其位置、字体、颜色等。
5.修改完成后,点工具栏中的命令,同样可以到剪贴板或文件。
二、组件AlignX运行AlignX后出现四个窗口,从上到下,从左到右分别为序列信息窗口,进化树窗口,同源比较图示窗口和序列比较窗口。
对于同源比较可以分为两类:一类是两个或几个序列(包括DNA/RNA和蛋白质序列)的比较,此时仅限于比较序列的相似性;另一类则是多个序列间的比较并得出系统进化树。
(一)导入外源序列的方法:点击Project菜单中Add Files命令,选择需要比较的序列文件,点击打开按钮,确认是DNA/RNA还是Protein Sequence后,点击Import按钮就可以完成序列的导入任务。
(二)我们以程序本身提供的演示Project来讲述AlignX的使用:1.选择Project菜单Open命令,找到Vector NTI的Demo Projects文件夹,打开DNA.apr文件;2.在文本窗口中,使用鼠标左键双击任一文件名,就可以得到该文件的所有基本信息;3.建立一个新的比较策略并进行比较:①在文本窗口中,按住Ctrl键选中四个序列:AF××××②点击Alignment菜单中Alignment Setup命令,出现Alignment Setup对话框,在此对话框中有30个选项来确定最终比较结果展示方式,这里我们使用默认值即可。
③点击Alignment菜单中的Align Selected Sequence命令,片刻后程序完成比较并给出结果和进化树;④在View菜单中,我们可以通过Edit Alignment命令来编辑比较结果,使用Display Setup命令来改变结果的展示方式和颜色等。
4.结果的导出:①进化树的导出:激活进化树窗口,点击工具栏中的Export Tree按钮即可将进化树保存为.Ph 文件,并可被其他树编辑软件所识别。
或者点击Edit菜单中的Camera命令,将图像保存到剪贴板后在Word等文档编辑软件中粘贴;②序列比较结果的导出;点击Project菜单中的Export MSF Format命令,将结果保存为.msf 文件后,用GeneDoc打开进一步进行编辑和修饰。
三、Contig Express该组件让您能够直接从测序仪或其它文件格式(如GenBank和Fasta等)中导入序列,并将这些阿序列片段拼接成一个长片段。
在拼接的过程中可以显示测序图谱,可以自动去除载体序列及测序模糊序列。
同时能在拼接的完成序列碱基的改动。
1.点击Project菜单中的Open命令,找到Demo Project文件夹中的DNA.cep文件。
当然我们也可以导入自己的序列,方法是点Project菜单中的Add Fragments命令,在此可以导入各种格式的文件。
2.建立拼接策略:点击Assemble菜单中的Assembly Setup命令,出现Assembly Setup对话框,在此我们使用程序的默认值,不作改动。
