pBI121酶切位点图
酶切位点汇总

酶切位点汇总
酶切位点,又称为限制性内切酶位点,是指DNA分子上特定的序列,这些序列是限制
性内切酶可以识别和切割的地方。
限制性内切酶是一种在细菌和其它生物中广泛存在的酶,能够切割或切除一个或多个DNA碱基对。
这些限制性内切酶在生物技术领域广泛应用,用
于DNA序列分析、DNA重组、基因工程等方面。
以下是常见的几种酶切位点:
1. EcoRI切割位点是5′-GAATTC-3′,这是一种广泛应用的限制性内切酶,通常用于DNA纯化、制备DNA载体等。
2. BamHI切割位点是5′-GGATCC-3′,BamHI能够切割链间,产生具有黏性末端的DNA 序列。
常被用于制备双链DNA的黏性末端。
4. PstI切割位点是5′-CTGCAG-3′,PstI是一种双切酶,可以切割成不同长度的DNA 序列,适用于构建多种不同长度的DNA分子。
总之,酶切位点及其对应的限制性内切酶在现代生物领域有着广泛的应用和重要的作用。
了解不同的酶切位点是有很大帮助的,它可以为实验设计和分子生物学研究提供基础。
同时,也让我们更好地理解限制性内切酶在DNA分子上的作用,帮助我们在生物技术领域
更加熟练地掌握其应用。
2022-2023学年山东省德州市高二下学期期中生物试题

2022-2023学年山东省德州市高二下学期期中生物试题1.《齐良要术》中有关发酵的记载有“作盐水,令极咸,于盐水中洗菜,即内(纳)瓮中。
其洗菜盐水,澄取清者,泻著瓮中,令没菜把即止”。
下列叙述错误的是()A.“令极咸”可以抑制杂菌的繁殖B.“洗菜盐水”中可能含有乳酸菌C.“令没菜把即止”更有利于乳酸菌的发酵D.发酵过程中乳酸菌的数量始终占据优势2.辣椒酱是由乳酸菌发酵得到的产品,制作时将辣椒洗净、去蒂、加盐﹑发酵、破碎。
发酵可以分为自然发酵和纯菌种发酵。
研究人员对自然发酵和纯菌种发酵条件下所得辣椒酱中亚硝酸盐的含量进行了测定,结果如下图。
下列有关叙述错误的是()A.将辣椒洗净后去蒂可防止杂菌的污染B.不同季节制作辣椒酱的周期可能不同C.自然发酵时,亚硝酸盐含量较高可能与杂菌有关D.纯菌种发酵后期,乳酸菌数量下降导致亚硝酸盐含量降低3.沙保氏培养基含4.0%的葡萄糖、1.0%的蛋白胨,pH为4.0~6.0,常用于真菌的培养。
某同学利用沙保氏培养基培养红酵母,结果如图所示。
下列有关叙述错误的是()A.培养细菌时也需要将培养基调至酸性B.该同学利用了平板划线法接种红酵母C.葡萄糖为红酵母提供碳源,蛋白胨提供碳源和氮源D.制备培养基时,应先调节pH再进行高压蒸汽灭菌4.王家园子醋以优质小麦、小米、高粱和金丝小枣为原料,采用了传统菌曲发酵工艺,其生产工艺流程如图。
下列叙述错误的是()A.糖化阶段需要淀粉酶的催化作用B.酒精发酵和醋酸发酵的最适温度不同C.酒精发酵和醋酸发酵过程中pH均降低D.酒精发酵和醋酸发酵过程中均需先通气后密闭5.下图表示利用胡萝卜植株进行植物组织培养的两种不同途径,有关叙述错误的是()A.外植体经过程Ⅰ形成不定形的薄壁组织块B.过程Ⅱ所使用的培养基也是固体培养基C.过程Ⅲ中诱导生芽和生根所需的培养基不同D.两种途径均体现了植物细胞具有全能性6.用拟南芥根尖进行植物组织培养诱导生芽的过程中,细胞分裂素(CK)通过ARRs(A)基因和WUS(W)基因起作用。
限制性内切酶酶切位点_方便搜索(word版本)讲解

CCRYGG
GGYRCC
BtgZI识别位点
GCGATG
CGCTAC
BtsCI识别位点
GCAGTGNN
CGTCACNN
BtsI识别位点
GCAGTGNN
CGTCACNN
Cac8I识别位点
GCNNGC
CGNNCG
ClaI识别位点
ATCGAT
TAGCTA
CspCI识别位点
NCAANGTGGN
TCCGGA
AGGCCT
BspHI识别位点
TCATGA
AGTACT
BspMI识别位点
ACCTGCN
TGGACGN
BspQI识别位点
GCTCTTCN
CGAGAAGN
BsrBI识别位点
CCGCTC
GGCGAG
BsrDI识别位点
GCAATGNN
CGTTACNN
BsrFI识别位点
RCCGGY
YGGCCR
BsrGI识别位点
CCCAGC
GGGTCG
BsgI识别位点
GTGCAG
CACGTC
BsiEI识别位点
CGRYCG
GCYRGC
BsiHKAI识别位点
GWGCWC
CWCGWG
BsiWI识别位点
CGTACG
GCATGC
BslI识别位点
CCNNNNNNNGG
GGNNNNNNNCC
BsmAI识别位点
GTCTCN
CAGAGN
BsmBI识别位点
BmrI识别位点
ACTGGGN
TGACCCN
BmtI识别位点
GCTAGC
CGATCG
BpmI识别位点
蛋白酶-酶切位点

or angiotensinase, or glutamyl-aminopeptidase Dipeptidyl-peptidases and tripeptidyl-peptidases Dipeptidyl-peptidase I, or cathepsin C or J
Dipeptidyl-peptidase IV
Cathepsin B
Clostripain, or endoproteinase Arg-C Calpain-1,
酶名称
中文
E.C.号
酶切位点
丝氨酸蛋白酶
胰凝乳蛋白酶,糜蛋白酶
胰朊酶,胰蛋白酶 胰弹性蛋白酶 凝血酶 胞质素,胞浆素
E.C.3.4.21
E.C.3.4.21.1
E.C.3.4.21.4 E.C.3.4.21.36 E.C.3.4.21.5 E.C.3.4.21.7
基氨肽酶
二肽基肽酶和三肽基肽酶 组织蛋白酶
E.C.3.4.14 E.C.3.4.14.1
二肽基肽酶 IV
C.3.4.14.5
脯氨酰三肽-肽酶
E.C.3.4.14.12
肽基-二肽酶
E.C.3.4.15
肽基-二肽酶 A,血管紧张素转化 E.C.3.4.15.1
酶
金属羧肽酶
E.C.3.4.17
羧肽酶 A
羧肽酶 N,羧基肽酶,激肽酶 I E.C.3.4.17.3 Xaa-|-Lys >>Xaa-|-Arg
羧肽酶 U 或 R 谷氨酸羧肽酶,叶酸水解酶
E.C.3.4.17.20 E.C.3.4.17.21
Xaa-|-Arg and Xaa-|-Lys Xaa-|-Glu, preferentially with Xaa=Asp or Glu
详细酶切位点及保护序列

酶切位点保护碱基-PCR引物设计用于限制性内切酶酶切反应本文给出了分子克隆中常用限制性内切酶的保护碱基序列,如AccI,AflIII,AscI,AvaI,BamHI,BglII,BssHII,BstEII,BstXI,ClaI,EcoRI,HaeIII,HindIII,K pnI,MluI,NcoI,NdeI,NheI,NotI,NsiI,PacI,PmeI,PstI,PvuI,SacI,S acII,SalI,ScaI,SmaI,SpeI,SphI,StuI,XbaI,XhoI,XmaI,为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。
酶切位点表——精选推荐

PCR 设计引物时酶切位点的保护碱基表 2
酶
寡核苷酸序列
BamH I
CGGATCCG CGGGATCCCG CGCGGATCCGCG
Bgl II
CAGATCTG GAAGATCTTC GGAAGATCTTCC
BssH II
GGCGCGCC AGGCGCGCCT TTGGCGCGCCAA
BstE II
25
0
10
10
10
0
0
50
>90
0
0
10
50
10
75
10
25
75
75
0
10
0
10
10
50
>90
>90
Spe I
GACTAGTC GGACTAGTCC CGGACTAGTCCG CTAGACTAGTCTAG
酶切位点保护碱基表 7
10
>90
10
>90
0
50
0
50
切割率%
酶
寡核苷酸序列
2 hr 20 hr
GAGTACTC AAAAGTACTTTT
CCCGGG CCCCGGGG CCCCCGGGGG TCCCCCGGGGGA
0
0
10
10
10
10
25
90
25
>90
10
>90
>90
>90
0
0
0
25
0
>90
0
0
0
25
0
50
75
>90
0
0
酶切位点的保护碱基原则
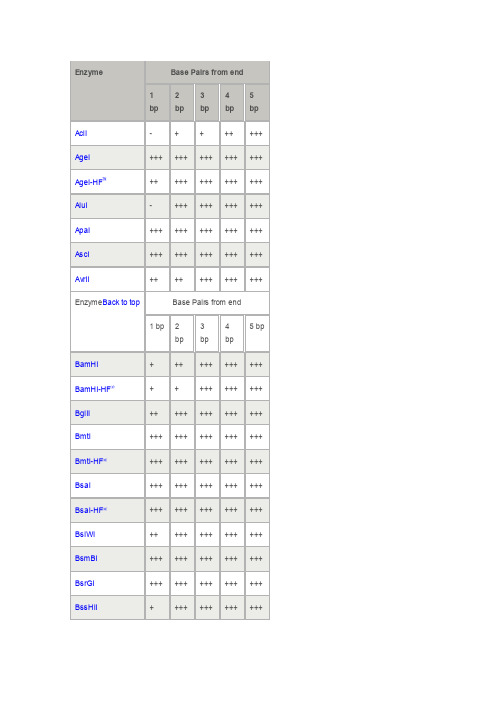
1 bp 2bp3bp4bp5bpAciI- + + ++ +++ AgeI+++ +++ +++ +++ +++ AgeI-HF™++ +++ +++ +++ +++ AluI- +++ +++ +++ +++ ApaI+++ +++ +++ +++ +++ AscI+++ +++ +++ +++ +++ AvrII++ ++ +++ +++ +++ Enzyme Back to top Base Pairs from end1 bp 2bp 3bp4bp5 bpBamHI+ ++ +++ +++ +++ BamHI-HF®+ + +++ +++ +++ BglII++ +++ +++ +++ +++ BmtI+++ +++ +++ +++ +++ BmtI-HF®+++ +++ +++ +++ +++ BsaI+++ +++ +++ +++ +++ BsaI-HF®+++ +++ +++ +++ +++ BsiWI++ +++ +++ +++ +++ BsmBI+++ +++ +++ +++ +++ BsrGI+++ +++ +++ +++ +++ BssHII+ +++ +++ +++ +++1 bp 2bp 3bp4bp5 bpClaI- - + +++ +++ Enzyme Back to top Base Pairs from end1 bp 2bp 3bp4bp5 bpDdeI+++ +++ +++ +++ +++ DpnI- ++ ++ nt nt DraIII+++ +++ +++ +++ +++ DraIII-HF®+++ +++ +++ +++ +++ Enzyme Back to top Base Pairs from end1 bp 2bp 3bp4bp5 bpEagI++ +++ +++ +++ +++ EagI-HF®+ +++ +++ +++ +++ EcoRI+ + ++ ++ +++ EcoRI-HF®+ + ++ +++ +++ EcoRV++ ++ ++ ++ +++ EcoRV-HF®+ ++ ++ ++ +++ Enzyme Back to top Base Pairs from end1 bp 2bp 3bp4bp5 bpFseI+ ++ +++ +++ +++ Enzyme Back to top Base Pairs from end1 bp2345 bpbp bp bpHindIII- + +++ +++ +++ HindIII-HF®- + +++ +++ +++ HpaI+++ +++ +++ +++ +++ Enzyme Back to top Base Pairs from end1 bp 2bp 3bp4bp5 bpKpnI+ +++ +++ +++ +++ KpnI-HF®+ +++ +++ +++ +++ Enzyme Back to top Base Pairs from end1 bp 2bp 3bp4bp5 bpMfeI+ ++ +++ +++ +++ MfeI-HF®+ ++ +++ +++ +++ MluI+ ++ +++ +++ +++ MseI+++ +++ +++ +++ +++ Enzyme Back to top Base Pairs from end1 bp 2bp 3bp4bp5 bpNcoI- ++ +++ +++ +++ NcoI-HF®+ ++ +++ +++ +++ NdeI+ + +++ +++ +++ NheI+ ++ +++ +++ +++ NheI-HF®++ ++ +++ +++ +++ NlaIII++ +++ +++ +++ +++NotI-HF®++ ++ ++ ++ ++ NsiI+ + +++ +++ +++ NspI- - + + +++ Enzyme Back to top Base Pairs from end1 bp 2bp 3bp4bp5 bpPacI+++ +++ +++ +++ +++ PciI+++ +++ +++ +++ +++ PmeI+++ +++ +++ +++ +++ PstI+ +++ +++ +++ +++ PstI-HF®++ +++ +++ +++ +++ PvuI+++ +++ ++ +++ +++ PvuI-HF®+++ +++ +++ +++ +++ PvuII++ ++ ++ +++ +++ PvuII-HF®- ++ ++ +++ +++ Enzyme Back to top Base Pairs from end1 bp 2bp 3bp4bp5 bpRsaI+ +++ +++ +++ +++ Enzyme Back to top Base Pairs from end1 bp 2bp 3bp4bp5 bpSacI- ++ +++ +++ +++ SacI-HF®- + +++ +++ +++SalI- ++ +++ +++ +++ SalI-HF®- ++ +++ +++ +++ SapI+++ +++ +++ +++ +++ Sau3AI+++ +++ +++ +++ +++ SbfI++ +++ +++ +++ +++ SbfI-HF®++ +++ +++ +++ +++ ScaI+++ +++ +++ +++ +++ ScaI-HF®+ +++ +++ +++ +++ SfiI+++ +++ +++ +++ +++ SmaI+++ +++ +++ +++ +++ SpeI+ ++ ++ ++ ++ SpeI-HF®+ ++ ++ ++ ++ SphI-HF®++ ++ +++ +++ +++ SphI+++ +++ +++ +++ +++ SspI+ +++ +++ +++ +++ SspI-HF®+ +++ +++ +++ +++ StuI+++ +++ +++ +++ +++ StyI+ ++ +++ +++ +++ StyI-HF®+ +++ +++ +++ +++ Enzyme Back to top Base Pairs from end1 bp 2bp 3bp4bp5 bpXbaI++ ++ ++ ++ ++XhoI++ ++ ++ +++ +++ XmaI+++ +++ +++ +++ +++。
常见限制性内切酶识别序列

常见限制性内切酶识别序列(酶切位点)(BamHI、EcoRI、HindII I、NdeI、XhoI等)Time:2009-10-22 PM 15:38Author:bioerHits: 7681 times在分子克隆实验中,限制性内切酶是必不可少的工具酶。
无论是构建克隆载体还是表达载体,要根据载体选择合适的内切酶(当然,使用T 载就不必考虑了)。
先将引物设计好,然后添加酶切识别序列到引物5' 端。
常用的内切酶比如Bam HI、EcoRI、HindII I、NdeI、XhoI等可能你都已经记住了它们的识别序列,不过为了保险起见,还是得查证一下。
下面是一些常用的II型内切酶的识别序列,仅供参考。
先介绍一下什么是II型内切酶吧。
The Type II restri ction system s typica lly contai n indivi dualrestri ction enzyme s and modifi catio n enzyme s encode d by separa te genes. The Type II restri ction enzyme s typica lly recogn ize specif ic DNA sequen ces and cleave at consta nt positi ons at or closeto that sequen ce to produc e 5-phosph atesand 3-hydrox yls. Usuall y they requir e Mg 2+ ions as a cofact or, althou gh some have more exotic requir ement s. The methyl trans feras es usuall y recogn ize the same sequen ce althou gh some are more promis cuous. Threetypesof DNA methyl trans feras es have been foundas part of Type II R-M system s formin g either C5-methyl cytos ine, N4-methyl cytos ine or N6-methyl adeni ne.酶类型识别序列ApaIType II restri ctionenzyme5'GGGCC^C 3'BamHIType II restri ctionenzyme5' G^GATCC3'BglIIType II restri ctionenzyme5' A^GATCT3'EcoRIType II restri ctionenzyme5' G^AATTC3'HindII IType II restri ctionenzyme5' A^AGCTT3'KpnIType II restri ctionenzyme5' GGTAC^C 3'NcoIType II restri ctionenzyme5' C^CATGG3' NdeIType II restri ctionenzyme5' CA^TATG 3'NheIType II restri ctionenzyme5' G^CTAGC3'NotIType II restri ctionenzyme5' GC^GGCCGC 3'SacIType II restri ctionenzyme5' GAGCT^C 3'SalIType II restri ctionenzyme5' G^TCGAC3' SphIType II restri ctionenzyme5' GCATG^C 3'XbaIType II restri ctionenzyme5' T^CTAGA3'XhoIType II restri ctionenzyme5' C^TCGAG3'要查找更多内切酶的识别序列,你还可以选择下面几种方法:1. 查你所使用的内切酶的公司的目录或者网站;2. 用软件如:Primer Premie r5.0或Bioe dit等,这些软件均提供了内切酶识别序列的信息;3. 推荐到NEB的REBA SE数据库去查(网址:http://rebas/rebase/rebase.html)当你设计好引物,添加上了内切酶识别序列,下一步或许是添加保护碱基了,可以参考:NEB公司网站提供的关于设计PC R引物保护碱基参考表下载(也可见图片)双酶切buf fer的选择(MBI、罗氏、NEB、Prom eg a、Takara)再给大家推荐一种新的不需要连接反应的分子克隆方法,优点包括:①设计引物不必考虑选择什么酶切位点;②不必考虑保护碱基的问题;③不必每次都选择合适的酶来酶切质粒制备载体;④而且不需要D NA连接酶;⑤假阳性几率低(因为没有连接反应这一步,载体自连的问题没有了)。
