群体进化-基于全基因组重测序
全基因组重测序数据分析详细说明

全基因组重测序数据分析详细说明全基因组重测序(whole genome sequencing, WGS)是一种高通量测序技术,用于获取个体的整个基因组信息。
全基因组重测序数据分析是指对这些数据进行处理、分析和解读,以获得有关个体的遗传变异、基因型、表达和功能等信息。
下面详细说明全基因组重测序数据分析的过程和方法。
首先,全基因组重测序数据的质量控制是必不可少的。
这一步骤包括对测序数据进行质量评估、剔除低质量序列,并进行去除接头序列和过滤序列等预处理操作,以确保后续分析的准确性和可靠性。
接下来,需要对全基因组重测序数据进行序列比对,将读取序列与参考基因组进行比对,以确定每个读取序列在参考基因组上的位置。
常用的比对工具包括Bowtie、BWA、BLAST等。
比对的结果将提供每个读取序列的基因组位置信息。
在序列比对完成后,就可以进行个体的变异检测。
变异检测的目的是识别个体的单核苷酸多态性(single nucleotide polymorphisms, SNPs)、插入缺失变异(insertions/deletions, indels)和结构变异(structural variations, SVs)等基因组变异。
通常,变异检测分为两个步骤:变异发现和变异筛选。
变异发现即根据比对结果,通过一定的算法和统计学原理,找到潜在的变异位点。
然后,利用临床数据库、已知变异数据库和基因功能注释数据库等,进行变异筛选,剔除假阳性和无功能变异,筛选出最有可能的致病变异。
接着,对筛选出的变异位点进行基因型確定。
基因型的确定可以通过直接从比对结果中读取碱基信息,或者通过再次测序来获取高度精确的基因型,以获得更可靠的变异信息。
随后,对变异位点进行注释和功能预测。
注释是指对变异位点进行功能和可能影响的基因、基因组区域和调控元件等进行注释。
常用的注释工具包括ANNOVAR、SnpEff、VEP等。
功能预测则是根据变异位点的位置和可能影响的功能进行预测,如是否影响蛋白质功能、是否在编码序列、是否在启动子或增强子区域等。
基因组重测序

基因组重测序
基因组重测序(Genome Resequencing)是一种研究族群遗传学和物种进化过程的常用分析方法,它包括对个体或物种基因组的重新测序,以及对基因组的遗传变异的进一步探讨。
基因组重测序可以用来研究物种进化,筛选便利性基因以及鉴定和分析基因组变异。
一、优势
1、基因组重测序的比较优势:重测序比利用芯片进行平面分析方法更加灵活。
能够快速鉴定多种类型的遗传变异,包括插入、缺失、临时变异,以及双倍体变异等。
2、复杂性大:由于重测序可以精细分析基因组中的染色体,因此可以更好地捕捉基因组变异的复杂性。
3、高效性:仪器分析周期短,该技术可以高效地获得基因组芯片和组装基因组变异的信息。
二、应用
1、种群遗传研究:基因组重测序能够针对个体或物种基因组的群体变异和单倍型进行分析,以发现先前未被准确定位的遗传标记和位点,有助于预测物种进入新环境时适应性和抗病性方面的变异。
2、育种研究:基因组重测序可以鉴定出品质和适应性相关的基因和位点,有助于精准育种。
3、公共健康:基因组重测序可以确定某种疾病的发病形态,有助于进
一步深入认识疾病的发生机理以及发病的根源,从而促进公共健康的发展。
三、前景
在未来,基因组重测序技术将会被广泛应用于基因组学中,例如用于进化生物学和疾病基因组学研究,它也可用于转基因技术和育种。
同时也会继续发展新的基因组重测序技术,更新、完善重测序技术,为科学家和科技工作者提供更多先进的应用技术。
群体重测序与大山雀的群体进化

诺禾致源最新“高性价比”群体进化研究成果继2013年合作完成地山雀基因组测序之后,北京诺禾致源重测序事业部团队与中国科学院动物研究所研究人员再次携手,通过对13个地区的32只大山雀进行全基因组重测序,解析了喜马拉雅山脉东部大山雀对随海拨变化的气候的适应机制。
研究成果发表于2015年9月的Scientific Reports杂志(IF:5.578)。
其中,中国科学院动物研究所屈延华研究员、诺禾致源田仕林为论文的共同第一作者。
群体重测序揭示大山雀适应随海拨而变的气候的机制NGS项目文章研究背景大山雀(Parus major )隶属于雀形目(Passeriformes)山雀科(Paridae)山雀属(Parus spilonotus )。
在东亚,主要分布在低海拔地区,也有部分种群生活在喜马拉雅山脉东部的高海拔地区。
有季节性迁徙习性,在海拔4000m地区繁殖,在海拔2000m地区越冬。
本研究采用群体重测序技术,从基因组水平上揭示了喜马拉雅山脉东部大山雀的起源及对这种季节性的、随海拨高度变化的气候的适应性机制。
研究方法基于Illumina HiSeq 2000 测序平台,对来自13个地区的32只大山雀进行全基因组重测序,其中,11只大山雀来自喜马拉雅山脉东部地区,11只来自中国中/东部地区,10只来自内蒙古和蒙古,测序深度5X/样。
以近缘物种地山雀(Pseudopodoces humilis )基因组作为参考基因组,对大山雀群体进行了遗传多样性、种群历史动态、选择消除等分析。
研究结果1. 大山雀的群体进化分析群体遗传多样性分析表明,来自蒙古(MON)、东喜马拉雅山脉(EH)和中国中/东地区(CE)的大山雀各自聚为一类。
EH和CE的亲缘关系较MON更近。
大山雀和地山雀约在5.8-13.3百万年前发生了分化;0.7-2.8百万年前大山雀中分化出了MON分支;0.4-1.9百万年前EH和CE发生了分化。
2. 大山雀种群历史动态分析EH、CE和MON在0.3-0.4百万年前种群遭遇了瓶颈效应。
动植物全基因组重测序简介

全基因组重测序是对已知基因组序列的物种进行不同个体的基因组测序,并在此基础上对个体或群体进行差异性分析。
基于全基因组重测序技术,人们可以快速进行资源普查筛选,寻找到大量遗传变异,实现遗传进化分析及重要性状候选基因的预测。
随着测序成本降低和拥有参考基因组序列物种增多,全基因组重测序成为动植物育种和群体进化研究迅速有效的方法。
简化基因组测序技术是对与限制性核酸内切酶识别位点相关的DNA进行高通量测序。
RAD-seq(Restriction-site Associated DNA Sequence)和GBS (Genotyping-by-Sequencing)技术是目前应用最为广泛的简化基因组技术,可大幅降低基因组的复杂度,操作简便,同时不受参考基因组的限制,可快速鉴定出高密度的SNP位点,从而实现遗传进化分析及重要性状候选基因的预测。
简化基因组技术尤其适合于大样本量的研究,可以为利用全基因组重测序技术做深度信息挖掘奠定坚实的基础。
全基因组重测序和简化基因组测序技术可广泛应用于变异检测、遗传图谱构建、功能基因挖掘、群体进化等研究,具有重大的科研和产业价值。
产品脉络图。
全基因组从头测序(de novo测序)

[3] Junjie Qin, Yujun Cui, et al. Open-Source Genomic Analysis of Shiga-Toxin–Producing E. coli O104:H4. N Engl J Med. 2011 Aug 25; 365(8): 718-24.
从头测序(de novo 测序)
从头测序即 de novo 测序,不需要任何参考序列资料即可对某个物种进行测序,用生物信息学分 析方法进行拼接、组装,从而获得该物种的基因组序列图谱。利用全基因组从头测序技术,可以获得 动物、植物、细菌、真菌的全基因组序列,从而推进该物种的研究。一个物种基因组序列图谱的完成, 意味着这个物种学科和产业的新开端!这也将带动这个物种下游一系列研究的开展。全基因组序列图 谱完成后,可以构建该物种的基因组数据库,为该物种的后基因组学研究搭建一个高效的平台;为后 续的基因挖掘、功能验证提供 DNA 序列信息。华大科技利用新一代高通量测序技术,可以高效、低 成本地完成所有物种的基因组序列图谱。
Medicine,NEJM)上在线发表。德国致病性大肠杆菌研究项目首次展示了快速的基因组测序
技术和及时的数据共享给全球各科研领域所带来的巨大贡献,证实了信息数据的快速共享在
公共卫生事件中可发挥至关重要的作用,同时也为应对全球重大突发性紧急公共卫生事件提
供了一个全新的解决思路。
德国肠出血性大肠杆菌项目进展时间轴
全基因组关联分析-基于全基因组重测序
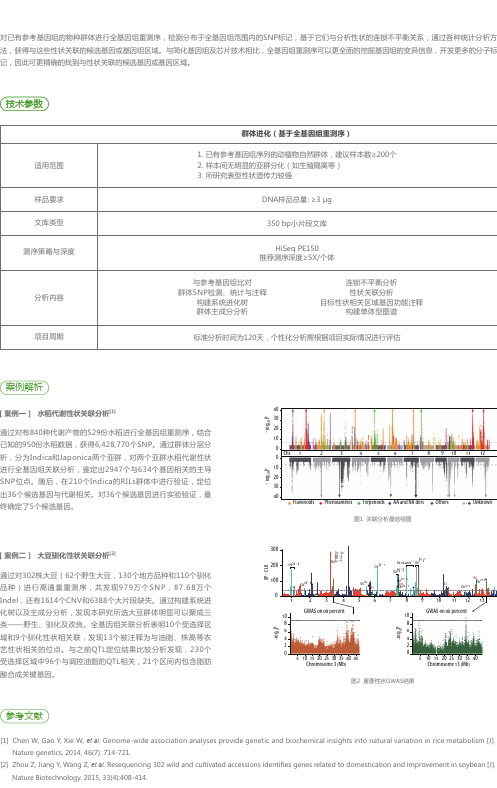
图2 重要性状GWAS结果
参考文献
[1] Chen W, Gao Y, Xie W, et al. Genome-wide association analyses provide genetic and biochemical insights into natural variation in rice metabolism [J]. Nature genetics, 2014, 46(7): 714-721.
对已有参考基因组的物种群体进行全基因组重测序,检测分布于全基因组范围内的SNP标记,基于它们与分析性状的连锁不平衡关系,通过各种统计分析方 法,获得与这些性状关联的候选基因或基因组区域。与简化基因组及芯片技术相比,全基因组重测序可以更全面的挖掘基因组的变异信息,开发更多的分子标 记,因此可更精确的找到与性状关联的候选基因或基因区域。
ቤተ መጻሕፍቲ ባይዱ
与参考基因组比对 群体SNP检测、统计与注释
构建系统进化树 群体主成分分析
连锁不平衡分析 性状关联分析
目标性状相关区域基因功能注释 构建单体型图谱
标准分析时间为120天,个性化分析需根据项目实际情况进行评估
案例解析
[案例一] 水稻代谢性状关联分析[1]
通过对有840种代谢产物的529份水稻进行全基因组重测序,结合 已知的950份水稻数据,获得6,428,770个SNP。通过群体分层分 析,分为Indica和Japonica两个亚群,对两个亚群水稻代谢性状 进行全基因组关联分析,鉴定出2947个与634个基因相关的主导 SNP位点。随后,在210个Indica的RILs群体中进行验证,定位 出36个候选基因与代谢相关。对36个候选基因进行实验验证,最 终确定了5个候选基因。
群体进化-基于简化基因组测序
X. malinche X. birchmanni
ቤተ መጻሕፍቲ ባይዱ
B
X. nezahualcoyotl
X. montezumae
X. nigrensis
X. multilineatus
X. pygmaeus
X. continens
C
X. alvarezi
X. signum
X. mayae
X. hellerii
X. kallmani X. mixei
minor allele frequency
Sword Intermediate No sword
X. meyeri
X. gordoni
X. couchianus
X. variatus
X. evelynae
A
X. milleri
X. xiphidium
X. andersi
X. maculatus
X. cortezi
参考基因组未知
tag聚类、局部组装 SNP检测及统计 构建系统进化树 群体主成分分析 群体遗传结构分析
标准分析为90天,个性化分析需根据项目实际情况进行评估
案例解析
[案例一] RAD-seq 研究剑尾鱼属的系统发生关系[1] 剑尾鱼属(花鳉科)包括26种来自中美洲的热带小型淡水鱼,外 型上最吸引人的莫过于雄鱼的剑尾。关于剑尾鱼属的系统发生关 系一直存在争议,本文用RAD-seq来解决这一问题。26种鱼每种 测5个个体,作为外群的3种鱼每种测2-7个个体,共测序143个个 体,平均每个个体每个位点覆盖深度为15X,找到约66,000个 SNP,以从未有的精度构建了剑尾鱼属的系统发育树。
全基因组重测序数据分析详细说明
全基因组重测序数据分析1. 简介(Introduction)通过高通量测序识别发现de novo的somatic和germ line 突变,结构变异-SNV,包括重排突变(deletioin, duplication 以及copy number variation)以及SNP的座位;针对重排突变和SNP的功能性进行综合分析;我们将分析基因功能(包括miRNA),重组率(Recombination)情况,杂合性缺失(LOH)以及进化选择与mutation之间的关系;以及这些关系将怎样使得在disease(cancer)genome中的mutation产生对应的易感机制和功能。
我们将在基因组学以及比较基因组学,群体遗传学综合层面上深入探索疾病基因组和癌症基因组。
实验设计与样本(1)Case-Control 对照组设计;(2)家庭成员组设计:父母-子女组(4人、3人组或多人);初级数据分析1.数据量产出:总碱基数量、Total Mapping Reads、Uniquely Mapping Reads统计,测序深度分析。
2.一致性序列组装:与参考基因组序列(Reference genome sequence)的比对分析,利用贝叶斯统计模型检测出每个碱基位点的最大可能性基因型,并组装出该个体基因组的一致序列。
3.SNP检测及在基因组中的分布:提取全基因组中所有多态性位点,结合质量值、测序深度、重复性等因素作进一步的过滤筛选,最终得到可信度高的SNP数据集。
并根据参考基因组信息对检测到的变异进行注释。
4.InDel检测及在基因组的分布: 在进行mapping的过程中,进行容gap的比对并检测可信的short InDel。
在检测过程中,gap的长度为1~5个碱基。
对于每个InDel的检测,至少需要3个Paired-End序列的支持。
5.Structure Variation检测及在基因组中的分布: 能够检测到的结构变异类型主要有:插入、缺失、复制、倒位、易位等。
群体进化文章-遗传研究与考古学证据揭示家犬的双重起源说 邮件
2005年伴随着家犬基因组测序的完成,对家犬的研究进入了一个新的阶段,
自此揭开了家犬驯化的神秘面纱。
在人类历史发展的过程中,狗的驯化是非常重要的事件,并且狗可能是第一个受驯化的动物。
狗的驯化过程可划分为两个阶段,第一个阶段是狗从狼中分化出来,
第二个阶段是现代各种各样品种狗的培育,用来满足人类不同的需求。
尽管狗在人类历史中具有重要的作用,但是它们的起源地和驯化时间仍存在争议。
研究背景
通过对古墓中的一条4800年前的爱尔兰犬和其他59条古代狗进行遗传研究表明,
东方的狗和西方的狗分别由当地的狼驯化而来,
后来东方的狗伴随着人类迁往西方,在那里东方狗的遗传基因取代了部分西方狗的遗传基因。
结合遗传研究和考古学证据证实了家犬的双重起源。
研究结论
研究方法
分 析
1. 家犬表型特征分析
2. 群体遗传多样性分析
3. 种群历史动态研究
4. 双起源说的提出
选 材
1条爱尔兰犬(4800年
前)、59条古代狗和
685条现代狗(数据已
报道)。
测 序
Illumina HiSeq2000
阅读原文 >>。
全基因组测序技术和重测序技术
全基因组测序技术和重测序技术全基因组测序技术和重测序技术是现代生物学领域中的两项重要技术,它们的出现和发展对于人类基因研究和生物医学领域的进展起到了重要的推动作用。
全基因组测序技术是指对一个生物体的全部基因组进行测序的技术。
在过去,由于测序技术的限制,只能对一小部分基因进行测序,而全基因组测序技术的出现,使得科学家们能够对整个基因组进行高通量的测序,从而更全面地了解生物体的基因组结构和功能。
全基因组测序技术的发展,不仅提供了大量的基因组数据,也为人类基因组计划等大规模基因组研究项目的实施提供了技术支持。
重测序技术是指对已经测序的基因组进行再次测序的技术。
由于全基因组测序技术的高通量和低成本,科学家们可以对同一个个体的基因组进行多次测序,从而获得更准确和可靠的基因组数据。
重测序技术的应用范围非常广泛,包括个体基因组的变异检测、疾病相关基因的筛查、基因组结构和功能的研究等。
通过重复测序,科学家们可以更好地理解基因组的变异和功能,为疾病的诊断和治疗提供更准确的依据。
全基因组测序技术和重测序技术的发展,对于人类基因研究和生物医学领域的进展带来了巨大的影响。
首先,全基因组测序技术的出现使得科学家们能够更全面地了解基因组的结构和功能,从而揭示了许多与疾病相关的基因变异和功能异常。
其次,重测序技术的应用使得基因组数据的准确性和可靠性得到了提高,为疾病的诊断和治疗提供了更可靠的依据。
此外,全基因组测序技术和重测序技术的发展也为个性化医学的实施提供了技术支持,使得医疗更加精准和个性化。
然而,全基因组测序技术和重测序技术的发展也面临着一些挑战和问题。
首先,由于全基因组测序技术的高通量和低成本,产生的基因组数据量巨大,对数据存储和分析能力提出了更高的要求。
其次,基因组数据的隐私和安全问题也需要引起重视,如何保护个体基因组数据的隐私和安全性是一个亟待解决的问题。
此外,全基因组测序技术和重测序技术的应用还需要进一步完善和标准化,以提高数据的可比性和可重复性。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
DNA样品总量: ≥3 μg 适用范围
样品要求
文库类型测序策略与深度
分析内容项目周期 群体进化(基于全基因组重测序)
标准分析时间为120天,个性化分析需根据项目实际情况进行评估
HiSeq PE150推荐测序深度≥5X/个体350 bp小片段DNA文库
1. 已有参考基因组序列的物种中不同亚群(自然群体)
2. 各亚群间划分明显,同一亚群内的个体有一定代表性
3. 每个亚群选取10个样本左右(推荐动物≥10个,植物≥15个)
4. 总体不少于30个样本与参考基因组比对群体SNP检测、注释及统计系统进化树构建群体遗传结构分析
群体主成分分析连锁不平衡分析选择消除分析候选基因GO和KEGG富集构建单体型图谱种群历史和有效群体大小
技术参数
针对已有参考基因组的物种,对其各亚种进行全基因组重测序获得基因组信息,通过与参考基因组比对,得到大量高准确性的SNP、InDel、SV等变异信息,讨论群体的遗传结构、遗传平衡和影响遗传平衡的因素,从而从分子层面揭示该物种的进化机制、环境适应性等系列问题。
该技术能精准地得到全基因组内所有遗传信息,最大程度地挖掘出群体内遗传变异。
诺禾具有丰富的群体遗传学项目经验,研究成果发表于Nature Genetics(Li, M, et al. 2013& Zhou, XM,
et al. 2014)等。
参考文献
[1] Li M, Tian S, Jin L, et al . Genomic analyses identify distinct patterns of selection in domesticated pigs and Tibetan wild boars [J]. Nature genetics, 2013, 45(12): 1431-1438.
[2] Zhan S, Zhang W, Niitepo ~ld K, et al . The genetics of monarch butterfly migration and warning colouration [J]. Nature, 2014.案例解析
[案例一] 家猪和藏猪的群体进化分析[1]
2013年,诺禾致源科技服务团队与四川农业大学研究者合作发表
该成果。
本研究对6个代表性藏猪群体、5个四川盆地特有猪种,
共48个样本进行全基因组重测序,并结合55个欧亚野猪及家猪的
基因组数据进行群体遗传学分析。
在藏猪中鉴定出低氧适应、能
量代谢等共268个适应高原环境的快速进化基因,揭示了藏猪高
原适应性的遗传机制。
与自然选择相比,人工选择可更有效地塑
造驯养动物基因组;欧亚猪种存在明显的遗传背景差异,欧亚地
理隔离造成的遗传结构差异甚至超过了野生和驯化的差异。
[案例二] 帝王蝶长距离迁飞遗传机制被解密[2]
北美地区的帝王蝶具有迁飞习性,而分布于热带地区的帝王蝶及
其近缘种不具有迁飞特性。
该研究从涵盖当今世界上主要的帝王
蝶分布区域中,选取了包括迁飞型和非迁飞型的22个地理种群、
5个近缘种的101只班蝶属蝴蝶进行了全基因组重测序和群体遗传
学分析。
结果表明,现存的帝王蝶起源于北美地区,且祖先属于
迁飞型,打破了先前认为包括鸟类等在内的迁飞物种均是热带起
源的普遍认知。
其次,利用群体遗传学分析对全基因组进行精细
扫描发现,与飞行相关的肌肉发育进化是帝王蝶实现长距离迁飞
的主要适应性选择。
图1 藏猪及其它猪种的群体遗传结构
图2 帝王蝶样本分布及系统进化树。
