微生物种群的鉴定方法选择
微生物菌种的选育方法
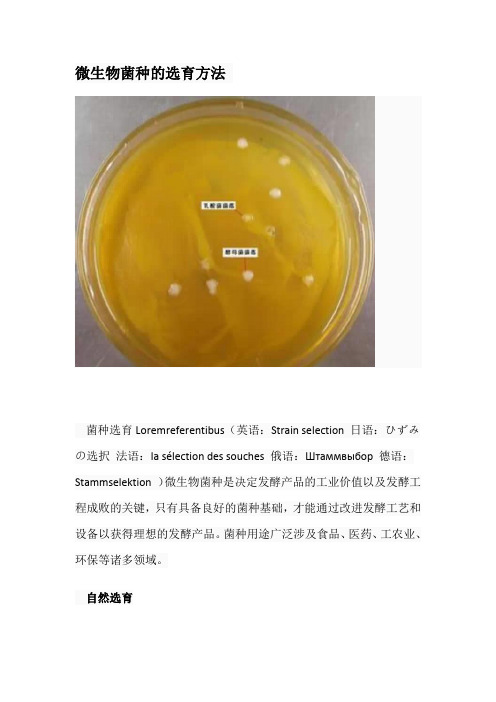
微生物菌种的选育方法菌种选育Loremreferentibus(英语:Strain selection 日语:ひずみの选択法语:la sélection des souches 俄语:Штаммвыбор 德语:Stammselektion )微生物菌种是决定发酵产品的工业价值以及发酵工程成败的关键,只有具备良好的菌种基础,才能通过改进发酵工艺和设备以获得理想的发酵产品。
菌种用途广泛涉及食品、医药、工农业、环保等诸多领域。
自然选育自然选育的菌种来源于自然界、菌种保藏机构或生产过程,从自然界中选育菌种的过程较为复杂,而从生产过程或菌种保藏机构得到菌种的自然选育过程较为简单。
自然选育的步骤主要是:采样,增长培养,培养分离和筛选等。
采样筛选的菌种采集的对象以土壤为主,也可以是植物、腐败物品和某些水域等。
土壤是微生物的汇集地,从土壤中几乎可以分离到任何所需的微生物,故土壤往往是首选的采集目标。
微生物的营养需求和代谢类型与生长环境有很大关系。
富集培养由于采集样品中各种微生物数量有很大差异,若估计到要分离的菌种数量不多时,就要人为增加分离的概率,增加该菌种的数量,称为富集培养。
纯种培养尽管通过增长培养的效果很好,但是得到的微生物还是处于混杂状态,因为样品中本身含有许多种类的微生物。
所以,为了取得所需的微生物纯种,增殖培养后必须进行分离。
平板分离法由接种环以无菌操作沾取少许待分离的材料,在无菌平板表面进行平行划线、扇形划线或其他形式的连续划线,微生物细胞数量将随着划线次数的增加而减少,并逐步分散开来。
如果划线适宜的话,微生物能一一分散,经培养后,可在平板表面得到单菌落。
分离方法有三种:即划线分离法、稀释法和组织分离法。
稀释分离法在溶液中再加入溶剂使溶液的浓度变小。
亦指加溶剂于溶液中以减小溶液浓度的过程。
浓溶液的质量×浓溶液的质量分数=稀溶液的质量×稀溶液的质量分数生产能力考察初筛一般通过平板稀释法获得单个菌落,然后对各个菌落进行有关性状的初步测定,从中选出具有优良性状的菌落。
高通量测序和DGGE分析土壤微生物群落的技术评价

高通量测序和DGGE分析土壤微生物群落的技术评价一、本文概述随着生物技术的发展和深入,土壤微生物群落的研究逐渐受到广泛关注。
作为土壤生态系统的重要组成部分,微生物群落的结构和多样性对土壤健康、生态平衡以及农业可持续发展具有重要影响。
高通量测序技术和DGGE(变性梯度凝胶电泳)分析是近年来在微生物生态学研究中常用的两种技术手段,它们各自具有独特的优势和局限性。
本文旨在全面评价这两种技术在土壤微生物群落研究中的应用效果,以期为相关领域的研究提供有益的参考和借鉴。
本文首先将对高通量测序技术和DGGE分析的基本原理、操作流程及其在土壤微生物群落研究中的应用进行详细介绍。
随后,通过综述已有文献和案例分析,评估这两种技术在揭示土壤微生物群落结构、多样性和动态变化方面的准确性和可靠性。
本文还将探讨这两种技术在实践应用中的优缺点、适用范围及限制条件,以期为研究者在选择合适的技术手段时提供有益的指导。
本文旨在通过深入评价高通量测序和DGGE分析在土壤微生物群落研究中的应用效果,为相关领域的研究提供有价值的参考信息,推动土壤微生物生态学研究的深入发展。
二、高通量测序技术及其在土壤微生物群落分析中的应用高通量测序技术,又被称为下一代测序技术(Next Generation Sequencing,NGS),是近年来生物学领域革命性的技术进步之一。
它允许在单次运行中同时对数百万至数十亿的DNA分子进行测序,极大地提高了测序通量和效率。
在土壤微生物群落分析中,高通量测序技术已成为不可或缺的工具。
高通量测序技术基于边合成边测序的原理,通过桥式PCR扩增生成DNA簇,并利用可逆性终止子的荧光标记核苷酸进行连续测序。
在测序过程中,每加入一种荧光标记的核苷酸,都会通过扫描记录下荧光信号,并切除荧光基团和终止基团,继续进行下一个核苷酸的添加和测序。
这一过程循环进行,直至获得完整的DNA序列信息。
土壤是一个极为复杂的生态系统,其中包含了大量的微生物种类和种群。
菌种鉴定方法大全

菌种鉴定方法大全一、金开瑞菌种鉴定服务简介在细菌/真菌的16/18SrDNA中有多个区段保守性,根据这些保守区可以设计出细菌/真菌通用引物,扩增出细菌/真菌的16/18SrDNA片。
因此,16/18SrDNA可以作为细菌/真菌群落结构分析最常用的系统进化标记分子。
该鉴定手段试用于单种鉴定或者少量混杂菌种鉴定。
金开瑞拥有多种配套的通用菌种鉴定引物,实验周期短,可以帮您快速实现菌种鉴定。
服务流程引物设计合成—PCR扩增16/18S rDNA/ITS—PCR产物纯化—测序,序列比对分析客户提供基因组DNA:要求基因组DNA的浓度≥100 ng/μl,总体积≥20 μl,且无明显降解;平板或斜面:经菌种分离后带有单菌落的新鲜平板或斜面。
最终交付测序结果,BLAST比对得到细菌种属名称服务内容及说明服务名称服务周期(工作日)原核生物/16SrDNA5-7真核生物/18SrDNA5-7真菌ITS序列分析5-7二、菌种鉴定方法介绍(1)常规鉴定常规鉴定内容有形态特征和生理生化特征。
形态特征包括显微形态和培养特征;理化特性包括营养类型、碳氮源利用能力、各种代谢反应、酶反应等。
(2)BIOLOG碳源自动分析鉴定BIOLOG鉴定系统以微生物对不同碳源的利用情况为基础,检测微生物的特征指纹图谱,建立与微生物种类相对应的数据库。
通过软件将待测微生物与数据库参比,得出鉴定结果。
该系统已获美国FDA认可,已逐步应用于食品和饮品企业、环保、海洋生物/水产品、制药、农业微生物、生物治理、化妆品、临床等领域的微生物鉴定试验中。
BIOLOG是一种微生物菌种快速鉴定系统,涉及革兰氏阴性菌、革兰氏阳性菌、厌氧菌、酵母、丝状真菌在内近2000种微生物。
(3)分子生物学鉴定应用分子生物学方法从遗传进化角度阐明微生物种群之间的分类学关系,是目前微生物分类学研究普遍采用的鉴定方法。
CICC拥有微生物菌种分类鉴定的分子生物学实验室,配有PCR仪、高速冷冻离心机、电泳仪、HPLC、凝胶成像系统、紫外控温分析系统等先进仪器设备,以及DNAMAN、BIOEDIT、CLUSTALX、TREEVIEW等序列分析软件。
微生物化验方法

微生物化验方法
微生物化验的方法有很多,以下为您推荐:
1.琼脂平板培养法:因培养基不同,琼脂平板法分为选择性培养基检测法和显色培养基检测法。
选择性培养基是在培养基中加入选择性抑制剂来抑制非目标微生物生长;显色培养基是在培养基中加入细菌特异性酶的显色底物,以菌落颜色区分目的菌落与非目的菌落。
2.显微镜镜检法:将待测样品中的微生物富集后,于油镜下直接计数。
显微镜镜检法通常与琼脂平板培养法结合使用,通过琼脂平板培养法对菌落进行定性分析,再用显微镜进行定量计数。
3.微生物测试片检测技术:一般情况下,微生物测试片由印有网格的聚丙烯薄膜和覆盖有培养基和显色物质的聚乙烯薄膜组成。
待测样品经过处理后可直接接种在微生物测试片上,然后放置在适宜的温度下培养——使固定在测试片上的显色物质与待检微生物生长产生的特异性酶发生显色反应,形成有颜色的菌落,通过对这些菌落进行计数便可实现检测。
微生物群落功能定量检测方法介绍

微生物群落功能定量检测方法介绍微生物是地球上最为广泛存在的生命形式之一,它们存在于各种自然环境中,从陆地到水体,从深海到高山,从北极到沙漠,几乎无处不在。
微生物群落是指一个特定环境中的所有微生物的总体群集,包括细菌、真菌、病毒等。
微生物群落的功能对于维持生态平衡、环境保护和人类健康具有重要意义。
因此,准确地定量检测和描述微生物群落功能是微生物生态学和环境科学领域的重要研究课题之一。
为了实现微生物群落功能的定量检测,研究人员开发了许多先进的技术和方法。
目前,常用的微生物群落功能定量检测方法主要包括基于高通量测序的方法和基于功能基因芯片的方法。
高通量测序方法是一种基于DNA测序技术的方法,可以快速、准确地检测和描述微生物群落的功能。
它利用先进的测序仪器和生物信息学分析工具,对微生物群落中的DNA进行扩增、测序和分析,从而了解其中微生物的种类和数量。
通过对特定功能基因的测序数据进行定量分析,可以有效评估微生物群落的功能水平。
功能基因芯片是另一种常用的微生物群落功能定量检测方法。
它是基于生物芯片技术的一种高通量平行分析技术,可以同时检测数千个功能基因。
功能基因芯片具有高灵敏度、高通量和高特异性的特点,可以准确测定微生物群落的功能丰度和多样性。
除了上述的主要方法外,还有一些其他的微生物群落功能定量检测方法,如荧光原位杂交技术(FISH)、荧光定量PCR(qPCR)等。
这些方法通过针对特定的微生物种群或功能基因的探针或引物进行定量检测,可以快速、高效地获得微生物群落的功能信息。
总结起来,微生物群落功能定量检测是研究微生物生态学和环境科学的关键议题。
目前,基于高通量测序、功能基因芯片和其他分子生物学技术的方法已经成为主流。
这些方法具有快速、准确、高通量和高特异性的特点,能够为我们深入了解微生物群落的功能及其对生态环境的影响提供有力的支持。
微生物群落功能定量检测的研究和应用在环境科学、生态学、医学和农业等领域都具有重要的意义。
微生物鉴别方法

微生物鉴别方法一、微生物鉴别方法——传统方法在传统的分类鉴定中,微生物分类鉴定的主要依据是形态学特征、生理生化反应特征、生态学特征以及血清学反应、对噬菌体的敏感性等。
在鉴定时,我们把这些依据作为鉴定项目,进行一系列的观察和鉴定工作。
1、形态学特征(1)细胞形态在显微镜下观察细胞外形大小、形状、排列等,细胞构造,革兰氏染色反应,能否运动、鞭毛着生部位和数目,有无芽抱和荚膜、芽抱的大小和位置,放线菌和真菌的繁殖器官的形状、构造,抱子的数目、形状、大小、颜色和表面特征等。
(2)群体形态群体形态通常是指以下情况的特征:在一定的固体培养基上生长的菌落特征,包括外形、大小、光泽、黏稠度、透明度、边缘、隆起情况、正反面颜色、质地、气味、是否分泌水溶性色素等;在一定的斜面培养基上生长的菌苔特征,包括生长程度、形状、边缘、隆起、颜色等;在半固体培养基上经穿刺接种后的生长情况;在液体培养基中生长情况,包括是否产生菌膜,均匀浑浊还是发生沉淀,有无气泡,培养基的颜色等。
如是酵母菌,还要注意是成醭状、环状还是岛状。
2、生理生化反应特征(1)利用物质的能力包括对各种碳源利用的能力(能否以C02为唯一碳源、各种糖类的利用情况等)、对各种氮源的利用能力(能否固氮、硝酸盐和铵盐利用情况等)、能源的要求(光能还是化能、氧化无机物还是氧化有机物等)、对生长因子的要求(是否需要生长因子以及需要什么生长因子等)。
(2)代谢产物的特殊性这方面的鉴定项目非常多,如是否产生H2S、吲哚、C02、醇、有机酸,能否还原硝酸盐,能否使牛奶凝固、冻化等。
(3)与温度和氧气的关系测出适合某种微生物生长的温度范围以及它的最适生长温度、最低生长温度和最高生长温度。
对氧气的关系,看它是好氧、微量好氧、兼性好氧、耐氧还是专性厌氧。
3、生态学特征生态学特征主要包括它与其他生物之间的关系(是寄生还是共生,寄主范围以及致病的情况)。
在自然界的分布情况(pH情况、水分程度等)、渗透压情况(是否耐高渗、是否有嗜盐性等)。
什么是微生物筛选方法(一)2024

什么是微生物筛选方法(一)引言概述:微生物筛选方法是指通过筛选作用,从大量微生物群体中选择出具有特定功能或性状的微生物。
微生物筛选方法是研究微生物多样性、发现和利用新的微生物资源、获得新的产物和功能等领域的重要手段。
本文将介绍微生物筛选方法的基本概念及其在科研与应用中的重要性。
正文:一、传统微生物筛选方法1. 传统培养筛选法a. 纯培养法:将微生物分离、纯培养并通过传统菌落特性进行筛选。
b. 特殊培养法:利用特殊培养基、培养条件等筛选特定类型的微生物。
c. 精准培养法:通过精确调控培养条件和培养基组成,获得特定产物或性状的微生物。
2. 生理和生化特性筛选法a. 生理特性筛选法:利用微生物在不同环境条件下的代谢特性进行筛选。
b. 生化特性筛选法:通过筛选微生物在特定生化反应中产生的特定产物。
3. 抗菌活性筛选法a. 抗菌活性筛选法:通过筛选微生物产生的具有抗菌活性的物质。
b. 抗生素产物筛选法:筛选微生物产生的抗生素。
4. 蛋白质和酶活性筛选法a. 蛋白质筛选法:通过筛选微生物产生的具有特定功能的蛋白质。
b. 酶活性筛选法:通过筛选微生物产生的具有特定酶活性的酶。
5. 基因工程筛选法a. 基因组筛选法:利用高通量测序和功能基因组学技术筛选具有特定基因组特征的微生物。
b. 基因表达筛选法:通过筛选具有特定基因表达产物的转基因微生物。
总结:微生物筛选方法是研究微生物多样性和发现新的微生物资源的重要手段。
传统微生物筛选方法主要有传统培养筛选法、生理和生化特性筛选法、抗菌活性筛选法、蛋白质和酶活性筛选法以及基因工程筛选法等。
未来随着技术的不断发展,微生物筛选方法将变得更加高效和精确,为微生物资源的发现和应用提供更多机会。
环境微生物的研究方法

环境微生物的研究方法研究环境微生物的方法可以分为以下几种:1. 培养方法:将环境样品如土壤、水体等放入培养基中,利用适当的条件(如温度、营养物等)培养微生物。
培养出的菌落可以进行分离纯化,并进行形态观察和生理生化特性研究。
2. 分子生物学方法:利用分子生物学技术可以直接从环境样品中提取微生物的核酸,如细菌16S rRNA基因、真菌ITS区域等。
通过PCR扩增、测序和序列分析,可以对微生物进行种类鉴定、多样性分析和进化关系研究。
3. 高通量测序:利用高通量测序技术如Illumina、PacBio等,可以在较短时间内获取大量微生物序列信息。
通过对样品DNA进行测序,可以得到微生物基因组序列、转录组序列等,从而对微生物进行功能、代谢和适应性等方面的研究。
4. 定量PCR:利用定量PCR技术可以对特定微生物种群进行定量分析。
通过选择适当的引物和探针,可以在环境样品中定量检测和监测微生物的数量和变化趋势。
5. 金标法和荧光原位杂交:利用特异性的探针标记微生物种群,可以直接观察微生物在环境中的分布和丰度。
金标法可以通过电镜等方法,将金标记记在目标微生物上,然后通过电子显微镜观察。
荧光原位杂交则利用荧光标记的核酸探针,结合荧光显微镜观察微生物的位置和数量。
6. 气相色谱-质谱联用和高效液相色谱-质谱联用:这些技术可以用于环境样品中微生物代谢物的检测和分析,如挥发性有机物、有机酸等。
通过检测微生物产生的代谢产物,可以了解微生物的生长状态和活性。
7. 其他辅助技术:如电子显微镜观察微生物的形态和结构,荧光显微镜观察微生物的活性和染色体分离等。
微生物学家还可以利用微生物类型文化集合(CCTCC)和国家微生物资源中心(CCTCC)等资源库,进行环境微生物的性状和功能研究。
需要根据研究目的和具体需求选择合适的方法,综合应用多种研究技术可以更全面地了解环境微生物的多样性和功能。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
目前用于湿地微生物群落结构分析的技术主要有平板计数法、荧光染色法、Biolog微平板分析、微生物醌指纹法、磷脂脂肪酸(PLFA)谱图、荧光原位杂交(FISH)技术等。
近年来,变性梯度凝胶电泳、随机扩增多态性DNA标记、单链构象多态性等方法在湿地研究中也开始应用,对湿地中微生物的研究起到显著的推动作用。
以期为利用现代分子技术研究人工湿地中微生物种群净化机理提供参考。
1 PCR-DGGE(Polymerase Chain Reac-tion and Denaturing Gradient Gel Electro-phoresis)1.1 DGGE 原理DGGE(变性梯度凝胶电泳)技术是由Fischer和Lerman于1979年最先提出的,主要是用于检测DNA突变的一种电泳技术。
1993年Muzyers等首次将DGGE 技术应用于分子微生物学研究领域。
DGGE利用序列不同的DNA片段在聚丙烯酰胺凝胶中解链温度不同的原理,通过梯度变性胶将DNA分开。
与其它电泳系统相比,它不是基于核酸分子量的不同将DNA片段分开,而是根据序列的不同将片段大小相同的DNA序列分开。
当双链DNA分子在含梯度变性剂(尿素、甲酰胺)的聚丙烯酰胺凝胶中进行电泳时,其解链的速度和程度与其序列密切相关,相同碱基对数目的双链DNA分子由于碱基对组成的不同,解链所需要的变性剂浓度也不同,当某一双链DNA序列迁移到变性凝胶的一定位置,并达到其解链温度时,即开始部分解链,解链程度越大,迁移阻力大,DNA分子的迁移速度随之减小,产生的迁移阻力与电场力相平衡时,具有不同序列的DNA片段则停留于凝胶的不同位置,形成相互分开的条带图谱。
理论上只要选择的电泳条件足够精细,最低可检测到只有1个碱基差异的DNA片段。
1.2 PCR-DGGE 在人工湿地研究中的应用1.2.1 微生物数量、丰度及多样性Dong等用PCR-DGGE技术鉴别分析用来处理猪圈废水的湿地基质中微生物的情况,得出菌种的分布与总磷、硝酸盐、磷酸盐的浓度显著相关,从进水到出水中微生物的多样性及丰度显著降低,其优势种为Pseudomonas sp.(假单胞菌), Arthrobac-ter sp.(节细菌属), Bacillus sp.(杆状菌)。
通过系统发育分析表明一部分16S rRNA的基因序列与不可培植的反硝化细菌具有极大的相关性,而这些反硝化细菌的活动对湿地中氮的去除起着重要的作用。
Yin等利用DGGE技术分析处理受污染景观湖水的三个水平潜流湿地中微生物的多样性、总的微生物群落的改变以及氨氧化细菌的组成。
通过PCR对携带单加氧酶的基因序列进行扩增,得出季节的变化对微生物群落的多样性和组成具有影响;序列分析说明湿地中氨氧化细菌是不可培养的,其菌群中含有大量的亚硝化单胞菌类似序列。
Guo等利用PCR-DGGE技术分析人工湿地处理二级废水后混合水中的微生物群落的结构,得出微生物菌群的多样性。
Gorra等利用PCR-DGGE技术鉴别处理污水的湿地基质对氨氧化细菌群落多样性和稳定性的影响,结果表明不同基质上群落的组成没有很大的区别,其中沸石最有利于氨氧化细菌的活动。
1.2.2 特定微生物功能群Drewe等通过嵌套的PCR对芦苇湿地进出水、基质及植物根区等15个位点上的Mycobacteriumavium(鸟分支杆菌)进行监测,得出芦苇湿地对鸟分支杆菌具有良好的去除作用,因而对鸟类肺结核具有有效的控制作用。
Park等(2009)通过利用实时PCR技术鉴别和定量测定分别种植菖蒲、黄睡莲、香蒲的表面流人工湿地中脱氮菌和除硫菌的活动状况。
Ruiz-Rueda等通过PCR-DGGE技术鉴别了湿地中阔叶香蒲和芦苇根际的硝化反硝化菌的活动及其群落结构,并判断它与湿地中的植物种类的相关性。
Huang等应用PCR-DGGE技术调查处理富营养观赏水的复合垂直流人工湿地中氨氧化细菌的多样性及活动分布的空间变更,并测定氨氧化细菌在湿地的不同层对富营养水的净化。
Sundberg等将氨氧化和反硝化菌群分别用目标16S rDNA基因探针和功能基因nosZ进行PCR扩增,结果由DGGE及核苷酸序列分析,以检测用来处理高氮浓度的垃圾掩埋浸析液的人工湿地中硝化和反硝化作用及其相应的菌群。
2 LH-PCR (Length Heterogeneity PCR)LH-PCR是于1998年在国外发展起来的一项新技术。
在LH-PCR中,荧光标记探针被用来决定来源于不同微生物扩增序列的相对数量,带标记的片段在电泳胶上被分离出来,而后被一个带有荧光性激光的自动基因顺序分析仪所监测到。
Nancy等(2000)利用LH-PCR技术评估土壤微生物群的组成,并发现LH-PCR 的结果具有可重复性。
Ahn等利用LH-PCR在不同湿地植物和磷负荷下采集人工湿地基质中微生物的指纹,通过对群落菌群克隆和排序来确定其微生物的多样性;并应用主坐标分析及相似性分析得出高磷负荷会使基质微生物群落结构发生显著改变且能降低其多样性,克隆排序表明不同的磷含量产生不同的微生物群落。
3 T-RFLP (Terminal-Restriction Frag-ment Length Polymorphism)T-RFLP(末端限制性酶切片段长度多态性)分析是在PCR技术和RFLP技术基础上发展起来的微生物群落分析技术,所不同的是在PCR扩增16SrRNA基因的过程中,其中一个引物用荧光标记,在计算机程序自动分析时仅分析带荧光标记的末端限制片段,这样使得限制片段长度多态性图谱更为简化,且每个可见的条带都代表一个“核型”或一个分类单元。
相对于其它分子生物学分析技术如RFLP、DGGE等,它具有分辨率高、易于实现自动化等特点。
Nicorarat等利用T-RFLP技术对处理酸化煤矿水的人工湿地系统中微生物的多样性进行分析。
将RFLP的结果与先前的有机体培养PCR-DGGE和FISH研究结合起来,说明了相对低的微生物多样性及嗜温硫杆菌在好氧湿地基质中具有优势。
Ishida等通过T-RFLP方法测定不同的水力负荷及水位波动对湿地底泥中微生物结构的影响,并根据营养物的去除情况确定湿地的性能。
Searcy等利用PCR 扩增每个生物菌膜样本上真核细菌的16SrDNA及亚硝酸还原酶基因,用T-RFLP技术分析扩增产物判断整个菌群及反硝化细菌群落的多样性。
Sleytr等利用T-RFLP技术分析比较芦苇湿地和芒竹湿地植物根区的微生物群落结构,发现不同的湿地在相似的条件下运行具有相似的菌群结构。
4 PCR-SSCP(Single Strand Conforma-tional Polymorphism)4.1 PCR-SSCP 基本原理单链构象多态性技术(简写为SSCP)是随着人类基因组的研究而发展起来用于检测碱基突变的技术。
通过与PCR方法相结合,PCR扩增产物经变性后,将得到具有二级构象的单链DNA置于非变性的聚丙烯酰胺凝胶上进行电泳,空间构象不同的单链DNA会因其迁移率不同而得到分离,最后根据电泳条带所显示的亮度、丰度、条带位置和清晰度等基本信息进行SSCP图谱分析,确定不同菌株类型及其相对数量关系等从而检测相应的遗传变异。
PCR-SSCP技术被认为是环境微生物分子生态学研究中,继变性梯度凝胶电泳(DGGE)和末端限制性片段长度多态性(T-RFLP)之后的又一技术。
4.2 PCR-SSCP 在人工湿地中的应用谢冰等(2007)采用PCR-SSCP技术对上海梦清园芦苇人工湿地的底泥微生物群落结构进行了研究,发现湿地中微生物优势种主要是七种芽孢杆菌。
XIE B 等(2009)再次利用PCR-SSCP图谱分析上海梦清园芦苇人工湿地进出口微生物的多样性,得出异养菌、硝化细菌和反硝化细菌各个季节的分布不一,微生物功能群的分布与湿地中不同营养水平有关。
Vacca等(2005)利用PCR-SSCP图谱分析六个处理生活污水的实验型人工湿地进出口、植物根区及基质不同深度的微生物群落的多样性,以判断湿地中填料类型、植物种植与否对菌群的影响,结果发现不同的微生物群落的存在与基质类型有关。
5 ARDRA (Amplified Ribosomal DNA Restriction Analysis)扩增rDNA限制性酶切片段分析法(简写为ARDRA)的技术原理是基于PCR 技术选择性扩增rDNA 片段( 如16S rDNA 、23S rDNA 、16S-23SrDNA片段),再对扩增的rDNA片段进行限制性酶切片段长度多态性分析(RFLP)。
该方法的步骤是:1)提取总DNA;2)以总DNA为模板,引物为rRNA基因的保守序列,采用PCR技术将特定的rDNA片段扩增出来;3)选择多种有四对碱基识别位点的限制性内切酶,对扩增产物进行限制性酶切;4)用低熔点琼脂糖凝胶电泳分离酶切的DNA片段;5)对ARDRA诊断谱带进行认定、特性的系统分析及计算机聚类。
ARDRA技术一般都需要同其它的分子技术如DGGE、T-RFLP等相结合使用。
Ibekwe等在变化植物密度和组成的湿地中研究微生物群落的组成与水质之间的关系,六个月中每星期对湿地的不同位点的水化学指标、硝酸盐、正磷酸盐、悬浮固体进行检测。
发现50%的植物覆盖率能将96.3%的硝酸盐去除。
采用DGGE获得总的DNA的群落图谱以测定湿地基质中、植物根区及水中主要的微生物组成,并建立菌克隆文库,其中300多个克隆体用ARDRA分析并整合到运算分类单位中,得出湿地基质中、植物根区及水中分别有35、31、36个不同的分类单位,根据香农威尔指数的计算说明50%植物覆盖率的微生物多样性比100%的要高。
土壤微生物群落和数量鉴定的方法在陆地生态系统中,生活在土壤中数量庞大的微生物种群,与植物和动物有着明确的分工,主要扮演“分解者”的角色,几乎参与土壤中一切生物和生物化学反应,担负着地球C、N、P、S 等物质循环的“调节器”土壤养分植物有效性的“转化器和污染环境的”净化器”等多方面生态功能[P]。
土壤微生物是气候和土壤环境条件变化的敏感指标,土壤微生物群落结构和多样性及其变化在一定程度上反映了土壤的质量。
然而,随着近代工农业的发展,大面积土壤受到严重污染,导致了土壤微生物群落结构和多样性的变化,而这种变化会如何影响地上部分和地下部分生态系统的变化尚不清楚。
目前,对污染土壤微生物多样性的研究已引起国内外学者的极大重视。
土壤微生物多样性一般包括微生物分类群的多样性、遗传(基因)多样性、生态特征多样性和功能多样性。
目前,对污染土壤微生物多样性方面的研究仍较薄弱。
一方面是由于测定微生物种类的方法和技术还不是很成熟,不能全面地、准确地研究土壤微生物多样性状况。
