Clussoft高性能软件使用手册
UV-Solution中文说明书1

1.软件功能及窗口介绍1.1. 软件功能本软件包含以下功能:●波长扫描功能●时间扫描功能●定波长测量波长扫描功能1.根据设定以指定波长间隔进行打印。
也可以指定波长进行打印(最高达12点)。
.2.扫描的方式有:透过率(%T);吸光度(Abs);样品光束能量(E(S));参比光束能量(E(R))(仅当连接U-2800和U-2810)3.提供扫描开始和结束的快捷键。
扫描开始:F4;扫描结束:F64.可以设置重复测量的间隔时间。
并可进行长达99次的重复测量。
时间扫描功能1.比率计算功能。
(动力学测定)2.根据设定以指定时间间隔进行打印。
也可以指定时间进行打印(最高达12点)。
定波长测量功能1.包含如下测量及计算功能:定波长分析;面积分析;峰高及峰面积分析;最多达20点的标准样品,一、二、三阶及折线方式回归;2.标准曲线重新测量功能;光标读取标准曲线功能。
3.标准曲线打印功能及保存功能。
4.标准曲线及样品测量数据同时显示,随时观察每个样品的测量结果。
5.显示标准曲线的相关系数值,可以很方便地判定所回归的标准曲线的质量。
并且提供双波长及三波长的回归功能。
6.统计计算功能。
可以进行样品的多次平均测定,可以进行标准样品偏离值(SD)的测定。
7.可以设定极限值,并自动标定出大于极限值的点。
8.定波长测量中可以显示光谱值。
用定波长测量的计算功能可以显示波长扫描中的峰高,峰面积及差值。
数据处理功能测量后经保存的数据可以进行各种数据处理。
1.图谱数据光标检索功能。
2.峰谷值自动判定功能。
3.图谱缩放功能。
4.数据平滑(数据滤波)功能。
5.数据微分处理功能。
6.图谱与图谱,图谱与常数的运算功能。
7.面积计算功能。
8.文件导出功能。
数据可转换成TXT(文本文件),图型可转换成Windows的位图文件。
9.显示字体及打印字体的设置功能。
10.数据列表功能。
11.设定参数打印功能。
12.数据分类打印功能。
如:峰值列表等。
13.坐标栅格打印功能。
ClusVis 1.2.0 用户手册说明书

Package‘ClusVis’October12,2022Type PackageTitle Gaussian-Based Visualization of Gaussian and Non-GaussianModel-Based ClusteringVersion1.2.0Author Christophe Biernacki[aut],Matthieu Marbac[aut,cre],Vincent Vandewalle[aut]Maintainer Matthieu Marbac<*************************>Description Gaussian-Based Visualization of Gaussian and Non-Gaussian Model-Based Cluster-ing done on any type of data.Visualization is based on the probabilities of classification. License GPL(>=2)Imports Rcpp,MASS,parallel,mgcv,mvtnorm,Rmixmod,VarSelLCM(>=2.1)LinkingTo Rcpp,RcppArmadillo,mvtnormByteCompile trueEncoding UTF-8LazyLoad yesLazyData trueCollate'clusvis.R''estimation.R''smartinit.R''RcppExports.R''plot.R''clusvismixmod.R''clusvisvarsellcm.R''modessearch.R'Depends R(>=3.4)RoxygenNote6.1.1NeedsCompilation yesRepository CRANDate/Publication2019-06-2408:30:15UTCR topics documented:ClusVis-package (2)clusvis (4)1clusvisMixmod (6)clusvisVarSelLCM (8)congress (9)plotDensityClusVisu (10)Index12 ClusVis-package Gaussian-Based Visualization of Gaussian and Non-Gaussian Model-Based Clustering.DescriptionThe main function for parameter inference is clusvis.Moreover,specific functions clusvisVarSelLCM and clusvisMixmod are implemented to visualize the results of the R package VarSelLCM and Rmixmod.After parameter inference,visualization is done with function plotDensityClusVisu. DetailsPackage:ClusVisType:PackageVersion: 1.1.0Date:2018-04-18License:GPL-3LazyLoad:yesAuthor(s)Biernacki,C.and Marbac,M.and Vandewalle,V.Examples##Not run:##First example:R package Rmixmod#Package loadingrequire(Rmixmod)#Data loading(categorical data)data("congress")#Model-based clustering with4componentsset.seed(123)res<-mixmodCluster(congress[,-1],4,strategy=mixmodStrategy(nbTryInInit=500,nbTry=25))#Inference of the parameters used for results visualization#(specific for Rmixmod results)#It is better because probabilities of classification are generated#by using the model parametersresvisu<-clusvisMixmod(res)#Component interpretation graphplotDensityClusVisu(resvisu)#Scatter-plot of the observation membershipsplotDensityClusVisu(resvisu,add.obs=TRUE)##Second example:R package Rmixmod#Package loadingrequire(Rmixmod)#Data loading(categorical data)data(birds)#Model-based clustering with3componentsresmixmod<-mixmodCluster(birds,3)#Inference of the parameters used for results visualization(general approach) #Probabilities of classification are not sampled from the model parameter,#but observed probabilities of classification are used for parameter estimation resvisu<-clusvis(log(resmixmod@bestResult@proba),resmixmod@bestResult@parameters@proportions)#Inference of the parameters used for results visualization#(specific for Rmixmod results)#It is better because probabilities of classification are generated#by using the model parametersresvisu<-clusvisMixmod(resmixmod)#Component interpretation graphplotDensityClusVisu(resvisu)#Scatter-plot of the observation membershipsplotDensityClusVisu(resvisu,add.obs=TRUE)##Third example:R package VarSelLCM#Package loadingrequire(VarSelLCM)#Data loading(categorical data)data("heart")#Model-based clustering with3componentsres<-VarSelCluster(heart[,-13],3)#Inference of the parameters used for results visualization#(specific for VarSelLCM results)#It is better because probabilities of classification are generated#by using the model parametersresvisu<-clusvisVarSelLCM(res)#Component interpretation graphplotDensityClusVisu(resvisu)#Scatter-plot of the observation membershipsplotDensityClusVisu(resvisu,add.obs=TRUE)##End(Not run)clusvis This function estimates the parameters used for visualizationDescriptionThis function estimates the parameters used for visualizationUsageclusvis(logtik.estim,prop=rep(1/ncol(logtik.estim),ncol(logtik.estim)),logtik.obs=NULL,maxit=10^3,nbrandomInit=12,nbcpu=1)Argumentslogtik.estim matrix.It contains the probabilities of classification used for parameter infer-ence(should be sampled from the model parameter or computed from the ob-servations).prop vector.It contains the class proportions(by default,classes have same propor-tion).logtik.obs matrix.It contains the probabilities of classification of the clustered sample.If missing,logtik.estim is used.maxit numeric.It limits the number of iterations for the Quasi-Newton algorithm(de-fault1000).nbrandomInit numeric.It defines the number of random initialization of the Quasi-Newton algorithm.nbcpu numeric.It specifies the number of CPU(only for linux)ValueReturns a listExamples##Not run:##First example:R package Rmixmod#Package loadingrequire(Rmixmod)#Data loading(categorical data)data("congress")#Model-based clustering with4componentsset.seed(123)res<-mixmodCluster(congress[,-1],4,strategy=mixmodStrategy(nbTryInInit=500,nbTry=25)) #Inference of the parameters used for results visualization#(specific for Rmixmod results)#It is better because probabilities of classification are generated#by using the model parametersresvisu<-clusvisMixmod(res)#Component interpretation graphplotDensityClusVisu(resvisu)#Scatter-plot of the observation membershipsplotDensityClusVisu(resvisu,add.obs=TRUE)##Second example:R package Rmixmod#Package loadingrequire(Rmixmod)#Data loading(categorical data)data(birds)#Model-based clustering with3componentsresmixmod<-mixmodCluster(birds,3)#Inference of the parameters used for results visualization(general approach)#Probabilities of classification are not sampled from the model parameter,#but observed probabilities of classification are used for parameter estimationresvisu<-clusvis(log(resmixmod@bestResult@proba),resmixmod@bestResult@parameters@proportions)#Inference of the parameters used for results visualization#(specific for Rmixmod results)#It is better because probabilities of classification are generated#by using the model parametersresvisu<-clusvisMixmod(resmixmod)#Component interpretation graphplotDensityClusVisu(resvisu)#Scatter-plot of the observation membershipsplotDensityClusVisu(resvisu,add.obs=TRUE)##Third example:R package VarSelLCM#Package loadingrequire(VarSelLCM)#Data loading(categorical data)data("heart")#Model-based clustering with3componentsres<-VarSelCluster(heart[,-13],3)#Inference of the parameters used for results visualization#(specific for VarSelLCM results)#It is better because probabilities of classification are generated#by using the model parametersresvisu<-clusvisVarSelLCM(res)#Component interpretation graphplotDensityClusVisu(resvisu)#Scatter-plot of the observation membershipsplotDensityClusVisu(resvisu,add.obs=TRUE)##End(Not run)clusvisMixmod This function estimates the parameters used for visualization of model-based clustering performs with R package Rmixmod.To achieve theparameter infernece,it automatically samples probabilities of classi-fication from the model parametersDescriptionThis function estimates the parameters used for visualization of model-based clustering performs with R package Rmixmod.To achieve the parameter infernece,it automatically samples probabili-ties of classification from the model parametersUsageclusvisMixmod(mixmodResult,sample.size=5000,maxit=10^3,nbrandomInit=4*mixmodResult@bestResult@nbCluster,nbcpu=1,loccont=NULL)ArgumentsmixmodResult[MixmodCluster]It is an instance of class MixmodCluster returned by function mixmodCluster of R package Rmixmod.sample.size numeric.Number of probabilities of classification sampled for parameter infer-ence.maxit numeric.It limits the number of iterations for the Quasi-Newton algorithm(de-fault1000).nbrandomInit numeric.It defines the number of random initialization of the Quasi-Newtonalgorithm.nbcpu numeric.It specifies the number of CPU(only for linux).loccont numeric.Index of the column containing continuous variables(only for mixed-type data).ValueReturns a listExamples##Not run:##First example:R package Rmixmod#Package loadingrequire(Rmixmod)#Data loading(categorical data)data("congress")#Model-based clustering with4componentsset.seed(123)res<-mixmodCluster(congress[,-1],4,strategy=mixmodStrategy(nbTryInInit=500,nbTry=25)) #Inference of the parameters used for results visualization#(specific for Rmixmod results)#It is better because probabilities of classification are generated#by using the model parametersresvisu<-clusvisMixmod(res)#Component interpretation graphplotDensityClusVisu(resvisu)#Scatter-plot of the observation membershipsplotDensityClusVisu(resvisu,add.obs=TRUE)##Second example:R package Rmixmod#Package loadingrequire(Rmixmod)#Data loading(categorical data)data(birds)#Model-based clustering with3componentsresmixmod<-mixmodCluster(birds,3)#Inference of the parameters used for results visualization(general approach)#Probabilities of classification are not sampled from the model parameter,8clusvisVarSelLCM #but observed probabilities of classification are used for parameter estimationresvisu<-clusvis(log(resmixmod@bestResult@proba),resmixmod@bestResult@parameters@proportions)#Inference of the parameters used for results visualization#(specific for Rmixmod results)#It is better because probabilities of classification are generated#by using the model parametersresvisu<-clusvisMixmod(resmixmod)#Component interpretation graphplotDensityClusVisu(resvisu)#Scatter-plot of the observation membershipsplotDensityClusVisu(resvisu,add.obs=TRUE)##End(Not run)clusvisVarSelLCM This function estimates the parameters used for visualization of model-based clustering performs with R package Rmixmod.To achieve theparameter infernece,it automatically samples probabilities of classi-fication from the model parametersDescriptionThis function estimates the parameters used for visualization of model-based clustering performs with R package Rmixmod.To achieve the parameter infernece,it automatically samples probabili-ties of classification from the model parametersUsageclusvisVarSelLCM(varselResult,sample.size=5000,maxit=10^3,nbrandomInit=4*varselResult@model@g,nbcpu=1,loccont=NULL) ArgumentsvarselResult[VSLCMresults]It is an instance of class VSLCMresults returned by function VarSelCluster of R package VarSelLCM.sample.size numeric.Number of probabilities of classification sampled for parameter infer-ence.maxit numeric.It limits the number of iterations for the Quasi-Newton algorithm(de-fault1000).nbrandomInit numeric.It defines the number of random initialization of the Quasi-Newton algorithm.nbcpu numeric.It specifies the number of CPU(only for linux).loccont numeric.Index of the column containing continuous variables(only for mixed-type data).congress9ValueReturns a listExamples##Not run:#Package loadingrequire(VarSelLCM)#Data loading(categorical data)data("heart")#Model-based clustering with3componentsres<-VarSelCluster(heart[,-13],3)#Inference of the parameters used for results visualization#(specific for VarSelLCM results)#It is better because probabilities of classification are generated#by using the model parametersresvisu<-clusvisVarSelLCM(res)#Component interpretation graphplotDensityClusVisu(resvisu)#Scatter-plot of the observation membershipsplotDensityClusVisu(resvisu,add.obs=TRUE)##End(Not run)congress Real categorical data set:Congressional Voting Records Data SetDescriptionThis data set includes votes for each of the U.S.House of Representatives Congressmen on the16 key votes identified by the CQA.The CQA lists nine different types of votes:voted for,paired for,and announced for(these three simplified to yea),voted against,paired against,and announced against(these three simplified to nay),voted present,voted present to avoid conflict of interest,and did not vote or otherwise make a position known(these three simplified to an unknown disposition). ReferencesCongressional Quarterly Almanac,98th Congress,2nd session1984,V olume XL:Congressional Quarterly Inc.Washington,D.C.,1985.Schlimmer,J.C.(1987).Concept acquisition through representational adjustment.Doctoral disser-tation,Department of Information and Computer Science,University of California,Irvine,CA.Website:https:///ml/datasets/congressional+voting+records10plotDensityClusVisuExamplesdata(congress)plotDensityClusVisu Function for visualizing the clustering resultsDescriptionFunction for visualizing the clustering resultsUsageplotDensityClusVisu(res,dim=c(1,2),threshold=0.95,add.obs=FALSE,positionlegend="topright",xlim=NULL,ylim=NULL,colset=c("darkorange1","dodgerblue2","black","chartreuse2","darkorchid2","gold2","deeppink2","deepskyblue1","firebrick2","cyan1","red","yellow"))Argumentsres object return by function clusvis or clusvisdim numeric.This vector of size two choose the axes to represent.threshold numeric.It contains the thersholds used for computing the level curves.add.obs boolean.If TRUE,coordinnates of the observations are plotted.positionlegend character.It specifies the legend location.xlim numeric.It specifies the range of x-axis.ylim numeric.It specifies the range of y-axis.colset character.It specifies the colors of the observations per class.Examples##Not run:#Package loadingrequire(Rmixmod)#Data loading(categorical data)data("congress")#Model-based clustering with4componentsset.seed(123)res<-mixmodCluster(congress[,-1],4,strategy=mixmodStrategy(nbTryInInit=500,nbTry=25))#Inference of the parameters used for results visualization#(specific for Rmixmod results)#It is better because probabilities of classification are generated#by using the model parametersresvisu<-clusvisMixmod(res)plotDensityClusVisu11 #Component interpretation graphplotDensityClusVisu(resvisu)#Scatter-plot of the observation membershipsplotDensityClusVisu(resvisu,add.obs=TRUE)##End(Not run)Index∗datasetscongress,9∗packageClusVis-package,2ClusVis(ClusVis-package),2clusvis,2,4,10ClusVis-package,2clusvisMixmod,2,6clusvisVarSelLCM,2,8congress,9MixmodCluster,6plotDensityClusVisu,2,10VSLCMresults,812。
CS3000 系统应用软件操作说明书

CS3000 系统应用软件操作说明书总目录一、前言-------------------------------------------------------------------------- -3-二、流程图画面总目录 ---------------------------------------------------------- -4-三、软件操作说明 --------------------------------------------------------------- -14-1、画面操作 ----------------------------------------------------------------- -14-1-1 总貌画面 ---------------------------------------------------------- -14-1-2 流程图画面 ------------------------------------------------------- -14-1-3 控制分组画面 ---------------------------------------------------- -15-2、仪表操作 ----------------------------------------------------------------- -16-2-1 开关仪表面板 ---------------------------------------------------- -16-2-2 调节仪表面板 ---------------------------------------------------- -173、报表操作 ----------------------------------------------------------------- -17-四、系统操作说明 --------------------------------------------------------------- -18-1、人机接口 ---------------------------------------------------------------- -18-1-1 人机接口功能 ----------------------------------------------------- -18-1-2 窗口类型 ----------------------------------------------------------- -181-3 窗口操作 ----------------------------------------------------------- -19-1-4 典型的操作监视窗口 -------------------------------------------- -192、系统基本维护 ---------------------------------------------------------- -30-2-1 系统维护窗口 ------------------------------------------------------ -30-2-2 过程报告书 --------------------------------------------------------- -332-3 历史报告书 --------------------------------------------------------- -33-3、操作员键盘---------------------------------------------------------------- -34-五、后记------------------------------------------------------------------------------ -35-前言神马特品工业丝计算机分散控制系统采用横河西仪有限公司提供的CENTUM-CS3000 控制系统。
IRSoft PC-Software 指导手册说明书

说明
使用
IRSoft 软件用于分析、处理和存放 testo 880 热成像仪 (下称“ 仪器” )所记录的图像。它还集成了清晰的数据表达报告 功能。 可通过仪器控制器对连接好的热成像仪进行设置。
系统要求
操作系统 软件可以在以下操作系统中运行: • Windows® XP ServicePack 2 (SP2) • Windows Vista 有限功能范围: • Windows® 2000 SP4(无仪器控制器) • Windows® XP,无 SP2 计算机 要使软件顺畅工作,应满足以下要求: • 1.2 GHz 以上的 Pentium 处理器或同等性能的处理器 • 256 MB RAM • 100 MB 硬盘剩余空间 • CD-ROM 驱动器 • USB 2.0 接口 • Internet Explorer 5.5 Service Pack 1 或更高版本
6.7.1. 用户界面 .................................................................................................33 6.7.2. 功能和命令..............................................................................................34 6.7.3. 编辑报告模板 ..........................................................................................34
3 说明 ...................................................................................................6 3.1. 使用 ........................................................................................ 6 3.2. 系统要求 .................................................................................... 6
岛津LCsolution软件快速参考说明书

8.1 查看和使用 [ 数据管理器 ]................................................................................. 32 8.2 查看数据 ........................................................................................................... 34 8.3 使用 CLASS-Agent 查看数据 ........................................................................... 36
岛津 LCsolution
色谱数据系统 快速参考说明书
使用产品前请仔细阅读本说明书。 请妥善保管本说明书以备今后参考。
岛津企业管理(中国)有限公司 分析仪器事业部 分析中心
1
本页空白。
目录
1 2
3 4 5
6 7 8
索引
在阅读此文档前....................................................................................................1
对羟基苯甲酸酯混合物
1.2
LCsolution 的数据结构
本节简要描述了 LCsolution 的数据结构。 各种 LCsolution 数据参数保存在数据文件中,例如在数据采集和数据分析 (元数据)中的仪器和分析条 件。由于可以从数据文件本身参照分析条件和参数,这样就确保了数据的可跟踪性。此外,仅使用数据文 件即可重新分析数据。
VISUAL SLOPE软件操作说明书

微软极品Sysinternals Suite工具包使用指南

注册表项和Windows 服务等资源具有哪种访问权限。
AccessChk 能够通过直观的界面和输出快速回答这些问题。
AccessEnum这一简单但强大的安全工具可以向您显示,谁可以用何种访问权限访问您系统中的目录、文件和注册表项。
使用此工具可查找权限漏洞。
AdExplorerActive Directory Explorer 是一个高级的Active Directory (AD) 查看器和编辑器。
AdInsight一种LDAP(轻型目录访问协议)实时监视工具,旨在对Active Directory 客户端应用程序进行故障排除。
AdRestore恢复已删除的Server 2003 Active Directory 对象。
Autologon登录过程中跳过密码屏幕。
Autoruns查看哪些程序被配置为在系统启动和您登录时自动启动。
Autoruns 还能够完整列出应用程序可以配置自动启动设置的注册表和文件位置。
BgInfo此完全可配置程序会自动生成桌面背景,其中包含有关系统的IP 地址、计算机名称、网络适配器及更多内容的重要信息。
BlueScreen此屏幕保护程序不仅精确模拟“蓝屏”,而且也模拟重新启动(完成CHKDSK),并可在Windows NT 4、Windows 2000、Windows XP、Server 2003 和Windows 9x 上工作。
CacheSetCacheSet 是一个允许您利用NT 提供的功能来控制缓存管理器的工作集大小的程序。
它与NT 的所有版本都兼容。
ClockRes查看系统时钟的分辨率,亦即计时器最大分辨率。
Contig您是否希望迅速对您频繁使用的文件进行碎片整理?使用Contig 优化单个的文件,或者创建连续的新文件。
CoreinfoCoreinfo 是一个新的命令行实用工具,可向您显示逻辑处理器与物理处理器之间的映射、NUMA 节点和它们所处的插槽,以及分配给每个逻辑处理器的缓存。
LXM26 调试软件Somove使用说明
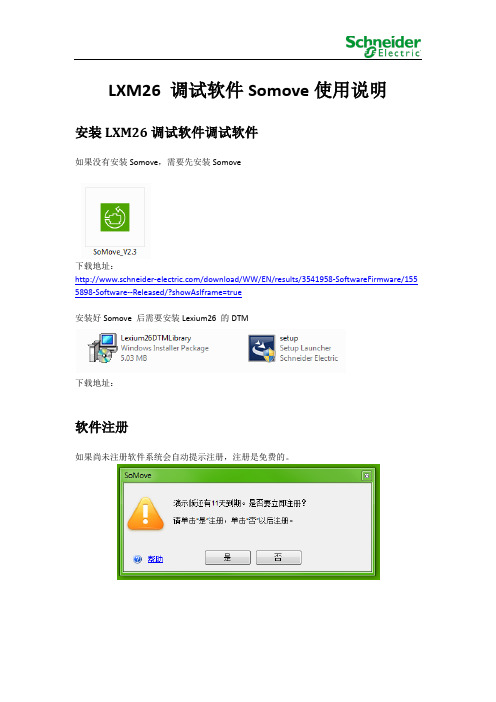
LXM26 调试软件Somove使用说明安装LXM26调试软件调试软件如果没有安装Somove,需要先安装Somove下载地址:/download/WW/EN/results/3541958-SoftwareFirmware/155 5898-Software--Released/?showAsIframe=true安装好Somove 后需要安装Lexium26 的DTM下载地址:软件注册如果尚未注册软件系统会自动提示注册,注册是免费的。
连接电脑Lexium26通过CN3口(modbus485)和电脑进行通讯,施耐德标准通讯线缆型号是TCSMCNAM3M002P,此电缆连接电脑一端为USB口在第一次进行调试时,需要查明电脑分配给此调试电缆的COM口,打开硬件管理器就可以看到,此处为COM4确认COM口后,需要在Somove里面设定对应的COM口,单击编辑链接选择modbus串行,并单击最右边编辑图标选择对应的COM端口,单击应用并确定然后电脑和伺服驱动器的通讯就可以开始了,单击连接选择Lexium26 ,并单击连接参数上载后后可以进行Somove在线调试我的设备用途:我的设备页面用于显示伺服驱动器和电机的基本信息,包括➢驱动器型号➢驱动器序列号➢驱动器固件版本➢电机型号➢驱动器额定/峰值电流➢电机额定/峰值转速➢电机额定/峰值扭矩…参数列表用于:参数列表页面用于设置驱动器P参数,可以按照P参数组开设置,也可以按照操作模式来设置。
相关参数说明可以在Lexium26手册第九章查询错误内存用途:错误内存界面用来查看伺服的故障历史,可以显示当前故障,已经5次历史故障,并且指明故障原因和处理方式。
可视化用途:可视化界面用于显示以下信号的状态或者数值➢数字输出/输出➢模拟量输入/输出➢指定参数的数值指定参数显示需要1.选定要显示的参数2.在右侧区域用鼠标选择显示区域示波器用途:可同时捕捉驱动器内部的最多4个变量,例如电流、电压、位置误差、实际速度等,并绘制成以时间为横坐标的曲线图,以此观察驱动器及电机的运行性能是否符合要求,并相应的做出控制环参数调整。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Clussoft高性能软件使用手册目录1 Clussoft简介 .............................................................................................................................- 3 -1.1 打印帮助说明................................................................................................................- 3 -1.2 查看所有可用软件包....................................................................................................- 3 -1.3 查看软件组....................................................................................................................- 4 -2 软件安装...................................................................................................................................- 6 -2.1 基本配置........................................................................................................................- 6 -2.1.1 设置安装根目录.................................................................................................- 6 -2.1.2 初始化集群环境.................................................................................................- 6 -2.2 根目录结构说明............................................................................................................- 6 -2.3 编译器安装说明............................................................................................................- 7 -2.4 MPI安装说明.................................................................................................................- 7 -2.5 数学库安装说明............................................................................................................- 7 -2.6 测试工具安装说明........................................................................................................- 8 -2.7 应用软件安装说明........................................................................................................- 8 -2.8 安装软件组....................................................................................................................- 9 -2.9 查看可用软件............................................................................................................. - 10 -3 软件使用说明........................................................................................................................ - 12 -3.1 设置软件环境变量..................................................................................................... - 12 -3.1.1 手动加载.......................................................................................................... - 12 -3.1.2 Moudle管理 ..................................................................................................... - 12 -3.2 clusbench使用说明..................................................................................................... - 12 -3.2.1 查看帮助.......................................................................................................... - 12 -3.2.2 单机Linpack测试........................................................................................... - 13 -3.2.3 整机Linpack测试........................................................................................... - 14 -3.2.4 内存测试.......................................................................................................... - 14 -3.3 appbench使用说明 ..................................................................................................... - 15 -3.3.1 查看帮助.......................................................................................................... - 15 -3.3.2 运行lammps测试 ........................................................................................... - 15 -4 Clussoft开发说明 .................................................................................................................. - 18 -4.1 架构说明..................................................................................................................... - 18 -4.2 开发说明..................................................................................................................... - 18 -1Clussoft简介Clussoft可以很方便的部署HPC比较流行的软件,目前该功能已集成在Gridview-3.2.*上,为了与Gridview 中的统一,我们重新编写了Clussoft,使用方式上与yum 比较类似,相比于上一版扩展性更强。
目前,Clussoft提供的自动化软件安装主要分为编译器、数学库、MPI、测试工具和应用软件等五大分类,为了简化安装又从上述几大类软件选取了部分软件分成Applications, base和benchmark三类软件组。
下面就最新版clussoft的使用作详细说明。
1.1打印帮助说明./clussoft --help # 查看基本帮助信息1.2查看所有可用软件包./clussoft -a结果如下图所示:第一列为软件名称和版本,第二列为软件分组,第三列为安装类型(share为共享安装,安装在共享目录下,如/public; distributed 为分布式安装,每个节点安装一份,放在本地磁盘)。
1.3查看软件组Clussoft提供类似yum组安装方式,可一键完成某个组中所有软件的安装,例如,./clussoft -L目前,clussoft分为Applications、base和benchmark三个分组,Applications包含常用应用软件,例如分子动力学、量子化学、开发工具和气象海洋等软件;base包含常用编译器、MPI和数学库等,benchmark包含集群性能测试工具包,如Linpack测试和应用测试。
