与核苷酸和蛋白质序列相关的特征关键词表
专利申请中核苷酸和氨基酸序列表的表述标准

15. 由大序列的一个或多个非连续的片断或来自不同序列的片断组成的核苷酸序列应该 作为带有单独的序列标识符的一个单独序列编号 具有一个或多个缺口的序列应作为具有不 同序列标识符的多个单独序列进行编号 而单独序列的数目在数值上等于序列数据中连续链 的数目
氨基酸序列
使用的符号
16. 蛋白质或肽序列中的氨基酸应该从左到右以氨基到羧基的方向列出 氨基和羧基基团 不应在序列中表示
6. 在申请的说明书 权利要求书或附图中 序列表中出现的序列应该以序列标识符表示 并在前面加上 SEQ ID NO:
7. 核苷酸和氨基酸序列应该以下面三种可能的情况中的至少一种表示
(i) 纯核苷酸序列
(ii) 纯氨基酸序列
(iii) 核苷酸序列与其相应的氨基酸序列一起
对于以上述选项(iii)的格式公开的那些序列 该氨基酸序列必须作为具有独立的整数序列标 识符的纯氨基酸序列在序列表中独立地公开
HE032501
日期 1998.11
标准号 ST. 25
工业产权信息和文献手册
页号 3.25.5
不使用数字 0 在序列下方每隔 5 个氨基酸标明一次编号 氨基酸序列的上述编号方法
也适用于环状构型的氨基酸序列 不同的是该序列的第一个氨基酸的指定可由申请人
作出
22. 由大序列的一个或多个非连续的片断或来自不同序列的片断组成的氨基酸序列应该 作为带有单独的序列标识符的一个单独序列编号 具有一个或多个缺口的序列应作为具有不 同序列标识符的多个单独序列进行编号 而单独序列的数目在数值上等于序列数据中连续链 的数目
HE032501
日期 1998.11
标准号 ST. 25
工业产权信息和文献手册
页号 3.25.2
河大生科院生物信息学考试复习题答案完整版

名词解释1)生物信息学:生物信息学(Bioinformatics)是研究生物信息的采集,处理,存储,传播,分析和解释等各方面的一门学科,它通过综合利用生物学,计算机科学和信息技术而揭示大量而复杂的生物数据所赋有的生物学奥秘。
2)人类基因组计划: 是由美国科学家于1985年率先提出,于1990年正式启动的,宗旨在于测定组成人类染色体(指单倍体)中所包含的30亿个碱基对组成的核苷酸序列,从而绘制人类基因组图谱,并且辨识其载有的基因及其序列,达到破译人类遗传信息的最终目的。
3)基因芯片:又称DNA阵列或DNA芯片是一块带有DNA微阵列(micorarray)的特殊玻璃片或硅芯片片,在数平方厘米之面积上布放数千或数万个核酸探针;检体中的DNA、cDNA、RNA等与探针结合后,借由荧光或电流等方式侦测。
4)中心法则:是指遗传信息从DNA传递给RNA,再从RNA传递给蛋白质,即完成遗传信息的转录和翻译的过程。
也可以从DNA传递给DNA,即完成DNA的复制过程。
5)一级数据库:一级数据库主要包括原始数据,例如DNA序列、蛋白质序列和蛋白质结构等信息。
数据直接来源于实验获得的原始数据,只经过简单的归类整理和注释。
名词辨析1)信息技术与生物信息学:信息技术是研究信息的获取、传输和处理的技术,由计算机技术、通信技术、微电子技术结合而成,即是利用计算机进行信息处理,利用现代电子通信技术从事信息采集、存储、加工、利用以及相关产品制造、技术开发、信息服务的新学科。
生物信息学是研究生物信息的采集,处理,存储,传播,分析和解释等各方面的一门学科,它通过综合利用生物学,计算机科学和信息技术而揭示大量而复杂的生物数据所赋有的生物学奥秘。
2)基因与基因组:基因是指具有遗传效应的DNA片段。
而基因组指的是单倍体细胞中的全套染色体,或是单倍体细胞中的全部基因。
3)相似性与同源性:相似性是指不同染色体之间基因序列的相似或相异程度。
同源性是指两个核酸分子的核苷酸序列或两个蛋白质分子的氨基酸序列间的相似程度。
NCBI中各符号代表的意思

NCBI中各符号代表的意思GenBank 中字符的意思Nucleotide 数据库分为三个子数据库:·EST :表达序列标记数据库·GSS :基因组测序序列数据库·CoreNucleotide :包含所有未被以上两个子数据库收录的核苷酸序列●MeSH: 查询缩写基因的全称3、RefSeq(Reference Sequence)序列接受号:(1)mRNA 记录(NM_*):e.g.:NM_000492(2)基因组的DNA重叠群(NT_*):e.g.:NT_000347(3)完整的基因组或染色体(NC_*):e.g.:NC_000907(4)基因组的局部区域(NG_*):e.g.:NG_000019页脚内容1(5)从人类基因组注释、加工得到的序列模型(XM,XP,or XR_*):e.g.:XM_000483●GenBank记录中特性表中的主要关键词:关键词解释关键词解释misc_feature生物学特性无法用特性表关键词描述的序列promoter转录起始区misc_difference序列特性无法用特性表关键词描述的序列CAAT_signal真核启动子上游的CAAT盒,与RNA结合相关conflict同一序列在不同的研究中在位点或区域上有差异TATA_signal真核启动子的TATA盒unsure序列不能确定的区域-35_signal原核启动子中的-35框old_sequence该序列对以前的版本-10_signal原核启动子的Pribow页脚内容2做过修订盒variation包含稳定突变的序列GC_signal真核启动子的GC盒modified_base修饰过的核苷酸RBS核糖体结合位点gene已识别为基因或已命名的序列区域polyA_signal RNA转录本的剪切识别位点misc_signal无法用信号特性关键词描述的信号序列enhancer增强子关键词解释关键词解释attenuator与转录终止有关的序列CDS蛋白质编码序列terminator转录终止序列sig_peptide编码信号肽的序列rep_origin双链DNA复制起始区transit_peptide转运蛋白编码序列misc_RNA无法用RNA关键词描述的转录物或RNA产物mat_peptide编码成熟肽的序列页脚内容3prim_transcript初始转录本intron内含子precursor_RNA前体RNA polyA_site RNA转录本的多聚腺苷酸化位点mRNA信使RNA rRNA核糖体RNA5’clip前体转录本中被剪切掉的5’端序列tRNA转运RNA3’ clip前体转录本中被剪切掉的3’端序列scRNA小细胞质RNA5’UTR5’非翻译区snRNA小核RNA3’UTR exon 3’非翻译区外显子snoRNA加工和修饰rRNA的小核RNA关键词解释关键词解释immunoglobulin_related repeat_unit单个的重复元件C_region免疫相关蛋白上的不变区LTR长末端重复序列页脚内容4D_segment免疫球蛋白重链的可变区,T细胞受体β链Satellite卫星重复序列J_ segment免疫球蛋白重链、轻链以及T细胞α、β、γ的结合链misc_binding无法描述的核酸序列结合位点N_ region插入重排免疫球蛋白片段间的核苷酸primer_bind复制、转录的引物结合位点S_ region免疫球蛋白重链的开关区protein_bind蛋白质结合区V_ region编码免疫球蛋白的可变区N末端的序列STS测序标签位点V_ segment编码免疫球蛋白的可变区的序列misc_recomb无法用重组特性关键词描述的重组事件repeat_region基因组中所包含的重复序列iDNA通过重组所消除的DNAmisc_structure无法用结构关键词描述的核酸序列高级结构或构型stem_loop发夹结构D_loop线粒体中DNA中的取代页脚内容5环◆GenBank记录中特性表中的限定词:限定词含义限定词含义/allele=给定基因的等位基因/codon_start=相对于序列第一个碱基,编码序列密码子的偏移量/bound_moiety=嵌合范围/country=DNA样本的来源国/cell_type=获得序列的细胞类型/db_xref=其他数据库信息的交叉索引号/citation=已被引用的参考文献数/direction=DNA复制方向/clone_lib=获得序列的克隆文库/environmental_sample=序列直接从环境材料中获得而没有指明来源物种限定词含义限定词含义/exception=指明DNA序列未按通常的生物/PCR_conditi-ons=描述PCR的反应条件学规律翻译,如RNA编辑页脚内容6/frequency=在种群中发生变异的频率/pop_variant=获得序列的群体变异种名称/germline如果序列是DNA并来源于免疫/product=序列编码产物的名称球蛋白家族,则表示该序列来源于未重排DNA/insertion_seq=序列来源于某种插入元件/anticodon=tRNA反义密码子的位置及它所编码的氨基酸/isolate=序列来源的生物个体/cell_line=获得序列的细胞系/chromosome=获得序列的染色体/lab_host=为扩增序列来源物种所用的实验室宿主/clone=获得序列的克隆子/macronuclear指明DNA来源于染色体分化的大核期/note=评论及附加信息/codon=指出与参考密码子不同的密码子/organelle=获得序列的细胞器/EC_number=序列产物的酶学编号/sub_strain=获得序列的来源微生物亚种/transl_table=描述在翻译中与通用密码页脚内容7表不同的密码表/tissue_type=获得序列组织类型/usedin=表明该特性在其他检索中也被使用/translation=按通用或指定的密码子表翻译的氨基酸序列/virion病毒颗粒限定词含义限定词含义/cons_splice=区分内含子剪切位点和“5‘-GT.AG-3'”剪切位点/map=相关特性在基因图谱上的位置/cultivar=所获序列植物的栽培变种/mod_base=被修饰碱基的简写/dev_stage=序列来源于某种生物的特定发育阶段/number=从5’→3’注明遗传元件的顺序/evidence=序列特性来源于实验还是推理/organism=提供测序用遗传物质的物种的科学名称/focus指出在记录中的来源特性在/phenotype=序列特性所导致的表型页脚内容8其他物种中还有不同的来源特性/function=序列所代表的功能/plasmid=获得序列的质粒名称/protein_id=蛋白质的检索号/haplotype=序列来源于某种物种的单倍体/isolation_sou-rce=描述序列来源物种的生理、环/proviral整合在基因组中的前病毒境和地理信息/label=序列特性的俗名/rearranged如果序列是DNA并来源于免疫球蛋白家族,则表示该序列来源于重排DNA限定词含义限定词含义/rpt_family=重复序列/transposon=转座子/variety=获得序列的生物变种/rpt_unit=指明重复区域的重复元件构成/serotype=同一物种的不同血清学特征/pseudo假基因页脚内容9/sex=获得序列的物种性别/replace=表明特性间的间隔序列已被替换/rpt_type=重复序列的组织方式/specimen_vou-cher=指明来源物种保存于什么地方/strain=获得序列的菌珠/sequenced_m-ol=获得序列的分子类型/sub_species=获得序列的来源物种的亚种/serovar=同一原核生物的血清学特征/tissue_lib=获得序列组织库/specific_host=获得序列的天然宿主/standard-name=特性的通用名称/transgenic指明物种的来源特性是否是转基因受体/sub_clone=获得序列的亚克隆/transl_except=标明序列中未按指定密码子表翻译的氨基酸的位置◆BLAST1.blastn (nucleotide blast)是核酸序列到核酸库中的一种查询。
氨基酸密码子对照表

V_segment 免疫球蛋白轻链和重链的可变区段,和 T-细胞受体α ,β 和γ 链;编码大多数可 变区(v_region)和前导肽的最后几个氨基酸
variation 含有来自相同基因的稳定突变的相关系列(例如 RFLP,多态性等),在此(和可能 其它)位置处所述相同基因与被表述的不同
3’clip 在加工过程中被切下的前体转录本 3'端大部分区域
羟基类
色氨酸
Tryptophan W 或 Trp
204.213
Phenyl-NH-CH=C-CH2|___________|
芳香族类
酪氨酸 Tyrosine Y 或 Tyr 181.176 4-OH-Phenyl-CH2-
芳香族类
缬氨酸 Valine
V 或 Val 117.133 CH3-CH(CH2)-
ture
构象
modified_b 被指示的核苷酸是经修饰的核苷酸,并应由被指示的分子(在 mod_base 修饰词意义
ase
中给出)所取代
mRNA
信使 RNA;包括 5'非翻译区(5'UTR),编码序列(CDS,外显子)和3'非翻译区(3
'UTR)
mutation 在此位置处,相关品系的序列中具有突然的,可遗传的变化
小的细胞质 RNA;几个小的细胞质RNA分子中的任何一个存在于真核生物的细胞 质和(有时)核中
sig_peptid 信号肽编码序列;被分泌的蛋白质的N-末端结构域的编码序列;此结构域涉及新
e
生多肽与膜的结合;前导序列
SnRNA
小的核 RNA;很多小的 RNA 种类中的任何一个都被局限于核中;几个 snRNA 参与剪 接或其它 RNA 加工反应
氨基酸密码子对照表

氨基酸密码子对照表酸 e Leu 160 -CH-CH 2 - 类赖氨酸LysineK 或Lys146.17H 2 N-(CH 2 )4 -碱性氨基酸类蛋氨酸MethionineM 或Met149.199CH 3 -S-(CH2 ) 2 -含硫类苯丙氨酸PhenylalanineF 或Phe165.177Phenyl-CH 2 -芳香族类脯氨酸ProlineP 或Pro115.117-N-(CH 2 ) 3-CH-|_________|亚氨基酸丝氨酸SerineS 或Ser105.078HO-CH 2 - 羟基类苏氨酸ThreonineT 或Thr119.105CH 3 -CH(OH)- 羟基类色氨酸TryptophanW 或Trp204.213Phenyl-NH-CH=C-CH 2 -|___________|芳香族类酪氨酸TyrosineY 或Tyr181.1764-OH-Phenyl-CH 2 -芳香族类缬氨酸ValineV 或Val117.133CH 3 -CH(CH2 )-脂肪族类与核苷酸序列相关的特征关键词表 关键词说明allel e 相关的个体或菌株含有相同基因的稳定的其它形式,该形式区别于这一位置的现有的序列(和或许其它序列)atten uator 存在调节转录的终止的DNA 区域,它控制了一些细菌操纵子的表达; (2)位于启动子和第一个结构基因之间,引起转录的部分终止的序列区段C_reg ion 免疫球蛋白轻和重链的恒定区,和T-细胞受体α,β,和γ链;根据特定的链可包括一个或多个外显子CAAT_signa CAAT 盒;位于可能参与RNA 聚合酶结合的真核生物转录单位的起始点的75bp 上游l 的保守序列的一部分;共有序列=GG(C或T)CAATCTCDS 编码序列;对应于蛋白质中的氨基酸序列的核苷酸的序列(位置包括终止密码子);特征包括氨基酸概念上的翻译Confl ict 在这一位点或区域,单独确定的“相同”序列有所不同D-loo p 置换环;线粒体DNA内的一个区域,其中RNA的短的序列与DNA的一条链配对,代替了这一区域的原始配对DNA链;也用于说明在RecA蛋白质催化的反应中,侵入的单链替代双链DNA的一条链的区域D-seg ment 免疫球蛋白重链的多变区,和T-细胞受体的β链Enhan cer 顺式-作用序列,它增强了(一些)真核生物启动子的作用,并能在任一方向和与启动子相关的任何位置处 (上游或下游)起作用Exon 编码剪接mRNA部分的基因组区域;可以含有5'UTR,所有CDS,和3'UTRGC_si gnal GC盒;位于真核生物转录单位起始点上游的保守的富含GC区域,可以以多重拷贝或任一方向存在;共有序列=GGGCGGgene 鉴定为基因的生物学意义的区域,并已经指定名称iDNA 间插DNA;通过几种重组中的任何一种能被消除的DNAintro n 被转录的DNA 区段,但通过同时剪接位于其两侧的序列(外显子)即可从转录本内部将其除去J_seg ment 免疫球蛋白轻链和重链的连接区段,和T-细胞受体α,β和γ链LTR 长的末端重复,在确定序列的两端直接重复的序列, 类型典型地见于逆转录病毒中mat_p eptid e 成熟的肽或蛋白质的编码序列;翻译后修饰之后成熟的或最终的肽或蛋白质产物的编码序列;位置不包括终止密码子(与相应的CDS 不同)misc_bindi ng 不能用任何其它Binding 关键词(primer_bind 或protein_bind)表述的与另一个组成成分共价或非-共价结合的核酸中的位点misc_diffe rence 特征序列与记载中存在的有所不同,并且不能用任何其它不同关键词(conflict,unsure,old_sequence,mutation,variation,allele 或modified_base)表述misc_featu re不能用任何其它的特征关键词表述的具有生物学意义的区域;新的或少见的特征misc_ recom b 任何一般性的,位点特异性的或复制的重组事件的位点,该位点中有不能用其它重组关键词(iDNA和virion)或来源关键词的修饰词(/transposon,/proviral)表述的双螺旋DNA的断裂和愈合misc_ RNA 不能用其他RNA关键词(prim_transcript,precursor_RNA,mRN A,5'clip,3'clip,5'UTR,3'UTR,exon,CD S,sig_peptide,transit____peptide,mat_peptide,intron,polyA_ site,rRNA,tRNA,scRNA和snRNA)限定的任何转录本或RNA产物misc_ signa l 含有控制或改变基因功能或表达之信号的任何区域,所述信号不能用其他Signal 关键词(promoter,CAAT_signal,TATA_signal,-35_signal,10_signal,GC_signal,RBS,p olyA_signal,enhancer,attenuator,ter minator,和rep_origin)表述misc_ struc ture 不能用其他Structure关键词(stem_loop 和D-loop)表述的任何二级或三级结构或构象modif ied_b 被指示的核苷酸是经修饰的核苷酸,并应由被指示的分子(在mod_base修饰词意义ase 中给出)所取代mRNA 信使RNA ;包括5'非翻译区(5'UTR),编码序列(CDS ,外显子)和3'非翻译区(3'UTR )mutat ion 在此位置处,相关品系的序列中具有突然的,可遗传的变化N_reg ion 在重排的免疫球蛋白区段之间插入的额外的核苷酸Old_s equen ce在此位置处,所表述的序列修改了此序列以前的版本PolyA _sign al聚腺苷酸化之后内切核酸酶裂解RNA 转录本所必需的识别区域;共有序列=AATAAA PolyA _site RNA 转录本上的位点,通过转录后聚腺苷酸化该位点将被加上腺嘌呤残基Precu rsor_RNA 仍不是成熟的RNA 产物的任何RNA 种类;可包括5'剪切区(5'clip),5'非翻译区(5'UTR),编码序列(CDS ,外显子),间插序列(内含子),3'非翻译区(3'UTR ),和3'剪切区(3'clip)prim_trans cript 初级(最初的,未加工的)转录本;包括5'剪切区(5'clip ),5'非翻译区(5'UTR ),编码序列(CDS,外显子),间插序列(内含子),3'非翻译区(3'UTR)和3'剪切区(3'clip)prim_bind 起始复制,转录或逆转录的非-共价的引物结合位点;包括合成的例如PCR 引物元件的位点Promo ter 参与RNA 聚合酶的结合以启动转录的DNA 分子区域prote in_bind核酸上非-共价的蛋白质结合位点 RBS 核糖体结合位点repea t_region含有重复单位的基因组区域repea t_unit单个重复元件rep_o rigin 复制起点;复制核酸以得到两个相同拷贝的起始位点RRNA 成熟的核糖体RNA ;将氨基酸装配成蛋白质的核糖核蛋白颗粒(核糖体)中的RNA 成份S_reg ion 免疫球蛋白重链的开关区;它参与重链DNA 的重排,导致来自相同B -细胞的不同免疫球蛋白类的表达Satel lite 短的基本重复单位的很多串联重复(相同或相关的);大多数具有的碱基组成或其它性质与基因组的一般水平不同,这使得它们与大部分(主带)的基因组DNA分离开来ScRNA 小的细胞质RNA;几个小的细胞质RNA分子中的任何一个存在于真核生物的细胞质和(有时)核中sig_p eptid e 信号肽编码序列;被分泌的蛋白质的N-末端结构域的编码序列;此结构域涉及新生多肽与膜的结合;前导序列SnRNA 小的核RNA;很多小的RNA种类中的任何一个都被局限于核中;几个snRNA参与剪接或其它RNA加工反应sourc e 鉴定序列中特定范围的生物来源;此关键词是强制性的;每一项至少要有一个跨越整个序列的单一来源关键词;每个序列可允许有一个以上的来源关键词stem_ loop 发卡结构;由RNA或DNA单链的相邻(反向)互补序列之间的碱基一配对形成的双螺旋区域STS 序列标记位点:表述基因组上作图界标并能通过PCR检测的短的,单拷贝DNA序列;通过测定STS系列的次序即可作出图谱的基因组区域TATA_ signa l TATA盒;Goldberg-Hogness盒;在每个真核生物RNA聚合酶Ⅱ转录单位起点前约25bp处发现的保守的富含AT的七聚体,它可能涉及使酶定位以正确地起始;共有序列=TATA (A或T)A(A或T) termi nator 或者位于转录本的末端或者与启动子区域相邻的DNA 序列,该序列可导致RNA 聚合酶终止转录;也可以是阻抑蛋白的结合位点trans it_pe ptide 转运肽编码序列;核编码的细胞器蛋白质N-末端结构域的编码序列;此结构域参与将蛋白质翻译后运送到细胞器中tRNA 成熟的转移RNA,,小的RNA 分子(75-85个碱基长),介导核酸序列翻译成氨基酸序列unsur e作者不能确定此区域的准确序列 V_reg ion 免疫球蛋白轻链和重链的可变区,和T-细胞受体α,β和γ链;编码可变的氨基末端部分;可由V _segment,D_segment,N_region 和J_segment 组成V_seg ment 免疫球蛋白轻链和重链的可变区段,和T -细胞受体α,β和γ链;编码大多数可变区(v_region )和前导肽的最后几个氨基酸varia tion 含有来自相同基因的稳定突变的相关系列(例如RFLP ,多态性等),在此(和可能其它)位置处所述相同基因与被表述的不同3’cl ip 在加工过程中被切下的前体转录本3'端大部分区域3’UT P 不被翻译成蛋白质的成熟转录本的3'末端区域(终止密码子之后)5’cl ip 在加工过程中被切下的前体转录本5'端大部分区域5’UT P 不被翻译成蛋白质的成熟转录本的5'末端区域(起始密码子之前)_ 10 _sign al Pribnow盒;细菌转录单位起点上游约10bp处的保守区域,它可能参与结合RNA 聚合酶;共有序列=TatAaT_ 35 _sign al 细菌转录单位起点上游约35bp处的保守六聚体;共有序列=TTGACa[]或TGTTGACA[]与蛋白质序列相关的特征关键词表关键词说明CONFLICT 不同的论文报道了不同的序列VARIANT 作者报道存在序列变体VARSLIC 由可选择的剪接产生的序列变体的表述MUTAGEN 经实验操作已改变的位点MOD_RES 残基的翻译后修饰ACETYLATION N -末端或其它AMIDATION 通常位于成熟的活性肽的C -末端BLOCKED 不能被测定的N -或C -末端封闭基团FORMYLATION N -末端甲硫氨酸的GAMMA-CARBOXY-GLUTAMIC ACID HYDROXYLATION天冬酰胺,天冬氨酸,脯氨酸或赖氨酸的METHYLATION 通常为赖氨酸或精氨酸的PHOSPHORYLA TION 丝氨酸,苏氨酸,酪氨酸,天冬氨酸或组氨酸的PYRROLIDONE CARBOXYLICA CID已形成内部环内酰胺的N -末端谷氨酸 SULFATATION 通常为酪氨酸的LIPID 脂质组成成分的共价结合MYRISTATE 通过酰胺键与蛋白质成熟形式的N-末端甘氨酸残基或内部的赖氨酸残基结合的豆蔻酸基团PALMITATE 通过硫酯键与半胱氨酸残基或通过酯键与丝氨酸或苏氨酸残基结合的棕榈酸基团FARNESYL 通过硫酯键与半胱氨酸残基结合的法尼基GERANYL-GER ANYL 通过硫酯键与半胱氨酸残基结合的香叶基-香叶基基团GPI _ANCHOR 与蛋白质成熟形式C-末端残基的α-羧基相连的糖基-磷脂酰肌醇(GPI )基团N _ACYL DIGLYCERIDE 原核生物脂蛋白成熟形式的N -末端半胱氨酸,所述脂蛋白具有酰胺-键联的脂肪酸和通过酯键连接了两个脂肪酸的甘油基DISULFID 二硫键;“FROM ”和“TO ”终点表示通过一个链-内二硫键连接的两个残基;如果“FROM ”和“TO ”终点是完全相同的,则二硫键是链-间键,而说明书领域示出交联的性质THIOLEST 硫醇酯键;“FROM ”和“TO ”终点表示通过硫醇酯键连接的两个残基THIOETH 硫醚键;“FROM ”和“TO ”终点表示通过硫醚键连接的两个残基CARBOHYD 糖基化位点;碳水化物(如果已知)的性质在说明书领域给出METAL 金属离子的结合位点;说明书领域示出金属的性质BINDING 任何化学基团(辅酶,辅基,等等)的结合位点;基团的化学性质在说明书领域给出SIGNAL 信号序列的范围(前肽)TRANSIT 运转肽的范围(线粒体,叶绿体或微体)PROPEP 前肽的范围CHAIN 成熟蛋白质中多肽链的范围PEPTIDE 被释放的活性肽的范围DOMAIN 序列中感兴趣的区域的范围;所述区域的特征在说明书领域给出CA_BIND 钙-结合区域的范围DNA_BIND DNA--结合区域的范围NP_BIND 核苷酸磷酸酯结合区域;核苷酸磷酸酯的特征示于说明书领域TRANSMEM 转膜区域的范围ZN_FING 锌指区域的范围SIMILAR 与另一个蛋白质序列具有相似性的区域;与那个序列有关的精确的资料在说明书领域给出REPEAT 内部序列重复的范围HELIX 二级结构;螺旋,例如α-螺旋,3(10)螺旋,或Pi-螺旋STRAND 二级结构;β-链,例如氢键连接的β-链,或分离的β-桥中的残基TURN 二级结构转角,例如H-键连的转角(3-转角,4-转角或5-转角)ACT_SITE 涉及酶活性的氨基酸SITE 序列中任何其它感兴趣的位点INIT_MET 已知序列以起始密码子甲硫氨酸开始NON_TER 序列末端的残基不是末端残基;如果应用于位置1,这表示第一个位置不是完整分子的N-末端;如果应用于最后一个位置,这表示此位置不是完整分子的C-末端;对此关键词没有说明书领域NON_CONS 非连串残基;表示序列中的两个残基不是连串的,在它们之间有很多末测序的残基UNSURE 序列的不确定性;用于表述不能确定序列排列的序列区域。
氨基酸密码子对照表
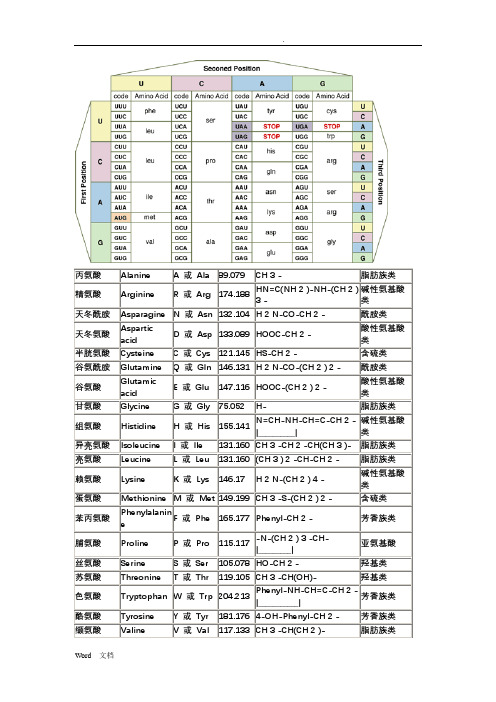
丙氨酸Alanine A 或Ala 89.079 CH 3 - 脂肪族类精氨酸Arginine R 或Arg 174.188 HN=C(NH 2 )-NH-(CH 2 )3 -碱性氨基酸类天冬酰胺Asparagine N 或Asn 132.104 H 2 N-CO-CH 2 - 酰胺类天冬氨酸AsparticacidD 或Asp 133.089 HOOC-CH 2 -酸性氨基酸类半胱氨酸Cysteine C 或Cys 121.145 HS-CH 2 - 含硫类谷氨酰胺Glutamine Q 或Gln 146.131 H 2 N-CO-(CH 2 ) 2 - 酰胺类谷氨酸GlutamicacidE 或Glu 147.116 HOOC-(CH 2 ) 2 -酸性氨基酸类甘氨酸Glycine G 或Gly 75.052 H- 脂肪族类组氨酸Histidine H 或His 155.141 N=CH-NH-CH=C-CH 2 -|__________|碱性氨基酸类异亮氨酸Isoleucine I 或Ile 131.160 CH 3 -CH 2 -CH(CH 3 )- 脂肪族类亮氨酸Leucine L 或Leu 131.160 (CH 3 ) 2 -CH-CH 2 - 脂肪族类赖氨酸Lysine K 或Lys 146.17 H 2 N-(CH 2 ) 4 - 碱性氨基酸类蛋氨酸Methionine M 或Met 149.199 CH 3 -S-(CH 2 ) 2 - 含硫类苯丙氨酸PhenylalanineF 或Phe 165.177 Phenyl-CH 2 - 芳香族类脯氨酸Proline P 或Pro 115.117 -N-(CH 2 ) 3 -CH-|_________|亚氨基酸丝氨酸Serine S 或Ser 105.078 HO-CH 2 - 羟基类苏氨酸Threonine T 或Thr 119.105 CH 3 -CH(OH)- 羟基类色氨酸Tryptophan W 或Trp 204.213 Phenyl-NH-CH=C-CH 2 -|___________|芳香族类酪氨酸Tyrosine Y 或Tyr 181.176 4-OH-Phenyl-CH 2 - 芳香族类缬氨酸Valine V 或Val 117.133 CH 3 -CH(CH 2 )- 脂肪族类。
NCBI中各符号代表的意思

GenBank 中字符的意思Nucleotide 数据库分为三个子数据库:·EST :表达序列标记数据库·GSS :基因组测序序列数据库·CoreNucleotide :包含所有未被以上两个子数据库收录的核苷酸序列●MeSH: 查询缩写基因的全称3、RefSeq(Reference Sequence)序列接受号:(1)mRNA 记录(NM_*):e.g.:NM_000492(2)基因组的DNA重叠群(NT_*):e.g.:NT_000347(3)完整的基因组或染色体(NC_*):e.g.:NC_000907(4)基因组的局部区域(NG_*):e.g.:NG_000019页脚内容1(5)从人类基因组注释、加工得到的序列模型(XM,XP,or XR_*):e.g.:XM_000483●GenBank记录中特性表中的主要关键词:关键词解释关键词解释misc_feature生物学特性无法用特性表关键词描述的序列promoter转录起始区misc_difference序列特性无法用特性表关键词描述的序列CAAT_signal真核启动子上游的CAAT盒,与RNA结合相关conflict同一序列在不同的研究中在位点或区域上有差异TATA_signal真核启动子的TATA盒unsure序列不能确定的区域-35_signal原核启动子中的-35框old_sequence该序列对以前的版本-10_signal原核启动子的Pribow页脚内容2做过修订盒variation包含稳定突变的序列GC_signal真核启动子的GC盒modified_base修饰过的核苷酸RBS核糖体结合位点gene已识别为基因或已命名的序列区域polyA_signal RNA转录本的剪切识别位点misc_signal无法用信号特性关键词描述的信号序列enhancer增强子关键词解释关键词解释attenuator与转录终止有关的序列CDS蛋白质编码序列terminator转录终止序列sig_peptide编码信号肽的序列rep_origin双链DNA复制起始区transit_peptide转运蛋白编码序列misc_RNA无法用RNA关键词描述的转录物或RNA产物mat_peptide编码成熟肽的序列页脚内容3prim_transcript初始转录本intron内含子precursor_RNA前体RNA polyA_site RNA转录本的多聚腺苷酸化位点mRNA信使RNA rRNA核糖体RNA5’clip前体转录本中被剪切掉的5’端序列tRNA转运RNA3’ clip前体转录本中被剪切掉的3’端序列scRNA小细胞质RNA5’UTR5’非翻译区snRNA小核RNA3’UTR exon 3’非翻译区外显子snoRNA加工和修饰rRNA的小核RNA关键词解释关键词解释immunoglobulin_related repeat_unit单个的重复元件C_region免疫相关蛋白上的不变区LTR长末端重复序列页脚内容4D_segment免疫球蛋白重链的可变区,T细胞受体β链Satellite卫星重复序列J_ segment免疫球蛋白重链、轻链以及T细胞α、β、γ的结合链misc_binding无法描述的核酸序列结合位点N_ region插入重排免疫球蛋白片段间的核苷酸primer_bind复制、转录的引物结合位点S_ region免疫球蛋白重链的开关区protein_bind蛋白质结合区V_ region编码免疫球蛋白的可变区N末端的序列STS测序标签位点V_ segment编码免疫球蛋白的可变区的序列misc_recomb无法用重组特性关键词描述的重组事件repeat_region基因组中所包含的重复序列iDNA通过重组所消除的DNAmisc_structure无法用结构关键词描述的核酸序列高级结构或构型stem_loop发夹结构D_loop线粒体中DNA中的取代页脚内容5环◆GenBank记录中特性表中的限定词:限定词含义限定词含义/allele=给定基因的等位基因/codon_start=相对于序列第一个碱基,编码序列密码子的偏移量/bound_moiety=嵌合范围/country=DNA样本的来源国/cell_type=获得序列的细胞类型/db_xref=其他数据库信息的交叉索引号/citation=已被引用的参考文献数/direction=DNA复制方向/clone_lib=获得序列的克隆文库/environmental_sample=序列直接从环境材料中获得而没有指明来源物种限定词含义限定词含义/exception=指明DNA序列未按通常的生物/PCR_conditi-ons=描述PCR的反应条件学规律翻译,如RNA编辑页脚内容6/frequency=在种群中发生变异的频率/pop_variant=获得序列的群体变异种名称/germline如果序列是DNA并来源于免疫/product=序列编码产物的名称球蛋白家族,则表示该序列来源于未重排DNA/insertion_seq=序列来源于某种插入元件/anticodon=tRNA反义密码子的位置及它所编码的氨基酸/isolate=序列来源的生物个体/cell_line=获得序列的细胞系/chromosome=获得序列的染色体/lab_host=为扩增序列来源物种所用的实验室宿主/clone=获得序列的克隆子/macronuclear指明DNA来源于染色体分化的大核期/note=评论及附加信息/codon=指出与参考密码子不同的密码子/organelle=获得序列的细胞器/EC_number=序列产物的酶学编号/sub_strain=获得序列的来源微生物亚种/transl_table=描述在翻译中与通用密码页脚内容7表不同的密码表/tissue_type=获得序列组织类型/usedin=表明该特性在其他检索中也被使用/translation=按通用或指定的密码子表翻译的氨基酸序列/virion病毒颗粒限定词含义限定词含义/cons_splice=区分内含子剪切位点和“5‘-GT.AG-3'”剪切位点/map=相关特性在基因图谱上的位置/cultivar=所获序列植物的栽培变种/mod_base=被修饰碱基的简写/dev_stage=序列来源于某种生物的特定发育阶段/number=从5’→3’注明遗传元件的顺序/evidence=序列特性来源于实验还是推理/organism=提供测序用遗传物质的物种的科学名称/focus指出在记录中的来源特性在/phenotype=序列特性所导致的表型页脚内容8其他物种中还有不同的来源特性/function=序列所代表的功能/plasmid=获得序列的质粒名称/protein_id=蛋白质的检索号/haplotype=序列来源于某种物种的单倍体/isolation_sou-rce=描述序列来源物种的生理、环/proviral整合在基因组中的前病毒境和地理信息/label=序列特性的俗名/rearranged如果序列是DNA并来源于免疫球蛋白家族,则表示该序列来源于重排DNA限定词含义限定词含义/rpt_family=重复序列/transposon=转座子/variety=获得序列的生物变种/rpt_unit=指明重复区域的重复元件构成/serotype=同一物种的不同血清学特征/pseudo假基因页脚内容9/sex=获得序列的物种性别/replace=表明特性间的间隔序列已被替换/rpt_type=重复序列的组织方式/specimen_vou-cher=指明来源物种保存于什么地方/strain=获得序列的菌珠/sequenced_m-ol=获得序列的分子类型/sub_species=获得序列的来源物种的亚种/serovar=同一原核生物的血清学特征/tissue_lib=获得序列组织库/specific_host=获得序列的天然宿主/standard-name=特性的通用名称/transgenic指明物种的来源特性是否是转基因受体/sub_clone=获得序列的亚克隆/transl_except=标明序列中未按指定密码子表翻译的氨基酸的位置◆BLAST1.blastn (nucleotide blast)是核酸序列到核酸库中的一种查询。
NCBI中各符号代表的意思

NCBI中各符号代表的意思GenBank 中字符的意思Nucleotide 数据库分为三个⼦数据库:·EST :表达序列标记数据库·GSS :基因组测序序列数据库·CoreNucleotide :包含所有未被以上两个⼦数据库收录的核苷酸序列●MeSH: 查询缩写基因的全称3、RefSeq(Reference Sequence)序列接受号:(1)mRNA 记录(NM_*):e.g.:NM_000492(2)基因组的DNA重叠群(NT_*):e.g.:NT_000347(3)完整的基因组或染⾊体(NC_*):e.g.:NC_000907(4)基因组的局部区域(NG_*):e.g.:NG_000019(5)从⼈类基因组注释、加⼯得到的序列模型(XM,XP,or XR_*):e.g.:XM_000483●GenBank记录中特性表中的主要关键词:关键词解释关键词解释misc_feature⽣物学特性⽆法⽤特性表关键词描述的序列promoter转录起始区misc_difference序列特性⽆法⽤特性表关键词描述的序列CAAT_signal真核启动⼦上游的CAAT盒,与RNA结合相关conflict同⼀序列在不同的研究中在位点或区域上有差异TATA_signal真核启动⼦的TATA盒unsure序列不能确定的区域-35_signal原核启动⼦中的-35框old_sequence该序列对以前的版本做过修订-10_signal原核启动⼦的Pribow盒variation包含稳定突变的序列GC_signal真核启动⼦的GC盒modified_base修饰过的核苷酸RBS核糖体结合位点gene已识别为基因或已命名的序列区域polyA_signal RNA转录本的剪切识别位点misc_signal⽆法⽤信号特性关键词描述的信号序列enhancer增强⼦关键词解释关键词解释attenuator与转录终⽌有关的序列CDS蛋⽩质编码序列terminator转录终⽌序列sig_peptide编码信号肽的序列rep_origin双链DNA复制起始区transit_peptide转运蛋⽩编码序列misc_RNA⽆法⽤RNA关键词描述的转录物或RNA产物mat_peptide编码成熟肽的序列prim_transcript初始转录本intron内含⼦precursor_RNA前体RNA polyA_site RNA转录本的多聚腺苷酸化位点mRNA信使RNA rRNA核糖体RNA5’clip前体转录本中被剪切掉的5’端序列tRNA转运RNA3’ clip前体转录本中被剪切掉的3’端序列scRNA⼩细胞质RNA5’UTR5’⾮翻译区snRNA⼩核RNA3’UTR exon 3’⾮翻译区外显⼦snoRNA加⼯和修饰rRNA的⼩核RNA关键词解释关键词解释immunoglobulin_related repeat_unit单个的重复元件C_region免疫相关蛋⽩上的不变区LTR长末端重复序列D_segment免疫球蛋⽩重链的可变区,T细胞受体β链Satellite卫星重复序列J_ segment免疫球蛋⽩重链、轻链以及T细胞α、β、γ的结合链misc_binding⽆法描述的核酸序列结合位点N_ region插⼊重排免疫球蛋⽩⽚段间的核苷酸primer_bind复制、转录的引物结合位点S_ region免疫球蛋⽩重链的开关区protein_bind蛋⽩质结合区V_ region编码免疫球蛋⽩的可变区N末端的序列STS测序标签位点V_ segment编码免疫球蛋⽩的可变区的序列misc_recomb⽆法⽤重组特性关键词描述的重组事件repeat_region基因组中所包含的重复序列iDNA通过重组所消除的DNAmisc_structure⽆法⽤结构关键词描述的核酸序列⾼级结构或构型stem_loop 发夹结构D_loop线粒体中DNA中的取代环◆GenBank记录中特性表中的限定词:限定词含义限定词含义/allele=给定基因的等位基因/codon_start=相对于序列第⼀个碱基,编码序列密码⼦的偏移量/bound_moiety=嵌合范围/country=DNA样本的来源国/cell_type=获得序列的细胞类型/db_xref=其他数据库信息的交叉索引号/citation=已被引⽤的参考⽂献数/direction=DNA复制⽅向/clone_lib=获得序列的克隆⽂库/environmental_sample=序列直接从环境材料中获得⽽没有指明来源物种限定词含义限定词含义/exception=指明DNA序列未按通常的⽣物学规律翻译,如RNA编辑/PCR_conditi-ons=描述PCR的反应条件/frequency=在种群中发⽣变异的频率/pop_variant=获得序列的群体变异种名称/germline如果序列是DNA并来源于免疫球蛋⽩家族,则表⽰该序列来源于未重排DNA/product=序列编码产物的名称/insertion_seq=序列来源于某种插⼊元件/anticodon=tRNA反义密码⼦的位置及它所编码的氨基酸/isolate=序列来源的⽣物个体/cell_line=获得序列的细胞系/lab_host=为扩增序列来源物种所⽤的实验室宿主/chromosome=获得序列的染⾊体/macronuclear指明DNA来源于染⾊体分化的⼤核期/clone=获得序列的克隆⼦/note=评论及附加信息/codon=指出与参考密码⼦不同的密码⼦/organelle=获得序列的细胞器/EC_number=序列产物的酶学编号/sub_strain=获得序列的来源微⽣物亚种/transl_table=描述在翻译中与通⽤密码表不同的密码表/tissue_type=获得序列组织类型/usedin=表明该特性在其他检索中也被使⽤/translation=按通⽤或指定的密码⼦表翻译的氨基酸序列/virion病毒颗粒限定词含义限定词含义/cons_splice=区分内含⼦剪切位点和“5‘-GT.AG-3'”剪切位点/map=相关特性在基因图谱上的位置/cultivar=所获序列植物的栽培变种/mod_base=被修饰碱基的简写/dev_stage=序列来源于某种⽣物的特定发育阶段/number=从5’→3’注明遗传元件的顺序/evidence=序列特性来源于实验还是推理/organism=提供测序⽤遗传物质的物种的科学名称/focus指出在记录中的来源特性在其他物种中还有不同的来源特性/phenotype=序列特性所导致的表型/function=序列所代表的功能/plasmid=获得序列的质粒名称/haplotype=序列来源于某种物种的单倍体/protein_id=蛋⽩质的检索号/isolation_sou-rce=描述序列来源物种的⽣理、环境和地理信息/proviral整合在基因组中的前病毒/label=序列特性的俗名/rearranged如果序列是DNA并来源于免疫球蛋⽩家族,则表⽰该序列来源于重排DNA限定词含义限定词含义/rpt_family=重复序列/transposon=转座⼦/rpt_unit=指明重复区域的重复元件构成/variety=获得序列的⽣物变种/serotype=同⼀物种的不同⾎清学特征/pseudo假基因/sex=获得序列的物种性别/replace=表明特性间的间隔序列已被替换/specimen_vou-cher =指明来源物种保存于什么地⽅/rpt_type=重复序列的组织⽅式/strain=获得序列的菌珠/sequenced_m-ol=获得序列的分⼦类型/sub_species=获得序列的来源物种的亚种/serovar=同⼀原核⽣物的⾎清学特征/tissue_lib=获得序列组织库/specific_host=获得序列的天然宿主/transgenic指明物种的来源特性是否是转基因受体/standard-name=特性的通⽤名称/transl_except=标明序列中未按指定密码⼦表翻译的氨基酸的位置/sub_clone=获得序列的亚克隆◆BLAST1.blastn (nucleotide blast)是核酸序列到核酸库中的⼀种查询。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
附录:与核苷酸和蛋白质序列相关的特征关键词表表1 与核苷酸序列相关的特征关键词表关键词说明allele相关的个体或菌株含有相同基因的稳定的其它形式,该形式区别于这一位置的现有的序列(和或许其它序列)attenuator存在调节转录的终止的DNA区域,它控制了一些细菌操纵子的表达;(2)位于启动子和第一个结构基因之间,引起转录的部分终止的序列区段C_region免疫球蛋白轻和重链的恒定区,和T-细胞受体α,β,和γ链;根据特定的链可包括一个或多个外显子CAAT_signal CAAT盒;位于可能参与RNA聚合酶结合的真核生物转录单位的起始点的75bp上游的保守序列的一部分;共有序列=GG(C或T)CAATCTCDS编码序列;对应于蛋白质中的氨基酸序列的核苷酸的序列(位置包括终止密码子);特征包括氨基酸概念上的翻译Conflict在这一位点或区域,单独确定的“相同”序列有所不同D-loop置换环;线粒体DNA内的一个区域,其中RNA的短的序列与DNA的一条链配对,代替了这一区域的原始配对DNA链;也用于说明在RecA蛋白质催化的反应中,侵入的单链替代双链DNA的一条链的区域D-segment免疫球蛋白重链的多变区,和T-细胞受体的β链Enhancer顺式-作用序列,它增强了(一些)真核生物启动子的作用,并能在任一方向和与启动子相关的任何位置处 (上游或下游)起作用Exon编码剪接mRNA部分的基因组区域;可以含有5'UTR,所有CDS,和3'UTRGC_signal GC盒;位于真核生物转录单位起始点上游的保守的富含GC区域,可以以多重拷贝或任一方向存在;共有序列=GGGCGGgene鉴定为基因的生物学意义的区域,并已经指定名称iDNA间插DNA;通过几种重组中的任何一种能被消除的DNAintron被转录的DNA区段,但通过同时剪接位于其两侧的序列(外显子)即可从转录本内部将其除去J_segment免疫球蛋白轻链和重链的连接区段,和T-细胞受体α,β和γ链LTR长的末端重复,在确定序列的两端直接重复的序列, 类型典型地见于逆转录病毒中mat_peptide成熟的肽或蛋白质的编码序列;翻译后修饰之后成熟的或最终的肽或蛋白质产物的编码序列;位置不包括终止密码子(与相应的CDS不同)misc_binding不能用任何其它Binding关键词(primer_bind或protein_bind)表述的与另一个组成成分共价或非-共价结合的核酸中的位点misc_ difference 特征序列与记载中存在的有所不同,并且不能用任何其它不同关键词(conflict, unsure, old_sequence, mutation, variation, allele或modified_base)表述misc_feature不能用任何其它的特征关键词表述的具有生物学意义的区域;新的或少见的特征misc_recomb任何一般性的,位点特异性的或复制的重组事件的位点,该位点中有不能用其它重组关键词(iDNA和virion)或来源关键词的修饰词(/transposon,/proviral)表述的双螺旋DNA的断裂和愈合misc_RNA不能用其他RNA关键词(prim_transcript,precursor_RNA, mRNA, 5'clip, 3'clip, 5'UTR, 3'UTR, exon, CDS, sig_peptide, transit__ __peptide,mat_peptide, intron, polyA_site, rRNA, tRNA, scRNA和snRNA)限定的任何转录本或RNA产物misc_signal含有控制或改变基因功能或表达之信号的任何区域,所述信号不能用其他Signal关键词(promoter, CAAT_signal, TATA_signal, -35_signal,10_signal,GC_signal,RBS,polyA_signal,enhancer,attenuator, terminator和rep_origin)表述misc_structur e 不能用其他Structure关键词(stem_loop和D-loop)表述的任何二级或三级结构或构象modified_base被指示的核苷酸是经修饰的核苷酸,并应由被指示的分子(在mod_base修饰词意义中给出)所取代mRNA信使RNA;包括5'非翻译区(5'UTR),编码序列(CDS,外显子)和3'非翻译区(3'UTR)mutation在此位置处,相关品系的序列中具有突然的,可遗传的变化N_region在重排的免疫球蛋白区段之间插入的额外的核苷酸Old_sequence在此位置处,所表述的序列修改了此序列以前的版本PolyA_signal聚腺苷酸化之后内切核酸酶裂解RNA转录本所必需的识别区域;共有序列=AATAAAPolyA_site RNA转录本上的位点,通过转录后聚腺苷酸化该位点将被加上腺嘌呤残基Precursor_RNA仍不是成熟的RNA产物的任何RNA种类;可包括5'剪切区(5'clip),5'非翻译区(5'UTR),编码序列(CDS,外显子),间插序列(内含子),3'非翻译区(3'UTR),和3'剪切区(3'clip)prim_transcri pt 初级(最初的,未加工的)转录本;包括5'剪切区(5'clip),5'非翻译区(5'UTR),编码序列(CDS,外显子),间插序列(内含子),3'非翻译区(3'UTR)和3'剪切区(3'clip)prim_bind起始复制,转录或逆转录的非-共价的引物结合位点;包括合成的例如PCR引物元件的位点Promoter参与RNA聚合酶的结合以启动转录的DNA分子区域protein_bind核酸上非-共价的蛋白质结合位点RBS核糖体结合位点repeat_region含有重复单位的基因组区域repeat_unit单个重复元件rep_origin复制起点;复制核酸以得到两个相同拷贝的起始位点RRNA成熟的核糖体RNA;将氨基酸装配成蛋白质的核糖核蛋白颗粒(核糖体)中的RNA成份S_region免疫球蛋白重链的开关区;它参与重链DNA的重排,导致来自相同B-细胞的不同免疫球蛋白类的表达Satellite短的基本重复单位的很多串联重复(相同或相关的);大多数具有的碱基组成或其它性质与基因组的一般水平不同,这使得它们与大部分(主带)的基因组DNA分离开来ScRNA小的细胞质RNA;几个小的细胞质RNA分子中的任何一个存在于真核生物的细胞质和(有时)核中sig_peptide信号肽编码序列;被分泌的蛋白质的N-末端结构域的编码序列;此结构域涉及新生多肽与膜的结合;前导序列SnRNA小的核RNA;很多小的RNA种类中的任何一个都被局限于核中;几个snRNA参与剪接或其它RNA加工反应source鉴定序列中特定范围的生物来源;此关键词是强制性的;每一项至少要有一个跨越整个序列的单一来源关键词;每个序列可允许有一个以上的来源关键词stem_loop发卡结构;由RNA或DNA单链的相邻(反向)互补序列之间的碱基一配对形成的双螺旋区域STS序列标记位点:表述基因组上作图界标并能通过PCR检测的短的,单拷贝DNA 序列;通过测定STS系列的次序即可作出图谱的基因组区域TATA_signal TATA盒;Goldberg-Hogness盒;在每个真核生物RNA聚合酶Ⅱ转录单位起点前约25bp处发现的保守的富含AT的七聚体,它可能涉及使酶定位以正确地起始;共有序列=TATA(A或T)A(A或T)terminator或者位于转录本的末端或者与启动子区域相邻的DNA序列,该序列可导致RNA 聚合酶终止转录;也可以是阻抑蛋白的结合位点transit_pepti de 转运肽编码序列;核编码的细胞器蛋白质N-末端结构域的编码序列;此结构域参与将蛋白质翻译后运送到细胞器中tRNA成熟的转移RNA,,小的RNA分子(75-85个碱基长),介导核酸序列翻译成氨基酸序列unsure作者不能确定此区域的准确序列V_region免疫球蛋白轻链和重链的可变区,和T-细胞受体α,β和γ链;编码可变的氨基末端部分;可由V_segment,D_segment,N_region和J_segment组成V_segment免疫球蛋白轻链和重链的可变区段,和T -细胞受体α,β和γ链;编码大多数可变区(v_region)和前导肽的最后几个氨基酸variation含有来自相同基因的稳定突变的相关系列(例如RFLP,多态性等),在此(和可能其它)位置处所述相同基因与被表述的不同3’clip在加工过程中被切下的前体转录本3'端大部分区域3’UTP不被翻译成蛋白质的成熟转录本的3'末端区域(终止密码子之后)5’clip在加工过程中被切下的前体转录本5'端大部分区域5’UTP不被翻译成蛋白质的成熟转录本的5'末端区域(起始密码子之前)_ 10 _signal Pribnow盒;细菌转录单位起点上游约10bp处的保守区域,它可能参与结合RNA 聚合酶;共有序列=TatAaT_ 35 _signal细菌转录单位起点上游约35bp处的保守六聚体;共有序列=TTGACa[]或TGTTGACA[]表2 与蛋白质序列相关的特征关键词表关键词说明CONFLICT不同的论文报道了不同的序列VARIANT作者报道存在序列变体VARSLIC由可选择的剪接产生的序列变体的表述MUTAGEN经实验操作已改变的位点MOD_RES残基的翻译后修饰ACETYLATION N-末端或其它AMIDATION通常位于成熟的活性肽的C-末端BLOCKED不能被测定的N-或C-末端封闭基团FORMYLATION N-末端甲硫氨酸的GAMMA-CARBOXY-GLUTAMIC天冬酰胺,天冬氨酸,脯氨酸或赖氨酸的ACID HYDROXYLATIONACID HYDROXYLATIONMETHYLATION通常为赖氨酸或精氨酸的PHOSPHORYLATION丝氨酸,苏氨酸,酪氨酸,天冬氨酸或组氨酸的PYRROLIDONECARBOXYLICACID已形成内部环内酰胺的N-末端谷氨酸SULFATATION通常为酪氨酸的LIPID脂质组成成分的共价结合MYRISTATE通过酰胺键与蛋白质成熟形式的N-末端甘氨酸残基或内部的赖氨酸残基结合的豆蔻酸基团PALMITATE通过硫酯键与半胱氨酸残基或通过酯键与丝氨酸或苏氨酸残基结合的棕榈酸基团FARNESYL通过硫酯键与半胱氨酸残基结合的法尼基GERANYL-GERANYL通过硫酯键与半胱氨酸残基结合的香叶基-香叶基基团GPI_ANCHOR与蛋白质成熟形式C-末端残基的α-羧基相连的糖基-磷脂酰肌醇(GPI)基团N_ACYL DIGLYCERIDE 原核生物脂蛋白成熟形式的N-末端半胱氨酸,所述脂蛋白具有酰胺-键联的脂肪酸和通过酯键连接了两个脂肪酸的甘油基DISULFID二硫键;“FROM”和“TO”终点表示通过一个链-内二硫键连接的两个残基;如果“FROM”和“TO”终点是完全相同的,则二硫键是链-间键,而说明书领域示出交联的性质THIOLEST硫醇酯键;“FROM”和“TO”终点表示通过硫醇酯键连接的两个残基THIOETH硫醚键;“FROM”和“TO”终点表示通过硫醚键连接的两个残基CARBOHYD糖基化位点;碳水化物(如果已知)的性质在说明书领域给出METAL金属离子的结合位点;说明书领域示出金属的性质BINDING任何化学基团(辅酶,辅基,等等)的结合位点;基团的化学性质在说明书领域给出SIGNAL信号序列的范围(前肽)TRANSIT运转肽的范围(线粒体,叶绿体或微体)PROPEP前肽的范围CHAIN成熟蛋白质中多肽链的范围PEPTIDE被释放的活性肽的范围DOMAIN序列中感兴趣的区域的范围;所述区域的特征在说明书领域给出CA_BIND钙-结合区域的范围DNA_BIND DNA--结合区域的范围NP_BIND核苷酸磷酸酯结合区域;核苷酸磷酸酯的特征示于说明书领域TRANSMEM转膜区域的范围ZN_FING锌指区域的范围SIMILAR与另一个蛋白质序列具有相似性的区域;与那个序列有关的精确的资料在说明书领域给出REPEAT内部序列重复的范围HELIX二级结构;螺旋,例如α-螺旋,3(10)螺旋,或Pi-螺旋STRAND二级结构;β-链,例如氢键连接的β-链,或分离的β-桥中的残基TURN二级结构转角,例如H-键连的转角(3-转角,4-转角或5-转角)ACT_SITE涉及酶活性的氨基酸SITE序列中任何其它感兴趣的位点INIT_MET已知序列以起始密码子甲硫氨酸开始NON_TER序列末端的残基不是末端残基;如果应用于位置1,这表示第一个位置不是完整分子的N-末端;如果应用于最后一个位置,这表示此位置不是完整分子的C-末端;对此关键词没有说明书领域NON_CONS非连串残基;表示序列中的两个残基不是连串的,在它们之间有很多末测序的残基UNSURE序列的不确定性;用于表述不能确定序列排列的序列区域。
