第6章 蛋白质生物信息学
生物信息学实验报告3(三)蛋白质序列分析

⽣物信息学实验报告3(三)蛋⽩质序列分析(三)蛋⽩质序列分析实验⽬的:掌握蛋⽩质序列检索的操作⽅法,熟悉蛋⽩质基本性质分析,了解蛋⽩质结构分析和预测。
实验内容:1、检索SOX-21蛋⽩质序列,利⽤ProParam⼯具进⾏蛋⽩质的氨基酸组成、分⼦质量、等电点、氨基酸组成、原⼦总数及疏⽔性(ProtScale⼯具)等理化性质的分析。
2、利⽤PredictProtein、PROF、HNN等软件预测分析蛋⽩质的⼆级结构;利⽤Scan Prosite软件对蛋⽩质进⾏结构域分析。
3、利⽤TMHMM、TMPRED、SOSUI等⼯具对蛋⽩质进⾏跨膜分析;采⽤PredictNLS进⾏核定位信号分析;利⽤PSORT进⾏蛋⽩质的亚细胞定位预测;利⽤CBS(http://www.cbs.dtu.dk/services/ProtFun/)⽹站⼯具预测蛋⽩的功能,将序列⽤Blocks、SMART、InterProScan、PFSCAN等搜索其保守序列的特征,进⾏motif 的结构分析。
4、利⽤Swiss-Model数据库软件预测该蛋⽩的三级结构,结果⽤蛋⽩质三维图象软件Jmol查看。
CPHmodels 也是利⽤神经⽹络进⾏同源模建预测蛋⽩质结构的⽅法和⽹络服务器I-TASSER预测所选蛋⽩质的空间结构。
5、分析蛋⽩质的翻译后修饰:分析信号肽及其剪切位点: SignalIP http://www.cbs.dtu.dk/services/SignalP/;分析糖链连接点:分析O-连接糖蛋⽩,NetOGlyc,http://www.cbs.dtu.dk/services/NetOGlyc/;分析N-连接糖蛋⽩,NetNGlyc,http://www.cbs.dtu.dk/services/NetNGlyc/。
6、利⽤检索的序列,进⾏同源⽐对,获得并分析⽐对结果。
实验步骤(⼀)1、在NCBI 蛋⽩质数据库中查找SOX-21蛋⽩质序列分别选择⽖蟾(Xenopus laevis)、⼩家⿏[Mus musculus]、猕猴[Macaca mulatt a]的SOX-21蛋⽩质序列,并保存其FASTA格式。
生物信息学 第六章 蛋白质结构预测及分子设计ppt课件

更多有用的链接
▪ PDB的外部链接中Compute pI Mw点击Chain B (可计算各链分子 量)
▪ 在打开的Compute pI/Mw页面中点击EX5B_ECOLI (ExPASy,大 量信息,链接)
▪ 在打开的UniProtKB/Swiss-Prot页面中点击EcoCyc:EG10824MONOMER (biocyc,参与的反应/路径图)
3、输入要找的蛋白名称或ID号等(如RecBCD, E. coli DNA repair)
4、点击”Go” 5、点击感兴趣的结果(1W36,进入MMDB) 结果列表中包含相关蛋白(powered by BLAST)、文献、结构域 (domain)、配体(ligand)、3D缩略图、三维查看器
在MMDB看搜到蛋白的结构(NCBI)
实验数据
数据库搜索
结构域匹配
已知结构的 同源蛋白?
有
同源 建模
无 二级
结构预测 有
串线法
三维结构模型
可用的折 叠模型?
无
从头 预测
蛋白质的基本性质
蛋白质的基本性质:
相对分子质量 氨基酸组成 等电点(pI) 消光系数
半衰期
不稳定系数 总平均亲水性 …….
工具 AACompldent
Compute pI/Mw
蛋白质跨膜区特性 ▪ 典型的跨膜螺旋区主要是由20~30个疏水性氨基酸(Leu、Ile、Val、Met、Gly、
Ala等)组成; ▪ 亲水残基往往出现在疏水残基之间,对功能有重要的作用; ▪ 基于亲/疏水量和蛋白质跨膜区每个氨基酸的统计学分布偏好性。 跨膜蛋白序列“边界”原则 ▪ 胞外末端:Asp(天冬氨酸)、Ser(丝氨酸)和Pro(脯氨酸) ▪ 胞外-内分界区:Trp(色氨酸) ▪ 跨膜区:Leu(亮氨酸)、Ile(异亮氨酸)、Val(缬氨酸)、Met(甲硫氨酸
生物信息学在蛋白质组学中的应用

生物信息学在蛋白质组学中的应用生物信息学是一门研究生物大分子信息的学科,通过计算机技术和信息科学的手段,对生物大分子的结构、功能和演化进行分析。
而蛋白质组学则是研究生物体内所有蛋白质的组成、结构和功能的学科。
两者的结合,引领着生命科学的革命。
生物信息学在蛋白质组学中的应用,让我们可以更加全面地了解和掌握蛋白质的结构和功能,而这对于科学研究和医学应用均有巨大的推动作用。
下面,我们将具体探讨生物信息学在蛋白质组学中的应用。
一. 蛋白质结构预测蛋白质的结构形态是其功能的决定因素之一,因此,预测蛋白质的结构形态,是理解其生物学功能的重要前提。
蛋白质结构预测作为生物信息学的一个重要分支,在很大程度上实现了无需实验即可预测蛋白质的结构。
生物信息学中,蛋白质结构预测主要通过构建三维结构预测模型,在预测蛋白的空间结构中发挥重要作用。
例如,alphaFold的发明使得结构预测的准确率大大提高,并促进了新型药物开发的进展。
二. 蛋白质分子演化研究蛋白质分子演化研究可揭示物种的进化历程、适应策略及其生物功能的变化,为研究生物进化提供了强有力的支持。
生物信息学中,通过基础序列、编码序列等方面的比对,可对蛋白质分子的演化进行系统研究。
蛋白质序列比对是生物信息学中的一项重要技术,可通过比对基因组任务与蛋白质的序列,确定蛋白质分子的演化历程。
而在基于比较基因组的全基因组分析上,生物信息技术能够通过分析基因间的各种相互作用、协同作用等,预测和分析蛋白质进化后的功能、异常活性等,为相关分子的研究提供了重要的启示。
三. 靶向药物设计靶向药物设计,是指通过研究靶点的结构、构象及其动态特征,设计新型药物分子以治疗相关疾病。
生物信息学在靶向药物设计中的应用主要包括分子对接、虚拟筛选、药物分子分析等方面。
分子对接技术能够基于生物分子的三维结构,预测其与其他分子之间的相互作用过程,从而验证确保新型药物,合理性以及药效稳定性。
而虚拟筛选是指在筛选化合物的过程中,通过计算机模拟技术进行模型建模,模拟实验与研究,选择出药物阶段,为临床的治疗进展理论基础提供了重要的保障。
蛋白质相互作用的生物信息学

Mass spectrometry of purified
complexes
• Benefits:
– several members of a complex can be tagged, giving an internal check for consistency;
– and it detects real complexes in physiological settings.
THE DIP DATABASE
• Database of Interacting Proteins • The DIP database catalogs
experimentally determined interactions between proteins.
DIP相互作用的表达
Nucleic Acids Research, 2000, 28, 289-291
Expression Profile Reliability
• EPR IndexExpression Profile Reliability Index (EPR Index) evaluates the quality of a large-scale protein-protein interaction data sets by comparing the expression profile of the interacting dataset with that of the high-quality subset of the DIP database.
• Drawbacks:
– it is a powerful method for discriminating cell states or disease outcomes, but is a relatively inaccurate predictor of direct physical interaction;
蛋白质生物信息学

蛋白质生物信息学
蛋白质生物信息学是指应用计算机科学和数学方法,研究蛋白质的结构、功能和互作关系,并将这些信息应用于生物学研究中的一门学科。
蛋白质是生命体中最重要的分子之一,具有广泛的生物功能,在疾病诊断、药物研发、食品安全等领域都有着重要的应用价值。
蛋白质生物信息学主要包括蛋白质序列分析、蛋白质结构预测、蛋白质功能预测、蛋白质相互作用网络分析等内容。
其中,蛋白质序列分析是研究蛋白质基本构成和序列特征的方法;蛋白质结构预测则是通过计算方法来预测蛋白质的三维结构;蛋白质功能预测则是根据蛋白质的序列、结构和互作关系等信息预测其功能。
此外,蛋白质相互作用网络分析则是研究蛋白质之间相互作用的方法,可以揭示蛋白质在细胞内的相互作用关系和生物过程的调控机制。
蛋白质生物信息学是一门交叉学科,需要具备生物学、计算机科学和数学等多方面的知识。
随着科技的发展,蛋白质生物信息学在生命科学领域中的应用越来越广泛,为深入了解生命体系、开发新药物和治疗疾病提供了新的思路和方法。
- 1 -。
生物信息学 实验六 蛋白质高级结构预测
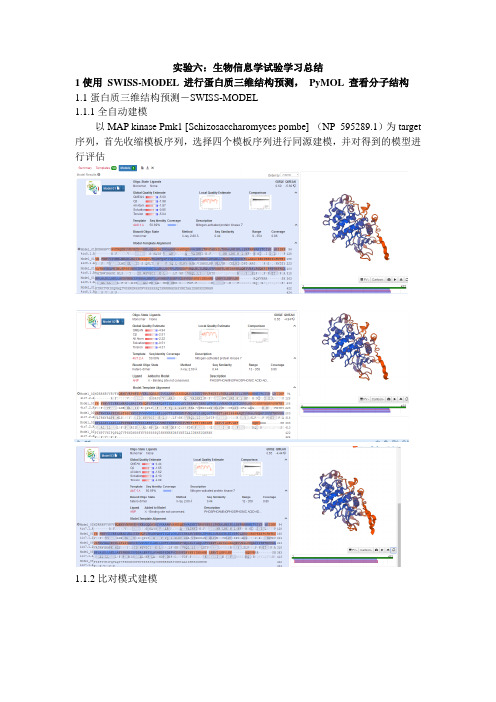
实验六:生物信息学试验学习总结1使用SWISS-MODEL 进行蛋白质三维结构预测,PyMOL 查看分子结构1.1蛋白质三维结构预测-SWISS-MODEL1.1.1全自动建模以MAP kinase Pmk1 [Schizosaccharomyces pombe] (NP_595289.1)为target 序列,首先收缩模板序列,选择四个模板序列进行同源建模,并对得到的模型进行评估1.1.2比对模式建模模型评估:GMQE(全局模型质量评估)是一种质量评估,它结合了目标-模板对齐和模板搜索方法的属性。
由此产生的GMQE分数表示为0到1之间的数字,反映了用该校准和模板构建的模型的预期精度。
较高的数字表明更高的可靠性。
一旦建立了模型,在这个特定的情况下,GMQE(1)就会得到更新,同时考虑到获得的模型的q 平均值,从而提高质量评估的可靠性。
QMEANQMEAN平均数(Benkert等)是基于不同几何属性的复合得分函数,并提供了全局的(即:对于整个结构)和局部(即每个残余物)绝对质量的估计是基于一个单一模型的。
z分数(2)提供了对模型中观察到的结构特征的“本土程度”的估计,并指出该模型是否具有与实验结构相似的质量。
较高的q均值z分数表明模型结构与相似尺寸的实验结构之间的一致性较好。
得分在0-4.0或以下的是一个质量很低的模型,这一点也可以通过在分数旁边的“拇指向下”符号的变化来突出显示。
QMEAN由四个单独的术语组成。
全球q平均值质量分数的四个单独术语也列在上面。
在巴图的白色区域(数值接近于零)表明这一特性与实验结构中所观察到的相似。
实证值表明,该模型平均得分高于实验结构,负数表明该模型平均得分低于实验结构。
q均值z分数本身显示在顶部。
单独的z分数比较了Cbeta原子之间的相互作用势,所有的原子,溶解势和扭转角度的潜力。
“局部质量”图显示了模型的每一个剩余部分(在x轴上报告),期望与本机结构(y轴)的相似性。
06第六章 常用生物信息学数据库简介

英国辛克斯顿
ID U00096 standard; circular genomic DNA; CON; 4639221 BP. AC U00096; SV U00096.1 DT 24-JUL-2003 (Rel. 76, Last updated, Version 3) DE Escherichia coli K-12 MG1655 complete genome. KW . OS Escherichia coli K12 OC Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; OC Enterobacteriaceae; Escherichia; Escherichia coli. RN [1] RP 1-4639221 RX MEDLINE; 97426617. RX PUBMED; 9278503. RA Blattner F.R., Plunkett G. III, Bloch C.A., Perna N.T., Burland V.,… RT "The complete genome sequence of Escherichia coli K-12"; RL Science 277(5331):1453-1474(1997). DR GOA; O32528. DR REMTREMBL; AAC74436; AAC74436. DR SPTREMBL; O32530; O32530. DR SWISS-PROT; O32528; YPDI_ECOLI. …
EMBL数据库简介
EMBL是最早的DNA序列 数据库,于1982年建立。
EMBL的数据来源主要有两条途径: 一是由序列发现者直接提交。几乎所有的国际权 威生物学刊物都要求作者在文章发表之前将所测定的 序列提交给EMBL、GenBank或DDBJ,得到数据库管 理系统所签发的登录注册号。 二是从生物医学期刊上收录已经发表的序列资料。
蛋白质生物信息学(共45张PPT)

利用生物信息学软件DNAman将VH-L-L的核苷酸序列翻译
为氨基酸序列
利用NCBI提供的ORF Finder预测VH-L-L的 ORF,从预测结果看出VH-L-L是一段连续 的较长的ORF,它可能是一个完整的编码 序列
利用ProtParam对VH-L-L的氨基酸序列及基本 理化性质进行了分析。
析,更加深入地理解DNA序列,结构,演化及其 与生物功能之间的关系。
研究课题涉及到分子生物学,分子演化及结构生 物学,统计学及计算机科学等许多领域。
研究过程
以数据(库)为核心 1 数据库的建立 2 生物学数据的检索 3 生物学数据的处理 4 生物学数据的利用:计算生物学
研究展望
由于生物信息学是基于分子生物学与多种学科交叉而成的 新学科,现有的形势仍表现为各种学科的简单堆砌,相互之 间的联系并不是特别的紧密。在处理大规模数据方面,没 有行之有效的一般性方法;而对于大规模数据内在的生成 机制也没有完全明了,这使得生物信息学的研究短期内很 难有突破性的结果。
第一节生物信息学与蛋白质工程 一、生物信息学概述
生物信息学是利用应用数学、信息学、统计 学和计算机科学的方法研究生物学的问题。
1987年,林华安首创Bioinformation 一词,被誉为”世界生物信息之父”。
概述
生物信息学分子生物学与信息技术(尤其是互联网 技术)的结合体。
研究材料和结果就是各种各样的生物学数据 研究工具是计算机
由于DNA自动测序技术的快速发展,
DNA数据库中的核酸序列公共数据量 以每天106bp速度增长,生物信息迅速 地膨胀成数据的海洋。毫无疑问,我们 正从一个积累数据向解释数据的时代转 变,数据量的巨大积累往往蕴含着潜 在突破性发现的可能。 “生物信息学” 正是从这一前提产生的交叉学科。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
轻链全长(L)DNA序列(708bp)
GGTGGTGGTGGCTCTGGCGGTGGTGGCT CTGGTGGCGGTGGTTCT 连接肽(G4S)3 蛋白质分子设计:VH-L-L
VH
linker
VL
CL
利用DNAman对VH-L-L的限制性内切酶位 点分析,结果显示VH-L-L有31个限制性酶 切位点,最多的是Eco57Ⅰ、TthlllⅠ分别 有三个酶切位点。
利用生物信息学软件DNAman将VH-L-L的核苷酸 序列翻译为氨基酸序列
利用NCBI提供的ORF Finder预测VH-L-L的 ORF,从预测结果看出VH-L-L是一段连续 的较长的ORF,它可能是一个完整的编码 序列
利用ProtParam对VH-L-L的氨基酸序列及基本 理化性质进行了分析。
第六章 生物信息学的应用
第一节生物信息学与蛋白质工程 一、生物信息学概述
生物信息学是利用应用数学、信息学、统计 学和计算机科学的方法研究生物学的问题。
1987年,林华安首创Bioinformation 一词,被誉为”世界生物信息之父”。
概述
生物信息学分子生物学与信息技术(尤其是互 联网技术)的结合体。 研究材料和结果就是各种各样的生物学数据 研究工具是计算机 研究方法包括对生物学数据的搜索(收集和 筛选)、处理(编辑、整理、管理和显示) 及利用(计算、模拟)。
利用Tmpred分析VH-L-L的跨膜区,分析表 明,该序列无跨膜区,不是跨膜蛋白。可 以预测该蛋白在膜外
利用NetPhos进行磷酸化位点分析,结果 显示磷酸化位点主要包括丝氨酸Ser位点: 28个,苏氨酸Thr: 5 个,酪氨酸Tyr: 3个
利用TargetP对VH-L-L蛋白的亚细胞定位 进行预测,结果表明,VH-L-L是分泌到 细胞周质的蛋白
利用SOPMA预测VH-L-L的二级结构,结 果显示,二级结构中α螺旋占15.56%,β 折叠34.95%,β转角12.24%,无规则卷 曲37.24%
蛋白质结构预测主要有两大类方法:
(1)理论分析方法
通过理论计算(如分子力学、分子动力学计算)进 行结构预测。
(2)统计的方法
对已知结构的蛋白质进行统计分析,建立序列到结 构的映射模型,进而对未知结构的蛋白质根据映射 模型直接从氨基酸序列预测结构。 包括:经验性方法(Chou-Fasman)、结构规律提 取方法(神经网络方法)、同源模型化方法
第二节、蛋白质常用数据库
一、核酸数据库 NCBI的Genbank、EMBL、DDBJ等SS-PROT、PIR 、TreEMBL、UniProt、GenPept
(二)蛋白序列二次数据库
蛋白保守区域和功能位点数据库,PROSITE、 PRINTS、BLOCKS
结果显示VH-L-L蛋白由392个氨基酸组成的稳定蛋白,分子 式为C1867H2894N494O596S11 ,分子量42149.1,等电点5.98。 理论推导半衰期为:30h(体外,哺乳动物的网织红细胞内)、 20h(体内,酵母细胞内)、10h(体内,大肠杆菌)。不稳定参 数是38.92,属于稳定蛋白。 含的氨基酸如图所示:Ser(S),Glu(G),Thr(T)最多,分别 占15.1%,9.9%,7.7%;不含Pyl(0),Sec(U)。总带正电 荷残基(Asp+Glu)为32,负电荷残基(Arg+Lys)为29。总的亲 水性平均系数-0.169,预测该蛋白属于亲水性蛋白。
每种氨基酸出现在各种二级结构中倾向或者 频率是不同的
例如:Glu主要出现在螺旋中 Asp和Gly主要分布在转角中 Pro也常出现在转角中,但是绝不会出现在 螺旋中
可以根据每种氨基酸残基形成二级结构的倾 向性或者统计规律进行二级结构预测
基本策略(1) 相似序列→相似结构
QLMGERIRARRKKLK QLMGAERIRARRKKLK
要真正解决这一问题,最终不能从计算机科学 得到,真正地解决可能还是得从生物学自身, 从数学上的新思路来获得本质性的动力。毫无 疑问,正如Dulbecco1986年所说:"DNA序列 是人类的真谛,这个世界上发生的一切事情, 都与这一序列息息相关"。但要完全破译这一 序列以及相关的内容,我们还有相当长的路要 走。
ATGGATTTTCAGGTGCAGATTTTCAGCTTCCTGCTAATCAGTGCCTCAGTCAT AATATCCAGAGGAGacatccagatgacccagtctccatcctccctgtctgcatctgtaggaga cagagtcACCATCACTTGCCAGGCGAGTCAGGACATCAGCAACTATTTAAATTG GTATCAGCAGAAACCAGGGAAAGCCCCTAAACTCCTGATCTACGATGCATCC AATTTGGAAACAGGGGTCCCATCAAGGTTCAGTGGAAGTGGATCTGGGACA GATTTTACTTTCACCATCAGCAGCCTGCAGCCTGAAGATATTGCAACATATTT CTGTCAACACTTTGATCATCTCCCGCTCGCTTTCGGCGGAGGGACCAAGGTG GAGATCAAACGT ACTGTGGCTGCACCATCTGTCTTCATCTTCC CGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCT GAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCC CTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGAC AGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAG AAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCC GTCACAAAGAGCTTCAACAGGGGAGAGTGT
III
蛋白质 序列:
蛋白质二级结构预测
↓
二级结构:
↓
1、二级结构预测概述
蛋白质的二级结构预测的基本依据是: 每一段相邻的氨基酸残基具有形成一定二 级结构的倾向。 二级结构预测问题是模式分类问题 二级结构预测的目标:
判断每一段中心的残基是否处于螺旋、折叠、 转角(或其它状态)之一的二级结构态,即三态。
研究内容
1、生物信息的收集、存储、管理与 提供 2、基因组序列信息的提取和分析 3、功能基因组相关信息分析 4、生物大分子结构模拟和药物设计 5、生物信息分析的技术与方法研究
发展条件
2001年2月,人类基因组工程测序 的完成,使生物信息学走向了一个 高潮。由于DNA自动测序技术的快 速发展,DNA数据库中的核酸序列 公共数据量以每天106bp速度增长, 生物信息迅速地膨胀成数据的海洋。 毫无疑问,我们正从一个积累数据 向解释数据的时代转变,数据量的 巨大积累往往蕴含着潜在突破性发 现的可能。 “生物信息学”正是从 这一前提产生的交叉学科。
一次数据库:实验获得的原始数据。简单归类 整理、注释。Genbank、Swiss-Prot、PDB 二次数据库:在一次数据库、实验数据和理论 分析的基础上,根据研究内容的需要,对相关 生物知识和信息进一步分析整理。包括人类基 因组图谱库GDB、转录因子和结合位点库 TRANSFAC、蛋白质结构家族分类库SCOP等。
核心内容是研究如何通过对DNA序列的统计计 算分析,更加深入地理解DNA序列,结构,演 化及其与生物功能之间的关系。 研究课题涉及到分子生物学,分子演化及结构 生物学,统计学及计算机科学等许多领域。
研究过程
以数据(库)为核心 1 数据库的建立 2 生物学数据的检索 3 生物学数据的处理 4 生物学数据的利用:计算生物学
生物信息学作为一门新的学科领域,把基 因组DNA序列信息分析作为源头,在获得蛋 白质编码区的信息后进行蛋白质空间结构 模拟和预测,然后依据特定蛋白质的功能 进行必要的药物设计。 基因组信息学,蛋白质空间结构模拟以及 药物设计构成了生物信息学的3个重要组成 部分。
第二节 蛋白质常用数据库及应用
有些蛋白质中含有大量的螺旋
如血红蛋白和肌红蛋白
而一些蛋白质中则不含或者仅含很少的螺旋
如铁氧蛋白
有些蛋白质的二级结构以折叠为主
如免疫球蛋白 例:肽链Ala(A)-Glu(E)-Leu(L)-Met(M) 倾向于形成 螺旋 肽链Pro(P)-Gly(G)-Tyr(Y)-Ser(S)则不会形成 螺旋
(三)蛋白结构数据库
三维结构数据库PBD、MMDB
全人源抗EGFR单克隆抗体
红色字体为信号肽,标黄部分为可变区重链可变区(HV) DNA序列(423bp)
ATGGATTTTCAGGTGCAGATTTTCAGCTTCCTGCTAATCAGTGCCTCAGT CATAATATCCAGAGGA CAGGTGCAGCTGCAGGAGTC GGGCCCAGGACTGGTGAAGCCTTCGGAGACCCTGTCCCTCACCTGCAC TGTCTCTGGTGGCTCCGTCAGCAGTGGTGATTACTACTGGACCTGGATT CGGCAGTCCCCAGGGAAGGGACTGGAGTGGATTGGACACATCTATTACA GTGGGAACACCAATTATAACCCCTCCCTCAAGAGCAGACTCACCATATCA ATTGACACGTCCAAGACTCAGTTCTCCCTGAAGCTGAGTTCTGTGACCG CTGCGGACACGGCCATTTATTACTGTGTGCGAGATCGAGTGACTGGTGC TTTTGATATCTGGGGCCAAGGGACAATGGTCACCGTCTCTTCA
