DNA拷贝数的计算方法
线粒体拷贝数 计算公式

线粒体拷贝数计算公式全文共四篇示例,供读者参考第一篇示例:线粒体是细胞中的一个重要细胞器,主要功能是产生细胞所需的能量。
线粒体内含有自己的DNA,与细胞核的DNA不同。
线粒体的DNA含有一些特殊的序列,可以用来计算细胞内线粒体的拷贝数。
线粒体拷贝数的计算可以帮助我们更好地了解细胞的代谢状态,以及细胞的功能活动。
在本文中,我们将介绍线粒体拷贝数的计算公式及其应用。
线粒体拷贝数的计算公式是基于线粒体DNA(mtDNA)的拷贝数和细胞总DNA的拷贝数之间的比值。
线粒体DNA的拷贝数通常通过PCR(聚合酶链反应)或实时荧光定量PCR来测量,而细胞总DNA的拷贝数则可以通过组织检测或细胞计数来确定。
线粒体拷贝数的计算公式如下:线粒体拷贝数= mtDNA拷贝数÷ 细胞总DNA拷贝数线粒体拷贝数的计算既可以用于单个细胞的研究,也可以用于整个组织或器官的研究。
通过测量线粒体拷贝数,我们可以了解细胞内线粒体的数量变化,预测细胞的代谢活动及能量需求。
线粒体拷贝数的变化与许多疾病的发生和发展有密切关系,如糖尿病、心脏病等。
线粒体拷贝数的计算对于研究疾病的发病机制及治疗方法具有重要的意义。
线粒体拷贝数的计算还可以用于研究不同细胞类型之间的线粒体数量差异。
在心肌细胞中线粒体拷贝数往往较高,因其需要大量能量来维持心肌的收缩功能。
而在肝细胞中,线粒体拷贝数也相对较高,因为肝细胞负责代谢和解毒。
不同细胞类型之间的线粒体拷贝数差异反映了细胞的功能特异性和代谢活动。
除了在生理学和疾病研究中的应用,线粒体拷贝数的计算还可以用于评估环境压力对生物体的影响。
环境因素如UV辐射、氧化压力等都可以影响线粒体的数量和功能,导致细胞代谢紊乱和细胞损伤。
通过测量线粒体拷贝数的变化,我们可以评估环境因素对细胞的影响,为环境保护和生物安全提供科学依据。
第二篇示例:线粒体是细胞内的一种细胞器,其主要功能是产生能量。
线粒体内含有自己的DNA,与细胞核DNA不同。
DNA拷贝数的计算方法

DNA拷贝数的计算方法1 A260 吸光度值= ds DNA 50 ug/ml= ss DNA 33 ug/ml= ss RNA 40 ug/ml核酸浓度=(OD260)×(dilution factor)×(33/40/50)= ng/ul平均分子量(MW)代表克/摩尔,单位道尔顿(dolton),即1dolton=1g/mol1摩尔=6.02×1023平均分子量(MW):dsDNA=(碱基数) x (660 道尔顿/碱基)ssDNA=(碱基数) x (330 道尔顿/碱基)ssRNA=(碱基数) x (340 道尔顿/碱基)拷贝数计算公式:(6.02 x 1023次拷贝数/摩尔) x (浓度g/ml) / (MW g/mol) = copies/ml.(6.02 x 1023次拷贝数/摩尔) x (浓度g/ml) / (DNA长度×660) = copies/ml.(6.02 x 1023次拷贝数/摩尔) x (ng/ul×10-9) / (DNA长度×660) = copies/ul.例:3000 碱基质粒,浓度100 ng/ml ,MW = 3000 bp x 660 dalton/bp = 1.98 x 106 daltons,1 mol = 1.98 x 106g.(6.02 x 10的23次拷贝数/摩尔) x (1x10的-7次克/微升) / (1.98 x 10的6次克/摩尔) = 3 x 1010次copies/ml.梯度配制方法:低浓度使用胎盘DNA 20ng/ul;高浓度使用灭菌水配制,20ng 基因组DNA所包含的拷贝数:6.02×3 x 1023 x20ng/2.91 x109 x660=6289个拷贝数。
计算copy数

做 qRT-PCR 的时候经常需要计算 DNA 拷贝数(Copy Number),比较烦,用下面的方法这就方便多了。
其实计算方法是相通的,只不过一个是带有分步推理过程,一个是直接计算而已!一、分步推理如何计算核酸拷贝数1A260吸光度值=dsDNA 50ug/ml=ssDNA 33ug/ml=ssRNA 40ug/ml核酸浓度=(OD260)×稀释倍数×(33 或 40 或 50)=ng/ulMW 代表克/摩尔,单位 dolton :1dolton 即表示 1g/mol 1 摩尔=6.02×1023摩尔分子(拷贝数)平均分子量(MW):dsDNA=碱基数×660 道尔顿/碱基ssDNA=碱基数×330 道尔顿/碱基ssRNA=碱基数×340 道尔顿/碱基得到拷贝数计算公式:mLmol g MW mL g mol mol copies /copies //)/copies 1002.6)/(1002.62323=⨯⨯=⨯⨯)()浓度((摩尔数 即(6.02×1023)×(g/ml)/(DNA length×660)=copies/ml.或(6.02×1023)×(ng/ul×10-9)/(DNA length×660)=copies/ul.例:3000 碱基质粒,浓度 100 ng/ulMW=3000bp×660dalton/bp=1.98×106daltonS=1.98×106g/mol ,即1mol=(100ng×10-9)g/1.98×106=摩尔数copy 数=摩尔数×6.02×1023=3×1010copies/ul.。
什么是基因拷贝数

什么是基因拷贝数实时荧光定量 PCR技术用于检测插入的外源基因拷贝数自 1994 年,第一例转基因植物产品 Flavr Savr TM 西红柿在美国上市,到 2003 年底,世界范围内转基因作物已遍布 18 个国家和地区,种植面积达167.2 万英亩 (6770 万公顷 )( Ewen SWB,1999; 北国网 ) 。
大量实践使得植物转基因技术已有了比较经典和成熟的技术路线。
除了在商业领域内获得抗性植株外,植物转基因技术还延伸到植物生理学研究(生化反应途径、植物抗病、抗逆性)以及生物反应器研究(药物、疫苗等)中( Khachatourians 2002 )。
一般经过上游表达载体的设计构建以及下游转化体系的建立、转化品系的筛选鉴定等一系列步骤后,即获得 T 0 代转基因植物。
在转化过程中,外源DNA 随机插入植物基因组内,插入的拷贝数和位点都不固定。
插入外源基因的拷贝数低( 1 或 2 个)能较好的表达,插入的拷贝数多则会导致表达的不稳定甚至基因沉默现象( Flavell 1994 , Vaucheret et al 1998 )。
因此,检测 T 0 代转基因植物的外源基因的拷贝数是研究其分子特性的基础步骤之一。
Southern blot 是一种常用的 DNA 定量的分子生物学方法。
其原理是将待测的 DNA 样品固定在固相载体(硝酸纤维膜或尼龙膜)上,与标记的核酸探针进行杂交,在与探针有同源序列的固相 DNA 的位置上显示出杂交信号,通过检测信号的有无、强弱可以对样品定性、定量,从而计算出转入的拷贝数。
Southern 法准确性高、特异性强,但存在费时费力的缺点。
另外,由于Southern 法检测不经过靶片段的扩增( PCR ),一般每个电泳通道需要 10-30 μ g 的 DNA ,在实际操作中就需要较大量的植物材料来提取 DNA ,而转基因植物的愈伤组织在无菌条件下经过筛选、重新分化后一般都比较细弱,不宜大量取样。
1定量PCR基本原理

1定量PCR基本原理定量PCR(Quantitative Polymerase Chain Reaction)是一种利用DNA复制技术进行定量测定DNA数量的方法。
它是PCR技术的一种改进,在遗传学、疾病诊断、药物研发等领域有广泛应用。
以下是定量PCR的基本原理的详细介绍:1.反应体系组成:定量PCR所需的反应体系包括DNA模板、引物、荧光探针、聚合酶、缓冲液、dNTPs和MgCl2等组分。
DNA模板是待测DNA的起始材料,引物是用于扩增特定DNA片段的寡核苷酸引物,荧光探针则会结合到PCR产物上生成荧光信号用于检测和计量。
2.PCR扩增步骤:定量PCR扩增过程包括三个主要步骤:变性、退火和延伸。
首先,将反应体系加热至95°C,使DNA双链解开变性为两条单链。
然后,将温度降低至特定的温度,使引物能够与DNA靶序列结合。
引物结合后,聚合酶开始在两条单链DNA上合成新的DNA链。
这一过程称为延伸。
随着循环的进行,PCR产物的数量呈指数增加。
3.荧光信号检测:定量PCR中使用特定的荧光探针来检测扩增产物的数量。
这些荧光探针一般由两个部分组成:探针序列和荧光染料。
探针序列与待测DNA的靶序列互补配对,而荧光染料则位于探针的末端。
在PCR过程中,引物扩增到接近探针的位置时,3'末端荧光染料会与5'末端的荧光信号发生共振能量转移(FRET),从而发出荧光信号。
通过测量这些信号的强度,可以推断PCR产物的数量。
4.标准曲线和计量:为了进行定量测定,标准曲线是必不可少的。
标准曲线是一系列已知浓度的标准品所构建的,通过对标准品进行PCR扩增并测量荧光信号的强度,可以建立起PCR产物浓度与荧光强度之间的关系。
通过测量待测样品的荧光强度,并根据标准曲线进行插值计算,可以得出待测样品中特定DNA序列的浓度。
5.分析和解释结果:定量PCR的结果可以通过多种途径进行分析。
一种常见的方法是计算待测样品和标准样品之间的CT值差,通过该差值来比较两者的浓度。
DNA拷贝数的计算方法

(6.02 x 10(23)) x (g/ml) / (DNA length x 660) = copies/ml.
或
(6.02 x 10(23)) x (ng/ul x 10(-9) ) / (DNA length x 660) = copies/ul.
例:
3000碱基质粒,浓度100 ng/ul,MW= 3000 bp x 660 dalton/bp =1.98 x 10(6) daltons,即1 mol =1.98 x 10(6) g。
[ 1010(6)=摩尔数copy数=摩尔数x 6.02 x 10(23)=3x 10(10) copies/ul.
感谢您使用本店文档 您的满意是我们的永恒的追求! (本句可删)
------------------------------------------------------------------------------------------------------------
1摩尔= 6.02 x 10(23)次摩尔分子(拷贝数)
平均分子量(MW):
dsDNA=(碱基数) x (660道尔顿/碱基)
ssDNA=(碱基数) x (330道尔顿/碱基)
ssRNA=(碱基数) x (340道尔顿/碱基)
得到拷贝数计算公式:
(6.02 x 10(23)次拷贝数/摩尔) x (浓度) / (MW g/mol) = copies/ml.
mw代表克摩尔单位dolton1dolt即表示1gmol50ngul1a260吸光度值dsdna50ugml二ssdna33ugml二ssrna40ugml核酸浓度od260dilutionfactor1023次摩尔分子拷贝数平均分子量mwdsdna碱基数x660道尔顿碱基ssdna碱基数x330道尔顿碱基ssrna碱基数x340道尔顿碱基得到拷贝数计算公式
southern blotting 确定拷贝数原理

southern blotting确定拷贝数原理
Southern blotting是一种分子生物学技术,主要用于检测DNA中特定序列的存在以及确定DNA拷贝数。
以下是Southern blotting确定拷贝数的基本原理:
1.DNA片段分离:首先,将待检测的DNA样本通过酶切等方法进行分离,生成一系列DNA片段。
这些片段的长度和序列将取决于所使用的酶。
2.凝胶电泳:将DNA片段加载到琼脂糖凝胶中,并通过电泳使其在凝胶中移动。
由于DNA片段的不同大小,它们在电场中会按照大小被分离开来,形成一个DNA带谱。
3.转移到膜上:将DNA凝胶上的分离片段转移到一块膜上,通常是硝酸纤维素或硝酸纸。
4.固定DNA到膜上:通过紫外线照射或烘烤等方法,将DNA片段固定在膜上。
5.杂交:将标记有放射性或荧光标记的DNA探针加到膜上。
这个探针是与待检测DNA 特定序列相互匹配的DNA片段。
6.探针与目标DNA杂交:探针与膜上DNA片段中与之互补的序列发生杂交。
这样,可以通过检测探针的位置来确定待检测DNA中特定序列的存在。
7.确定拷贝数:通过观察带谱的强度和数量,可以推断出待检测DNA中特定序列的拷贝数。
如果一个序列在DNA中存在多个拷贝,相应的带谱会更强烈。
总体而言,Southern blotting提供了一种分子水平上检测DNA片段的方法,并且可以用来确定目标序列在基因组中的拷贝数。
荧光定量PCR之绝对定量分析——标准曲线的绘制
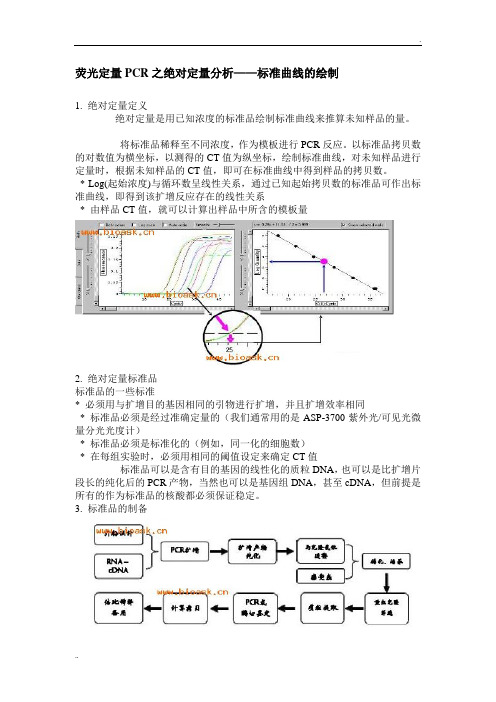
荧光定量PCR之绝对定量分析——标准曲线的绘制1. 绝对定量定义绝对定量是用已知浓度的标准品绘制标准曲线来推算未知样品的量。
将标准品稀释至不同浓度,作为模板进行PCR反应。
以标准品拷贝数的对数值为横坐标,以测得的CT值为纵坐标,绘制标准曲线,对未知样品进行定量时,根据未知样品的CT值,即可在标准曲线中得到样品的拷贝数。
* Log(起始浓度)与循环数呈线性关系,通过已知起始拷贝数的标准品可作出标准曲线,即得到该扩增反应存在的线性关系* 由样品CT值,就可以计算出样品中所含的模板量2. 绝对定量标准品标准品的一些标准* 必须用与扩增目的基因相同的引物进行扩增,并且扩增效率相同* 标准品必须是经过准确定量的(我们通常用的是ASP-3700紫外光/可见光微量分光光度计)* 标准品必须是标准化的(例如,同一化的细胞数)* 在每组实验时,必须用相同的阈值设定来确定CT值标准品可以是含有目的基因的线性化的质粒DNA,也可以是比扩增片段长的纯化后的PCR产物,当然也可以是基因组DNA,甚至cDNA,但前提是所有的作为标准品的核酸都必须保证稳定。
3. 标准品的制备一般一条标准曲线取四到五个点,浓度范围要能覆盖样品的浓度区间,以保证定量的准确性。
一般一个点重复三至五次,对于常期稳定使用的标准品可以适当减少重复的次数。
倍比梯度稀释方法:1v原液(标准品i)+9v稀释缓冲液,得标准品ii1v标准品ii+9v稀释缓冲液,得标准品iii1v标准品iii+9v稀释缓冲液,得标准品iv1v标准品iv+9v稀释缓冲液,得标准品v依次倍比稀释拷贝数的计算:详见核酸拷贝数的计算4. 实例标准品的制作:将标准品依次进行10倍稀释,ASP-3700 测得其拷贝数1.55×108copy /ul标准曲线的绘制(1cycle=1min)设置对照:浓度为1.55×107、1.55×106、1.55×105、1.55×104、1.55×103、1.55×102、1.55×101的标准样品各一个,设空白对照PCR反应:以不同浓度标准品作为模板标准品的扩增曲线标准品的溶解曲线标准品的标准曲线图X Log 7 6 5 4 3 2 1Y CT值12.01 14.89 17.92 21.18 24.56 27.89 31.25扩增效率(E)计算:E=10-1/斜率=10-1/-3.23=2.04,E%=(2.04-1)×100%=104%若未知样本的CT值为19.11,将CT值代入线性方程:即19.11=34.29-3.23X,所以X=(19.11-34.29)/(-3.23)=4.7Quantityunknow=104.7=50118 copies核酸拷贝数的计算一、分步推理如何计算核酸拷贝数1A260吸光度值=dsDNA 50ug/ml=ssDNA 33ug/ml=ssRNA 40ug/ml核酸浓度=(OD260)×(dilution factor)×[33或40或50]=ng/ulMW代表克/摩尔,单位dolton:1dolton即表示1g/mol1摩尔=6.02x1023摩尔分子(拷贝数)平均分子量(MW):dsDNA=(碱基数)×(660道尔顿/碱基)ssDNA = (碱基数)×(330道尔顿/碱基) ssRNA=(碱基数)×(340道尔顿/碱基)得到拷贝数计算公式:(6.02x1023拷贝数/摩尔)×(浓度)/(MW g/mol)= copies/ml. 即(6.02x1023)×(g/ml)/(DNA length×660)=copies/ml.或(6.02×1023)×(ng/ul×10-9)/(DNA length×660)=copies/ul.例:3000碱基质粒,浓度100 ng/ulMW=3000bp×660dalton/bp=1.98×106daltons,即1mol=1.98×106g(100ng×10-9)g/1.98×106=摩尔数copy数=摩尔数×6.02×1023=3×1010copies/ul.二、这是一个小小的计算器,使你计算更加方便快捷,免去算数之苦……什么是拷贝数?如何计算拷贝数?计算方法:(6.02×1023拷贝数/摩尔) ×(浓度g/ml) / (MW g/mol) = copies/ml [平均分子量(MW g/mol):dsDNA=(碱基数)×(660道尔顿/碱基);ssDNA=(碱基数)×(330道尔顿/碱基);ssRNA=(碱基数)×(340道尔顿/碱基)]。
