构建本地 blast - 成长博客博客教育博客教师博客学生博客
本地Blast

本地Blast使用说明一、软件的下载安装1.1安装流程建议安装在非系统盘,如将下载的 BLAST 程序安装到 E:\blast,生成bin、doc 两个子目录,其中 bin 是程序目录,doc 是文档目录,这样就安装完毕了。
1.2 设置环境变量右键点击“我的电脑”-“属性”,然后选择“高级系统设置”标签-“环境变量”(图1),在用户变量下方“Path”随安装过程已自动添加其变量值,即“E:\Blast\bin”。
此时点击“新建”-变量名“BLASTDB”,变量值为“E:\Blast\db”(即数据库路径,图2)。
二、查看程序版本信息点击 Windows 的“开始”菜单下的“运行”,输入“cmd”调出 MS-DOS 命令行,转到 Blast 安装目录,输入命令“blastn -version”即可查看版本,若能显示说明本地blast 已经安装成功。
三、使用3.1本地数据库的构建下载所需的数据(Fasta格式),将X 放到E:\blast\db 文件夹下,然后调出MS-DOS 命令行,转到E:\blast\db 文件夹下运行以下命令:格式化数据库,命令为:makeblastdb -in 数据库文件 -dbtype 序列类型(核酸:nul;蛋白:prot)-title database_title-parse_seqids -out database_name-logfile File_Name格式化数据库后,创建三个主要的文件——库索引(indices),序列(sequences)和头(headers)文件。
生成的文件的扩展名分别是:.pin、.psq、.phr(对蛋白质序列)或.nin、.nsq、.nhr(对核酸序列)。
而其他的序列识别符和索引则包含在.psi和.psd(或.nsi 和.nsd)中。
3.2核酸序列相似性搜索blastn -db database_name -query input_file -out output_file-outfmt "7 qacc sacc qstart qend sstart send length bitscore evalue pident ppos"备注:qacc:查询序列Acession号;sacc:目标序列Acession号;qstart qend:分别表示查询序列比对上的起始、终止位置;sstart send:分别表示目标序列比对上的起始、终止位置;length:长度; bitscore:得分; evalue:E-Value值;pident:一致性; ppos:相似性3.3 查看并获取目标序列:blastdbcmd -db refseq_rna -entry 224071016 -out test.fa可以从数据库中提取gi号为224071016的序列,并且以fasta格式存入文件3.4蛋白质序列相似性搜索Blastp -db database_name-query input_file -out output_file-outfmt "7 qacc sacc qstart qend sstart send length bitscore evalue pident ppos"3.5 查看并获取目标序列:重复3.3。
Blast使用方法攻略

Blast使⽤⽅法攻略结果12列Query id,Subject id,% identity,alignment length,mismatches,gap openings,q. start,q. end,s. start,s. end,e-value,bit scoreBlast,全称Basic Local Alignment Search Tool,即"基于局部⽐对算法的搜索⼯具",由Altschul等⼈于1990年发布。
Blast能够实现⽐较两段核酸或者蛋⽩序列之间的同源性的功能,它能够快速的找到两段序列之间的同源序列并对⽐对区域进⾏打分以确定同源性的⾼低。
Blast的运⾏⽅式是先⽤⽬标序列建数据库(这种数据库称为database,⾥⾯的每⼀条序列称为subject),然后⽤待查的序列(称为 query)在database中搜索,每⼀条query与database中的每⼀条subject都要进⾏双序列⽐对,从⽽得出全部⽐对结果。
Blast是⼀个集成的程序包,通过调⽤不同的⽐对模块,blast实现了五种可能的序列⽐对⽅式:blastp:蛋⽩序列与蛋⽩库做⽐对,直接⽐对蛋⽩序列的同源性。
blastx:核酸序列对蛋⽩库的⽐对,先将核酸序列翻译成蛋⽩序列(根据相位可以翻译为6种可能的蛋⽩序列),然后再与蛋⽩库做⽐对。
blastn:核酸序列对核酸库的⽐对,直接⽐较核酸序列的同源性。
tblastn:蛋⽩序列对核酸库的⽐对,将库中的核酸翻译成蛋⽩序列,然后进⾏⽐对。
tblastx:核酸序列对核酸库在蛋⽩级别的⽐对,将库和待查序列都翻译成蛋⽩序列,然后对蛋⽩序列进⾏⽐对。
Blast提供了核酸和蛋⽩序列之间所有可能的⽐对⽅式,同时具有较快的⽐对速度和较⾼的⽐对精度,因此在常规双序列⽐对分析中应⽤最为⼴泛。
可以毫不夸张的说,blast是做⽐较基因组学乃⾄整个⽣物信息学研究所必须掌握的⼀种⽐对⼯具。
Blast使用教程详解

Blast相关的问题
怎么获得blast服务,怎么使用的问题?
为什么使用blast,可以获得什么样的信息? 其他问题:实际使用时选择哪种方式(网 络,本地化),参数的选择,结果的解 释…
11
Blast资源
1.NCBI主站点:
/BLAST/(网络版) ftp:///blast/ (单机版)
13
两种版本的Blast比较(一)
网络版本 包括NCBI在内的很多网站都提供了在线 的blast服务,这也是我们最经常用到的 blast服务。网络版本的blast服务就有方便, 容易操作,数据库同步更新等优点。但是 缺点是不利于操作大批量的数据,同时也 不能自己定义搜索的数据库。
14
两种版本的Blast比较(二)
单机版 单机版的blast可以通过NCBI的ftp站点获得, 有适合不同平台的版本(包括linux,dos 等)。获得程序的同时必须获取相应的数 据库才能在本地进行blast分析。单机版的 优点是可以处理大批的数据,可以自己定 义数据库,但是需要耗费本地机的大量资 源,此外操作也没有网络版直观、方便, 需要一定的计算机操作水平。
生物序列的相似性搜索
-blast简介及其应用
2005年3月
生物信息学常见的应用与软件
序列数据的保存格式与相关数据库资源 在数据库中进行序列相似性搜索
多序列比对
进化树构建与分子进化分析 Motif的寻找与序列的模式识别 RNA二级结构,蛋白质二、三级结构的预测 基因芯片的数据分析
2
内容提要
1.基本概念 相似性,同源性 2.Blast介绍 Blast资源和相关问题 3.Blast的应用 网络版,单机版 4.深入了解Blast(改进程序,算法基础) 5.其他的序列相似性搜索工具(fasta)
Blast软件及常用数据库的具体介绍

Blast软件及常用数据库的具体介绍
17
2021/1/25
blast常用的一些参数
-p: 执行的程序名称
-d: 检索的数据库名称
-i : 要查询的序列文件名
-o :查询结果输出文件名
-m: 比对结果显示格式选项,缺省值为0 ,即pairwise格式。另外
还可以根据不同的需要选择1~6等不同的格式。
新建一个名为“text” 的文本文档
打开,输入序列,如
>Text AGCTTTTCATTCTGACTGCAACGGGCAATATGTCTCTGTG TGGATTAAAAAAAGAGTGTCTGATAGCAGC TTCTGAACTGGTTACCTGCCGTGAGTAAATTAAAATTTTAT TGACTTAGGTCACTAAATACTTTAACCAA TATAGGCATAGCGCACAGACAGATAAAAATTACAGAGTAC ACAACATCCATGAAACGCATTAGCACCACC ATTACCACCACCATCACCATTACCACAGGTAACGGTGCG GGCTGACGCGTACAGGAAACACAGAAAAAAG CCCGCACCTGACAGTGCGGGCTTTTTTTTTCGACCAAAG GTAACGAGGTAACAACCATGCGAGTGTTGAA GTTCGGCGGTACATCAGTGGCAAATGCAGAACGTTTTCT GCGTGTTGCCGATATTCTGGAAAGCAATGCC AGGCAGGGGCAGGTGGCCACCGTCCTCTCTGCCCCCG CCAAAATCACCAACCACCTGGTGGCGATGATTG AAAAAACCATTAGCGGCCAGGATGCTTTACCCAATATCAG CGATGCCGAACGTATTTTTGCCGAACTTTT
构建NCBI本地BLAST数据库(NRNT等)blastxdiamond使用方法blast。。。
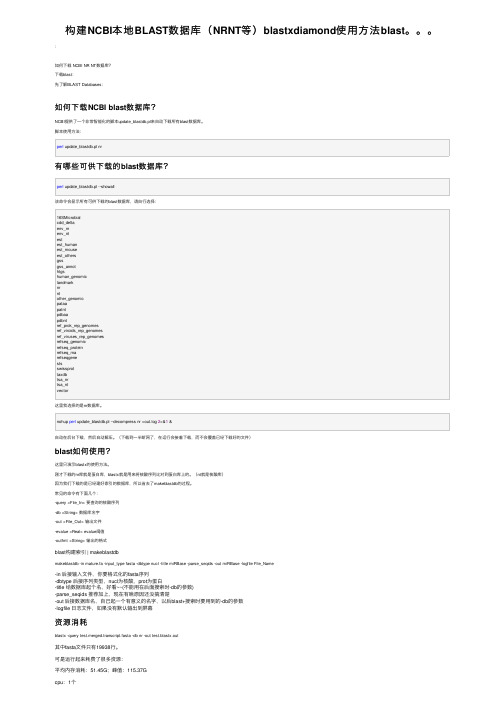
构建NCBI本地BLAST数据库(NRNT等)blastxdiamond使⽤⽅法blast。
:如何下载 NCBI NR NT数据库?下载blast:先了解BLAST Databases:如何下载NCBI blast数据库?NCBI提供了⼀个⾮常智能化的脚本update_blastdb.pl来⾃动下载所有blast数据库。
脚本使⽤⽅法:perl update_blastdb.pl nr有哪些可供下载的blast数据库?perl update_blastdb.pl --showall该命令会显⽰所有可供下载的blast数据库,请⾃⾏选择:16SMicrobialcdd_deltaenv_nrenv_ntestest_humanest_mouseest_othersgssgss_annothtgshuman_genomiclandmarknrntother_genomicpataapatntpdbaapdbntref_prok_rep_genomesref_viroids_rep_genomesref_viruses_rep_genomesrefseq_genomicrefseq_proteinrefseq_rnarefseqgenestsswissprottaxdbtsa_nrtsa_ntvector这⾥我选择的是nr数据库。
nohup perl update_blastdb.pl --decompress nr >out.log 2>&1 &⾃动在后台下载,然后⾃动解压。
(下载到⼀半断⽹了,在运⾏会接着下载,⽽不会覆盖已经下载好的⽂件)blast如何使⽤?这⾥只演⽰blastx的使⽤⽅法。
刚才下载的nr库就是蛋⽩库,blastx就是⽤来将核酸序列⽐对到蛋⽩库上的。
(nt就是核酸库)因为我们下载的是已经建好索引的数据库,所以省去了makeblastdb的过程。
常见的命令有下⾯⼏个:-query <File_In> 要查询的核酸序列-db <String> 数据库名字-out <File_Out> 输出⽂件-evalue <Real> evalue阈值-outfmt <String> 输出的格式blast构建索引 | makeblastdbmakeblastdb -in mature.fa -input_type fasta -dbtype nucl -title miRBase -parse_seqids -out miRBase -logfile File_Name-in 后接输⼊⽂件,你要格式化的fasta序列-dbtype 后接序列类型,nucl为核酸,prot为蛋⽩-title 给数据库起个名,好看~~(不能⽤在后⾯搜索时-db的参数)-parse_seqids 推荐加上,现在有啥原因还没搞清楚-out 后接数据库名,⾃⼰起⼀个有意义的名字,以后blast+搜索时要⽤到的-db的参数-logfile ⽇志⽂件,如果没有默认输出到屏幕资源消耗blastx -query test.merged.transcript.fasta -db nr -out test.blastx.out其中fasta⽂件只有19938⾏。
如何构建属于自己的序列比对库 本地Blast比对库构建教程

序列比对库的构建教程如何将大量的基因序列或基因组序列文件建成一个属于自己的系列比对库,方便我们随时进行本地Blast比对呢?构建本地Blast比对库的好处是:1.不用再担心停电而导致无法比对。
2.不用再担心打不开基因组序列的窘境了。
3.可以实现对批量序列进行比对。
4.如果NCBI里没有某物种的基因组,恰恰你手中有,可以建库进行基因比对。
5.能够快速知道序列与基因组里哪些部位有匹配,且匹配率有多大。
6.通过与基因组库比对,能帮我们验证所扩增的基因是否是正确的,而不是其它基因。
7.能够将收集到的对自己研究方向有用的大量序列构建成库,针对性更强,方便随时进行序列比对。
基于以上原因,我们知道构建本地Blast序列比对库的好处会很多,接下来的教程将教大家如何构建本地Blast序列比对数据库,为我们的研究营造更多方便。
本教程以构建基因序列的比对库为例(注:蛋白序列比对库构建方法与此相同)建库及使用的前提:如需要将本地比对库建在D盘,我们应先在D盘下载并安装一下BLAST软件,下载地址可以点击如下链接(也可在NCBI点击BLAST下载):https:///blast/executables/blast+/2.9.0/然后下载界面如下图,我们需要根据自己的电脑版本来下载,Windows 64位系统下载下图圈出来的那个(注:切记不要下载ncbi-blast-2.10.1+-win64,这个新版本在建库时会出现磁盘空间不足的现象)。
安装好BLAST软件后接下来开始建库:1.首先我们需要准备用于建库的基因序列,比如自己下载的某个类群的大量基因序列或基因组序列(需为fasta文件),或自己手里测得的基因组序列等,本教程演示一个基因组序列建库。
2.准备好序列以后,我们将建库序列fasta文件放到电脑的非系统盘,如D盘内建一个名为“dna”的文件夹,以后所有的文件都在这个名为dna的文件夹内。
接下来就可以构建库了。
3.先点击电脑桌面最左下角的Windows图标,再在搜索框中输入“cmd”然后按键盘上的“Enter”键,操作如下图4.进入后的界面如下图5.因为我们的建库序列在D盘内,所以我们要先进到这个盘内的dna这个文件夹内,操作是先输入“D:”再加键盘的Enter键,这样就进入到D盘了,如下图。
Blast使用教程详解
生物序列的同源性
同源性(homology): 指从一些数据中推断出的两个基因或蛋
白质序列具而共同祖先的结论,属于质的 判断。就是说A和B的关系上,只有是同 源序列,或者非同源序列两种关系。而说 A和B的同源性为80%都是不科学的。
5
相似性和同源性关系
序列的相似性和序列的同源性有一定的关系,一 般来说序列间的相似性越高的话,它们是同源序 列的可能性就更高,所以经常可以通过序列的相 似性来推测序列是否同源。
单机版的Blast程序包,把基本的blast分析, 包括blastn,blastp,blastx等都整合到了 blastall一个程序里面。
44
单机版的Blast使用(六)
以下是一个典型的blastn分析命令: (待分析序列seq.fa,数据库nt_db)
我们选上
30
分析过程(三)
6.限制条件,我们限制 在病毒里面找。
7.其他选项保持默认值
打分矩阵
31
分析过程(五)
10.查询序列的一些 相关信息 在cdd库里面找到 两个保守区域, 点击可以进入
32
33
分析过程(六)
图形结果
34
分析过程(七)
匹配序列列表
35
分析过程(八)
具体匹配情况
36
37
…
12
Blast结果给出的信息
Blast结果会列出跟查询序列相似性比较 高,符合限定要求的序列结果,根据这些 结果可以获取以下一些信息。 1.查询序列可能具有某种功能 2.查询序列可能是来源于某个物种 3.查询序列可能是某种功能基因的同源基因 … 这些信息都可以应用到后续分析中。
13
Hale Waihona Puke 两种版本的Blast比较(一)
本地blast使用说明
Microsoft Windows XP [版本 5.1.2600]
(C) 版权所有 1985-2001 Microsoft Corp.
C:\Documents and Settings\Administrator>cd c:\
C:\>dir
驱动器 C 中的卷没有标签。
2007-08-23 05:38 1,662,976 impala.exe
2007-08-23 05:38 1,572,864 makemat.exe
2007-08-23 05:38 2,088,960 megablast.exe
2009-03-12 15:41 20,998 result.txt
卷的序列号是 A427-A482
C:\ 的目录
2009-03-09 08:48 39,087 1.txt
2009-03-09 14:36 159 2.txt
2008-11-10 12:46 0 AUTOEXEC.BAT
2008-11-10 12:50 <DIR> Documents and Settings
2008-11-10 12:35 <DIR> Driver
2009-03-06 15:39 18,292 miRNA定位的原始序列.txt
2007-08-23 05:38 1,634,304 formatdb.exe
2009-03-12 15:40 348 formatdb.log
2007-08-23 05:38 1,744,896 formatrpsdb.exe
2007-08-23 05:39 2,068,480 rpsblast.exe
如何本地化进行blast序列比对
如何本地化进行blast序列比对1、基本概念相似性(Similarity)是指序列比对过程中用来描述检测序列和目标序列之间相同或相似碱基或氨基酸残基占全部比对碱基或氨基酸残基的比例的高低,属于量的判断。
同源性(Homology)是指从某一共同祖先经趋异进化而形成的不同序列。
只有当两个蛋白质在进化关系上具有共同的祖先时,才可称它们为同源的,属于质的判断。
相似性和同源性的关系当相似程度高于50%时,比较容易推测检测序列和目标序列可能是同源序列;而当相似性程度低于20%时,就难以确定或者根本无法确定其是否具有同源性。
总之不能把相似性和同源性混为一谈。
所谓“具有50%同源性”,或“这些序列高度同源”等说法,都是不确切的,应避免使用。
序列相似性比较和同源性分析序列相似性分析:就是用来计算待研究序列与某序列之间的相似性程度,常用的软件包有BLAST、FASTA等;序列同源性分析:是将待研究与来自不同物种的序列中进行进化分析,以确定该序列与其它序列间的亲源关系。
常用的程序包有Phylip及Mega等进化分析软件;全局比对与局部比对全局比对寻找序列在全长范围内最佳比对。
常用算法如:Needleman-Wunsch algorithm(Needle)在线程序如: Needle局部比对寻找序列在局部区域的最高比对打分。
常用算法如:Smith-Waterman algorithm, blast,fasta等在线程序如: WaterNeedle及Water的在线程序也可以本地安装Emboss执行以上程序局部相似性比对的生物学基础蛋白质功能位点往往是由较短的序列片段组成的,尽管在序列的其它部位可能有插入、删除等突变,但这些关键的功能部位的序列往往具有相当大的保守性。
而局部比对往往比整体比对对这些功能区段具有更高的灵敏度,因此其结果更具生物学意义。
BLAST程序常用的两个评价指标Score:使用打分矩阵对匹配的片段进行打分,这是对各对氨基酸残基(或碱基)打分求和的结果,一般来说,匹配片段越长、相似性越高则Score值越大,结果越可信。
Blast分析报告
Blast分析报告1. 简介Blast(Basic Local Alignment Search Tool)是一种常用的生物信息学工具,用于比对和对比两个或多个生物序列。
它可以帮助研究人员在生物信息学研究中进行序列比对、寻找同源序列以及进行功能注释等工作。
本文将引导您详细了解和使用Blast进行分析。
2. 安装和配置Blast软件首先,您需要从NCBI(National Center for Biotechnology Information)官方网站下载并安装Blast软件。
一旦安装完成,您需要设置Blast的环境变量,以便在命令行中能够直接调用Blast命令。
3. 准备序列数据在进行Blast分析之前,您需要准备好待比对的序列数据。
这些序列可以是蛋白质序列或核酸序列,可以从NCBI数据库或其他来源获取。
确保您已经将这些序列保存在合适的文件中,并准备好进行分析。
4. 运行Blast分析接下来,您将使用命令行界面来运行Blast进行分析。
以下是一个基本的Blast命令示例:blastn -query query.fasta -db database.fasta -out result.txt在这个示例中,blastn表示您要运行的Blast程序,query.fasta是您的查询序列文件,database.fasta是您的数据库文件,result.txt是结果输出文件。
您可以根据需要调整Blast命令的参数,例如,您可以指定比对算法、设置阈值、选择输出格式等。
详细的命令选项和参数可以通过blastn -help命令来查看。
5. 解读Blast结果当Blast分析完成后,您将获得一个结果文件,其中包含了比对结果的详细信息。
您可以使用文本编辑器或其他工具打开这个结果文件,并解读其中的内容。
在结果文件中,您将看到每个查询序列和数据库序列的比对结果,包括比对得分、相似度、匹配位置等信息。
根据这些信息,您可以判断查询序列与数据库序列之间的关系,进一步分析和解释结果。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
构建本地blast比对常用软件blast和clustal(多序列比对)。
本地blast的构建及使用步骤如下:程序下载>程序安装>进入Dos命令系统>数据库格式化>程序运行(及进行比对)>查看分析结果。
1.程序下载:连接到:ftp:///blast/executables/release/LATEST/按自己的OS进行下载;2.现在D盘建立新文件夹并命名为“blast”,然后下载“win32-ia32”(windows)到blast文件夹中;3.双击win32-ia32.exe安装程序,产生3个新文件:bin、data和doc4.以blastp记录本地blast的使用(目的:用番茄的TFT1蛋白序列跟拟南芥中的每一条14-3-3蛋白序列比较,以查看该序列与拟南芥中的哪条序列相似性最高)4.1建库在bin中建一个txt文件,将拟南芥的13条序列(必须为fasta格式)粘贴进去:>AtGF14 chiMATPGASSARDEFVYMAKLAEQAERYEEMVEFMEKV AKA VDKDELTVEERNLLSV AYK NV IGARRASWRIISSIEQKEESRGNDDHVSLIRDYRSKIETELSDICDGILKLLDTILVPAA ASGDSKVFYLKMKGDYHRYLAEFKSGQERKDAAEHTLTAYKAAQDIANSELAPTHPIRLG LALNFSVFYYEILNSPDRACNLAKQAFDEAIAELDTLGEESYKDSTLIMQLLRDNLTLWT SDMQDDV ADDIKEAAPAAAKPADEQQS>AtGF14 psiMSTREENVYMAKLAEQAERYEEMVEFMEKV AKTVDVEELSVEERNLLSV AYKNVIGARR ASWRIISSIEQKEESKGNEDHV AIIKDYRGKIESELSKICDGILNVLEAHLIPSASPAESK VFYLKMKGDYHRYLAEFKAGAERKEAAESTLV AYKSASDIATAELAPTHPIRLGLALNFS VFYYEILNSPDRACSLAKQAFDDAIAELDTLGEESYKDSTLIMQLLRDNLTLWTSDMTDE AGDEIKEASKPDGAE>AtGF14 omegaMASGREEFVYMAKLAEQAERYEEMVEFMEKVSAA VDGDELTVEERNLLSV AYKNVIGA RR ASWRIISSIEQKEESRGNDDHVTAIREYRSKIETELSGICDGILKLLDSRLIPAAASGDS KVFYLKMKGDYHRYLAEFKTGQERKDAAEHTLAAYKSAQDIANAELAPTHPIRLGLALN F SVFYYEILNSPDRACNLAKQAFDEAIAELDTLGEESYKDSTLIMQLLRDNLTLWTSDMQD DAADEIKEAAAPKPTEEQQ>AtGF14 phiMAAPPASSSAREEFVYLAKLAEQAERYEEMVEFMEKV AEA VDKDELTVEERNLLSV AYKN VIGARRASWRIISSIEQKEESRGNDDHVTTIRDYRSKIESELSKICDGILKLLDTRLVPA SANGDSKVFYLKMKGDYHRYLAEFKTGQERKDAAEHTLTAYKAAQDIANAELAPTHPIR L GLALNFSVFYYEILNSPDRACNLAKQAFDEAIAELDTLGEESYKDSTLIMQLLRDNLTLWTSDMQDESPEEIKEAAAPKPAEEQKEI>AtGF14 upsilonMSSDSSREENVYLAKLAEQAERYEEMVEFMEKV AKTVETEELTVEERNLLSV AYKNVIGA RRASWRIISSIEQKEDSRGNSDHVSIIKDYRGKIETELSKICDGILNLLEAHLIPAASLA ESKVFYLKMKGDYHRYLAEFKTGAERKEAAESTLV AYKSAQDIALADLAPTHPIRLGLAL NFSVFYYEILNSSDRACSLAKQAFDEAISELDTLGEESYKDSTLIMQLLRDNLTLWTSDL NDEAGDDIKEAPKEVQKVDEQAQPPPSQ>AtGF14 lambda MAATLGRDQYVYMAKLAEQAERYEEMVQFMEQLVTGATPAEELTVEERNLLSV AYKNVI G SLRAAWRIVSSIEQKEESRKNDEHVSLVKDYRSKVESELSSVCSGILKLLDSHLIPSAGA SESKVFYLKMKGDYHRYMAEFKSGDERKTAAEDTMLAYKAAQDIAAADMAPTHPIRLG LA LNFSVFYYEILNSSDKACNMAKQAFEEAIAELDTLGEESYKDSTLIMQLLRDNLTLWTSD MQEQMDEA>AtGF14 nuMSSSREENVYLAKLAEQAERYEEMVEFMEKV AKTVDTDELTVEERNLLSV AYKNVIGAR R ASWRIISSIEQKEESRGNDDHVSIIKDYRGKIETELSKICDGILNLLDSHLVPTASLAES KVFYLKMKGDYHRYLAEFKTGAERKEAAESTLV AYKSAQDIALADLAPTHPIRLGLALNF SVFYYEILNSPDRACSLAKQAFDEAISELDTLGEESYKDSTLIMQLLRDNLTLWNSDIND EAGGDEIKEASKHEPEEGKPAETGQ>GF14 kappa MATTLSRDQYVYMAKLAEQAERYEEMVQFMEQLVSGATPAGELTVEERNLLSV AYKNVI G SLRAAWRIVSSIEQKEESRKNEEHVSLVKDYRSKVETELSSICSGILRLLDSHLIPSATA SESKVFYLKMKGDYHRYLAEFKSGDERKTAAEDTMIAYKAAQDV A V ADLAPTHPIRLGL A LNFSVFYYEILNSSEKACSMAKQAFEEAIAELDTLGEESYKDSTLIMQLLRDNLTLWTSD MQEQMDEA>AtGF14 epsilonMENEREKQVYLAKLSEQTERYDEMVEAMKKV AQLDVELTVEERNLVSVGYKNVIGARR AS WRILSSIEQKEESKGNDENVKRLKNYRKRVEDELAKVCNDILSVIDKHLIPSSNA VESTV FFYKMKGDYYRYLAEFSSGAERKEAADQSLEAYKAA V AAAENGLAPTHPVRLGLALNFS V FYYEILNSPESACQLAKQAFDDAIAELDSLNEESYKDSTLIMQLLRDNLTLWTSDLNEEG DERTKGADEPQDEN>AtGF14 iotaMSSSGSDKERETFVYMAKLSEQAERYDEMVETMKKV ARVNSELTVEERNLLSVGYKNVI G ARRASWRIMSSIEQKEESKGNESNVKQIKGYRQKVEDELANICQDILTIIDQHLIPHATS GEATVFYYKMKGDYYRYLAEFKTEQERKEAAEQSLKGYEAATQAASTELPSTHPIRLGLA LNFSVFYYEIMNSPERACHLAKQAFDEAIAELDTLSEESYKDSTLIMQLLRDNLTLWTSDLPEDGGEDNIKTEESKQEQAKPADATEN>AtGF14 omicronMENERAKQVYLAKLNEQAERYDEMVEAMKKV AALDVELTIEERNLLSVGYKNVIGARR ASWRILSSIEQKEESKGNEQNAKRIKDYRTKVEEELSKICYDILA VIDKHLVPFATSGESTV FYYKMKGDYFRYLAEFKSGADREEAADLSLKAYEAATSSASTELSTTHPIRLGLALNFSV FYYEILNSPERACHLAKRAFDEAIAELDSLNEDSYKDSTLIMQLLRDNLTLWTSDLEEGG K>AtGF14 muMGSGKERDTFVYLAKLSEQAERYEEMVESMKSV AKLNVDLTVEERNLLSVGYKNVIGSR RASWRIFSSIEQKEA VKGNDVNVKRIKEYMEKVELELSNICIDIMSVLDEHLIPSASEGES TVFFNKMKGDYYRYLAEFKSGNERKEAADQSLKAYEIATTAAEAKLPPTHPIRLGLALNF SVFYYEIMNAPERACHLAKQAFDEAISELDTLNEESYKDSTLIMQLLRDNLTLWTSDISE EGGDDAHKTNGSAKPGAGGDDAE>AtGF14 Pi MENEREKLIYLAKLGCQAGRYDDVMKSMRKVCELDIELSEEERDLLTTGYKNVMEAKRV S LRVISSIEKMEDSKGNDQNVKLIKGQQEMVKYEFFNVCNDILSLIDSHLIPSTTTNVESI VLFNRVKGDYFRYMAEFGSDAERKENADNSLDAYKV AMEMAENSLVPTNMVRLGLALN FS IFNYEIHKSIESACKLVKKAYDEAITELDGLDKNICEESMYIIEMLKFNLSTWTSGDGNG NKTDG完成后重命名为“db”并去掉扩展名(这就是所建的库,相当于在NCBI中你所选择的搜索库)4.2在bin中再建一个txt文件>TFT1(tomato) MALPENLTREQCLYLAKLAEQAERYEEMVKFMDKLVIGSGSSELTVEERNLLSV AYKNVIGSLRAAWRIVSSIEQKEEGR KNDEHVVLVKDYRSKVESELSDVCAGILKILDQYLIPSAS AGESKVFYLKMKGDYYRYLAEFKVGNERKEAAEDTMLAYKAAQDIA V AELAPTHPIRLGLALNFSVFYYEILNASEKACS MAKQAFEEAIAELDTMGEESYKDSTLIMQLLRDNLTLWTS重命名为“in”,并去掉扩展名(相当于在NCBI中输入的query序列)。
