mdict 格式解析
FANUC系统MDI面板详解

第一课加工中心 FANUC 系统操作面板功能键及开关的使用方法一、 FANUC 系统操作面板功能键的含义三、开关的使用方法:设定自动运行方式 .:设定程序编辑方式 .:设定 MDI方式: 设定 DNC 运行方式 .: 单程序段运行方式 .:可选程序段跳过运行方式 , 跳过程序段开头带有 / 的程序 . : 程序停止 .: 手动示教 ( 手轮示教 ) 方式 .: 程序重启 .:机床机械锁住 .: 空运行方式 .: 循环停止 .( 自动操作停止 ).: 循环启动 .( 自动操作开始 ).: 程序停 .( 进给保持 ).: 返回参考点方式 .:手动进给方式 .: 手轮进给方式 .: 手轮进给倍率: 手动进给轴选择 .: 快速进给 .: 移动方向选择 .: 主轴 , 正转 , 停止 , 反转 .: 进给倍率 .:主轴转速倍率: 紧急停止: 程序保护开关六、小结:加工中心慨况及面板操作七、作业:操作面板,熟练面板各个功能键第二课加工中心加工工件的安装、对刀与换刀加工中心加工定位基准的选择:1.选择基准的三个基本要求:(1)所选基准应能保证工件定位准确装卸方便方便可靠。
(2)所选基准与各加工部位的的尺寸计算简单。
(3)保证加工精度。
2.选择定位基准6原则:(1)尽量选择设计基准作为定位基准;(2)定位基准与设计基准不能统一时,应严格控制定位误差保证加工精度;(3)工件需两次以上装夹加工时,所选基准在一次装夹定位能完成全部关键精度部位的加工;(4)所选基准要保证完成尽可能多的加工内容;(5)批量加工时,零件定位基准应尽可能与建立工件坐标系的对刀基准重合;(6)需要多次装夹时,基准应该前后统一。
加工中心夹具的确定:1.对夹具的基本要求:(1)夹紧机构不得影响进给,加工部位要敞开;(2)夹具在机床上能实现定向安装;(3)夹具的刚性与稳定性要好。
2.常用夹具种类:(1)通用夹具:如虎钳、分度头、卡盘等;(2)组合夹具:组合夹具由一套结构已经标准化、尺寸已经规格化的通用元件组合元件所构成;(3)专用夹具:专为某一项或类似的几项加工设计制造的夹具;(4)可调整夹具:组合夹具与专用夹具的结合,既能保证加工的精度,装夹更具灵活性;(5)多工位夹具:可同时装夹多个工件的夹具;(6)成组夹具:专门用于形状相似、尺寸相近且定位、夹紧、加工方法相同或相似的工件的装夹。
对于TMS320F2812的CMD文件的理解分解

对于TMS320F2812的CMD文件的理解1.COFF格式要谈CMD文件,首先不可避免的要谈下COFF格式,COFF格式是通用目标文件格式(Common Object File Format)的缩写,它是一种流行的二进制可执行文件格式,在DSP里二进制可执行文件包括库文件(.lib)、目标文件(.obj)和最终可执行文件(.out)。
详细的COFF格式文件包括段头、可执行代码、初始化数据、可重定位信息、行号入口、符号表、字符串。
对于DSP的C语言编程我们只需要了解定义段和给段分配空间即可。
采用COFF格式更利于我们对其进行模块化编程,我们可以自由的把哪些段分配到哪些空间。
2.Section(1)其次,在编写CMD文件得时候要碰到SectionS命令,SectionS命令的英文理解就有区域的意思,我们利用SectionS来将目标文件的代码放到指定的区域中。
SectionS目标文件中最小的单位我们称之为块,一个块就是最终在存储器映像中占据连续空间的一段代码或者数据。
COFF文件格式默认有三个块:.text 存放可执行代码;.data 存放已初始化数据;.bss 为未初始化数据留下的保留空间。
(2)汇编器对块的处理和设置未初始化块的设置:.bss 变量存放空间;.usect 用户自定义的未初始化段;初始化块的设置:.text 汇编指令代码.data 常数数据(比如对变量的初始化数据).sect 用户自定义的已初始化段.asect 类似于.sect,多了绝对地址定位功能,一般不用(3)C语言对块得的设置和处理未初始化块(data).bss 存放全局和静态变量.ebss 长调用的.bss(超过了64K地址限制).stack 存放C语言的栈.sysmem 存放C语言的堆.esysmem 长调用的.sysmem(超过了64K地址限制)初始化块.text 可执行代码和常数(program).switch switch语句产生的常数表格(program/低64K数据空间).pinit Tables for global constructors (C++)(program).cinit 用来存放对全局和静态变量的初始化常数值(program).const 全局和静态的const变量初始化值和字符串常数,(data).econst 长.const(可定位到任何地方)(data)(4)C语言自定义块#pragma DATA_SECTION(函数名或者全局变量名,“用户自定义在数据空间的段名”);#pragma CODE_SECTION(函数名或者全局变量名,“用户自定义在程序空间的段名”);必须注意:不能在函数体内声明,必须在定义和使用前声明。
【常见的各种文件类型】各种常用文件类型的详细讲解

【常见的各种文件类型】各种常用文件类型的详细讲解各种常用文件类型的详细讲解会昌天宇高科电脑培训教程之文件类型acamicrosoft的代理使用的角色文档acf系统管理配置acm音频压缩管理驱动程序,为windows系统提供各种声音格式的编码和解码功能aif声音文件,支持压缩,可以使用windowsmediaPlayer和QuickTimePlayer播放aiF音频文件,使用windowsmediaPlayer播放aiFc音频文件,使用windowsmediaPlayer播放aiFF音频文件,使用windowsmediaPlayer播放ani动画光标文件扩展名,例如动画沙漏。
ansaScii字符图形动画文件arc一种较早的压缩文件,可以使用winzip,winRaR,PKaRc等软件打开arj压缩文件。
可以使用winzip,winRaR,PKaRc等软件打开asf微软的媒体播放器支持的视频流,可以使用windowsmediaPlayer 播放asp微软的视频流文件,可以使用windowsmediaPlayer打开asp微软提出的activeServerPage,是服务器端脚本,常用于大型网站开发,支持数据库连接,类似PHP。
可以使用Visualinterdev编写,是目前的大热门asxwindowsmedia媒体文件的快捷方式au是internet中常用的声音文件格式,多由Sun工作站创建,可使用软件waveformHoldandmodify播放。
netscapenavigator中的Liveaudio 也可以播放.au文件avi一种使用microsoftRiFF规范的windows多媒体文件格式,用于存储声音和移动的图片bak备份文件,一般是被自动或是通过命令创建的辅助文件,它包含某个文件的最近一个版本,并且具有于该文件相同的文件名basBasic语言源程序文件,可编译成可执行文件,目前使用Basic开发系统的是VisualBasicbat批处理文件,在mS-doS中,.bat文件是可执行文件,有一系列命令构成,其中可以包含对其他程序的调用bbs电子告示板系统文章信息文件bfcwindows的公文包文件bin二进制文件,其用途依系统或应用而定bmpBitmap位图文件,这是微软公司开发Paint的自身格式,可以被多种windows和windowsnT平台及许多应用程序支持,支持32位颜色,用于为windows界面创建图标的资源文件格式。
各种文件格式含义

DWG文件是CA D图形,不是图片文件,如果高版本的DWG文件,用低版本的可能无法打开L RC歌词是一种通过编辑器把歌词按歌曲歌词出现的时间编辑成一个文件,在播放歌曲时同步依次显示出来的一种歌词文件。
把歌曲和L RC歌词命为相同的文件名放在同一目录下,用带显示歌词功能的播放器播放歌曲时歌词就可以同步显示显示。
*.dat文件一般是VCD中的视频。
文件如果是文本格式的.d at文件,用记事本之类的软件就可以直接打开,而且无需转换。
如果是VCD中的.da t文件则需要用VCD播放软件打开,而且不能转换为文本文件。
还有某些软件的一些数据文件也是.dat扩展名,这些文件不能直接打开,也不能转换成文本文件。
*.dat文件如果不能用系统自带的媒体播放机打开,也可以从网上下载VCD专用播放器——迷你V CD播放器。
swf是flas h文件,浏览器可以打开,fla shpla yer也可以打开,另外还有一些flash播放器也可以打开。
APK是A ndroi d Pac kage的缩写,即A ndroi d安装包(anapk)。
)。
A PK是类似Symbi an Si s或Sis x的文件格式。
通过将APK文件直接传到A ndroi d模拟器或Andro id手机中执行即可安装。
apk文件和sis一样最终把a ndroi d sdk编译的工程打包成一个安装程序文件格式为a pk。
A PK文件其实是zip格式。
t mp 的文件大部分都是因为不正常关机、或死机后所留下的文件,这些临时的暂存盘,在你重新开机后,已经没有任何的用途,可以放心删除。
另外,Win dows在长年累月的使用之下,在 Wi ndows\Temp里一定会多出很多的「垃圾」,其实这T emp 里的所有的文件、或文件夹都是可以删除的,有空不妨常常清理下这个Temp文件夹,但是要留意一下,我指的是删除Temp里的东西(Temp文件夹还是保留的),不是要你把整个 Te mp 连文件夹一起删掉。
FANUC系统MDI面板详解
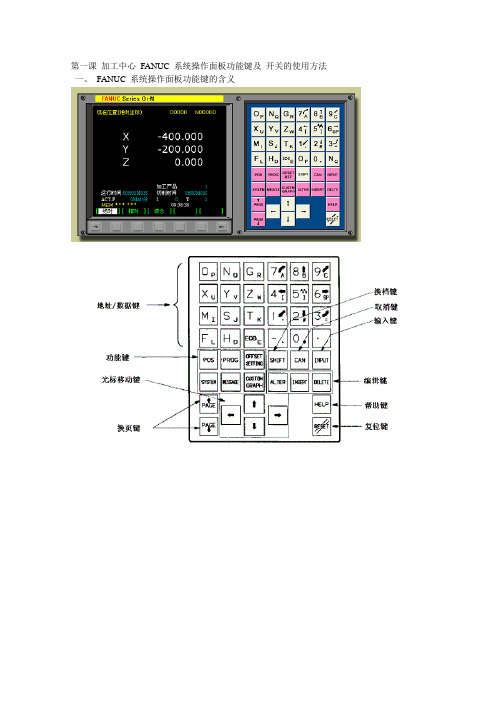
第一课加工中心FANUC 系统操作面板功能键及开关的使用方法一、FANUC 系统操作面板功能键的含义三、开关的使用方法:设定自动运行方式.:设定程序编辑方式.:设定MDI方式: 设定DNC 运行方式 .: 单程序段运行方式.:可选程序段跳过运行方式, 跳过程序段开头带有/ 的程序. : 程序停止.: 手动示教( 手轮示教) 方式 .: 程序重启.:机床机械锁住.: 空运行方式.: 循环停止.( 自动操作停止).: 循环启动.( 自动操作开始).: 程序停 .( 进给保持).: 返回参考点方式.:手动进给方式.: 手轮进给方式.: 手轮进给倍率: 手动进给轴选择.: 快速进给.: 移动方向选择.: 主轴, 正转, 停止, 反转.: 进给倍率.:主轴转速倍率: 紧急停止: 程序保护开关六、小结:加工中心慨况及面板操作七、作业:操作面板,熟练面板各个功能键第二课加工中心加工工件的安装、对刀与换刀加工中心加工定位基准的选择:1.选择基准的三个基本要求:(1)所选基准应能保证工件定位准确装卸方便方便可靠。
(2)所选基准与各加工部位的的尺寸计算简单。
(3)保证加工精度。
2.选择定位基准6原则:(1)尽量选择设计基准作为定位基准;(2)定位基准与设计基准不能统一时,应严格控制定位误差保证加工精度;(3)工件需两次以上装夹加工时,所选基准在一次装夹定位能完成全部关键精度部位的加工;(4)所选基准要保证完成尽可能多的加工内容;(5)批量加工时,零件定位基准应尽可能与建立工件坐标系的对刀基准重合;(6)需要多次装夹时,基准应该前后统一。
加工中心夹具的确定:1.对夹具的基本要求:(1)夹紧机构不得影响进给,加工部位要敞开;(2)夹具在机床上能实现定向安装;(3)夹具的刚性与稳定性要好。
2.常用夹具种类:(1)通用夹具:如虎钳、分度头、卡盘等;(2)组合夹具:组合夹具由一套结构已经标准化、尺寸已经规格化的通用元件组合元件所构成;(3)专用夹具:专为某一项或类似的几项加工设计制造的夹具;(4)可调整夹具:组合夹具与专用夹具的结合,既能保证加工的精度,装夹更具灵活性;(5)多工位夹具:可同时装夹多个工件的夹具;(6)成组夹具:专门用于形状相似、尺寸相近且定位、夹紧、加工方法相同或相似的工件的装夹。
牛津英语搭配词典mdict

牛津英语搭配词典mdict
牛津英语搭配词典(Oxford Collocations Dictionary)是一
本权威的英语词典,旨在帮助学习者准确地使用英语单词的搭配。
该词典以Mdict格式提供,Mdict是一种常见的电子词典格式,可
在各种平台上使用。
牛津英语搭配词典的Mdict版本具有以下特点和优势:
1. 丰富的词汇搭配信息,该词典收录了大量的英语单词,并提
供了详细的词汇搭配信息,包括常见的动词、名词、形容词和副词
的搭配用法,帮助学习者更准确地表达自己的意思。
2. 多角度的搭配示例,词典中提供了丰富的例句,展示了单词
在不同语境中的搭配用法,帮助学习者理解和掌握单词的实际用法。
3. 系统化的搭配分类,词典中的搭配信息按照不同的语法和语
义类别进行了分类,使学习者更好地理解和记忆搭配用法。
4. 用户友好的界面和搜索功能,Mdict格式的词典通常具有用
户友好的界面和强大的搜索功能,可以快速准确地查找所需的搭配
信息。
使用牛津英语搭配词典的Mdict版本,学习者可以更好地掌握
英语单词的搭配用法,提升自己的语言表达能力。
通过详细的搭配
信息和丰富的例句,学习者可以更加准确地选择和运用单词,使自
己的表达更加地自然和地道。
同时,Mdict格式的词典还具有便捷
的特点,可以随时随地在电子设备上使用,满足学习者的各种需求。
总之,牛津英语搭配词典的Mdict版本是一款功能强大、实用
性高的英语学习工具,可以帮助学习者全面准确地掌握英语单词的
搭配用法,提升自己的语言水平。
FANUC系统MDI面板详解2

数控系统操作面板也称CRT(或LCD)/ MDI面板,是数控系统的主要组成部分,如图1所示,操作面板的左侧是CRT(即荧光屏显示器)显示器或LCD显示器(即液晶显示器),在显示器下面的一行键称为软键,每个软键的用途在不同的显示界面中是不同的。
操作面板右侧是MDI键盘,MDI键盘上的键按用途分为功能键、编辑键、数据输入键等。
图1 数控系统操作面板数控机床操作面板一般分两大部分,除了数控系统操作面板外,还有一部分是机床操作面板,机床的各项操作大部分通过机床操作面板来完成。
一般来说机床操作面板比较容易上手,而数控系统操作面板属于数控系统的输入/输出装置,其功能是完成人与数控机床之间的人机交互,相当于个人电脑配置中的键盘与显示器。
MDI面板与机床面板在操作上是不能独立分开的,一般来说,机床面板是为MDI面板服务的。
FANUC系统各种版本的MDI键盘在布局上虽然有差别,但是按键的名称与用途基本上差别不大,下面以FANUC 0i -TC系统为例介绍FANUC系统的MDI面板。
FANUC 0i -TC系统的MDI面板布局如图2所示。
MDI键盘的布局如图3所示。
图2 FANUC 0i -TC系统的MDI面板布局图3 FANUC 0i -TC系统MDI键盘布局显示器下面软键的具体名称随着按下的功能键而改变,如果按下相应的软键,没有出现所需的画面,可通过按“翻页键”进行翻页查找。
㈠功能键。
在操作MDI键盘时,应先按相应的功能键,然后再按该功能键所对应的软键。
在MDI键盘上共有六个功能键,如图4所示,分别是“POS键”、“PROG键”、“OFS/SET 键”、“SYSTEN键”、“MESSAGE键”以及“CSTM/GR键”。
图4 功能键1.“POS键”。
按此键显示位置画面。
按下“POS键”后,对应的软键主要有三个,即“绝对(ABS)”,显示绝对坐标画面;“相对(REL)”,显示相对坐标画面;“综合(ALL)”,显示所有坐标画面。
SMITIDstruct 数据结构和操作工具说明说明书

Package‘SMITIDstruct’October12,2022Type PackageEncoding UTF-8Title Data Structure and Manipulations Tool for Host and ViralPopulationVersion0.0.5Date2019-06-14Author Jean-Francois Rey[aut,cre]Maintainer Jean-Francois Rey<*************************>Description Statistical Methods for Inferring Transmissions of Infectious Diseases from deep se-quencing data(SMITID).It allow sequence-space-time host and viral population data storage,indexation and querying. License GPL(>=2)|file LICENSELazyData trueBuildVignettes trueNeedsCompilation noBiarch trueURL https://informatique-mia.inra.fr/biosp/anr-smitid-project,https://gitlab.paca.inra.fr/SMITID/structRBugReports https://gitlab.paca.inra.fr/SMITID/structR/issuesDepends methods,utils,grDevices(>=3.0.0),graphics(>=3.0.0),R(>=3.3.0)DependsNote BioC(>=3.0)Imports ggplot2,sf(>=0.6.3),stats(>=3.0.2),Biostrings(>=2.0.0)ImportsNote BioC(>=3.0),Recommended:BiostringsSuggests testthat(>=2.0)Collate'Class-Host.R''Class-ViralPop.R''Methods-Host.R''Methods-ViralPop.R''Methods-time.R''SMITIDstruct.R''demo.R''diversity.R''index.R'12R topics documented:RoxygenNote6.1.1Repository CRANDate/Publication2019-06-1411:30:11UTCR topics documented:SMITIDstruct-package (3)addcode (4)addHost (4)addIndex (5)addViralObs (5)alleleCount (6)concatViralPop (6)createA ViralPop (7)createHost (7)createIndex (8)demo.SMITIDstruct.run (8)diversity.pDistance (8)diversity.sfs (9)getCov (9)getDate (10)getDiversity.pDistance (10)getDiversity.sfs (11)getInfosByHostAndTime (11)getStates (12)getTimeLine (12)getTimestamp (13)getTransmissionTree (13)Host (14)is.juliendate (14)is.StringDate (15)is.timestamp (15)isInCode (16)loadCoords (16)loadCovs (17)loadHost (17)loadStates (18)loadTree (18)loadTreeDF (19)loadViralObs (20)loadViralPop (20)loadViralPopSet (21)mergeCode (21)plotDiversity.pDistance (22)plotDiversity.sfs (22)setStates (23)simulateStates (23)SMITIDstruct-package3 ViralPop (24)Index25 SMITIDstruct-package Data Structure and Manipulation Tool for Host and Viral PopulationDescriptionStatistical Methods for Inferring Transmissions of Infectious Diseases from deep sequencing data (SMITID).It allow sequence-space-time host and viral population data storage,indexation and querying.DetailsPackage:SMITIDstructType:PackageVersion:0.0.5Date:2019-06-14License:GPL(>=2)The SMITIDstruct package contains functions and methods for manipulating Host and Viral popu-lation genotico-space-time data.Author(s)Jean-Francois Rey<*************************>Maintainer:Jean-Francois Rey<*************************>See Alsodemo.SMITIDstruct.runExamples##Run a simulationlibrary("SMITIDstruct")demo.SMITIDstruct.run()4addHost addcode addcodeDescriptionadd a code event to an anotherUsageaddcode(code,code.add)Argumentscode an existing codecode.add the code to addValuemerge of the two codeaddHost addHostDescriptionadd an Host to a HostSetUsageaddHost(lhost,id)Argumentslhost a hostSet Objectid a character of host IDValuea HostSet of host object with there IDExampleslhost<-list()lhost<-addHost(lhost,"42")addIndex5 addIndex addIndexDescriptionadd to an index a new eventcodeUsageaddIndex(index,id_host,time,code)Argumentsindex an indexid_host an host index in HostSettime a timecode an event codeValuethe index updated(add a row or update one)addViralObs addViralObsDescriptionload Viral pop observation in Host objectUsageaddViralObs(lhost,lvpop)Argumentslhost a HostSetlvpop a ViralPopSetValuelhost update with viral population observed6concatViralPop alleleCount alleleCountDescriptioncount allele at each positionUsagealleleCount(mat,seq.char=c("A","T","G","C"))Argumentsmat a genomique seq list as matrix by rowseq.char allele alphabetValuea matrix,each row as a unique seq and col as allele count by positionconcatViralPop concatViralPopDescriptionconcat several Viral population in one ViralPop objectUsageconcatViralPop(lvpop,lid)Argumentslvpop a ViralPop Setlid vector of viralpop id to concatValuea ViralPop object with ID concatenation from all IDs and time at0.createA ViralPop7 createAViralPop createAViralPopDescriptionCreate a new ViralPop objectUsagecreateAViralPop(host_id,obs_time,seq,id_seq="seq_ID",seq_value="seq",prop="prop",compact=FALSE)Argumentshost_id host ID which viral pop is observedobs_time time of the observation(numeric or date)seq a data.frame of sequences ID,sequences and countsid_seq column name containing the sequences IDseq_value column name containing the sequencesprop column name containing the count of each sequencescompact boolean,default FALSE,if TRUE will try group identicals sequences(not im-plemented yet)createHost createHostDescriptioncreate a list of Host class objectUsagecreateHost(list_host)Argumentslist_host a character vector of host IDValuea HostSet of host object with there IDExampleslh<-seq(1,30,1)lhost<-createHost(lh)8diversity.pDistance createIndex createIndexDescriptioncreate an index of time id_host and event codeUsagecreateIndex(hostlist)Argumentshostlist a HostsetValuea data.frame with TIME,ID_HOST and EVENTCODE as columnsdemo.SMITIDstruct.run demo.SMITIDstruct.runDescriptionrun a demo to load HostSet,ViralPopSet and indexUsagedemo.SMITIDstruct.run()diversity.pDistance diversity.pDistanceDescriptiondiversity calculation using Mean Pairwise DistanceUsagediversity.pDistance(vpop)Argumentsvpop a ViralPop objectValueresultdiversity.sfs9 diversity.sfs diversity.sfsDescriptionAllele frequency spectrum or Site frequency spectra:the distribution of alternative allele frequen-cies across all sites of genetic sequencesUsagediversity.sfs(vpop)Argumentsvpop a viralPop classValuethe site frequency spectragetCov getCovDescriptionget Host(s)covariatesUsagegetCov(lhost,id=NA)Argumentslhost a HostSetid a vector of host id(default NA:all lhost)Valuea data.frame10getDiversity.pDistance getDate getDateDescriptionConverte timestamp to Date(string)UsagegetDate(time,format="%Y-%m-%dT%H:%M:%S")Argumentstime a timestamp or vector offormat Date format output(default%Y-%m-%dT%H:%M:%S)Valuetime as string dategetDiversity.pDistancegetDiversity.pDistanceDescriptionget pairwise distance of an host over viral population observatedUsagegetDiversity.pDistance(host,lvpop)Argumentshost an Host objectlvpop a ViralPopSet objectValuea data.frame with col as time of observation and p_distancegetDiversity.sfs11 getDiversity.sfs getDiversity.sfsDescriptionget Allele Frequency Spectrum or Site Frequency spectra for observated viral pop of an hostUsagegetDiversity.sfs(host,lvpop)Argumentshost an Host objectlvpop an ViralPopSet objectValuea list indexed by time that contains allele.time and countgetInfosByHostAndTime getInfosByHostAndTimeDescriptionget hosts informations,status,infectedby,coordinates and timeUsagegetInfosByHostAndTime(index,lhost)Argumentsindex an indexlhost a hosts listValuea data.frame with colnames(id,time,infectedby,status,probabilities,X,Y)12getTimeLine getStates getStatesDescriptionget Host(s)statesUsagegetStates(lhost,id=NA)Argumentslhost a HostSetid a vector of host id(default NA:all lhost)Valuea data.framegetTimeLine getTimeLineDescriptionget the time line of an hostUsagegetTimeLine(lhost,id)Argumentslhost a hostSetid a host IDValuea data.framegetTimestamp13 getTimestamp getTimestampDescriptionGet the timestamp of DateUsagegetTimestamp(date,format="%Y-%m-%dT%H:%M:%S")Argumentsdate a date(as string)or vector offormat the date format(default%Y-%m-%dT%H:%M:%S)Valuetimestamp of the date(s)getTransmissionTree getTransmissionTreeDescriptionget a transmission tree as a data.frameUsagegetTransmissionTree(lhost,id=NA)Argumentslhost a hostSetid a vector of hosts ids(default NA:all host)Valuea data.frame as source|target|time in columnsExamplespath=system.file("extdata","data-simul/",package="SMITIDstruct")lhost<-list()lhost<-loadTree(lhost,paste(path,"/tree.txt",sep= ))print(getTransmissionTree(lhost))14is.juliendate Host Class HostDescriptionSpatio-temporal information about Host.DetailsObject can be created by calling...rdname Host-classSlotsID Host identifiercoordinates Host coordinates in time(as sf)states Host States/Status(dob,Inf...)sources data.frame of time and host id who infected this hostoffsprings data.frame of time and host id who has been contamined by this hostID_V_POP data.frame of time and index of Viral population Observationcovariates data.frame of time,cavariate and value of this host.is.juliendate is.juliendateDescriptionChekc if a numeric is not a timestampUsageis.juliendate(time)Argumentstime a numericValueTRUE if time is a julien day,otherwise FALSEis.StringDate15 is.StringDate is.StringDateDescriptionCheck if a string represent a dateUsageis.StringDate(date)Argumentsdate a string or a vector of string(without NA)ValueTRUE if date contains date formatis.timestamp is.timestampDescriptionCheck if a numeric represent a timestampUsageis.timestamp(time)Argumentstime a numericValueTRUE if time>=197116loadCoords isInCode isInCodeDescriptioncheck a code contains a specific codeUsageisInCode(code,thecode)Argumentscode list of code to testthecode the real codeValueTRUE if code contain thecode otherwise FLASEloadCoords loadCoordsDescriptionLoad Hosts statesUsageloadCoords(lhost,dfCoords,id="ID")Argumentslhost a HostSetdfCoords a data.frame with host ID,time and longitude latitude valuesid colname for host IDValuelhost updatedExamplespath=system.file("extdata","data-simul/",package="SMITIDstruct")lhost<-list()lhost<-loadTree(lhost,paste(path,"/tree.txt",sep= ))coords<-read.table(file=paste(path,"/hosts_coords.txt",sep= ),header=TRUE,s=FALSE) lhost<-loadCoords(lhost,coords)loadCovs17 loadCovs loadCovsDescriptionLoad Hosts covariatesUsageloadCovs(lhost,dfCovs,id="ID",colCovs)Argumentslhost a HostSetdfCovs a data.frame with host ID in rows and covariates in columnsid colname for host IDcolCovs colnames of covariates columnsValuelhost updated with covariatesloadHost loadHostDescriptionload host object from afileUsageloadHost(file="host.txt")Argumentsfile afile containing hosts dataValuea list of Host object(HostSet)include Class-Host.R18loadTree loadStates loadStatesDescriptionLoad Hosts statesUsageloadStates(lhost,dfStates,id="ID",colStates)Argumentslhost a HostSetdfStates a data.frame with host ID and states in columns and time as valueid colname for host IDcolStates colnames of States columnsValuelhost updatedExamplespath=system.file("extdata","data-simul/",package="SMITIDstruct")lhost<-list()class(lhost)<-"hostSet"lhost<-loadTree(lhost,paste(path,"/tree.txt",sep= ))obs<-read.table(paste(path,"/obs.txt",sep= ),header=TRUE,s=FALSE)obs.states<-c(colnames(obs[-grep("ID|Tobs.*",colnames(obs))]))lhost<-loadStates(lhost,obs,colStates=obs.states)loadTree loadTreeDescriptionload sources and offsprings fromfileUsageloadTree(lhost=list(),file="tree.txt",source="ID-source",receptor="ID-receptor",tinf="Tinf",weight="Weight")loadTreeDF19Argumentslhost a HostSetfile afile containing tree datasource column name for source IDreceptor column name for receptor IDtinf column name for infection Timeweight column name of infection weightValuethe lhost param update with sources and offspringsExamplespath=system.file("extdata","data-simul/",package="SMITIDstruct")lhost<-list()class(lhost)<-"hostSet"lhost<-loadTree(lhost,paste(path,"/tree.txt",sep= ))loadTreeDF loadTreeDFDescriptionload sources and offsprings from a data.frameUsageloadTreeDF(lhost=list(),df=data.frame(),source="ID-source",receptor="ID-receptor",tinf="Tinf",weight="Weight")Argumentslhost a HostSetdf a data.frame containing tree datasource column name for source IDreceptor column name for receptor IDtinf column name for infection Timeweight infection links probabilityValuethe lhost param update with sources and offsprings20loadViralPop loadViralObs loadViralObsDescriptionload a ViralPop objectUsageloadViralObs(id,time,file)Argumentsid host pathogen IDtime time of the observation(numeric or Date)file a fastafileValuea new ViralPop objectloadViralPop loadViralPopDescriptionLoad all ViralPop observated in thefile.obsUsageloadViralPop(directory,listFiles,listCol=list(id="id",timeObs="time",filename="filename"),file.extension="fasta")Argumentsdirectory path where is datalistFiles a dataframe with host ID,time observation andfile name(filename.fasta)listCol a list of listFiles colomns names("id","timeObs","filename")file.extension genotypefile extensionValuea vector of VirlaPop objectloadViralPopSet21 Examplespath=system.file("extdata","data-simul/",package="SMITIDstruct")files<-list.files(path,pattern=".*.fasta",s=FALSE)lfileinfo<-sapply(files,function(x){return(substr(x,1,nchar(x)-6))})splitFiles<-strsplit(lfileinfo,"_");listF<-cbind(data.frame(matrix(unlist(splitFiles),nrow=length(splitFiles),byrow=TRUE), stringsAsFactors=FALSE),names(splitFiles))colnames(listF)<-c("id","time","filename")lvpop<-loadViralPop(path,listF)loadViralPopSet loadViralPopSetDescriptionload a list of viral populationsUsageloadViralPopSet(lvpop=list(),list)Argumentslvpop a viralPopSet(default new one)list a list(see details)DetailsThe list have to be on this format:list$HOST_ID$TIME$list$seq_id$seq$prop A list indexed by host ID,follow by a list indexed by time(of observation).The last list contains an array of seq_ID (sequence ID),an array of seq(sequence as characters),and an array of the count of seq.exam-ple:$’HOST_42’$’2014-01-01T00:00:00’$seq_ID["SEQ_1","SEQ_2"]$’HOST_42’$’2014-01-01T00:00:00’$seq["ACGT","TGCA"]$’HOST_42’$’2014-01-01T00:00:00’$seq_ID["46","6"]mergeCode mergeCodeDescriptionmerge a list of event codeUsagemergeCode(listcode)22plotDiversity.sfsArgumentslistcode a list of event code*Valuea codeplotDiversity.pDistanceplotDiversity.pDistanceDescriptionplot Mean Pairwise Distance for an host viralpop over timeUsageplotDiversity.pDistance(host,lvpop)Argumentshost an Host objectlvpop a ViralPopSet objectplotDiversity.sfs plotDiversity.sfsDescriptionplot Allele frequency spetrum for an host viralpop over timeUsageplotDiversity.sfs(host,lvpop)Argumentshost an Host objectlvpop an ViralPopSet objectsetStates23 setStates setStatesDescriptionset hosts states from a data.frameUsagesetStates(lhost,dfStates,colStates=c(id="ID",time="time",states="value"))Argumentslhost a HostSetdfStates a data.frame with host ID and states and time in columnscolStates vector of the columns name,id,time and statesValuethe HostSet updatedsimulateStates simulateStatesDescriptionsimulate states from sources infectionUsagesimulateStates(lhost)Argumentslhost a HostSetValuelhost update with states from sources time~24ViralPop ViralPop Class ViralPopDescriptionViral population data containing genotypesSlotsID Host identifiertime Observation time as numeric since1970/01/01size Qt of variantsnames list of variants id with same sequencegenotypes all variants genotypes(as DNAStringSet)proportions proportions of each variantsIndex_PACKAGE(SMITIDstruct-package),3 addcode,4addHost,4addIndex,5addViralObs,5alleleCount,6 concatViralPop,6 createAViralPop,7 createHost,7createIndex,8demo.SMITIDstruct.run,3,8 diversity.pDistance,8 diversity.sfs,9getCov,9getDate,10getDiversity.pDistance,10 getDiversity.sfs,11 getInfosByHostAndTime,11 getStates,12getTimeLine,12 getTimestamp,13 getTransmissionTree,13Host,14Host-class(Host),14is.juliendate,14is.StringDate,15is.timestamp,15isInCode,16loadCoords,16loadCovs,17loadHost,17loadStates,18loadTree,18loadTreeDF,19loadViralObs,20loadViralPop,20loadViralPopSet,21mergeCode,21plotDiversity.pDistance,22plotDiversity.sfs,22setStates,23simulateStates,23SMITIDstruct(SMITIDstruct-package),3SMITIDstruct-package,3ViralPop,24ViralPop-class(ViralPop),2425。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
mdict 格式解析
mdict 格式是一种用于存储和管理英语词典的电子词典格式。
它以二进制方式存储,通常使用 .mdx 和 .mdd 文件扩展名。
.md(x) 文件包含词典的索引信息和单词的定义,而.mdd 文件则包含音频、图片或其他多媒体资源。
在 mdict 格式中,词典的索引采用了特定的数据结构,如压缩字典树(Compressed Trie)或者 Hash Table,以实现快速的单词查询。
通常,为了访问 mdict 格式的词典内容,需要使用专门的软件或应用程序,例如GoldenDict、MDict 等。
mdict 格式广泛应用于英语学习和翻译工具中,因为它可以提供离线查询、快速检索和丰富的多媒体资源支持。
1。
