2010-4-22-微生物分子生态学
微生物学中的微生物资源和微生物分子生态学

微生物学中的微生物资源和微生物分子生态学微生物是地球上最早出现的生物体,祖先最可能是非细胞体的原核生物,具有非常灵活和多样的基因,随着演化逐渐演变成目前细菌、古菌、真核生物等三个领域,同时又派生出不计其数的亚型、种等。
虽然人类已经大规模研究微生物很多年,但微生物的汇聚现象、多样性和功能仍不为人类所完全掌握。
微生物资源是指自然界中的微生物,它们有利于人类的生产和生活。
微生物界种类多样,数量极其庞大,它们生存的环境和方式也非常复杂多样。
微生物资源主要包括抗生素、生物农药、生物肥料、微生物修复剂、微生物工程菌等。
微生物资源的主要价值在于它们能代替人类繁琐的工作,同时在很多领域中,微生物资源能为我们创造更好的生活。
而同样重要的是,微生物对于环境和生态的重要作用,其中微生物分子生态学就是一个十分重要的研究方向。
微生物分子生态学是指以基于微生物分子的研究方法为主的生态学,它通过对微生物群体结构和功能变化进行研究,探讨微生物的生态演化和种群动态变化,为研究各种生态环境提供生物学基础数据。
微生物分子生态学的研究对象包括环境微生物、肠道微生物、人体微生物群落、土壤微生物生态系统等。
研究方法包括PCR- DGGE 技术、微生物组学、微生物群落构成分析、功能基因组学等多种方法。
其中PCR-DGGE 技术是当前研究微生物分子生态学中使用较为广泛的一种,它可以检测微生物群体内已知或未知的细菌主要峰,通过检测各种峰的强度确定微生物群体的多样性指数、丰度等指标,为研究微生物群体评估生态演化及其生态重要性提供了一个快速和可靠的方法。
微生物分子生态学的研究发现,微生物通过累积基因的变异和重组等遗传方式,形成了非常复杂的群体结构,产生了一系列的群体效应。
而生态系统中生物与环境之间的相互关系非常复杂,微生物的活动对环境的影响不单单是直接生长和代谢所产生的物质变化,还包括在多种生物和非生物因素作用下产生的种群和交互效应,如融合、竞争和共生等。
【国家自然科学基金】_微生物分子生态学_基金支持热词逐年推荐_【万方软件创新助手】_20140801
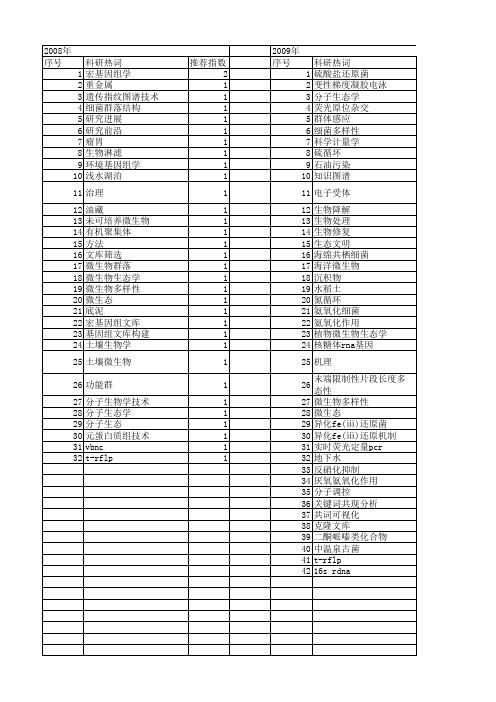
2012年 序号 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36
科研热词 推荐指数 长江口 1 铁氧化还原 1 胃肠道微生物 1 群落多样性 1 组成特征 1 碳-硫-氮循环 1 生物可利用性磷 1 浮游细菌类群 1 氨氧化古菌 1 无机磷细菌 1 扩增核糖体限制性酶切片段分析 1 微生物 1 干旱 1 巢湖 1 小鼠肠道菌群 1 宏基因组dna 1 宏基因组 1 奇古菌 1 外生菌根共生体 1 地质突变期 1 地质微生物功能群 1 地球生物学 1 响应 1 呼吸型异化铁还原菌 1 发酵型异化铁还原菌 1 功能基因 1 分子生态学 1 分子化石 1 光合作用 1 元基因组学 1 中温泉古菌 1 shewanella 1 rrna基因 1 pcr 1 geobacter 1 16s rdna 1
推荐指数 2 2 2 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
2010年 序号 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39
科研热词 多样性 分子生态学 食品微生物学 革兰阴性菌结合蛋白 锰结核 调控机制 虫害诱导的植物挥发物 芽胞杆菌 肽聚糖识别蛋白 肥胖 肠道菌群 肠道微生态 群落 纤维降解 益生菌 瘤胃微生物 甲烷 生物化学技术 特定腐败菌 激发子 海洋生物学 水产品 氢化 模式识别受体 植物微生物生态学 微生物鉴定 微生物群落结构 反硝化菌群落 功能基因 分子生物学技术 分子技术 内毒素 先天免疫 信号转导 保鲜技术 代谢性疾病 代谢 中国多金属结核区 东太平洋
微生物分子生态学及其应用

微生物分子生态学及其应用随着科技的不断进步和生物学研究的深入,微生物分子生态学逐渐成为了一个热门的研究领域。
微生物分子生态学是指通过分析微生物的分子组成和动态变化,揭示微生物间的相互作用及其与环境的关联,探索微生物生态系统的演变和调控机制的学科。
相较于传统的微生物学研究,微生物分子生态学能够更准确、更全面地研究微生物与环境间的关联,使得微生物的研究更具针对性。
微生物分子生态学通过分析微生物的分子生物学信息,可以深入探究微生物的生理、代谢、生态等各个方面,并进一步揭示微生物的生境分布、演化和生态功能。
这不仅有助于更深入地理解微生物的生态系统,也为微生物的应用研究提供了有力的支撑。
1. 微生物分子生态学的研究方法微生物分子生态学一般通过以下方法进行研究:(1)高通量测序技术高通量测序技术大大提高了微生物分子生态学研究的效率和准确度,尤其在微生物群落结构和功能的研究中应用广泛。
基于高通量测序技术,不仅能够分析微生物群落的构成,还可以揭示微生物间的相互作用及其与环境的关联。
(2)荧光原位杂交技术荧光原位杂交技术常用于微生物群落结构和空间分布的研究。
该技术通过使用荧光标记引物,能够将特定细菌、真菌或病毒等微生物直接标记并固定在试样中,观察其在不同空间中的分布情况,进而分析微生物间的相互作用。
(3)质谱分析技术质谱分析技术可以分析微生物的代谢产物,并结合高通量测序技术或荧光原位杂交技术等技术,深入探究微生物的代谢途径和功能。
2. 微生物分子生态学在环境保护中的应用微生物在环境保护中有着重要的作用,而微生物分子生态学则为环境保护提供了更加有效的手段。
(1)土壤污染修复土壤污染是一个长期而严重的问题,微生物可以分解或转化污染物,促进土壤的简易修复。
通过微生物分子生态学的研究,不仅可以深入了解微生物的生理代谢机制,还能针对特定污染物的生态功能和代谢途径,实现更加精准的修复。
(2)环境监测微生物群落是环境中的重要组成部分,通过对微生物群落的组成、分布和转化过程的研究,可以更加精准地评估环境状况。
微生物多样性的分子生态学研究

微生物多样性的分子生态学研究微生物多样性是指各种形态、类型、数量和功能各异的微生物在自然环境中存在的程度和组成,包括细菌、真菌、病毒等。
微生物是地球上存在时间最长,数量最多,功能最丰富的物种。
微生物多样性是自然生态系统的重要组成部分,对于维持自然生态平衡、促进农业、医药、环保等方面都具有重要的价值。
因此,微生物多样性的研究一直是生态学和环境科学中的重要研究方向。
分子生态学是生态学的一个分支学科,主要是利用分子生物学技术解决生态学问题的一种方法。
分子生态学的关键是将生物多样性和生态系统的结构、功能及其相互作用联系起来,通过研究DNA、RNA、蛋白质和代谢物等分子水平的细节,从而更加全面地了解生态系统的复杂性。
微生物多样性的研究需要从分子生态学的角度进行,利用现代分子生物学技术,对细菌、真菌、病毒等微生物进行分离、纯化、鉴定以及对其功能进行分析。
在微生物多样性的研究中,分子生态学扮演了重要的角色。
在过去,人们从微生物的外在形态、结构、生长特性等宏观特征入手,来进行微生物多样性的研究。
但是,由于微生物的数量巨大,形态、特征、环境适应能力高度多样,因此无法用传统的分类学方法来进行鉴定和分类。
而分子生态学的出现,则提供了新的思路和技术手段。
目前,分子生态学在微生物多样性研究中的应用主要有以下几个方面。
一、16S rRNA测序16S rRNA是所有细菌和古菌都具有的基因,与其它部位不同的是,16S rRNA序列具有相对保守和相对变异的两个区域。
利用PCR方法扩增16S rRNA序列,根据序列分析可以区分菌种、菌株、类系等信息。
16S rRNA测序是微生物分类学中一种现代的化学发展出来的技术,通过在不同生态系统中分离出的微生物,提取出它们的16S rRNA序列,利用生物信息学分析手段对其进行分类、鉴定和多样性研究。
通过16S rRNA测序,可以系统地研究微生物的多样性,探究微生物在不同环境中的分布和变化规律,探明微生物群落的组成和结构,揭示不同微生物之间的生态关系。
第5讲 环境微生物分子生态

高等环境微生物学
2、茎、叶和果实上的微生物
植物的茎、叶和果实为附生微生物种群提供了良好的栖息场所, 在植物的这些部分也发现有大量的异养细菌、光合细菌、真菌(特别 是酵母)、地衣和藻类等。
高等环境微生物学
3、植物的微生物病害——植物病原体
植物的绝大多数病害都与微生物有关,也就是说很多 微生物(病毒、细菌和真菌)可引起植物疾病,不仅会产生 严重的生态问题,也会造成重大的经济损失。植物病害甚 至还会引起饥荒和人口迁移。如1845年发生在欧洲特别是 在爱尔兰的马铃薯软腐病就引起了大规模的饥荒,造成了 约1/4人口的死亡,大量的移民从爱尔兰涌入北美。
高等环境微生物学
很多研究发现,根际周围微生物的数量远远高于周围土壤 中的微生物数量,同时,根际微生物的种类受植物的种类和根 分泌物的影响, 例如,在黄瓜和玉米的根际土壤中, 荧光假单胞 菌较高, 而在大麦根际, 恶臭假单胞菌的数量较高。73%-91% 的根的分泌物可被周围微生物用做碳源和能源。苜蓿的生长促 进根际假单胞菌的生长,而假单胞菌能够合成假单胞菌素 (pseudobactin),进一步刺激根瘤菌的生长,加强微生物- 植物的共生固氮作用,假单胞菌不仅仅生活在根的周围土壤中, 还能侵入根的表皮以下组织 。
根瘤(Nodules)、根瘤菌及联合固氮作用
固氮菌可以与许多植物,特别是豆科植物形成根瘤结构的共生关系。 根瘤中的固氮菌从植物根系中获取其他所需营养,但其最重要的作 用是可以将大气中的氮气转化成氨,以供植物及其本身生长所需。根瘤 中根瘤菌的固氮作用对于维持土壤肥力是极为重要的。在农业生产上, 可以用于提高作物产量。根瘤形成过程是根瘤菌与植物根系一系列复杂 的相互作用的结果。
高等环境微生物学
(二)种群内部的相互作用
分子生态学期末重点哈师大

分子生态学期末复习重点总结一、名词解释1.分子生态学:应用分子生物学的方法来解决生态学问题的学科。
2.中性进化:一个选择中性(即选择等价)的等位基因在不断的突变压力作用下,通过随机漂变的相互取代过程。
3.操纵子:编码功能上相关的蛋白质或酶的基因与一个共同的调控顺序相串联,形成基因表达和调控的功能单位。
4.负性调节:负调节蛋白(阻遏物)将基因关闭,使其不能转录的调节方式。
5.转座子:基因组中存在的能够自发地在基因组内移动,从染色体的一个区段转移到另一区段或从一条染色体转入另一条染色体的DNA片段。
6.基因突变:是指基因的核苷酸序列发生改变,造成基因表达产物的变化,从而引起表型的改变。
7.移码突变:某些化合物与DNA结合后,可使DNA分子插入或缺失一个或几个碱基对,使该碱基对以下的读码顺序发生移动,从而改变其遗传信息。
8.PCR技术:聚合酶链式反应,又称体外DNA扩增技术。
9.蛋白质组:是指由一个基因组,或一个细胞、组织表达的所有蛋白质。
10.物种(BSC):同种生物个体间相互进行杂交并可以产生可育的后代。
11.渐渗杂交:指两物种的杂交后代与亲本反复回交,把某一亲本的性状带至另一亲本。
12.隐存种:是指一组物种,他们符合生物学对物种的定义,也就是说彼此相互隔离,但是它们在形态学是非常相似的,甚至有些时候完全一致。
13.行为生态学:主要是研究生态学中的行为机制和动物行为的生态学意义和进化意义,即研究动物的行为功能、存活值、适合度和进化过程。
14.异双亲:(很多食肉动物、啮齿动物和大约300种鸟类中)参与抚育幼小动物的非双亲成年动物叫异双亲或帮手。
15.种群(population):居住在某特定区域单个物种的一群个体。
16.基因频率(gene frequecy):指基因组中某特定位点的等位基因数量占种群中该位点全部等位基因总数的比率,也就是该等位基因在种群中出现的概率。
17.基因型频率(genotype frequency):指某基因位点特定基因型占种群中该位点全部基因型的比率,也就是该位点特定基因型在种群中出现的概率。
北京大学生命科学学院

北京大学生命科学学院生物科学专业辅修2010年招生简章经北京大学教务部批准,北京大学生物科学专业辅修2010年秋季招生。
现将申请报名、课程安排以及教学管理的相关情况通报如下:一.申请报名:1.报名资格:(1)在校本科生(专科起点本科除外),没有不及格课程且平均学分绩点在2.0以上,学有余力者;(2)对辅修专业有兴趣并有一定基础者;(3)在校各专业、未选修其他专业的辅修/双学位的08、09级本科生,且已修先修课程如下:高等数学C 类以上、物理学B类以上、普通化学B类以上。
(4)英语语种。
2.报名办法:(1) 时间:2009年5月14日(周五)向我院提交报名材料。
(2)地点:北京大学东门生命科学大楼116房间。
(3)申报材料:向本人所在院系提出申请并填写《北京大学攻读辅修专业申请表》(教务部网页的日常工作项中可以下载)一份,由所在院系主管领导批准并加盖公章。
附《北京大学攻读辅修/双学位专业学籍表》一份及本人学生证复印件一份(请注明联系方式)。
二.考试、录取事项:1.录取办法:我院5月21日(周五)下午2:00-5:00在生命科学院208房间对报名同学进行综合面试,决定录取名单。
2.录取人数:10名3. 录取查询:电话查询62751854杨老师。
三.教学管理:1、教学计划(1)专业简介北京大学生命科学学院的前身是创办于1925年的北京大学生物学系,是我国高等学校最早建立的生物学系之一。
北大生命科学学院拥有的国家重点学科、研究生、硕士点、博士点、中科院院士、长江特聘教授、博士生导师数目均居全国高校之首。
在此基础上,我们设立生物科学辅修,希望培养出具有跨学科从事生物科学研究能力的人才。
(2)专业培养要求、目标北京大学生物科学辅修,要求学生具有扎实的数、理、化基础。
经过系统的学习,使学生初步掌握生物学的基础理论,以及基本生物科学的研究方法和实验技术;具有良好的科学作风和科学素质,以及理论联系实际、实事求是、独立思考、勇于创新的科学精神;初步具有跨学科从事生物科学研究和应用的能力。
分子生态学名词解释

一、翻译并解释名词:10x4分1.allele 等位基因一个位点的序列变异.2.Effective population size Ne 有效种群大小在一个具有相等性比、随机交配的理想种群中表现出与特定统计全部成体数目规模相对应的真实的种群杂合性随时间丧失的速率相同的个体数.3.F-statistics F 统计检验用于评估个体间、亚种群间和整个种群间杂合性的分布的统计方法,被广泛应用于定量亚种群的遗传分化.4.Genetic load 遗传负荷相对于理论最佳值来说降低了的基因型适合度.5.Hardy-Weiberg equilibrium哈温平衡当所有等位基因频率是已知的时候,在一个大的随机交配种群中的纯合子和杂合子的预期比例.假设没有迁移、突变或选择作用,哈温平衡定律则认为等位基因频率从一个世代到下一个世代应该保持不变.6.Bottleneck effect瓶颈效应种群的规模大为缩小,随后常常有一个种群的恢复.7.Selection sweep选择扫荡.课件:Occurrence of a beneficial mutation,Only individuals carrying the mutation reproduce,‘Population bottleneck’,Mainly affects linked loci.8.IAM 无限等位基因模型其中突变不是以可预料的方式一个接一个发生,而大多数突变是像产生SNP单核苷酸多态性那样出现的.9.Linkage disequilibrium LD 连锁不平衡.术语表:Linkage equilibrium 连锁平衡:由重组促成的情形,其中遗传位点在繁殖期相互独立分离.当两个位点上的等位基因一起分离时,如他们在同一个染色体上的物理位置太接近时,则发生不平衡.百度:连锁平衡:不同的各在人群中以一定的出现.在某一群体中,不同座位上某两个出现在同一条染色体上的高于预期的随机频率的现象,称连锁不平衡 linkage disequilibrium .由于 HLA 不同的某些经常连锁在一起遗传,而连锁的基因并非完全随机地组成单体型,有些基因总是较多地在一起出现,致使某些单体型在群体中呈现较高的,从而引起连锁不平衡.10.Metapopulation复合种群种群再分为多个同类群,至少其中的一些偶尔灭绝,随后通过从其他同类群迁入再建立种群.11.Microsatellite微卫星带有单序列通常为2-,3-或4-核苷酸重复多次的遗传位点.12.MtDNA 线粒体DNA存在于线粒体中的环状染色体.13.Non-synomous mutation非同义突变由一个三联密码变化使特定氨基酸改变的突变.14.PCR聚合酶链式反应用寡核苷酸引物和耐热的DNA聚合酶扩增大量DNA序列的一种方法.15.SNP单核苷酸多态性在DNA序列中一个特殊位点上出现不同核苷酸碱基的等位基因.16.RFLP限制性片段长度多态性用限制性酶和凝胶电泳鉴定DNA序列多态性的方法.17.Transition转换一个嘌呤核苷酸被另一个嘌呤核苷酸替代,或一个嘧啶核苷酸被另一个嘧啶核苷酸取代的突变.18.Transversion颠换一个嘌呤核苷酸被另一个嘧啶核苷酸取代或相反过程的突变.19.Molecular ecology分子生态学课件:分子生物学是应用分子生物学的原理和方法来研究生命系统与环境系统相互作用的生态机理及其分子机制的科学.它是生态学与分子生物学相互渗透而形成的一门新兴交叉学科,其研究内容包括种群在分子水平的遗传多样性及遗传结构,生物器官变异的分子机制、生物体内有机大分子对环境因子变化的响应、生物大分子结构、功能演变与环境长期变化的关系以及其它生命层次生态现象的分子机理等.分子生态学的理论和方法对传统学科有巨大的促进作用,同时,对解决诸如转基因、克隆技术应用中的生态安全、环境与人类健康等重大问题将产生深刻的影响.20.Functional ecological and evolutionary genomicsFEEG生态和进化基因组学21.PhylogeographicPhylogeographiy 亲缘地理学研究调控系谱世系的地理分布的原理和过程的科学.22.Monophyly 单系类群中的所有个体是从同一个祖先来的,并且从这个祖先来的所有存活的后代都在这个类群中.23.Intron 内含子真核生物的结构基因间的非编码DNA序列.内含子被转录但它们的RNA拷贝在功能产生期间被切除.24.Introgression 渐渗杂交等位基因从一个种群或物种向另一个种群或者物种的扩散,而产生像种群间或物种间的近交或杂交这样的结果.25.Genetic drift 遗传漂变生物多样性导论:在有性生殖的群体中,每个世代的基因库是对上一个世代基因库的随机抽样和复制.在世代交替过程中,不同的等位基因遗传到下一代的偶然性对种群遗传结构有可能产生显着影响. Wright把这种由于配子产生及结合过程中的随机性导致的基因频率的波动称为遗传漂变genetic drift,也称随机漂变、遗传偏离或Wright效应.26.Haplotype 单倍体型源于同一染色体或染色体单倍体组的一套等位基因.27.ESUEvolutionary Significant Unit 进化显着单元分类学上对保护重要类群的一种尝试性定义.28.Metagenomics 宏基因组学百度:宏基因组学Metagenomics又叫微生物环境基因组学、元基因组学.它通过直接从环境样品中提取全部微生物的DNA,构建宏基因组文库,利用基因组学的研究策略研究环境样品所包含的全部微生物的遗传组成及其群落功能.它是在微生物基因组学的基础上发展起来的一种研究微生物多样性、开发新的生理活性物质或获得新基因的新理念和新方法.其主要含义是:对特定环境中全部为生物的总DNA也称宏基因组,metagenomic进行克隆,并通过构建宏基因组文库和筛选等手段获得新的生理活性物质;或者根据rDNA数据库设计引物,通过系统学分析获得该环境中微生物的遗传多样性和分子生态学信息.29.CpDNA 叶绿体DNA存在于叶绿体内的环状染色体.30.Heterosis 杂种优势,杂合体优势杂合子比纯合子有较高的适合度的情形.31.Recombination 重组二倍体生物中减数分裂期间同源配对染色体间的DNA交换.32.rRNA 核糖体RNA核糖体中的RNA分子…………33.SNP 单核苷酸多态性在DNA序列中一个特殊位点上出现不同核苷酸碱基的等位基因.34.Sympatric speciation 同域发生物种形成生活在同一地区的个体中形成的新物种.35.Vicariance 地理隔离种群或物种被环境事件,如山脉形成造成的物理隔离注:描黄部分为术语表中无相关解释的,仅供参考.分子生态学简答题1. 什么是分子生态学, 分子生态学的主要研究内容是什么教材的定义:分子生物学是应用分子生物学的原理和方法来研究生命系统与环境系统相互作用的生态机理及其分子机制的科学.它是生态学与分子生物学相互渗透而形成的一门新兴交叉学科.研究内容:包括种群在分子水平的遗传多样性及遗传结构,生物器官变异的分子机制、生物体内有机大分子对环境因子变化的响应、生物大分子结构、功能演变与环境长期变化的关系以及其它生命层次生态现象的分子机理等.分子生态学的理论和方法对传统学科有巨大的促进作用,同时,对解决诸如转基因、克隆技术应用中的生态安全、环境与人类健康等重大问题将产生深刻的影响.2. 什么是宏基因组学,其主要研究过程如何宏基因组学Metagenomics又叫微生物环境基因组学、元基因组学.它通过直接从环境样品中提取全部微生物的DNA,构建宏基因组文库,利用基因组学的研究策略研究环境样品所包含的全部微生物的遗传组成及其群落功能.宏基因组学的研究过程:对特定环境中全部为生物的总DNA也称宏基因组,metagenomic进行克隆,并通过构建宏基因组文库和筛选等手段获得新的生理活性物质;或者根据rDNA数据库设计引物,通过系统学分析获得该环境中微生物的遗传多样性和分子生态学信息.3. 什么是生态和进化基因组学,其主要研究内容如何研究环境条件与基因组结构、功能、动态及进化相互关系的学科.4. 什么是亲缘地理学,其研究内容是什么研究调控系谱世系的地理分布的原理和过程的科学.5. 什么是数量性状数量性状有那些类型数量性状又称为适应性性状,通常由互相影响的多个基因控制,并会明显受到环境的影响.在足够大的群体中,数量性状的分布基本符合正态规律.数量性状的类型:连续型、阈值型、间断型或离散型连续型:数量性状的表现为连续分布,群体中个体数足够多时,连续型数量性状基本符合正态分布,如:身高、体重、奶牛产奶量等.阈值型数量性状:此种数量性状的表现会受到潜在风险因素的影响,具有最低或最高阈值,例如:生物的性成熟年龄,疾病的易感率等.间断型数量性状:只能用离散的数值或数组表示其表现型的数量性状,如动物每胎产仔个数、果蝇足上的刚毛数等.6. 影响种群等位基因频率变化的进化过程有哪些种群进化的主要动力有:突变、遗传漂变、选择、基因的迁移基因流动.突变:生物核酸序列上碱基种类或数目发生改变,导致新等位基因产生或原有等位基因丧失.遗传漂变:由于进行有性生殖的生物的配子形成过程具有随机性,亲代的部分遗传信息在通过配子传递给子代时会由于这种不确定性而丢失,导致部分等位基因在整个种群的下一代中频率降低甚至消失.遗传漂变现象在小种群中表现尤其明显.选择:外部自然环境对种群等位基因的选择往往是定向的,若等位基因所控制的形状不能使生物较好地适应生存环境,自然选择就会逐步淘汰这些等位基因.基因流动:种群内个体在不同种群之间进行迁移时,会造成不同种群之间等位基因的混合.整体而言,大规模的种群之间个体迁移能够调和等位基因频率分布的不均衡现象,有利于提高种群等位基因多样性.7. 什么叫选择扫荡是如何产生的A selective sweep is the reduction or elimination of variation among the nucleotides in neighboring DNA of a mutation as the result of recent and strong positive natural selection.A selective sweep can occur when a new mutation occurs that increases the fitness of the carrier relative to other members of the population. Natural selection will favour individuals that have a higher fitness and with time the newly mutated variant allele will increase in frequency relative to other alleles. As its prevalence increases, neutral and nearly neutral genetic variation linked to the new mutation will also become more prevalent. This phenomenon is called genetic hitchhiking.A strong selective sweep results in a region of the genome where the positively selected haplotype the mutated allele and its neighbours is essentially the onlyone that exists in the population, resulting in a large reduction of the total genetic variation in that chromosome region.选择扫荡或选择性清除:种群中产生了能够提高个体适合度的有利变异后,该有利变异以及与之连锁的中性和近中性变异的等位基因频率逐渐提高,从而导致其他等位基因频率的降低乃至丧失,最终造成种群遗传多样性整体显着降低的现象.当种群中产生了可提高个体适合度的新变异即有利变异时,选择性扫荡就可能发生.携带了这种有利变异的个体在自然选择中更具优势,拥有更高繁殖成功率,因此随着时间推移,该有利变异在同位点等位基因中的频率逐渐提高,并且与其可连锁遗传的其他中性和近中性等位基因频率也会相应提高.强烈的选择性扫荡作用会导致基因组整体遗传多样性的大幅降低乃至丧失,因为它往往会最终导致种群中只剩下具有少数几种有利突变基因型的个体.8. 什么是瓶颈效应产生瓶颈效应的原因有哪些规模较大、具有高度遗传多样性的种群,由于干扰等因素导致种群个体数量急剧减少后,虽然个体数目可以在后来逐渐恢复至原来的水平,但由于大量个体丧失而同时导致的种群遗传多样性的损失却再也无法恢复.导致瓶颈效应产生的原因:破坏性自然灾害、传染病、大规模捕杀或采集、栖息地的破坏等均可造成种群数量急剧减少,从而导致瓶颈效应.9. 什么是FST,怎样用FST 衡量种群分化程度FST 是用于衡量种群分化程度的F-统计方法所用的统计量之一,定义为:T S ST H H F -=1 ,其中:HS 为亚种群期望杂合度,HT 为整个种群的期望杂合度.它反映的是一个大种群内部各个亚种群之间的分化程度.FST 取值范围为:0≤FST <1若FST=0,则有HS=HT,原种群无分化现象;若FST→1,则有HS→0,表示亚种群杂合度极低,原种群已发生高度分化.10. 举例说明常用的分子标记及其在分子生态学研究中的应用.常用分子标记有:RFLP Restriction fragment length polymorphism限制性片段长度多态性SSLP Simple sequence length polymorphism简单序列长度多态性AFLP Amplified fragment length polymorphism扩增片段长度多态性RAPD Random amplification of polymorphic DNA随机扩增的多态性DNAVNTR Variable number tandem repeat可变数目串联重复序列Microsatellite polymorphism, SSR Simple sequence repeat简单重复序列多态性微卫星SNP Single nucleotide polymorphism简单核苷酸多态性STR Short tandem repeat短串联重复序列SFP Single feature polymorphism单一特征多态性DArT Diversity Arrays Technology多样性阵列技术RAD markers Restriction site associated DNA markers与DNA相关的限制性位点分子标记分子标记的应用:研究动植物交配机制、遗传漂变、亲缘地理学、种群生态学、保护生物学、宏基因组学研究例如微生物群落组成与功能的研究等.分子标记的特点:普遍性、稳定性、高度多态性、可遗传性、可区分由于遗传和生存环境导致的相似性区分同源性与相似性;对于绝大多数种类难以进行培养的微生物,可利用分子标记,直接建立环境样品中的微生物宏基因组文库,以便研究环境中微生物群落的组成、结构与功能.此外,分子标记还是基因组学分析的重要工具.11. 什么叫适应,适应性状有哪些特点定义:使生物个体对其生境适应性提高的现象.适应性状:又称为数量性状,其表现型一般由多个基因共同控制,并且受环境影响较明显.补充:群体很大时,数量性状的表型分布规律基本为正态的.12. 什么叫突变突变有那些种类定义:生物核酸序列上核苷酸种类的改变或少数几个核苷酸的插入或缺失现象.突变种类:按突变发生机制,可以分为碱基替换Substitution、插入与删除Indels碱基替换:又称错误配对.分为转换一种嘌呤替换为另一种嘌呤,或一种嘧啶替换为另一种嘧啶和颠换嘌呤和嘧啶之间的替换替换不改变核苷酸数目,可能导致对应密码子和氨基酸的改变.按突变结果,可以分为:同义突变或沉默突变、错义突变对应氨基酸改变、无义突变对应的密码子变为终止子,使转录停止.插入与删除:即在DNA新链合成时,由于复制错误,在新链中添加或遗漏了若干个核苷酸.这将导致新链核苷酸数目改变,造成密码子读取顺序随之发生变化,又称为移码突变.按突变发生场所,亦可分为体细胞突变与生殖细胞突变.分子生态学论述题回答参考思路1. 举例说明分子生态学在解决经典生态学问题中的优势和局限是什么经典生态学的研究层次:个体、种群、群落、生态系统.个体生态学:个体的形态结构、生理功能、生态习性多样性及分布;种群生态学:种群组成与结构、种群动态出生与死亡、迁移、种群行为交配机制、种间关系等;群落生态学:群落的物种组成与结构、群落动态群落演替、群落稳定性;生态系统生态学:生态系统的组成与结构生产者、消费者、分解者、生态过程物质循环、能量流动、信息传递※分子生态学的直接研究对象是生物大分子如核酸序列、蛋白质.优势:从分子生态学的主要研究手段方面考虑,分子生态学的最大特点是应用分子生物学原理和技术手段,上述研究方向,哪些可以应用局限性:起步相对较晚,缺乏原始数据没有建立完善的数据库,因此进行基础性研究仍存在一定难度.2. 举例说明分子生态学方法在微生物学中的应用.1宏基因组学:提取环境样品中的全部微生物基因组信息,构建环境微生物群落的宏基因组文库.传统研究方法:微生物培养;仅能培养约1%的微生物种类2分子标记技术:进行微生物的分类学研究.使用微生物的16SrRNA中的可变序列,利用PCR技术、凝胶电泳、放射自显影的方法对环境品种的微生物进行遗传序列鉴定,可以有效地进行微生物的分类学研究.传统分类学通过对生物个体表性特征的鉴别进行分类,对于个体及其微小的微生物,传统方法显然是不适合的.3基因工程与微生物育种:利用生物突变原理,对微生物进行诱变育种;利用基因工程技术培育杂交菌种等.3. 从种群遗传学和保育生物学角度来说明小种群为什么易于灭绝、遗传漂变、瓶颈效应、奠基者效应、近交现象4. 人类谱系地理学证据如何表明人类起源于非洲如何解释用线粒体和Y-染色体来推断人类最近共同祖先进化时间上的差异科学家利用遵循母系遗传的线粒体DNA和遵循父系遗传的Y染色体片段作为分子标记,对采集自世界各地的人类样本进行了遗传序列比对,分析出样本之间遗传序列的差异情况,并根据分子钟原理和系统发育树的建立原理,得到了人类的大致谱系发育图.大量这方面的相关研究表明,除非洲外的其他区域采集到的样本都曾来源于共同的祖先同一个谱系,而只有在非洲采集到的样本中,发现有的样本不属于上述共同的谱系,因此科学家推断,人类可能更早起源于非洲,后通过迁移扩散分布于世界各地.进化时间差异:用线粒体DNA估计出的人类母系祖先出现的时间为距今约20万年前,早于用Y染色体估计出的人类父系祖先出现时间为距今约6~14万年<=¥▽¥=>.产生差异的主要原因:线粒体DNA和Y染色体的突变速率不同,线粒体DNA的突变速率更快.建议大家背题的时候,遇到不懂的地方自己查找一下有关的资料.论述题只需要建立清晰的条理,明白地表达自己的观点即可,因此建议:对上述四个问题,请大家选择自己容易理解的角度,尽可能完全弄懂.考试时有得写才是最重要的_:37∠_。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Sufficient amount, length and coverage
α-Proteobacteria Less than products from whole cells
Library
Constructing Vector transformation Verifying functional screening sequence-based screening substrate induced gene expression screening (SIGEX) Other screening methods
!! Expression of the metagenomics library.
Sequencing DNA extraction clone library (plasmid) shotgun sequencing Assembly poisson distribution manual curation evaluation Sort into organisms Results contigs, scaffolds, unassembled singletons diversity some cannot be readily separated.
Environmental genomics
By: Shang, Lei School of Life Sciences 2010-4-22
Outline
1
Definition
2
Main techniques & examples ★
3
Applications
4ConcBiblioteka usionDefinition
DNA extraction
Sequencing (Shotgun)
assembly
【Whole Genome Amplificatio n (WGA)】
Metagenomic library construction
Verification
WGA
- Multiple displacement amplification (MDA)
Conclusion
A promising method
Future work
metagenomic library construction and expression single-cell genomics functional genes and pathways protocol improvement new bioactive compounds
Little difference
Quantity
DOP-PCR > PEP
DNA polymerase activity Increased annealing and extension time
Develo pment
Applications
Academic microbe resources – classification & diversity mechanism and evolution structure and function of microorganism community relationship between microbe and environment Practical active materials pollution control [water and soil] Preimplantation Genetic Diagnosis & Prenatal Diagnosis
DOP-PCR
Exponential amplification
No
Detect variance of different loci
Yes
The starting template has no effect on product
Merits & Pitfalls
Coverage of genome
WGA from single cells
MDA Primer extension preamplification (PEP) Degenerate oligonucleotide primed PCR (DOP-PCR) Tagged random primers (T-PCR) Linker-adaptor PCR
This is the first article on the applicability of MDA to metagenome application from scleractinian coral.
Aims:(1) evaluate the coverage and biases (2) amplify metagenomes
1991 Pace
“ environmental genomics” first bacteriophage library
1998 EGP
(Environmental Genome Project)
1998 Handelsman
“ metagenome” the genome of total microbiota found in nature
Broad sense Narrow sense
total biota
all bacteria and fungi
Techniques
- For uncultured microbes(★)
Ⅰ total DNA extraction Environmental samples Gene discovery [PCR\ORF] Function analysis
Ⅱ single-cell genomics
Problems & Solutions
(Hutchison and Venter, 2006)
Problems & Solutions
background synthesis remove sources of contaminating DNA template. reduce the reaction volume.
(Venter et al., 2004)
ORF
- Subtractive hybridization magnetic bead capture
Merits multi gene targets high specificity
Pitfalls requires library not always fulllength ORF
bias [“plones”]
chimeric clones in sequencing libraries enzymatic treatment before cloning into vector computational methods 454 technology
breakage of large chromosomal DNA several copies of chromosome
Efficiency/ADO Short products
Most complete (not all)
With each additional cycle Shorter PEP protocol Improved PEP PCR
Most complete (1/3)
500 bp in average
Anammox
Nitrogen cycle wastewater treatment
K.stuttgartiensis
> 200 genes metal-respiration Nirs NO acetyl-CoA pathway
(Strous et al., 2006)
Thanks!
Phylum Planctomycetes
deepest branching normal phylum related to the Chlamudiae
Phylogenetic analysis Orthologous groups
gene transfer superphylum
(Peng et al., 2006)
Comparison
PEP
Princip le
Sequence-independent Primer Temperature (T) Random mixture 92℃-37 ℃-55 ℃ Yes Partially degenerate Annealing T increase
