基因组学-课件-4基因组测序与序列组装
合集下载
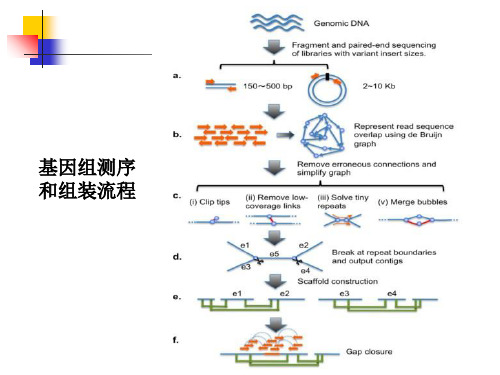
第二讲基因组测序与序列组装

解序:是指将一段染色体区段的DNA 序 列进行组装. 一些已绘制了遗传图与物理图的微生物基 因组测序中也采用这一方法. 如高等植物拟南芥基因组的测序完全依据 克隆重叠群,先进行各个BAC克隆的随机测序, 再进行序列组装; 水稻基因组测序计划采取的策略与此相同.
基因等; 编码蛋白质的基因
基因的不连续性
Intron 和Exon:
大多数真核生物蛋 白质基因的编码顺 序(Exon)都被或长 或短的非编码顺序 (Intron)隔开
基因家族
一群具有一致的或相似顺序的基因,有的还担负 类似的生物学功能, 可以相互补偿, 比如:E2f transcription factor
12 4.6
什么是C 值?
通常是指一种生物单倍体基因组DNA 的总量.
在真核生物中,C值一般随着生物的进化而 增加,高等生物C值一般大于低等生物。 C值悖理:
生物的复杂性与基因组的大小并不完全成比 例增加
阴影部分为一个门内C-值的范围
动物
真菌 等 细菌
重复顺序
高度重复顺序: 长度:几个——几千个bp 拷贝数:几百个——上百万个 首尾相连,串联排列 集中分布于染色体的特定区段(如端粒,着丝粒等) 也称卫星DNA
4.3 指导测序与序列组装
建立在基因组图谱基础上的”鸟枪法”,即所谓”指 导鸟枪法”或”指导测序”。
在人类基因组进入测序组装阶段就采用此方法,其基 本步骤如下 参考人类基因组图,特别是大量的STS位标作为基点,进 行序列组装,排成重叠克隆群.
中度重复顺序: 一般分散于整个基因组中; 长度和拷贝数差别很大
单一顺序: 基因主要位于单一顺序 动物中单一顺序约占50% 植物中单一顺序约占20%
2. 什么是基因?
细菌全基因组测序 ppt课件
基因家族(gene family) 和基因簇(gene cluster)分析
基因组中来源相同,结构和功能相关的基因 聚集在一起形成基因家族。
基因家族的各个成员紧密成簇排列成大段的串联 重复单位,分布在某一条染色体的特殊区域
genefamily.xls
基因家族聚类结果
genefamily.stat
各基因家族统计信息
培养条件① 培养条件②
或活性较低
测定转录 组mRNA
细菌全基因组测序
比较 新 差异 基因
其他方面的应用研究
❖ 应用NMR、FTIR、UV, 14C标记的木质 素降解机理方面的研究; ❖农药残留物以及其他一些难降解有机物的 降解; ❖ 重金属有机物化合物的降解。
② 木质素降解过程中涉及到的细胞外酶主要有:木质素过氧化物酶
(LiP)和锰过氧化物酶(MnP),以及漆酶(Lac)。此外,一 些附属酶参与过氧化氢的产生,乙二醛氧化酶(glyoxal oxidase, 缩写作GLOX)和芳基醇氧化酶(aryl alcohol oxidase,缩写作 AAO)属于这类酶。
对4株菌的亲缘关系进行分析,确定菌株之间的相互关 系;
通过对4株菌进行进化分析,判定是否为古菌或新的菌 种。
细菌全基因组测序
基因分离
下一步的实验安排
对已注释出的基因进行验证
载体
酶切
酶切
连接
转化
筛选 表达
细菌全基因组测序
未注释出功能的基因鉴定,挖掘新基因
DNA 转录 RNA 翻译 Protein
细菌全基因组测序
“一个物种基因组计划的完成, 就意味着这一物种学科和产业 发展的新开端”
向仲怀院士
谢谢!!
细菌全基因组测序
微生物基因组学 ppt课件

39
PPT课件
六、研究基因组功能的意义 1. 加速致病基因的研究 2. 寻找灵敏而特异性的病原分子标记 病原微生物的特异性DNA序列可以作为分子标记用于疾病的诊断。 3. 促进新药的发现和疫苗的发展 (1)促进新药的发现 (2)疫苗的研究 4. 促进微生物分类的发展
40
PPT课件
5. 提高对人类相关基因功能的认识
(1)一些人类的遗传性疾病,如结肠癌、肝豆状核变性、肾上腺脑白质 营养不良等,在细菌的基因组分析中,也存在类似的蛋白物。
(2)可以利用微生物做模拟,去检测高等生物的基因性状和功能。 (3)从基因水平去揭发人类疾病与病原微生物之间关系,如发病机理, 人类与病原微生物之间相互作用的基因机理等。
41
PPT课件
30
PPT课件
三.微生物基因组的注释 (一)概念:在微生物基因测序的基础上,对其基本 结构和部件进行认定,以进一步研究其功能。
31
PPT课件
(二)微生物基因组注释的内容 1.碱基组成分析,即G+C Mol%测定。 G+C含量是物种的一个重要特征,在微生物的分类上具有重要意义,是 重要参数之一。 2.开放阅读框的鉴定: 3.编码序列分析
消化 (4)分子杂交 (5)Southern十字杂交法
38
PPT课件
五、微生物基因组功能分析 1、根据目的基因组的性状而推测可能的基因组功能。 如致病岛的G+C mol%与细菌本身的G+C mol%有很大差异。致病岛或耐 药岛等。 2、根据已知的数据库进行同源性搜索。 美国NIH的GenBank;欧洲的分子生物学实验数据库(FMBL)日本的 DNA数据库(DDBJ) 3、利用不同条件、不同作用因素的影响而鉴定未知基因的功能。 如用过氧化氢酶处理沙门氏菌而获得该菌的对H2O2氧化应激反应的基因。 4、采用基因敲除的方法来推测或确定基因的功能。
基因组测序技术PPT课件

1 ATACGTTA
2 2GCTGTATGTAAGTCAT
4 C4GATCTGATGTAATGA
3 3TACGTTAG A
5 GTTAGATC
1 ATACGTTA
3 TACGTTAG
4 ACGTTAGA
2
CGTTAGAT
5
GTTAGATC
计算机分析杂交图象 并由探针的重叠情况 推导样品的核酸序列
互补序列为:ATACGTTAGATC 样品序列为:TATGCAATCTAG
在人类基因组进入测序组装阶段就采用此方法, 其基本步骤如下 参考人类基因组图,特别是大量的STS位标作为基点, 进行序列组装,排成重叠克隆群.
先将染色体打成比较大的片段(几十-几百Kb), 利用 分子标记将这些大片段排成重叠的克隆群(Contig), 分别 测序后拼装. 这种策略叫基于克隆群(contig-based)的策 略.
2000年 12 月,第一个植物基因组—— 拟南芥(Arabidopsis thaliana)基因组 被全部测序 ,大小为125 Mb.
一、测序流程1.构建生物基因组或cDNA DNA的提取和制备→酶切制备克隆用DNA片段 →与载体连接→转化受体细胞 →筛用超声波(或限制性内切酶)切成能够测序 的小片断(200-500bp)→小片断和载体结合,植入 细菌中进行扩增(或用PCR扩增) →从细菌中提 取出繁殖好的质粒→ 酶切,制取测序的DNA片段
A
B
C
大片段contig
小片段测序拼装
A
B
C
两种策略的比较
鸟枪法策略
指导测序策略
不需背景信息
时间短 需要大型计算机 得到的是草图(Draft)
构建克隆群 (遗传、物理图谱) 需要几年的时间
基因组学-课件-4基因组测序与序列组装

中英联合实验室
4.1 DNA测序的方法
• DNA测序技术主要有两种方法,都是在20 世纪70年代中期发明的。 • A. 双脱氧链终止法(the chain termination method),是通过合成与单 链DNA互补的多核苷酸链来读取待测DNA 分子的顺序。 • B. 化学降解法(chemical degradation method),是将双链DNA分子用化学试剂 处理,产生切口,用同位素标记进行测序。
A.通用引物:与载体DNA中 附近插入片段的顺序退火, 可引入新链的合成。 B.内部引物:提供一系列端 部以及内部可完成长序列顺 序的克隆。
中英联合实验室
B.化学降解法
• 基本原理:在选 定的核苷酸碱基中 引入化学基团,再 用哌啶处理使DNA 分子在被修饰的核 苷酸位置降解。
中英联合实验室
同时完成4组反应,A、G、C、T 链终止法:主流技术,易于机械化自动控制 化学降解法:试剂含有毒性主要原因是链终止法。
测序技术的发展
• 放射性同位素标记底物:灵敏度高 • 荧光标记物:灵敏度与分辨力,便于仪器阅 读。一种碱基一种颜色(A红色,G黄色,C 蓝色,T绿色) • 毛细管电泳:取代聚丙烯凝胶平板电泳,96 个泳道,每次可同时进行96次测序,每轮实 验不到2小时,1天可完成近千次反应
• 光点测序pyrosequencing:无须电泳,每 连接dNTP时释放1个焦磷酸,焦磷酸在磷 酸化酶的作用下转换为化学能,发出光亮 。每次只加入1种dNTP • DNA芯片测序:将各种排列顺序的寡聚核 苷酸点在芯片上,DNA分子与芯片温浴, 能杂交的寡聚核苷酸都会在确定位置发出 信号,获取信息进行对比组装。
A.双脱氧链终止法
• 天然的DNA聚合酶不能满足,需要进行改造
4.1 DNA测序的方法
• DNA测序技术主要有两种方法,都是在20 世纪70年代中期发明的。 • A. 双脱氧链终止法(the chain termination method),是通过合成与单 链DNA互补的多核苷酸链来读取待测DNA 分子的顺序。 • B. 化学降解法(chemical degradation method),是将双链DNA分子用化学试剂 处理,产生切口,用同位素标记进行测序。
A.通用引物:与载体DNA中 附近插入片段的顺序退火, 可引入新链的合成。 B.内部引物:提供一系列端 部以及内部可完成长序列顺 序的克隆。
中英联合实验室
B.化学降解法
• 基本原理:在选 定的核苷酸碱基中 引入化学基团,再 用哌啶处理使DNA 分子在被修饰的核 苷酸位置降解。
中英联合实验室
同时完成4组反应,A、G、C、T 链终止法:主流技术,易于机械化自动控制 化学降解法:试剂含有毒性主要原因是链终止法。
测序技术的发展
• 放射性同位素标记底物:灵敏度高 • 荧光标记物:灵敏度与分辨力,便于仪器阅 读。一种碱基一种颜色(A红色,G黄色,C 蓝色,T绿色) • 毛细管电泳:取代聚丙烯凝胶平板电泳,96 个泳道,每次可同时进行96次测序,每轮实 验不到2小时,1天可完成近千次反应
• 光点测序pyrosequencing:无须电泳,每 连接dNTP时释放1个焦磷酸,焦磷酸在磷 酸化酶的作用下转换为化学能,发出光亮 。每次只加入1种dNTP • DNA芯片测序:将各种排列顺序的寡聚核 苷酸点在芯片上,DNA分子与芯片温浴, 能杂交的寡聚核苷酸都会在确定位置发出 信号,获取信息进行对比组装。
A.双脱氧链终止法
• 天然的DNA聚合酶不能满足,需要进行改造
基因组测序和序列组装-文档资料

得到精细图谱
Thank you!
Sanger 双脱氧链终止 DNA测序法:
利用DNA聚合酶和 双脱氧链终止物测定 DNA核苷酸顺序的方法, 是由英国剑桥分子生物 学实验室的生物化学家 F. Sanger等人于1977年 发明的。
1980年诺贝尔奖金获得者F. Sanger
Sanger 双脱氧链终止DNA 测序法的基本原理:
•聚丙烯酰胺凝胶电泳可以区分 长度只差一个核苷酸的DNA分子。 • 利用DNA聚合酶不能够区分 dNTP和ddNTP的特性,使 ddNTP参入到寡核苷酸链的3’末端。因为ddNTP 3’不是-OH, 不能与下一个核苷酸聚合延伸, 从而终止DNA链的增长。
DNA 样 品 TATGCAATCTAG 与基因芯片上 65,000 种可能的 八聚体进行杂交从而形成特定 的结合图形 1 ATACGTTA CGTTAGAT 22 GTTAGATC
4 CGTTAGAT 4 ACGTTAGA 33 TACGTTAG ACGTTAGA 5 GTTAGATC 1 ATACGTTA 3 TACGTTAG 4 2 5 ACGTTAGA CGTTAGAT GTTAGATC 计算机分析杂交图象 并由探针的重叠情况 推导样品的核酸序列 互补序列为:ATACGTTAGATC 样品序列为:TATGCAATCTAG
Maxam-Gilbert化学降解法的原理: 用化学试剂处理末端放射性标记的DNA片段,造成碱基 的特异性切割。由此产生的一组具有各种不同长度的 DNA链的反应混合物,经凝胶电泳和放射自显影后,直 接读出待测DNA片段的核苷酸顺序。
碱基特异的化学切割反应
碱基 G A+G
特异修饰方法 Ph8.0,用硫酸二甲酯对 N7进行甲基化,使 C8-C9键对碱基裂解有特殊敏感性 pH2.0 哌啶甲酸可使嘌呤环的N原子化,从 而导致脱嘌呤,并因此消弱腺嘌呤和鸟嘌呤 的糖苷键 肼可打开嘧啶环,后者重新环化成五元环后 易除去 1.5mol/L NaCl存在时,可用肼除去胞嘧啶
基因组序列组装的理论与方法简介学习PPT教案

Shotgun法序列拼接
Low Base Quality
Single Stranded Region
Sequence Gap
Consensus
Mis-Assembly (Inverted)
正反向测序对: 从同一个克隆片段两端分别测序所得到的一对序列。. 插入片段长度: 克隆载体中插入的外源DNA片段长度。 片段连接群(contig):用识别互相重叠的方法对测序数据进行拼接的结果。. Scaffold: 用正反向测序对连接的非重叠片段连接群。 LW-洞:由于没有测序数据覆盖而在组装结果中留下的洞。
genome sequence of size G, with 4 repeats shotgun library construction
O(G2) pair-wise comparison
R = 3 segments w/ repeat-termination
“overlap-layout-consensus” algorithm explores R! = exponential number of possible solutions
重复序列分析
覆盖度: 基因组被测序数据覆盖的次数。 重复数: 一段DNA序列在基因组中出现的次数。 深度:一段DNA序列在鸟枪法测序数据集中出现次数。例如一个转座子在基因组中 出现N次,测序数据集的覆盖度为C, 则这个转座子的平均深度为NC。 20-mer 重复序列:任何深度超过为该数据集确定的重复序列标准的20-bpDNA片段。 是数学定义的重复序列。 重复序列洞: 由于屏蔽重复序列而在组装结果中留下的洞。
exact 20-mer repeats fraction masked, by size fully-masked reads
一讲基因组测序与序列组装

感谢您的观看
THANKS
03
基因组序列组装
序列组装的基本流程
序列读取
通过测序技术获取基 因组序列的原始数据。
序列质量评估
对原始数据进行质量 评估,去除低质量序 列和错误序列。
序列比对
将高质量序列比对到 参考基因组或组装到 独立的基因组上。
序列拼接
将比对或独立基因组 上的序列片段拼接成 完整的基因组。
组装后验证
对组装得到的基因组 进行验证,确保其完 整性、准确性和一致 性。
下一代测序技术
总结词
更高通量、更低成本、更短周期的测序技术。
详细描述
下一代测序技术是一种尚未完全成熟的测序技术,目 前正处于研究和发展阶段。相比于前几代测序技术, 下一代测序技术将具有更高的通量、更低的成本和更 短的周期等特点。它可能采用更加先进的纳米技术、 光学技术和生物信息学技术等手段,以提高测序的准 确性和速度。下一代测序技术的出现将为基因组学和 生物医学领域的研究提供更加高效装得到的基因组的完整性,包括染色 体水平的完整性和基因水平的完整性。
准确性评估
评估组装得到的基因组的准确性,包括单核苷酸水 平上的准确性和结构变异上的准确性。
一致性评估
评估组装得到的基因组的一致性,包括不同 组装方法或不同数据集之间的一致性和内部 的一致性。
04
基因组测序与序列组装的挑 战与前景
例如,通过研究水稻基因组,科学家们发现了与抗旱、耐盐等抗逆性状相关的基因,为培育抗逆性更强的水稻品种提供了重 要的理论依据。
病原微生物基因组研究
病原微生物基因组研究是利用基因组测序和序列组装技术来了解病原微生物的基因组结构和功能,旨 在发现新的药物靶点、疫苗候选基因和诊断标记物等。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
中英联合实验室
4.2 DNA顺序的组装
• 噬菌体P1载体:与λ载体相似,由于P1载体的基 因组要大于λ噬菌体基因组,并且其颗粒也比噬 菌体大,故可以P1载体克隆更长的DNA片段。 • BAC:是基于大肠杆菌的野生型F质粒设计的。BAC 可用于克隆300kb左右的片段。 • PAC:结合P1载体和BAC所设计的,其容量也可以 达到300kb。 • 在搭建克romosome walking)
鸟枪法的运用
• 2002年中国科学家在绘制籼稻基因组工作框架图的过程中, 采用的是全基因组鸟枪测序法。植物基因组有大量的重复 序列,重复序列的正确识别和组装,因此需要开发特殊的 计算软件。 • 首先在整个水稻基因组上生成许多已知长度的DNA切片, 然后使它们按DNA序列的重合区域进行排列。这些切片数 量足以覆盖水稻基因组4次。接着,确定每个切片的碱基 对序列,并用计算机程序将其组装成更长的片段,然后将 这些片段排序、装配成10万多个被称为支架的更大组件。 • 设计出的软件重点是通过支架水平上的接近来进行组装, 并采取了独特的重复序列处理算法,可识别并暂时屏蔽占 水稻基因组约40%的重复序列。这样做的好处是既能减少 计算量,又最大限度降低了错误拼接的可能性。
中英联合实验室
4.2 DNA顺序的组装
染色体步移的原理:在开始时,验室
4.2 DNA顺序的组装
的DNA序列查找与之连接的克 隆建立重叠群,直到覆盖整个DNA片段,甚至染色 体 • 拟南芥基因组的测序完全依据克隆重叠群,先进 行各个BAC克隆的随机测序,再进行序列组装
中英联合实验室
4.2 DNA序列的组装
• 将大量短的DNA序列组装
– 直接鸟枪法 – 克隆重叠群法
– 引导鸟枪法
中英联合实验室
• “全基因组鸟枪法测序”也称“霰弹法”,是将基因组DNA 打成小片段进行测序,随机测序
直接鸟枪法的原理和特点
• 直接鸟枪法序列组装:从已测序的小片段中寻找彼此
重叠的测序克隆,依次向两侧邻接的序列延伸,组装成一 个完整的基因组。 不需预先了解任何基因组的情况,即 使缺少遗传图或物理图也可完成整个基因组顺序组装。 • 鸟枪法的最大优点是经济、快速、高效,但对高性能计算 的方法和设备要求非常高。 DNA
中英联合实验室
A. 双脱氧链终止法
• 原理:
–DNA复制: 模板DNA、DNA聚合酶、引物、4种dNTP –ddNTP的加入:在反应系统中加入双脱氧(碱基)核苷三 磷酸(ddNTP),只要双脱氧核苷酸掺入3’ 端,该链就 停止延伸,链端掺入单脱氧核苷酸的片段可继续延伸。 如此得到3’端终止于ddNTP的一系列长度不等的核酸片段 –4个反应体系中分别加入4种不同的ddNTP,浓度低于 dNTP。反应终止后,分四个泳道进行电泳(聚丙烯酰胺 凝胶),分离长短不一的核酸片段(长度相邻者仅差一个 碱基),根据片段3’端的双脱氧碱基,可依次阅读合成片 段的碱基排列顺序。
中英联合实验室
PCR制备单链DNA
A.双脱氧链终止法
• 通过M13载体获得单链 DNA以测序: 是利用M13噬菌体在 体外能以单链形式存 在。将待测序DNA片段 连接到M13载体中去, 再利用引物,进行DNA 合成,并在合成系统 中加入ddNTP
中英联合实验室
A.双脱氧链终止法
• 双脱氧链终止法中引物的影响:引物决定 了模板链的测序起点,一般来说,是采用 克隆位点附近载体上的一段顺序。
中英联合实验室
A.双脱氧链终止法
双 脱 氧 链 终 止 法 测 序 的 原 理
中英联合实验室
• 双脱氧链终止法对DNA多聚酶的要求:
– 高酶活性 – 无5 ’-3 ’外切核酸酶活性,5 ’-3 ’外切核酸酶使 DNA聚合酶去除已存在的链 – 无3 ’-5 ’ 外切核酸酶活性, 3 ’-5 ’外切核酸酶 使DNA聚合酶校正自身错误
A.通用引物:与载体DNA中 附近插入片段的顺序退火, 可引入新链的合成。 B.内部引物:提供一系列端 部以及内部可完成长序列顺 序的克隆。
中英联合实验室
B.化学降解法
• 基本原理:在选 定的核苷酸碱基中 引入化学基团,再 用哌啶处理使DNA 分子在被修饰的核 苷酸位置降解。
中英联合实验室
同时完成4组反应,A、G、C、T 链终止法:主流技术,易于机械化自动控制 化学降解法:试剂含有毒性主要原因是链终止法。
中英联合实验室
• 2000年6月26日,美国总统克林顿(中)、塞莱拉公司董事 长兼首席科学家克雷格· 文特尔(左)和人类基因组计划首 席科学家弗胡西斯· 柯林斯(右)在华盛顿白宫参加庆祝人 类基因组草图绘制成功。
实例: TIGR测定流感嗜血杆菌基因组
• 主要步骤: • 1.建立高克隆片段的碱基 总数应达到基因组5倍以菌基因组的测定完成,使用了14台 测序仪,用三个月时间完成了必需的28,463个测序反应, 测序总长度达6倍基因组(1830kb,11631kb)。 • 3.序列集合。发展了新的软件,修改了序列集合规则以最 大限度地排除错误的连锁匹配。 • 4.填补缺口。有两种待填补的缺口,一是没有相应模板 DNA的物理缺口,二是有模板DNA但未测序的序列 DNA
组装的大分子 DNA
完整的基因组 DNA
中英联合实验室
鸟枪法的运用
• “鸟枪法”是美国塞莱拉遗传公司创始人克 雷格· 文特尔发明的,是目前常用的两种DNA 测序法中较为快捷的一种。传统的DNA测序 法要将DNA片段放大,并在克隆的细菌中对 其测序,过程繁琐复杂。 • 2000年塞莱拉公司和国际合作项目“人类 基因组计划”分别通过这两种方法绘制出 人类基因组草图。
中英联合实验室
鸟枪法的缺陷
• 对比利用“鸟枪法”绘制的人类基因组草图和 “人类基因组计划”公布的最新草图进行了,发 现“鸟枪法”无法测到人类基因组中重复出现的 DNA片段,这些片段占到基因组的3%至5%,对于理 解遗传性疾病具有重要意义。 • 但在进行快速DNA测序时,“鸟枪法”仍然不失为 一种可取的手段。最佳的DNA测序法是,将两种测 序方法相结合:用“鸟枪法”进行整体测序,识 别出“鸟枪法”无法测序的区域,再通过传统方 法对这些区域测序。
中英联合实验室
实例:果蝇基因组的鸟枪法测序
• 第一个完全采取鸟枪法完成基因组测序的真30kb
–10kb长度足以覆盖果蝇基因组中大多数重复顺序,可 在序列组装时找到单一顺序连接真实的重叠克隆 –130kb便于大范围序列组装,有效建立全基因组的物理 图框架
A.双脱氧链终止法
• 天然的DNA聚合酶不能满足,需要进行改造
– Klenow聚合酶:来自大肠杆菌DNA聚合酶Ⅰ,外 切酶活性去除,酶活性不够,250bp – 测序酶Sequenase:噬菌体T7编码
中英联合实验室
A.双脱氧链终止法
• 双脱氧链终止法要求单链作为模板:
– 将DNA克隆到质粒载体中:碱变性或热变性变为单链 ,双向测序;污染,干扰 – 以M13载体克隆单链DNA:无需变性,直接测序; >3kb,扩增时容易发生丢失与重排,小片段DNA测序 – 以噬菌粒(phagemid)克隆DNA:改造的质粒载体 ,2个复制起始点(质粒自身和 M13单链噬菌体), 在大肠杆菌细胞中产生单链噬菌粒, >10kb DNA测序 – PCR产生单链DNA
中英联合实验室
列通 拼过 接覆 流盖 感不 嗜同 血序 杆列 菌重 的叠 全群 基之 因间 组间 序隙 列的 序
中英联合实验室
实例: TIGR测定流感嗜血杆菌基因组
• 鸟枪法测序的缺点 随着所测基因组总量增大,所需测序的片段大量 增加。高等真核生物(如人类)基因组中有大量 重复序列,导致判断失误。 • 对鸟枪法的改进 (1) Clone contig法。首先用稀有内切酶把待测 基因组降解为数百kb以上的片段,再分别测序。 (2) 靶标鸟枪法(directed shotgun)。首先根据 染色体上已知基因和标记的位臵来确定部分DNA片 段的相对位臵,再逐步缩小各片段之间的缺口。
测序技术的发展
• 放射性同位素标记底物:灵敏度高 • 荧光标记物:灵敏度与分辨力,便于仪器阅 读。一种碱基一种颜色(A红色,G黄色,C 蓝色,T绿色) • 毛细管电泳:取代聚丙烯凝胶平板电泳,96 个泳道,每次可同时进行96次测序,每轮实 验不到2小时,1天可完成近千次反应
• 光点测序pyrosequencing:无须电泳,每 连接dNTP时释放1个焦磷酸,焦磷酸在磷 酸化酶的作用下转换为化学能,发出光亮 。每次只加入1种dNTP • DNA芯片测序:将各种排列顺序的寡聚核 苷酸点在芯片上,DNA分子与芯片温浴, 能杂交的寡聚核苷酸都会在确定位置发出 信号,获取信息进行对比组装。
基因组测序与序列组装
中英联合实验室
基因组测序与序列组装
• 基因组计划的最终目标是获得所研究的生物的完 整的DNA顺序。最佳状况是将物理图谱和遗传图谱 进行有机整合,以确定基因以及其他重要的序列 在DNA顺序中的位置。 • 主要内容: • 1.DNA测序的方法 • 2.DNA序列的组装 • 3.基因组测序的其他路线 • 4.人类基因组的测序和组装
最新进展:经济快捷的DNA染色测序法
2005年4月,美国columbia大学的科学家给4种碱基染 上不同的颜色,然后从人类p53基因中抽取一个由12个碱 基组成的片段,运用分子生物学技术使染色的碱基排列成 与该DNA片段互补的一个新片段。 • 传统DNA测序的方法有毛细管微阵列电泳测序和焦磷酸测 序等,这些方法技术要求较高、成本昂贵,而且容易出错。 相比而言,新方法通过逐个观察DNA片段上碱基的颜色来 确定其序列,精确度极高。 • 对这项研究提供资助的美国国家人类基因组研究所希望, 在10年内将哺乳动物的基因测序成本从1000万美元降低 到1000美元,这样就有可能以廉价的成本对患者进行“量 身定做”的基因治疗。
中英联合实验室
4.2 DNA序列的Biblioteka 装• 克隆重叠群的序列组装
4.2 DNA顺序的组装
• 噬菌体P1载体:与λ载体相似,由于P1载体的基 因组要大于λ噬菌体基因组,并且其颗粒也比噬 菌体大,故可以P1载体克隆更长的DNA片段。 • BAC:是基于大肠杆菌的野生型F质粒设计的。BAC 可用于克隆300kb左右的片段。 • PAC:结合P1载体和BAC所设计的,其容量也可以 达到300kb。 • 在搭建克romosome walking)
鸟枪法的运用
• 2002年中国科学家在绘制籼稻基因组工作框架图的过程中, 采用的是全基因组鸟枪测序法。植物基因组有大量的重复 序列,重复序列的正确识别和组装,因此需要开发特殊的 计算软件。 • 首先在整个水稻基因组上生成许多已知长度的DNA切片, 然后使它们按DNA序列的重合区域进行排列。这些切片数 量足以覆盖水稻基因组4次。接着,确定每个切片的碱基 对序列,并用计算机程序将其组装成更长的片段,然后将 这些片段排序、装配成10万多个被称为支架的更大组件。 • 设计出的软件重点是通过支架水平上的接近来进行组装, 并采取了独特的重复序列处理算法,可识别并暂时屏蔽占 水稻基因组约40%的重复序列。这样做的好处是既能减少 计算量,又最大限度降低了错误拼接的可能性。
中英联合实验室
4.2 DNA顺序的组装
染色体步移的原理:在开始时,验室
4.2 DNA顺序的组装
的DNA序列查找与之连接的克 隆建立重叠群,直到覆盖整个DNA片段,甚至染色 体 • 拟南芥基因组的测序完全依据克隆重叠群,先进 行各个BAC克隆的随机测序,再进行序列组装
中英联合实验室
4.2 DNA序列的组装
• 将大量短的DNA序列组装
– 直接鸟枪法 – 克隆重叠群法
– 引导鸟枪法
中英联合实验室
• “全基因组鸟枪法测序”也称“霰弹法”,是将基因组DNA 打成小片段进行测序,随机测序
直接鸟枪法的原理和特点
• 直接鸟枪法序列组装:从已测序的小片段中寻找彼此
重叠的测序克隆,依次向两侧邻接的序列延伸,组装成一 个完整的基因组。 不需预先了解任何基因组的情况,即 使缺少遗传图或物理图也可完成整个基因组顺序组装。 • 鸟枪法的最大优点是经济、快速、高效,但对高性能计算 的方法和设备要求非常高。 DNA
中英联合实验室
A. 双脱氧链终止法
• 原理:
–DNA复制: 模板DNA、DNA聚合酶、引物、4种dNTP –ddNTP的加入:在反应系统中加入双脱氧(碱基)核苷三 磷酸(ddNTP),只要双脱氧核苷酸掺入3’ 端,该链就 停止延伸,链端掺入单脱氧核苷酸的片段可继续延伸。 如此得到3’端终止于ddNTP的一系列长度不等的核酸片段 –4个反应体系中分别加入4种不同的ddNTP,浓度低于 dNTP。反应终止后,分四个泳道进行电泳(聚丙烯酰胺 凝胶),分离长短不一的核酸片段(长度相邻者仅差一个 碱基),根据片段3’端的双脱氧碱基,可依次阅读合成片 段的碱基排列顺序。
中英联合实验室
PCR制备单链DNA
A.双脱氧链终止法
• 通过M13载体获得单链 DNA以测序: 是利用M13噬菌体在 体外能以单链形式存 在。将待测序DNA片段 连接到M13载体中去, 再利用引物,进行DNA 合成,并在合成系统 中加入ddNTP
中英联合实验室
A.双脱氧链终止法
• 双脱氧链终止法中引物的影响:引物决定 了模板链的测序起点,一般来说,是采用 克隆位点附近载体上的一段顺序。
中英联合实验室
A.双脱氧链终止法
双 脱 氧 链 终 止 法 测 序 的 原 理
中英联合实验室
• 双脱氧链终止法对DNA多聚酶的要求:
– 高酶活性 – 无5 ’-3 ’外切核酸酶活性,5 ’-3 ’外切核酸酶使 DNA聚合酶去除已存在的链 – 无3 ’-5 ’ 外切核酸酶活性, 3 ’-5 ’外切核酸酶 使DNA聚合酶校正自身错误
A.通用引物:与载体DNA中 附近插入片段的顺序退火, 可引入新链的合成。 B.内部引物:提供一系列端 部以及内部可完成长序列顺 序的克隆。
中英联合实验室
B.化学降解法
• 基本原理:在选 定的核苷酸碱基中 引入化学基团,再 用哌啶处理使DNA 分子在被修饰的核 苷酸位置降解。
中英联合实验室
同时完成4组反应,A、G、C、T 链终止法:主流技术,易于机械化自动控制 化学降解法:试剂含有毒性主要原因是链终止法。
中英联合实验室
• 2000年6月26日,美国总统克林顿(中)、塞莱拉公司董事 长兼首席科学家克雷格· 文特尔(左)和人类基因组计划首 席科学家弗胡西斯· 柯林斯(右)在华盛顿白宫参加庆祝人 类基因组草图绘制成功。
实例: TIGR测定流感嗜血杆菌基因组
• 主要步骤: • 1.建立高克隆片段的碱基 总数应达到基因组5倍以菌基因组的测定完成,使用了14台 测序仪,用三个月时间完成了必需的28,463个测序反应, 测序总长度达6倍基因组(1830kb,11631kb)。 • 3.序列集合。发展了新的软件,修改了序列集合规则以最 大限度地排除错误的连锁匹配。 • 4.填补缺口。有两种待填补的缺口,一是没有相应模板 DNA的物理缺口,二是有模板DNA但未测序的序列 DNA
组装的大分子 DNA
完整的基因组 DNA
中英联合实验室
鸟枪法的运用
• “鸟枪法”是美国塞莱拉遗传公司创始人克 雷格· 文特尔发明的,是目前常用的两种DNA 测序法中较为快捷的一种。传统的DNA测序 法要将DNA片段放大,并在克隆的细菌中对 其测序,过程繁琐复杂。 • 2000年塞莱拉公司和国际合作项目“人类 基因组计划”分别通过这两种方法绘制出 人类基因组草图。
中英联合实验室
鸟枪法的缺陷
• 对比利用“鸟枪法”绘制的人类基因组草图和 “人类基因组计划”公布的最新草图进行了,发 现“鸟枪法”无法测到人类基因组中重复出现的 DNA片段,这些片段占到基因组的3%至5%,对于理 解遗传性疾病具有重要意义。 • 但在进行快速DNA测序时,“鸟枪法”仍然不失为 一种可取的手段。最佳的DNA测序法是,将两种测 序方法相结合:用“鸟枪法”进行整体测序,识 别出“鸟枪法”无法测序的区域,再通过传统方 法对这些区域测序。
中英联合实验室
实例:果蝇基因组的鸟枪法测序
• 第一个完全采取鸟枪法完成基因组测序的真30kb
–10kb长度足以覆盖果蝇基因组中大多数重复顺序,可 在序列组装时找到单一顺序连接真实的重叠克隆 –130kb便于大范围序列组装,有效建立全基因组的物理 图框架
A.双脱氧链终止法
• 天然的DNA聚合酶不能满足,需要进行改造
– Klenow聚合酶:来自大肠杆菌DNA聚合酶Ⅰ,外 切酶活性去除,酶活性不够,250bp – 测序酶Sequenase:噬菌体T7编码
中英联合实验室
A.双脱氧链终止法
• 双脱氧链终止法要求单链作为模板:
– 将DNA克隆到质粒载体中:碱变性或热变性变为单链 ,双向测序;污染,干扰 – 以M13载体克隆单链DNA:无需变性,直接测序; >3kb,扩增时容易发生丢失与重排,小片段DNA测序 – 以噬菌粒(phagemid)克隆DNA:改造的质粒载体 ,2个复制起始点(质粒自身和 M13单链噬菌体), 在大肠杆菌细胞中产生单链噬菌粒, >10kb DNA测序 – PCR产生单链DNA
中英联合实验室
列通 拼过 接覆 流盖 感不 嗜同 血序 杆列 菌重 的叠 全群 基之 因间 组间 序隙 列的 序
中英联合实验室
实例: TIGR测定流感嗜血杆菌基因组
• 鸟枪法测序的缺点 随着所测基因组总量增大,所需测序的片段大量 增加。高等真核生物(如人类)基因组中有大量 重复序列,导致判断失误。 • 对鸟枪法的改进 (1) Clone contig法。首先用稀有内切酶把待测 基因组降解为数百kb以上的片段,再分别测序。 (2) 靶标鸟枪法(directed shotgun)。首先根据 染色体上已知基因和标记的位臵来确定部分DNA片 段的相对位臵,再逐步缩小各片段之间的缺口。
测序技术的发展
• 放射性同位素标记底物:灵敏度高 • 荧光标记物:灵敏度与分辨力,便于仪器阅 读。一种碱基一种颜色(A红色,G黄色,C 蓝色,T绿色) • 毛细管电泳:取代聚丙烯凝胶平板电泳,96 个泳道,每次可同时进行96次测序,每轮实 验不到2小时,1天可完成近千次反应
• 光点测序pyrosequencing:无须电泳,每 连接dNTP时释放1个焦磷酸,焦磷酸在磷 酸化酶的作用下转换为化学能,发出光亮 。每次只加入1种dNTP • DNA芯片测序:将各种排列顺序的寡聚核 苷酸点在芯片上,DNA分子与芯片温浴, 能杂交的寡聚核苷酸都会在确定位置发出 信号,获取信息进行对比组装。
基因组测序与序列组装
中英联合实验室
基因组测序与序列组装
• 基因组计划的最终目标是获得所研究的生物的完 整的DNA顺序。最佳状况是将物理图谱和遗传图谱 进行有机整合,以确定基因以及其他重要的序列 在DNA顺序中的位置。 • 主要内容: • 1.DNA测序的方法 • 2.DNA序列的组装 • 3.基因组测序的其他路线 • 4.人类基因组的测序和组装
最新进展:经济快捷的DNA染色测序法
2005年4月,美国columbia大学的科学家给4种碱基染 上不同的颜色,然后从人类p53基因中抽取一个由12个碱 基组成的片段,运用分子生物学技术使染色的碱基排列成 与该DNA片段互补的一个新片段。 • 传统DNA测序的方法有毛细管微阵列电泳测序和焦磷酸测 序等,这些方法技术要求较高、成本昂贵,而且容易出错。 相比而言,新方法通过逐个观察DNA片段上碱基的颜色来 确定其序列,精确度极高。 • 对这项研究提供资助的美国国家人类基因组研究所希望, 在10年内将哺乳动物的基因测序成本从1000万美元降低 到1000美元,这样就有可能以廉价的成本对患者进行“量 身定做”的基因治疗。
中英联合实验室
4.2 DNA序列的Biblioteka 装• 克隆重叠群的序列组装
