基因组和转录组高通量测序数据分析流程和分析平台
RNA-seq(转录组学)的分析流程和原理

RNA-seq(转录组学)的分析流程和原理在开始详细讲解RNA测序之前,我们先来了解一下它的基本步骤:1.建库:提取RNA,富集mRNA或消除rRNA,合成cDNA和构建测序文库。
2.测序:然后在高通量平台(通常是Illumina)上进行测序(每个样本测序reads在DNA测序中,读数是对应于单个DNA片段的全部或部分的碱基对(或碱基对概率)的推断序列。
深度为10-30 Million reads。
)3.分析:先比对/拼装测序片段到转录本,通过计数、定量,样本间过滤和标准化,以进行样本组间基因/转录本统计差异分析。
大致了解这个过程之后,我们就先从建库开始了解建库的难点在于提纯出mRNA, 一般在我们抽离出的RNA中rRNA占比很大,其他还会有tRNA、microRNA等。
我们需要从抽离出的RNA中提取出mRNA,并建立cDNA文库。
这里以应用最广泛的Illumina公司的Truseq RNA的建库方法为例来进行介绍。
首先,利用高等生物的mRNA通常有poly(A)尾的(使mRNA更稳定,翻译不容易出错)特点,用带有poly(T)探针的磁珠与总RNA进行杂交,这样磁珠就和带poly(A)尾巴的mRNA结合在一起了。
接下来,就回收磁珠,把这些带poly(A)的mRNA从磁珠上洗脱下来。
再用镁离子溶液(或者超声波)进行处理,把mRNA打成小段。
然后,利用这些被打断的mRNA片段,以随机引物进行逆转录,得到第一链cDNA。
再根据第一链cDNA合成出ds-cDNA。
对cDNA在平末端进行3’端加A碱基(腺苷酸)(adapter接头上带了T碱基头,为了和adapter配对)在双链cDNA的两端加分别上Y型接头再经PCR扩增经筛选的目的基因,就得到可以上机的测序文库了。
这个建库方法对RNA的完整度有较高的要求。
也就是说,只有在mRNA大部分是完整的状态下,才能得到比较好的效果。
因为带Poly(T)的磁珠,它所吸附的是带有Poly(A)的那些序列。
生物信息学中的转录组测序数据分析流程解析

生物信息学中的转录组测序数据分析流程解析转录组测序是一种基于高通量测序技术的生物学研究方法,用于研究特定物种在特定生理或环境条件下所产生的所有转录本(mRNA)。
转录组测序数据分析是将原始的测序数据转化为有意义的生物学信息的过程。
本文将解析转录组测序数据分析的基本流程。
1. 数据质量控制(Quality Control,QC)数据质量控制是在转录组测序数据分析中非常重要的一步,它能够及早发现并剔除测序过程中产生的低质量测序数据,保证后续分析的准确性。
常用的QC工具包括FastQC和Trimmomatic。
FastQC用于检查测序数据的质量分布情况,发现可能存在的测序错误和污染问题。
Trimmomatic则用于去除低质量的测序片段和接头,提高数据的质量。
2. 数据比对数据比对是将测序数据与参考基因组进行比对的过程。
比对的目的是将测序片段精确地定位到基因组上,并获得每个基因组区域的覆盖度和深度等信息。
常用的比对工具包括Bowtie2和TopHat。
Bowtie2是一种基于Burrows-Wheeler Transform的短序列比对工具,适用于低错配率的比对。
TopHat则是一种用于对转录组数据进行比对和注释的工具,可以检测新基因和外显子剪接事件。
3. 定量分析定量分析是研究不同转录本在不同条件下的表达水平差异的过程。
常用的定量工具包括Cufflinks和HTSeq。
Cufflinks是一种用于估计转录本表达水平和发现新的转录本的工具。
它可以根据RNA-Seq数据拼接转录本,并计算不同基因或转录本的表达水平。
HTSeq则是一种用于计算不同基因的读数的工具,读数可以用来估计基因的表达水平。
4. 差异分析差异分析是研究在不同处理条件下,基因或转录本的表达水平是否存在显著差异的过程。
常用的差异分析工具包括DESeq2和edgeR。
DESeq2是一种基于负二项分布模型的差异表达分析工具,它可以对转录本进行差异分析,并计算基因的表达水平在不同条件下的折叠变化。
基因组和转录组高通量测序数据分析流程和分析平台

基因组和转录组高通量测序数据分析流程和分析平台基因组和转录组高通量测序数据分析是生物信息学领域中的一个重要研究方向。
随着高通量测序技术的发展,获取大规模基因组和转录组数据已经成为可能。
通过对这些数据的分析,可以深入了解生物体内基因的表达和功能等相关信息。
本文将介绍基因组和转录组高通量测序数据分析的基本流程和常用的分析平台。
数据预处理是分析流程的第一步,主要包括测序数据的质控和去除低质量序列。
常用的质控工具包括FastQC和Trim Galore等,它们可以评估测序数据的质量和检测可能的污染。
在质控的基础上,可以使用Trimmomatic等工具去除低质量序列和适配体序列,保证后续分析的准确性和可靠性。
基因定量是分析流程的第二步,用于评估基因的表达水平。
常用的基因定量工具包括kallisto、Salmon和STAR等。
这些工具可以根据测序数据和已知的转录组序列,计算基因的表达水平。
基因定量的结果一般以表达矩阵的形式输出,该矩阵包含了每个样本中每个基因的表达值。
差异表达基因分析是分析流程的第三步,用于寻找基因表达水平在不同样本中存在显著差异的基因。
常用的差异表达基因分析工具包括DESeq2、edgeR和limma等。
这些工具可以对表达矩阵进行统计学分析,找出在不同样本之间具有显著差异的基因。
差异表达基因分析的结果一般以差异表达基因列表的形式输出。
富集分析是分析流程的第四步,用于寻找差异表达基因中富集的生物学功能或通路。
常用的富集分析工具包括GOseq、KEGG和enrichR等。
这些工具可以根据差异表达基因列表,基于GO注释和KEGG通路等数据库,计算差异表达基因在特定功能或通路上的富集度。
生物学注释是分析流程的最后一步,用于解释基因的功能和相关信息。
常用的生物学注释工具包括DAVID、GSEA和STRING等。
这些工具可以根据差异表达基因列表,提供关于基因功能、互作关系和代谢通路等信息。
除了上述基本流程外,还有一些附加的分析步骤,如差异剪接分析、外显子计数等。
生物信息学分析工具的使用教程

生物信息学分析工具的使用教程导言:在生物学领域中,随着高通量测序技术的快速发展,生物信息学分析工具的应用变得越来越重要。
这些工具能够帮助研究人员进行基因组、转录组、蛋白质组等大规模数据的分析和解释。
本文将为您介绍几种常用的生物信息学工具,并提供详细的使用指南。
一、BLAST(基因序列比对工具)BLAST(Basic Local Alignment Search Tool)是最常用的生物信息学工具之一,用于比对基因或蛋白质序列中的相似性。
以下是使用BLAST的步骤:1. 打开NCBI网站的BLAST页面,并选择适当的BLAST程序(如BLASTn、BLASTp等)。
2. 将查询序列粘贴到"Enter Query Sequence"框中,或者上传一个FASTA格式的文件。
3. 选择适当的数据库,如"nr"(非冗余序列数据库)或"refseq_rna"(已注释的RNA序列数据库)。
4. 设置相似性阈值、期望值和其他参数。
5. 点击"BLAST"按钮开始比对。
6. 结果页面会显示比对结果的列表和详细信息,包括匹配上的序列、相似性得分等。
二、DESeq2(差异表达基因分析工具)DESeq2是一种用于差异表达基因分析的R包。
以下是使用DESeq2的步骤:1. 安装R语言和DESeq2包。
2. 将基因表达矩阵导入R环境中,并进行预处理(如去除低表达基因)。
3. 根据实验设计设置条件和组别。
4. 进行差异分析,计算基因的表达差异和显著性。
5. 可视化差异表达基因的结果,如绘制散点图、MA图、热图等。
三、GSEA(基因集富集分析工具)GSEA(Gene Set Enrichment Analysis)是一种基于基因集的富集分析方法,用于识别与特定性状或实验条件相关的生物学功能。
以下是使用GSEA的步骤:1. 准备基因表达矩阵和相关的分组信息。
转录组学分析流程及常用软件介绍

转录组学分析流程及常用软件介绍转录组学是研究在特定条件下生物体内转录的所有RNA分子的总体,包括信使RNA(mRNA)、转运RNA(tRNA)、核糖体RNA(rRNA)和小核RNA(snRNA)等。
转录组学研究可以通过分析转录组中的基因表达水平和调控机制,揭示基因功能和调控网络,从而深入了解生物体的生命活动和适应能力。
转录组学分析流程包括实验设计、RNA提取、RNA测序、数据分析和结果解释等环节,并依赖于一系列的软件工具来完成。
下面将介绍转录组学分析的流程以及常用的软件。
1.实验设计:确定研究目的和假设,设计实验方案,包括样本的选择和处理方式等。
2.RNA提取:从样本中提取总RNA,并进行纯化和富集,去除DNA和其他杂质。
3. RNA测序:将提取得到的RNA反转录成cDNA,然后通过高通量测序技术进行测序。
常用的测序技术包括Illumina HiSeq、Ion Torrent Proton等。
4.数据分析:对测序得到的数据进行质控、比对和定量等处理。
这一步通常需要使用一系列的转录组学分析软件。
5.结果解释:根据数据分析的结果,进行差异表达基因的筛选、基因富集分析和信号通路分析,以探索转录组的生物学意义。
常用的转录组学分析软件包括:1. 基因表达微阵列分析:在早期的转录组学研究中,基因表达微阵列是常用的分析方法。
常用的分析软件有Affymetrix Expression Console、Partek Genomics Suite等。
2. RNA测序数据分析:随着高通量测序技术的发展,RNA测序已成为转录组学研究的主要方法。
RNA测序数据的分析可以分为质控、比对和定量等环节。
常用的软件工具有Trimmomatic、FastQC、STAR、HISAT等。
3. 差异表达基因分析:差异表达基因是通过比较不同样本之间的基因表达水平而筛选出来的。
常用的软件包括DESeq2、edgeR、limma等。
4. 基因富集分析:基因富集分析可以帮助我们了解不同基因集之间的功能和通路差异,从而揭示转录组的生物学意义。
转录组测序数据分析流程

转录组测序数据分析流程1.样品准备:根据研究需求,选择适当的样品,如病人和对照组组织、不同发育阶段的样品等。
提取总RNA,并通过凝胶电泳、紫外线分析、比色法等方法鉴定RNA的完整性和浓度。
2. 测序:使用高通量测序技术,如Illumina HiSeq、Ion Torrent等对RNA样品进行测序。
根据实验的需要,可以采用不同的测序策略,如单端测序或双端测序,以及测序长度的选择。
3. 质控:对测序数据进行质量控制,包括去除低质量Reads、修剪接头序列、去除低复杂度序列、过滤低质量的碱基等,以确保后续分析的准确性和可靠性。
4. 数据预处理:根据测序平台的要求,对测序数据进行数据切分、过滤低质量read、去除低质量碱基等。
同时,进行去除rRNA、tRNA等非编码RNA的对应序列,以提高分析效果。
5. 比对:将得到的测序reads与参考基因组进行比对。
常用的比对工具有Bowtie、Tophat、STAR等,通过比对可以找到reads在参考基因组中的位置,为后续的表达量计算提供支持。
6. 表达量计算:根据比对结果,统计每个基因的reads数或覆盖度来计算其表达量。
可以使用RSEM、HTSeq、Cufflinks等工具进行表达量的计算,得到基因表达量矩阵。
7. 差异表达基因分析:根据不同条件下的样品表达量矩阵,使用统计学方法分析基因的差异表达情况。
常用的差异分析工具有DESeq2、edgeR、Limma等,通过计算差异表达基因的显著性水平,筛选出差异表达的基因。
8. 功能注释:对差异表达基因进行生物学功能注释,包括基因本体论(Gene Ontology, GO)、KEGG通路分析等。
可以通过数据库如DAVID、GSEA、KEGG等进行功能注释,以进一步了解差异表达基因在生物学过程中的功能。
9. 富集分析:对差异表达基因进行富集分析,即确定差异表达基因是否富集在特定的功能类别中。
可以使用Fisher精确检验、超几何检验等方法,从而发现与特定疾病或生物过程相关的富集基因集。
高通量测序技术的数据分析方法教程

高通量测序技术的数据分析方法教程随着生物技术的发展,高通量测序技术(high-throughput sequencing technology)已成为生物学、医学和生物信息学研究中的重要工具。
高通量测序技术可以快速而准确地测定DNA或RNA序列,透过大量的数据来揭示生物体的基因组、转录组以及其他生物学过程中的变化。
然而,正确且高效地分析测序数据是高通量测序技术应用的关键一步。
本文将介绍高通量测序技术的数据分析方法教程。
首先,分析高通量测序数据前,我们需要了解常见的测序平台和数据格式。
当前常用的高通量测序平台包括Illumina、ABI SOLiD、Ion Torrent等,而测序数据通常以FASTQ、SAM/BAM和VCF等格式存储。
FASTQ格式用于存储原始测序数据,其中包含了每个测序读段的序列信息及其对应的质量分数。
而SAM/BAM格式则是将测序读段比对到参考基因组之后的结果,其中SAM是比对结果的文本格式,而BAM则是对应的二进制格式。
VCF(Variant Call Format)格式则用于存储基因型变异信息。
接下来,我们将介绍高通量测序数据的基本分析流程。
通常,测序数据分析可以分为质控、比对、变异检测和功能注释几个主要步骤。
在质控步骤中,我们需要对测序数据进行质量评估和过滤。
质量评估可以通过查看测序数据的质量分数、GC含量、碱基分布和测序错误率等指标来判断测序数据的质量。
使用质量评估工具如FastQC和NGS QC Toolkit可以帮助我们快速准确地评估测序数据的质量,并进行相应的过滤工作,去除低质量的测序读段。
接下来,我们需要将测序读段比对到参考基因组上。
比对工作可以通过软件如Bowtie、BWA和HISAT等进行。
比对结果通常以SAM格式存储,然后可以进行排序、去重和索引等处理,生成最终的BAM格式文件。
在变异检测步骤中,我们需要从比对后的BAM文件中检测样本中存在的变异信息。
变异检测可以通过多种工具来实现,如GATK、Samtools和VarScan等。
转录组测序数据分析流程
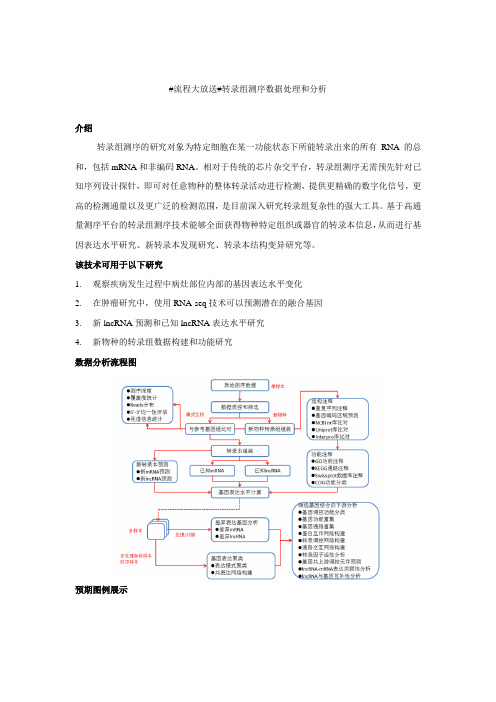
#流程大放送#转录组测序数据处理和分析
介绍
转录组测序的研究对象为特定细胞在某一功能状态下所能转录出来的所有RNA的总和,包括mRNA和非编码RNA。
相对于传统的芯片杂交平台,转录组测序无需预先针对已知序列设计探针,即可对任意物种的整体转录活动进行检测,提供更精确的数字化信号,更高的检测通量以及更广泛的检测范围,是目前深入研究转录组复杂性的强大工具。
基于高通量测序平台的转录组测序技术能够全面获得物种特定组织或器官的转录本信息,从而进行基因表达水平研究、新转录本发现研究、转录本结构变异研究等。
该技术可用于以下研究
1.观察疾病发生过程中病灶部位内部的基因表达水平变化
2.在肿瘤研究中,使用RNA-seq技术可以预测潜在的融合基因
3.新lncRNA预测和已知lncRNA表达水平研究
4.新物种的转录组数据构建和功能研究
数据分析流程图
预期图例展示
示例图1 差异表达基因筛选示例2 基因聚类分析heatmap图
示例3 差异基因互作网络图示例4 lncRNA、基因与上游共有miRNA网络图。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
基因功能(Gene function)
差异表达分析和富集分析(DEG & enrichment) 同源基因(Homology gene)
系统发育树和进化( Phylogeny tree & evolution )
比较基因组学( Comparative genomics )
Sequencing quality control
178_49 2.21E-12 76 58 95_88 2.48E-51 2.17E-14 4.97E-41
|CDS|O orf19. RF| 6115
|ORF|int orf19. ron| 57 |long_ter gamm + minal a-1a
13717 105673 G_T 13728 105121 A_A
m mm fu as ta re ne Score a c r 0 0 0 0 0 0 2 0 0 0 0 -10 0 0 0 0 0 0 2 0 0 0 0 3 0 0 0 0 0 3 0 0 3 0 1 0 0 0 0 0 3 0 0 1 0 0 0 0 0 0 0 3 0 0 3 0 0 0 0 0 0 0 2 0 0 2 0 1 0 0 0 1 0 3 0 1 1 0 0 0 0 0 0 0 2 0 0 3 0 -10 -5.9 -5.6 -4.5 -4.5 -3.7 -3.4
Comparative genomics --- LCB
Genome A: Genome B:
0
1
2
3 1
4 6
5
6
7
8 5
9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 2 7 9 -10 -12 11 14 16 15 17 20 22 -24 21 23 25 8
高通量测序数据(NGS) 数据分析平台
NGS Data Analysis Strategy
Wet lab Database Homology Feature Variation
3. System biology
1. Functional biology
Bio-function Annotation Assembly
Gene prediction result assessment
DEG & enrichment
Phylogeny tree & evolution time
Comparative genomics --- Dot Plot
translocation
inverted repeats
0 0 0 1 0 1 3 0 0 3 0
0 0 0 0 1 0 2 0 0 3 0
-3.1
-2.7
SNP/mutation identification
Alignment based SNP identification and Fisher’s Exact Test:
ID 13 14 17 18 126 Positio Allea n target 954 963 1129 1144 4061 A_A T_C T_G T_C C_G Quality target 31 29_8 29_2 27_31 32_31 30_30 31 Freq target 8 90_1 92_1 171_1 111_2 Allea Quality Freq referenc referenc referenc P-value e e e G_G C_C T_G T_C G_G 31 31 30_27 27_30 32 29 31_32 141 147 78_54 2.01E-13 4.87E-66 4.12E-14 Chromos Annotat Str Gene ome ion and Chr1 Chr1 Chr1 Chr1 Chr1 Chr1 Chr2 |ORF| |ORF| |ORF| |ORF| orf19. + 6115 orf19. + 6115 orf19. + 6115 orf19. + 6115
0 -18 -19
3 -4 13
Locally Collinear Blocks (LCBs)
Orthologous gene visualization
Conserved region in an ortholog family
ortholog link betweogy in functional view
126_113 G_G 255 A_G
SNP visualization
Genome overview
Features Data Assembly Size(Mb) 37.5 Scaffold N50 (kb) 178 Coverage(fold) 78 G+C content (%) 46.01 GC Exonic (%) 51.73 GC Intronic (%) 47.05 Repeat rate(%) 1.68 Protein-coding genes 9405 Gene density (per Mbp) 250.8 Exons per genes 2.53 tRNAs 72 rRNAs 19 SM(Secondary 28 Metabolism)genes TE 15%
DB_Desc ve us sa
Database Gene Ontology (GO) Funcat KEGG KOG/COG IPRSCAN Protein family PKS/NRPS Others
SUBFAMILY NOT NAMED Flavodoxin, conserved site Salmonella virulence plasmid 65kDa B protein SpvB Chromo domain subgroup Myelin P0 protein Insecticide toxin TcdB middle/N-terminal Integrin alpha betapropellor PUTATIVE UNCHARACTERIZED PROTEIN Rhs repeat-associated core
NGS Genomics data NC AN AF MO MAA MAC AO … Meta
Transcriptomics Proteomics Metabonomics
genomics
2. Comparative biology
基因组/转录组 分析流程
质控(QC) 组装(Assembly) 基因预测(Gene prediction) 基因组特征(Genome feature)
