数字基因表达谱
花生干旱胁迫响应基因的数字表达谱分析

作物学报 ACTA AGRONOMICA SINICA 2013, 39(6): 1045−1053/zwxb/ ISSN 0496-3490; CODEN TSHPA9E-mail: xbzw@本研究由国家现代农业产业技术体系建设专项(CARS-14-07B), 国家自然科学基金项目(31101177)和山东省自然科学基金项目(ZR2011CQ027)资助。
*通讯作者(Corresponding author): 万勇善, E-mail: yswan@, Tel: +86 (0)538 8241540第一作者联系方式: E-mail: saqsshh@ **同等贡献(Contributed equally to this work)Received(收稿日期): 2012-07-06; Accepted(接受日期): 2013-01-15; Published online(网络出版日期): 2013-02-19. URL: /kcms/detail/11.1809.S.20130219.1020.007.htmlDOI: 10.3724/SP.J.1006.2013.01045花生干旱胁迫响应基因的数字表达谱分析孙爱清1,** 张杰道2,** 万勇善1,* 刘风珍1 张 昆1 孙 利11山东农业大学农学院 / 作物生物学国家重点实验室 / 山东省作物生物学重点实验室, 山东泰安 271018; 2山东农业大学生命科学学院, 山东泰安 271018摘 要: 以抗旱性强的花生品种丰花5号为材料, 利用Solexa 高通量测序技术对15% PEG 处理后的花生叶片cDNA 文库进行差异基因表达谱分析。
结果表明, 转录组基因表达表现出高度的不均一性和冗余性, 低于10个拷贝的标签占总标签种类的73.1%, 但其表达量只占总标签表达量的9.0%。
根据已知序列信息鉴定出935个差异表达基因, 其中64.5%下调表达。
整合数字基因表达谱与全基因组关联分析鉴定猪血液性状候选基因

整合数字基因表达谱与全基因组关联分析鉴定猪血液性状候选基因徐盼;张震;章峰;杨斌;段艳宇【期刊名称】《中国农业科学》【年(卷),期】2016(000)002【摘要】【目的】整合数字基因表达谱与全基因组关联分析鉴定白色杜洛克×二花脸 F2资源群体的血液性状候选基因。
【方法】白色杜洛克×二花脸F2资源群体在(240±3)d屠宰,收集血液于抗凝管中进行血常规检测。
利用Illumina 60K SNP芯片对1020头F2资源群体进行基因分型。
剔除基因型检出率<90%和孟德尔错误检出率>5%的个体。
检出率<95%、次等位基因频率<5%、哈代-温伯格检验(HWE)P <5×10-6、与性染色体连锁疑似常染色体的SNP被筛除。
利用Illumina GA II 测序仪测序对502头F2资源群体的肝脏进行数字基因表达谱测序。
测序得到的原始数据经过滤获得清洁标签后与参考标签数据库比对,将能唯一比对到参考基因序列的清洁标签数量进行标准化处理以获得标准化的基因表达量。
每个转录本的表达水平进一步转化为 lg2值。
在少于20%的个体中表达的转录本被滤去。
表型性状和基因表达性状使用R程序包中GenABEL内polygentic功能进行性别、批次和亲缘关系的校正。
其残差使用R程序包中斯皮尔曼系数评估基因表达水平与表型数据的关联性,设定保守阈值P<5×10-4时调整多重检验。
将检测到的表达数量性状位点(eQTL)及其对应基因根据其位置相对照的关系进行绘图。
搜寻前期GWAS最高点5.0 Mb区域内eQTL结合GWAS结果进行综合分析。
Gene Ontology & KEGG pathway富集分析使用在线工具DAVID。
基因共表达网络使用在线工具GeneMANIA进行构建。
【结果】白色杜洛克×二花脸F2资源群体中502个个体的20108个肝脏转录本通过了质检。
基因表达谱分析在药物研究中的应用

基因表达谱分析在药物研究中的应用在众多的药物研究中,基因表达谱分析已经逐渐成为一种能够有效提高药物研究效率的工具。
作为一种新颖的基因组学技术,它可以快速地分析人体内基因的表达情况,并识别与特定疾病相关的基因。
这种技术已经在许多的药物研究中成功应用,这里将会具体介绍基因表达谱分析在药物研究中的应用。
基因表达谱分析简介基因表达谱分析是一种可以追踪特定基因在特定条件下的转录活动量的方法。
该方法结合基因组学、生物信息学、计算机科学和生物学于一体,可以为研究人员提供一系列有关基因表达的数据,包括基因转录过程中产生的mRNA量。
此外,基因表达谱分析还可以通过测量RNA分子在细胞内的存在量,从而识别细胞类型、状态以及其所在环境。
总体来说,基因表达谱分析可以为药物研究提供大量的基础信息。
基因表达谱分析的应用1.寻找新的药物靶点通过基因表达谱分析,可以了解到特定疾病患者基因的表达情况。
这让科学家们有了更深层次的认识和了解相关病理生理特征。
比如,目前就有很多疾病是由于基因表达失调导致的,比如乳腺癌、大肠癌、肝炎等。
通过基因表达谱分析,药物研究人员可以识别新的药物靶点及开发新的药物治疗方法。
2.评估药物疗效药物的疗效是影响药物研究的重要因素之一。
通过基因表达谱分析,科学家们可以获得药物与靶点蛋白相互作用所涉及到的相关基因信息,这样就能对药物的疗效做出更加准确的评估。
比如,科学家们发现使用某种特定 Compound A治疗非小细胞肺癌患者可以降低基因P13K/AKT/mTOR的表达量,这些表达量的下降是由Compound A对癌症细胞中PI3K/Akt/mTOR的电荷阻断所引起的,可以更好的评估Compound A的疗效。
3.抗药性研究许多患者在使用药物治疗之后会形成抗药性,这是药物研究人员在后续工作中需关注和解决的一个问题。
通过基因表达谱分析,可以发现在基因层面上抗药性基因的表达量增多。
这些基因是抗药性形成的重要因素,对其进行研究并找出相应药物突破可以使药物研究有所突破。
转录组RNAseq术语解释

转录组RNAseq术语解释RNA-Seq名词解释1.inde某2.碱基质量值(QualityScore或Q-core)是碱基识别(BaeCalling)出错的概率的整数映射。
碱基质量值越高表明碱基识别越可靠,碱基测错的可能性越小。
3.Q30碱基质量值为Q30代表碱基的精确度在99.9%。
4.FPKM (FragmentPerKilobaeoftrancriptperMillionfragmentmapped)每1百万个map上的read中map到外显子的每1K个碱基上的fragment个数。
计算公式为公式中,cDNAFragment表示比对到某一转录本上的片段数目,即双端Read数目;MappedRead(Million)表示MappedRead总数,以10为单位;TrancriptLength(kb):转录本长度,以kb个碱基为单位。
5.FC(FoldChange)即差异表达倍数。
6.FDR(FaleDicoveryRate)即错误发现率,定义为在多重假设检验过程中,错误拒绝(拒绝真的原(零)假设)的个数占所有被拒绝的原假设个数的比例的期望值。
通过控制FDR来决定P值的阈值。
7.P值(P-value)即概率,反映某一事件发生的可能性大小。
统计学根据显著性检验方法所得到的P值,一般以P<0.05为显著,P<0.01为非常显著,其含义是样本间的差异由抽样误差所致的概率小于0.05或0.01。
8.可变剪接(Alternativeplicing)有些基因的一个mRNA前体通过不同的剪接方式(选择不同的剪接位点)产生不同的mRNA剪接异构体,这一过程称为可变剪接(或选择性剪接,alternativeplicing)。
可变剪接是调节基因表达和产生蛋白质组多样性的重要机制,是导致真核生物基因和蛋白质数量较大差异的重要原因。
在生物体内,主要存在7种可变剪接类型:A)E某onkipping;B)Intronretention;C)Alternative5'pliceite;D)Alternative3'pliceite;E)Alternativefirte某on;F)Alternativelate某on;G)Mutuallye某cluivee某on。
表达谱数据

表达谱数据表达谱数据是一种描述基因在特定条件下表达水平的数据,可以反映基因在生物体内的活跃程度和重要作用。
表达谱数据通常是通过高通量测序技术获得的,例如RNA-Seq或microarray技术。
这些数据对研究基因功能及其对生物过程的贡献非常重要,因为它们可以揭示基因的功能、代谢途径和信号通路等复杂的生物过程。
表达谱是生物数据科学领域的主流,它可以用于构建生物系统的基因调控网络图,从而去了解生物物种内部的调控机制,从而识别出治疗基因表达异常疾病的潜在靶点。
表达谱数据的数据类型和学科领域是多如牛毛的,涵盖了癌症、神经学、生殖生物学、免疫学和感染病理学等众多领域。
表达谱数据可以用于许多不同的目的,例如预测细胞的类型、研究细胞的生物学特性、发现新的医疗目标等。
表达谱数据的分析包括预处理、特征提取和建模等步骤。
预处理步骤包括数据过滤、归一化、批次效应调整和缺失数据填充等,这是优化数据质量的必要步骤。
特征提取步骤包括细胞类型、生物过程、调节机制和代谢途径等关键生物学特征的提取。
建模是指将表达谱数据与其他类型的数据整合,例如蛋白质互作数据或基因组测序数据。
这就是基因表达,调控网络和系统生物学等领域的研究方向。
表达谱数据的应用范围非常广泛,从基础科学到应用科学都有着重要作用。
例如,在基础研究方面,表达谱数据可以用于研究基因调控以及基因的功能,从而进一步了解遗传学机制和生物进化的过程。
在应用科学中,表达谱数据可以用于分析药物的作用和安全性,发现影响药物代谢和毒性的基因,并为药物研发提供重要的参考信息。
另外,表达谱数据也可以用于生产领域,例如利用表达谱数据优化工业培养基和工业生物发酵生产过程,提高生产效率。
在生物安全领域,表达谱数据可以用于分析致病菌基因的表达水平,从而开发针对它们的抗菌药物。
在农业领域,基于表达谱数据,可以研究农作物的病害抵抗性、调节所属基因功能等方向进行研究。
总的来说,表达谱数据是重要的生物信息学研究领域,有着广泛的应用前景和潜在的医疗价值。
RNA-Seq(数字基因表达谱)
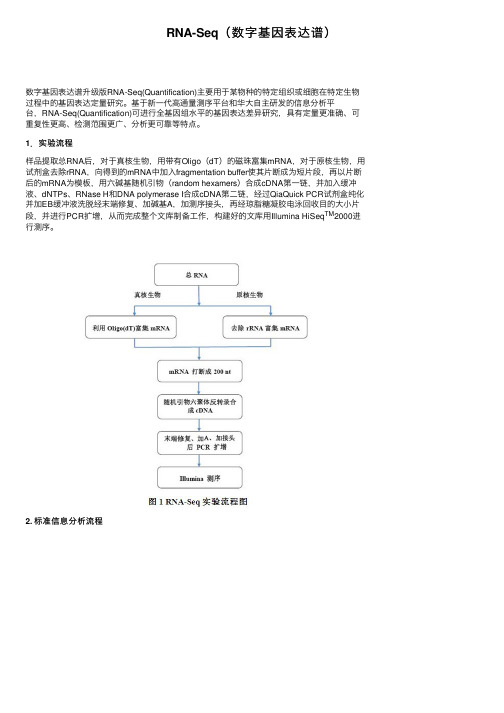
RNA-Seq(数字基因表达谱)数字基因表达谱升级版RNA-Seq(Quantification)主要⽤于某物种的特定组织或细胞在特定⽣物过程中的基因表达定量研究。
基于新⼀代⾼通量测序平台和华⼤⾃主研发的信息分析平台,RNA-Seq(Quantification)可进⾏全基因组⽔平的基因表达差异研究,具有定量更准确、可重复性更⾼、检测范围更⼴、分析更可靠等特点。
1.实验流程样品提取总RNA后,对于真核⽣物,⽤带有Oligo(dT)的磁珠富集mRNA,对于原核⽣物,⽤试剂盒去除rRNA,向得到的mRNA中加⼊fragmentation buffer使其⽚断成为短⽚段,再以⽚断后的mRNA为模板,⽤六碱基随机引物(random hexamers)合成cDNA第⼀链,并加⼊缓冲液、dNTPs、RNase H和DNA polymerase I合成cDNA第⼆链,经过QiaQuick PCR试剂盒纯化并加EB缓冲液洗脱经末端修复、加碱基A,加测序接头,再经琼脂糖凝胶电泳回收⽬的⼤⼩⽚段,并进⾏PCR扩增,从⽽完成整个⽂库制备⼯作,构建好的⽂库⽤Illumina HiSeq TM2000进⾏测序。
2. 标准信息分析流程3.技术优势⾼重复性通过对UHRR和HBRR标准品进⾏重复性研究(如图3)证明,两个样本的技术重复相关系数均可达0.99以上,可见,RNA-Seq(Quantification)具有极好的技术重复性。
检测阈值宽从图4中基因表达量检测范围看,RNA-Seq(Quantification)不仅检测范围⽐Affymetrix芯⽚宽,⽽且⽐Affymetrix更易检测到低丰度的基因。
定量准确⽤qRT-PCR和RNA-Seq(Quantification)两种⽅法进⾏UHRR和HBRR基因表达差异研究,结果相关性如图5所⽰: RNA-Seq (Quantification)与qPCR研究结果相关系数为0.915,表明RNA-Seq (Quantification)技术定量准确性⾼。
使用生物大数据中心数据库进行基因表达谱分析的步骤

使用生物大数据中心数据库进行基因表达谱分析的步骤生物大数据中心数据库是一个强大的工具,可以用于分析基因表达谱。
在进行基因表达谱分析之前,我们需要明确几个步骤。
本文将详细介绍如何使用生物大数据中心数据库进行基因表达谱分析。
第一步是向生物大数据中心数据库注册账号并登录。
注册账号是使用生物大数据中心数据库进行基因表达谱分析的第一步。
可以访问该数据库的官方网站进行注册。
填写个人信息、用户名和密码后,您将获得一个账号。
登录之后,您可以访问数据库的各个功能和工具。
第二步是选择合适的基因表达数据集。
生物大数据中心数据库拥有众多的基因表达数据集,您可以根据自己的研究需求选择合适的数据集。
数据集通常被分类为不同的物种、组织类型和疾病状态。
例如,如果您的研究关注人类心脏组织的基因表达谱,您可以选择包含心脏组织样本的数据集。
第三步是导入和预处理基因表达数据。
一旦选择了适当的数据集,您可以根据需要下载数据集中的原始数据。
原始数据通常以文本文件或Excel文件的形式提供。
在导入数据之前,您可能需要进行一些预处理步骤,例如去除噪声、归一化或筛选不感兴趣的基因。
这些预处理步骤可以使用生物大数据中心数据库中的工具完成。
第四步是进行基因表达谱分析。
生物大数据中心数据库提供了各种分析工具,可以帮助您更好地理解基因表达谱。
其中包括差异表达基因分析、基因共表达网络分析、功能富集分析等。
差异表达基因分析可以帮助您识别在不同样本之间表达水平显著不同的基因。
基因共表达网络分析可以帮助您发现在相似组织或条件下共同表达的基因模块。
功能富集分析可以帮助您理解哪些生物学过程和信号通路参与了基因的调控。
这些工具可以根据您的研究需求进行灵活的组合和调整。
第五步是解释和呈现分析结果。
一旦完成了基因表达谱分析,您将得到大量的结果,包括差异表达基因列表、共表达基因模块和功能富集结果。
解释和呈现这些结果对于得到有意义的结论至关重要。
生物大数据中心数据库通常提供了数据可视化和分析结果导出的功能。
基因组学研究中的表达谱数据分析方法解析

基因组学研究中的表达谱数据分析方法解析概述:基因组学研究是研究生物体基因组的编码和非编码序列的科学。
在基因组学研究中,表达谱数据是一种重要的数据类型,由于其高维度和复杂性,需要采用一系列的分析方法和技术来解析。
本文将介绍基因组表达谱数据的分析方法,包括数据预处理、差异表达分析、聚类分析、富集分析以及网络分析。
一、数据预处理:数据预处理是基因组表达谱数据分析的第一步,目的是清除原始数据中的噪声、去除非生物学的变异以及纠正技术上的偏见。
常用的数据预处理步骤包括数据质量控制、归一化和基因过滤。
1. 数据质量控制:首先需要对原始数据进行质量控制,该步骤可通过查看测序质量分数和测序错误率来评估。
常用的工具有FastQC和Trimmomatic等。
该步骤的目的是排除测序引入的噪声。
2. 归一化:由于不同样本之间的表达量存在显著的差异,我们需要对数据进行归一化处理,以消除样本间的偏差。
常用的归一化方法有TPM、FPKM和RPKM等。
归一化后的数据便于后续的比较和统计分析。
3. 基因过滤:在分析表达谱数据时,一些基因的表达量非常低,对分析结果产生较小的影响并增加运算复杂性。
因此,我们通常会对表达量低于一定阈值的基因进行过滤处理,从而提高分析效率。
常用的过滤标准包括表达量百分位数和表达量阈值。
二、差异表达分析:差异表达分析是基因表达谱数据分析的核心内容之一,旨在发现不同条件下存在差异表达的基因。
通常,差异表达分析包括基于假设检验的方法和机器学习方法。
1. 基于假设检验的方法:这类方法通常基于统计学原理,将样本分组,通过计算差异表达的显著性水平来判断基因是否差异表达。
常用的方法包括Student's t-test、Wilcoxon秩和检验和Fisher's确切检验等。
这些方法基于不同的假设,在数据有明确的分布前提下,可以得到比较可靠的差异表达结果。
2. 机器学习方法:机器学习方法对差异表达分析具有较高的灵活性和预测能力。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
图 1 WGCNA 展示的所有基因表达关系并以此得到共表达基因模块
图 2 基因模块间可视化网络及相关功能关系
参考文献
Xue Z, Huang K, Cai C, et al. Genetic programs in human and mouse early embryos revealed by single-cell RNA sequencing [J]. Nature, 2013, 500(7464): 593-597.
测序策略 Illumina HiSeq 测序平台;SE50/PE125
整体质量评估 基因表达水平分析
差异基因筛选 差异基因 GO 富集分析 KEGG pathway 富集分析
转录因子注释 癌症基因注释 差异基因蛋白互作网络分析
……
数据量 SE50-6 M clean reads; PE125-3 Gb clean data
1
项目周期 SE50-30 天;PE125-35 天
案例解析
案例一ห้องสมุดไป่ตู้人和小鼠早期胚胎发育的关键基因筛选
哺乳动物植入前胚胎发育的程序化过程,是发育生物学的核心问题,研究这一过程对于研究哺乳动物早期发育具有重 要意义。本研究利用 RNA-seq 技术对人胚胎早期发育各阶段转录组进行了系统分析,并结合共表达网络以期发现驱动胚 胎早期发育各阶段的关键候选基因。
数字基因表达谱
数字基因表达谱(DGE) 是基于 HiSeq 平台,研究特定组织在特定状态下的基因表达情况,全面、经济、 快速地检测不同材料间的差异表达基因,挖掘调控某一性状的关键靶点基因。
考基因组比对
技术参数
样本要求 样品类型:total RNA 样品总量:≧ 1.5 μg 样品浓度:≧ 50 ng/ul
