生物信息学课程设计实验报告—典型的生物信息学分析[小编推荐]
生物仿真分析实验报告(3篇)

第1篇一、实验名称生物仿真分析实验二、实验目的1. 了解生物仿真的基本概念和原理。
2. 掌握使用仿真软件进行生物系统建模和模拟的方法。
3. 分析仿真结果,验证生物系统的行为和机制。
三、实验原理生物仿真是指利用计算机技术对生物系统进行建模和模拟的过程。
通过构建数学模型,模拟生物体的生理、生化过程,分析其行为和机制。
本实验采用仿真软件对某一生物系统进行建模和模拟,通过调整模型参数,观察系统行为的变化。
四、实验设备1. 仿真软件:如MATLAB、Simulink等。
2. 生物数据:实验所需的相关生物数据。
3. 计算机:运行仿真软件的计算机。
五、实验步骤1. 数据准备:收集实验所需的生物数据,包括生理参数、生化参数等。
2. 模型构建:利用仿真软件,根据实验数据构建生物系统的数学模型。
3. 模型验证:通过调整模型参数,验证模型在特定条件下的准确性和可靠性。
4. 模拟实验:在验证模型的基础上,进行模拟实验,观察系统行为的变化。
5. 结果分析:分析仿真结果,验证生物系统的行为和机制。
六、实验结果1. 模型构建:根据实验数据,成功构建了某一生物系统的数学模型。
2. 模型验证:通过调整模型参数,验证了模型在特定条件下的准确性和可靠性。
3. 模拟实验:在模型验证的基础上,进行了模拟实验,观察到了系统行为的变化。
4. 结果分析:通过分析仿真结果,验证了生物系统的行为和机制。
七、讨论和分析1. 模型构建:在构建生物系统模型时,充分考虑了实验数据的准确性和可靠性。
通过调整模型参数,验证了模型的准确性和可靠性。
2. 模拟实验:通过模拟实验,观察到了系统行为的变化,进一步验证了生物系统的行为和机制。
3. 结果分析:仿真结果与实验数据基本一致,验证了生物系统的行为和机制。
八、注意事项1. 数据收集:在收集实验数据时,应注意数据的准确性和可靠性。
2. 模型构建:在构建生物系统模型时,应充分考虑生物系统的复杂性和动态性。
3. 模拟实验:在模拟实验过程中,应注意调整模型参数,以观察系统行为的变化。
生物信息学分析

生物信息学分析随着科技的不断进步,生物信息学已成为现代生物学研究的重要工具。
生物信息学分析不仅帮助我们更好地理解生命现象,还在疾病诊断、药物研发等领域发挥着重要作用。
本文将介绍生物信息学分析的基本概念、方法和应用。
一、生物信息学分析的基本概念生物信息学分析是指利用计算机技术、数学和统计学方法对生物数据进行分析、处理和解释的过程。
生物数据包括基因组序列、蛋白质序列、基因表达谱、蛋白质蛋白质相互作用等。
通过对这些数据进行生物信息学分析,我们可以揭示生物分子之间的相互关系,了解生命现象的内在规律。
二、生物信息学分析的方法1. 序列比对:序列比对是生物信息学分析中最基本的方法,用于比较不同生物分子之间的相似性。
常用的序列比对工具有BLAST、Clustal Omega等。
2. 蛋白质结构预测:蛋白质结构预测是根据蛋白质序列预测其三维结构的过程。
常用的蛋白质结构预测工具有AlphaFold、Rosetta等。
3. 基因表达谱分析:基因表达谱分析用于研究基因在不同生物过程、不同环境条件下的表达水平变化。
常用的基因表达谱分析工具有DESeq2、EdgeR等。
4. 蛋白质蛋白质相互作用网络分析:蛋白质蛋白质相互作用网络分析用于研究蛋白质之间的相互作用关系,揭示生命活动的分子机制。
常用的蛋白质蛋白质相互作用网络分析工具有Cytoscape、Gephi等。
三、生物信息学分析的应用2. 药物研发:生物信息学分析可以帮助我们筛选潜在的药物靶点,预测药物分子的生物活性,加速药物研发过程。
例如,通过蛋白质结构预测,可以筛选出具有特定功能的蛋白质作为药物靶点。
3. 个性化医疗:生物信息学分析可以帮助我们了解个体的基因组、蛋白质组等信息,为个性化医疗提供依据。
例如,通过对个体基因组的分析,可以预测个体对特定药物的反应,为临床用药提供指导。
生物信息学分析在生命科学研究中发挥着越来越重要的作用。
随着生物数据量的不断增加和计算技术的不断进步,生物信息学分析将为我们揭示生命现象的奥秘提供更多有力工具。
生信分析实例[G]
![生信分析实例[G]](https://img.taocdn.com/s3/m/e01d75c45fbfc77da269b1f2.png)
Blast result of amino acid sequence for Major ORF of RGDV Seg8
核苷酸序列=>氨基酸序列
• ExPASy 上的Translate tool /tools/dna.html
• 生物学软件BioEdit a) 查看密码子用法(Codon Usage Database ) b) 整理制作密码子使用频率表 c) 翻译成氨基酸
或Oligonucleotide Properties Calculator /biotools/oligocalc.html
序列同源分析 (By BLAST 2 SEQUENCES)
序列矩阵图示意
序列同源 序列易位 序列交换 序列插入
蛋白功能分析
理化性质
理论pI、亲/疏水性、 不稳定系数等
功能域
跨膜螺旋、信号肽、 结构功能域、亚细胞 定位等
M
Motif 搜索
-PORSITE数据库、 -ProfileScan数据库
三维结构模拟
-同源模建 -折叠识别
二级结构预测
PHD 神经网络
蛋白质理化性质分析
• 在线分析 • ExPasy服务器上的ProtParam • /tools/protparam.html
• 核苷酸序列的基本分析 • 分子量、碱基组成 • 序列变换-反向、反向互补、转换为RNA序列等 • 限制性酶切分析
• 基因结构分析: • 启动子及转录因子结合位点 • 重复序列 • CpG Island (HTF Island) • ORF分析(内含子/外显子分析)
• 电子克隆(e-PCR) • 利用EST数据库的重叠序列克隆新基因
>20h >10h 37.11 85.34 -0.243
生物信息学技术的使用教程与分析步骤解析

生物信息学技术的使用教程与分析步骤解析生物信息学是生物学领域的重要分支,它应用于基因组学、转录组学、蛋白质组学等领域的研究中。
在当前的大数据时代,生物信息学技术的发展为解决生物科学研究中的复杂问题提供了便利和支持。
本文将为您介绍生物信息学技术的使用教程与分析步骤解析。
一、生物信息学技术的使用教程生物信息学技术的使用过程包括数据获取、数据处理和数据分析等步骤。
下面将详细介绍这些步骤的内容及相关工具的选择。
1. 数据获取生物信息学研究常用的数据主要来源于公共数据库,如NCBI、ENA、GenBank等。
在获取数据时,需要根据研究需求选择合适的数据库,并确定所需的数据类型,如基因组、转录组或蛋白质组等。
此外,需要掌握相应的搜索和下载技巧,如使用关键词、过滤条件和下载工具等。
2. 数据处理数据处理是将原始数据转化为可分析的格式,通常包括数据清洗、格式转换和数据预处理等步骤。
为了提高数据质量,需要对原始数据进行去噪、去冗余、去重复等处理,并将数据转换为常用的格式,如FASTA、GFF、BAM等。
此外,还需要进行数据预处理,如基因组组装、序列比对和变异检测等。
3. 数据分析数据分析是生物信息学研究的核心内容,主要涉及序列分析、结构分析和功能分析等方面。
在序列分析方面,常见的技术包括序列比对、序列聚类和序列比较等。
在结构分析方面,可以利用已知的结构数据进行比对和模拟,以预测蛋白质的结构和功能。
而在功能分析方面,可以运用基于GO注释的功能富集分析和基于KEGG数据库的代谢通路分析等方法来揭示基因和蛋白质的功能。
二、分析步骤解析在进行生物信息学研究时,需要经过一系列的分析步骤来获取有意义的结果。
下面将介绍常见的分析步骤及其解析。
1. 基因组组装基因组组装是将高通量测序生成的reads拼接成完整的基因组序列的过程。
该步骤的关键是选择合适的组装工具,如SOAPdenovo、Velvet和SPAdes等,并根据测序产出的数据类型,如illumina、PacBio或OXFORD NANOPORE等来制定合适的参数设置。
高通量测序的生物信息学分析报告
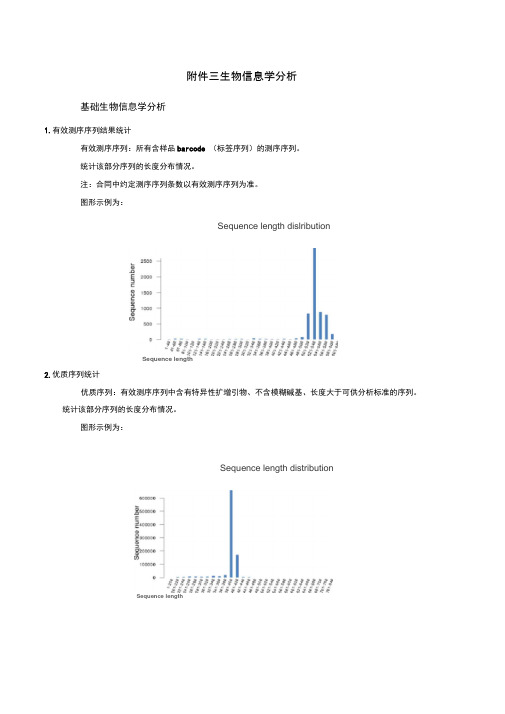
附件三生物信息学分析基础生物信息学分析1.有效测序序列结果统计有效测序序列:所有含样品barcode (标签序列)的测序序列。
统计该部分序列的长度分布情况。
注:合同中约定测序序列条数以有效测序序列为准。
图形示例为:Sequence length dislributionSequence length2.优质序列统计优质序列:有效测序序列中含有特异性扩增引物、不含模糊碱基、长度大于可供分析标准的序列。
统计该部分序列的长度分布情况。
图形示例为:Sequence length distributionSequence length3.各样本序列数目统计:统计各个样本所含有效测序序列和优质序列数目。
结果示例为:4. OTU生成:根据序列的相似性,将序列归为多个OTU(操作分类单元),以便后续分析。
5. 稀释曲线(rarefaction 分析)根据第4条中获得的OTU数据,做出每个样品的Rarefaction 曲线。
本合同默认生成OTU相似水平为0.03 的rarefaction 曲线。
rarefaction 曲线结果示例:Number of R«ads SampladM; 0.036. 指数分析计算各个样品的相关分析指数,包括:丰度指数:ace'chao多样性指数: sha nnon\simps on本合同默认生成OTU 相似水平为0.03的上述指数值。
多样性指数分析结果示例:IDR HM 4H机MOTU■chM|A2W0MO ICNMtlOOO.iOM}S(KS) J O L WCO,UM亠帼血期’ th注:默认分析以上所列指数,如有特殊需要请说明7. Shannon-Wiener 曲线利用各样品的测序量在不同测序深度时的微生物多样性指数构建曲线, 反映各样本在不同测序数量 时的微生物多样性。
当曲线趋向平坦时, 说明测序数据量足够大, 可以反映样品中绝大多数的微生物信 息。
绘制默认水平为:0.03。
生物信息学分析方法

生物信息学分析方法生物信息学是一门综合应用信息学、生物学和统计学等相关知识和技术的学科,旨在通过利用计算机和信息技术处理和分析生物学数据,揭示生物系统的结构和功能,并解决生物学研究中的问题。
生物信息学分析方法主要包括序列比对、基因预测、蛋白质结构与功能预测、基因表达谱分析、基因调控网络构建和演化分析等。
以下将对其中几种常见的生物信息学分析方法进行详细介绍。
1. 序列比对:序列比对是生物信息学中最基本、最常用的方法之一、通过将待比对的序列与已知数据库中的序列进行比对,可以判断序列的相似性和进化关系,从而推断序列的功能和结构。
序列比对方法主要包括全局比对、局部比对和多序列比对等。
常用的序列比对工具有BLAST、ClustalW等。
2.基因预测:基因预测是指通过对DNA序列进行分析和预测,确定其中的基因位置和结构。
基因预测方法主要包括基于序列、基于比对和基于表达等方法。
其中,基于序列的方法依据基因的核苷酸组成、序列保守性和启动子顺应性等特征进行预测;基于比对的方法通过将待预测序列与已知基因进行比对,从而确定基因位置和结构;基于表达的方法则通过分析基因的表达模式和转录组数据,推断基因的存在和功能。
3.蛋白质结构与功能预测:蛋白质结构与功能预测是指通过分析蛋白质序列和结构,预测其二级结构、三级结构和功能。
蛋白质结构预测方法主要包括同源建模、蛋白质折叠动力学和序列匹配等方法。
同源建模是最常用的蛋白质结构预测方法,其基本原理是通过将待预测蛋白质序列与已知结构的同源蛋白质进行比对,并从中找到最佳匹配。
蛋白质功能预测方法主要包括结构域分析、功能域预测和功能注释等方法。
4.基因表达谱分析:基因表达谱分析是通过对基因在不同组织或条件下的表达水平进行比较和分析,揭示基因在生物体内的功能和调控机制。
常见的基因表达谱分析方法有RT-PCR、微阵列和高通量测序等。
RT-PCR是一种常用的基因表达定量方法,可以通过测定特定基因在RNA水平的表达量推断基因的转录水平;微阵列技术则可以同时检测数千个基因的表达水平,从而了解基因在不同组织和条件下的表达情况;高通量测序技术可以对整个转录组进行测序,从而揭示基因的全局表达谱。
生物信息学实验指导书_新版本

生物信息学实验指导书重庆邮电大学生物信息学实验指导书生物信息教学部谭军编重庆邮电大学生物信息学院前言生物信息学是上世纪90年代初人类基因组计划(HGP)依赖,随着基因组学、蛋白组学等新兴学科的建立,逐渐发展起来的生物学、数学和计算机信息科学的一门交叉应用学科。
目前生物信息学的研究领域主要包括基于生物序列数据的整理和注释、生物信息挖掘工具开发及利用这些工具揭示生物学基础理论知识等领域。
生物信息学作为新型交叉应用学科,可以依托本校已有的计算机科学、信息学、生物学和数学等学科优势,充分展现投入少、见效快、起点高的特色,推动学校学科建设和本科教学水平。
本实验指导书中的8个实验均设计为综合性开发实验,面向生物信息学院全体本科学生和研究生,以及全校对生物信息学感兴趣的其他专业学生开放。
生物信息学实验室将提供系统的保障,包括采用mail服务器和linux帐号管理等进行实验过程管理和支持。
限选《生物信息学及实验》的生物技术专业本科生至少选择其中5个实验,并不少于8个学时,即为课程要求的0.5个学分。
其他选修者按照课时和学校相关规定计算创新学分。
实验一熟悉生物信息学网站及其数据的生物学意义实验目的:培养学生利用互联网资源获取生物信息学研究前沿和相关数据的能力,熟悉生物信息学相关的一些重要国内外网站,及其核酸序列、蛋白质序列及代谢途径等功能相关数据库,学会下载生物相关的信息数据,了解不同的数据文件格式和其中重要的生物学意义。
实验原理:利用互联网资源检索相关的国内外生物信息学相关网站,如:NCBI、SANGER、TIGR、KEGG、SWISSPORT、Ensemble、中科院北京基因组研究所、北大生物信息学中心等,下载其中相关的数据,如fasta、genbank格式的核算和蛋白质序列、pathway等数据,理解其重要的生物学意义。
实验内容:1.浏览和搜索至少10个国外和至少5个国内生物信息学相关网站,并描述网站特征;2.下载各网站的代表性数据各10条(组)以上,并说明其生物学意义;3.讨论各网站适合做何种生物信息学研究的平台,并设计一个研究设想。
生信论文——精选推荐

关于生物信息学及其研究进展摘要生物信息学是一门新兴的学科,是20世纪80年代末随着人类基因组计划的启动而兴起的,其研究内容紧随基因组的研究进展而发展。
应用计算机技术及相应软件对生物学上的一些数据进行处理以得到精确的结论分析是这门学科出现的使命。
长时间以来,其一直被认为是一个建立在对DNA 和蛋白质序列比较基础上的学科。
我国的生物信息学由于起步晚,投入资金少等原因,发展比较缓慢,技术有待进一步提高。
本文就生物信息学的发展历程,研究方向及研究进展进行了较为深入的探讨与学习。
关键词:生物信息学信息技术软件人类基因组计划数据库The abstractBioinformatics is an young discipline which is rised by the human genome project the late 80s of the 20th century, Its researching content is followed by the progress in genome research . It’s the discipline’s mission using computer technology and the corresponding softwares to organize some datas on the biological for getting more accurate results . For a long time, it has been considered to be a subject established on the DNA and protein’s sequence comparison. Bioinformatics in China are developed slowly due to late starting, lower input costs and other reasons,so this technology is need be further improved. In this paper, we are discussing and learning about the development of bioinformatics ,course of studying and the direction of researching.Key word:Bioinformatics software of information technology, human genome project database一、前言上个世纪50年代,计算机技术发展的非常迅速,伴随着其发展加上生物技术的应用需要,一门以计算机应用技术为依托新的学科领域诞生了,这就是生物信息学。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
生物信息学课程设计实验报告—典型的生物信息学分析[小编推荐]第一篇:生物信息学课程设计实验报告—典型的生物信息学分析[小编推荐]搜索感兴趣的基因找出自己想要的基因片段找出FASTA格式的基因序列,复制下来,保存在文本文档中水稻瘤矮病发生与危害水稻瘤矮病于1976年在广东湛江地区发现,局部县市危害严重,近年在两广陆续有此病危害的报告,且有逐年加重的趋势,我国广东茂名地区曾大面积发生危害,近年在福建福州以南的一些县零星发生。
症状识别水稻瘤矮病是由电光叶蝉、黑尾叶蝉和二点黑尾叶蝉传播的一种病毒病。
病苗明显矮缩,叶色深绿,叶背和叶鞘长有淡黄绿色近球形小瘤状突起,有时沿叶脉连成长条,叶尖卷转,个别新叶的一边叶缘灰白坏死,形成2-3个缺刻。
病株根细弱,抽穗迟、细小、空粒多。
水稻瘤矮病感病植株病原及发病条件为水稻瘤矮病毒 [Rice gall dwarf Virus(RGDV)]。
病毒粒体球状,直径65nm,由单一粒体组分和十二个片段的双链RNA组成。
此病可由电光叶蝉、二条黑尾叶蝉;二点黑尾叶蝉、黑尾叶蝉和马来亚黑尾叶蝉以持久性方式传播,也能通过二条黑尾叶蝉的卵传给下一代。
国内以电光叶蝉和二点黑尾叶蝉为有效介体。
二点黑尾叶蝉亦可经卵传播。
防治方法:1)治虫防病,力争将传毒媒介昆虫电光叶蝉、二条黑尾叶蝉;二点黑尾叶蝉、黑尾叶蝉和马来亚黑尾叶蝉消灭在传毒前。
杀虫药剂可用25%喹硫磷或40%乐果1000-1500倍稀释液,或菊酯类农药5000倍稀释液喷雾。
2)及早毁除病株,或踩入泥土,或集中烧毁,以防止蔓延。
3)如插后不久发病,还可立即补苗。
4)稻株大胎期用“九二0”纯品50000倍稀释液喷雾,使病株提早抽穗,可减轻为害。
5)每亩用10%叶蝉散可湿性粉剂200克;或每亩用25%速灭威可湿性粉剂150克;每亩用50%杀螟松乳油 + 40%稻温净乳油各50毫升均加水50千克喷雾搜索对应的蛋白质序列Proparam软件分析蛋白质理化性质从分析结果可知:RGDV p8 各个氨基酸所占的比重,如上图。
分子质量为47422da,氨基酸数目为426,正负电荷残基总数30/30,分子式为C2126H3316O623S15,在M-1 cm-1单位在280海里的水里测量的消光系数为48610和48360,脂肪指数为92.68,组氨酸His(H)最少为0.5%,丝氨酸含量最多Ser(S)9.9%。
疏水性分析:氨基酸的疏水性=各种氨基酸的疏水性—甘氨酸的疏水性疏水性氨基酸在蛋白质的内部,由于其疏水性的像相互作用,在保持蛋白质三级结构的形成和稳定中起着重要的作用。
疏水性分析结果由图可知在P8蛋白C端位置有一个典型的疏水区域。
Bioedit 分析结果跨膜区域分析膜蛋白是一类结构独特的蛋白质,执行着重要的细胞生物学功能。
蛋白质序列含有跨膜区,提示他可能作为膜受体起作用,也可能定位在膜上的锚定蛋白或离子通道蛋白。
对膜蛋白的跨膜螺旋进行预测是生物信息学的重要应用。
对RGDV P8蛋白使用TMHMM的跨膜区域分析结果1信号肽预测从分析结果可以看出,剪切位点分值24,信号肽分值为3,综合剪切位点的分值4 Coil 区分析卷曲螺旋主要是控制蛋白质寡聚化的元件,含有卷曲结构的蛋白质主要是一些转录因子通过分析发现,在P8蛋白在三种不同的窗口下尽然没有非常高的置信值,说明没有卷曲重复序列和七肽重复区。
亚细胞定位1、通过分析可知最终预测的叶绿体转运肽,线粒体导肽,信号肽的其他类型的分值分别为0.178、0.066,0.148,0.660。
信号的定位可能是他们中的最高值。
2、“Loc”表示上面分值所预测的可能定位,C表示定位于叶绿体,可能是cTP;M表示定位于线粒体,可能是mTP;S表示分泌通路,即分泌到细胞周质,可能是SP;_表示除前面三处外的其他位置。
3、“RC”是可靠性级别,分为五级。
表示输出结果最高值与次高值之间的差异大小,具体五级如下:1.Diff>0.800;2.0.800>diff>0.600;3.0.600>diff>0.400;4.0.400>diff >0.200;0.200>diff.4.TPlen的预测剪切位点的序列长度。
有结果可知目的蛋白P8的分泌途径为_型,定位在其他细胞器,预测剪切位点序列为0个氨基酸。
结构域的分析及motif的搜索常见的结构域的5种类型:全平行结构域、反平型结构域、α+β结构域、α/β结构域及其他结构域类型。
结构域是蛋白质的功能结构和进化单元,结构功能域分析对于蛋白质的结构的分类和预测由重要的作用。
结果解读:综合上诉结果可知,RGDV p8蛋白的第1~426位之间是个高度保守的结构功能域——Phytoerto_P8,即Phytoerto_P8家族成员共有的典型结构域,该结构功能域由多个植物呼肠孤病毒属外层衣壳蛋白P8序列组成,具有结构分子活性。
Motif 搜索蛋白质二级结构预测预测结果如下:CCHHHHHHHHHHHHHHHHHEEECCCCCEEEEEHCCHHHHH HHHHHHEEHCCCECCCCCCCHHHHHHHHHHHHCCHHHHHHHEC CCCCCCCCCCCCCCCCHHHHEECCCCCCCCHHHHHHHHCCCCCCC CCHHHHHHHHHHHHHCCCCCCCCCCCCHHHHHHCCCCCCCHHH HHHCCCCCHHHHCCCEEEECCCEEEEEECCCCCCCCCCEEEECCCCH HEEEECCEEEEEECCCCCEEEEEEEEEEECCCCCEEEEEECCCCCCCCCCC CCEEEEECCCCEEEEEEEECCCCEEEEEECCEEEEECCCCCEEEEEEEEECC CECCCCCHHHHHHHHHHHHHHCCHHHHHHHHHHHHHHHHHH HHCCCCCCCCEEEECCHHHHHHHHHHHHHHCCCCCCHHHHHHH HHHHHHHHHHHHHHCCEECCC其中C表示coil无规则卷曲,H表示heilx(α-螺旋),E表示extended(β—折叠)蛋白质三级结构预测同源建模三级结构预结果对三级结构的全局模式质量分析和局部模式质量分析,分析的结果第二篇:生物信息学生物信息学是上世纪90年代初人类基因组计划(HGP)依赖,随着基因组学、蛋白组学等新兴学科的建立,逐渐发展起来的生物学、数学和计算机信息科学的一门交叉应用学科。
目前生物信息学的研究领域主要包括基于生物序列数据的整理和注释、生物信息挖掘工具开发及利用这些工具揭示生物学基础理论知识等领域。
生物信息学作为新型交叉应用学科,可以依托本校已有的计算机科学、信息学、生物学和数学等学科优势,充分展现投入少、见效快、起点高的特色,推动学校学科建设和本科教学水平。
本实验指导书中的8个实验均设计为综合性开发实验,面向生物信息学院全体本科学生和研究生,以及全校对生物信息学感兴趣的其他专业学生开放。
生物信息学实验室将提供系统的保障,包括采用mail服务器和linux帐号管理等进行实验过程管理和支持。
限选《生物信息学及实验》的生物技术专业本科生至少选择其中5个实验,并不少于8个学时,即为课程要求的0.5个学分。
其他选修者按照课时和学校相关规定计算创新学分。
实验一熟悉生物信息学网站及其数据的生物学意义实验目的:培养学生利用互联网资源获取生物信息学研究前沿和相关数据的能力,熟悉生物信息学相关的一些重要国内外网站,及其核酸序列、蛋白质序列及代谢途径等功能相关数据库,学会下载生物相关的信息数据,了解不同的数据文件格式和其中重要的生物学意义。
实验原理:利用互联网资源检索相关的国内外生物信息学相关网站,如:NCBI、SANGER、TIGR、KEGG、SWISSPORT、Ensemble、中科院北京基因组研究所、北大生物信息学中心等,下载其中相关的数据,如fasta、genbank格式的核算和蛋白质序列、pathway等数据,理解其重要的生物学意义。
实验内容:1.浏览和搜索至少10个国外和至少5个国内生物信息学相关网站,并描述网站特征;2.下载各网站的代表性数据各10条(组)以上,并说明其生物学意义;3.讨论各网站适合做何种生物信息学研究的平台,并设计一个研究设想。
实验报告:1.各网站网址及特征描述;2.代表性数据的下载和生物学意义的描述;3.讨论:这些生物信息学相关网站的信息资源,可以被那些生物信息学研究所利用。
参考书目:《生物信息学概论》罗静初等译,北京大学出版社,2002;《生物信息学手册》郝柏林等著,上海科技出版社,2004;《生物信息学实验指导》胡松年等著,浙江大学出版社,2003。
实验二利用BLAST进行序列比对实验目的:了解BLAST及其子程序的原理和基本参数,熟练地应用网络平台和Linux计算平台进行本地BLAST序列比对,熟悉BLAST结果的格式和内容并能描述其主要意义,同时比较网上平台和本地平台的优缺点。
实验原理:利用实验一下载的核算和蛋白质序列,提交到NCBI或者其他拥有BLAST运算平台的网页上,观察其基本参数设定库文件类型,并得到计算结果;同时在本地服务器上学会用formatdb格式化库文件,并输入BLAST命令进行计算,获得结果文件。
实验内容:1.向网上BLAST服务器提交序列,得到匹配结果;2.本地使用BLAST,格式化库文件,输入命令行得到匹配结果;3.对结果文件进行简要描述,阐述生物学意义。
实验报告:1.阐述BLAST原理和比对步骤;2.不同类型BLAST的结果及其说明;3.讨论:不同平台运行BLAST的需求比较。
参考书目:《生物信息学概论》罗静初等译,北京大学出版社,2002;《生物信息学实验指导》胡松年等著,浙江大学出版社,2003。
实验三利用ClustalX(W)进行多序列联配实验目的:掌握用Clustal X(W)工具及其基本参数,对具有一定同源性和相似性的核酸与蛋白质序列进行联配和聚类分析,由此对这些物种的亲缘关系进行判断,并且对这些序列在分子进化过程中的保守性做出估计。
实验原理:首先对于输入的每一条序列,两两之间进行联配,总共进行n*(n-1)/2次联配,这一步通过一种快速的近似算法实现,其得分用来计算指导树,系统树图能用于指导后面进行的多序列联配的过程。
系统树图是通过UPGMA方法计算的。
在系统树图绘制完以后,输入的所有序列按照得分高低被分成n-1个组,然后再对组与组之间进行联配,这一步用Myers和Miller算法实现。
实验内容:1.明确软件所支持的输入文件格式,搜集整理出合适的数据;2.在Windows环境运行Clustal X,在Linux环境运行Clustal W;3.实验结果及分析,用TREEV32或Njplotwin95生成NJ聚类图。
